2. 鹏城实验室 广东 深圳 518055;
3. 北京大学 计算语言学教育部重点实验室 北京 100871
2. Peng Cheng Laboratory, Shenzhen 518055, China;
3. Key Laboratory of Computational Linguistics of Ministry of Education, Peking University, Beijing 100871, China
医疗数据是以患者为中心,在医生对患者诊疗和治疗过程中产生的数据,包括患者的基本数据、电子病历数据、诊疗数据、医学影像数据、医学管理数据、经济数据、医疗设备和仪器数据等。医疗数据的应用价值,存在于医学研究、公共卫生、个人健康、远程会诊以及医学诊断等方面。例如,在医学研究方面,通过搜集、整理与新冠病毒相关的科研基础数据和科研文献,可用于预测新冠肺炎易感染人群、病毒变异性和潜在的治疗药物,如老药新用等;在公共卫生方面,2008年推出的谷歌流感预测(GFT)上线后受到广泛关注,其原理是采用流感趋势系统监控全美的网络搜索,寻找与流感相关的词语,如“咳嗽”和“发烧”等,通过检测一个地区与流感相关词的数量,就可以估计出该地区流感流行的情况。
本文所关注的医学文本包括医学教材、临床实践、电子病历等,通常由非结构化或半结构化文本组成。非结构化和半结构化文本难以直接使用,而面对海量的医学文本数据,显然无法全部依赖人工进行处理。因此,如何自动从海量文本中发现知识,使得人们可以低成本地理解、使用这些数据就显得尤为重要,实体关系抽取就是在此背景下应运而生的。本文从实体关系抽取的相关概念出发,探讨了深度学习方法在医学领域实体关系抽取任务上的发展历程,根据数据集的构建方式对监督学习和远程监督的多实例学习模型进行了分析,并展望了面向医学文本的实体关系抽取的未来研究方向。
1 面向医学文本的信息抽取 1.1 医学知识图谱现有的大型知识图谱,诸如Wikidata[1]、DBpedia[2]、Freebase[3]、CTD[4]和CMeKG[5]等,富含海量知识并以结构化形式存储。以支气管肺癌相关知识为例,如图 1所示,图中每个节点代表实体,并用连线标记实体间的关系。这样,支气管肺癌的相关知识就以结构化的形式记录下来。
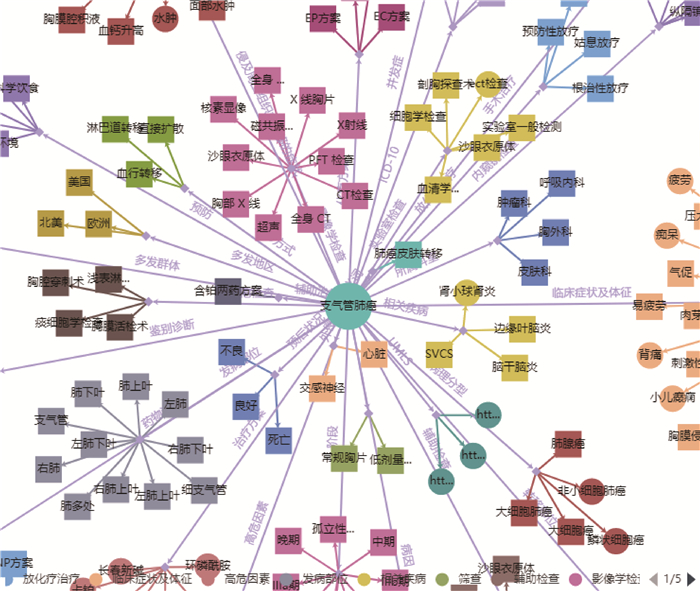
|
图 1 支气管肺癌相关知识 Fig. 1 Knowledge of bronchial lung cancer |
目前,将结构化的知识应用于搜索引擎、问答系统等自然语言处理应用中,可以提升系统语义理解能力。但与现实世界快速增长的知识量相比,知识图谱覆盖度仍力有未逮。由于知识规模巨大且人工标注昂贵,这些新知识单靠人力标注添加难以完成。为尽可能及时准确地为知识图谱增添更加丰富的世界知识,研究者们努力探索自动获取世界知识的办法,即实体关系抽取技术。实体关系抽取是一个经典任务,特征工程、核函数、图模型都曾被广泛应用于其中,并取得了一些阶段性成果。随着深度学习时代的来临,神经网络模型和预训练模型为实体关系抽取带来了新的突破。
1.2 相关概念非结构化的医学文本,如医学教材的每一个自然段落,临床实践中每种疾病的主题,电子病历数据中的主诉、现病史、鉴别诊断等,都是由中文自然语言句子或句子集合组成的。关系是指两个医学实体之间的关系事实,关系抽取是从非结构化医学文本中找出关系事实的过程。
1996年,在消息理解研讨会上首次提出了命名实体识别(named entity recognition,NER)任务[6],任务目标是识别文章中特定类型的实体名称或符号,如实体名称(组织名、人名、地点),时间表达(日期、时间)等。医学领域的信息抽取是从非结构化医学文本中抽取出结构化的信息,主要包括实体抽取(entity extraction, EE)、关系抽取(relation extraction,RE)和属性抽取(attribute extraction,AE)等。医学实体指文本中名词性短语,如疾病、药物、治疗方式、症状、病因、高危因素等。实体抽取又称命名实体识别,即从文本中抽取上述医学实体。关系抽取则是判断实体对之间的语义关系,如临床体征、并发症、辅助检查、药物治疗等。属性抽取需要识别实体的属性名和属性值,属性名通常是一个名词短语,但是属性值可以是词或句子。具体来说,给定一个句子,模型需要根据句子语义信息抽取出实体,并推测实体间的关系。例如,“小细胞肺癌@小细胞肺癌主要发生于老年吸烟者。最常见的主要症状是咳嗽、胸痛、咯血、呼吸困难和体重减轻。”模型需要识别出实体“小细胞肺癌”“老年吸烟者”“咳嗽”“胸痛”“咯血”“呼吸困难”“体重减轻”,进而通过语义得到“多发群体”和“临床症状”的关系,并最终抽取出〈小细胞肺癌,多发群体,老年吸烟者〉,〈小细胞肺癌,临床症状,咳嗽〉,〈小细胞肺癌,临床症状,胸痛〉,〈小细胞肺癌,临床症状,咯血〉,〈小细胞肺癌,临床症状,呼吸困难〉和〈小细胞肺癌,临床症状,体重减轻〉的知识三元组。
1.2.1 命名实体识别在医学领域,传统的命名实体识别方法可以分为基于规则的方法、基于字典的方法和基于机器学习的方法,其中基于规则的方法和基于字典的方法往往需要医学专家手工构建规则模板和字典。
基于规则的方法需要根据医学文本的特点制定对应的抽取规则,并且所制定的规则仅在特定数据集中有效,无法应用于其他数据集[7]。Hanisch等[8]通过基于规则的方法和预处理的同义词词典构建ProMiner系统,用于识别生物医学文本中可能出现的实体名,并将识别结果对齐至蛋白质和基因数据库。Savova等[9]提出临床文本分析和知识提取系统,采用流水线结构以及基于规则和机器学习技术组成的模块化系统,用于从电子病历和临床记录中提取信息。
基于字典的方法适合于精确搜索,广泛应用于大规模医学临床文本标注和检索。由于医学术语存在多种表述,很难用单个词典涵盖所有的可能表述,因此很容易忽略未记录在字典中的实体。针对这一问题,比较流行的方法是模糊字典匹配法和后处理法。Yang等[10]提出一种基于字典的生物医学实体名称识别方法,包括构建和扩充生物医学词典、匹配近似字符串以及后处理步骤,并设计了医学术语缩写识别算法对生物医学实体词典进行扩充,通过改进的编辑距离算法提高查全率。
基于机器学习的方法在识别临床记录中的实体方面表现出良好的性能,其中算法设计和特征选择是影响系统识别效果的关键。与基于规则的方法和基于字典的方法不同,机器学习方法需要标准的标注数据集以及合适的算法,然后利用样本数据的统计特征和参数来构建模型。根据训练数据的不同特点,可以使用的机器学习方法有隐马尔可夫模型(hidden Markov model,HMM)、支持向量机(support vector machine,SVM)、条件随机场(conditional random field,CRF)[11]和最大熵(maximum entropy,ME)等。在这些机器学习算法中,CRF方法由于能够添加对序列标注过程有帮助的各种辅助特征而受到欢迎。Tang等[12]提出结构支持向量机(structural SVMs,SSVMs)算法用于识别临床记录中实体名,该算法结合了CRF和SVM的优点以及单词嵌入的有效表示,与使用相同特征的传统CRF相比,识别效果更好。Chang等[13]将基于规则的方法和基于ME模型相结合,用于提取出院总结中实体之间的时间关系,梳理相关事件的顺序,帮助医务人员做出准确合理的临床决策。
1.2.2 关系抽取医学领域关系抽取用于识别医学实体对之间的关系事实,常见关系如疾病与症状的关系以及疾病与药物的关系等。由于医学文本的来源不同,疾病的描述详细程度存在差异,往往需要根据实际标注文本制定符合当前数据的关系事实。2011年Uzuner等[14]在关系分类(relation classification,RC)评测任务中,考虑了电子病历记录中的三类关系:疾病与疾病之间的关系;疾病与检查之间的关系;疾病与治疗药物之间的关系。此外,该任务仅考虑句子级关系事实抽取,即从给定的一个句子中抽取其中的关系事实。2015年Wei等[15]提出一项从生物医学文献中自动抽取化学药品和疾病之间关系的任务,该任务由疾病名称识别子任务和化学药品致病关系抽取(chemical-induced diseases relation extraction,CID)子任务组成。其中,疾病名称识别子任务是识别疾病名称并标准化为MeSH词表中的概念实体,CID子任务是识别化学药品与疾病之间的致病关系。
传统的实体关系抽取方法包括基于共现的方法[16]、基于模式的方法和机器学习方法。基于共现的方法是指当两个实体出现在同一个句子时,说明实体之间存在一定的关系,实体对共同出现的频率越高,两者的联系也越强。机器学习方法的应用最为广泛,不过越来越多的学者选择将两种或多种方法结合使用,用于处理更加复杂的句子结构。
自动抽取患者病情记录中临床事件之间和时间表达式之间的时间关系,可以帮助医生了解疾病进展和患者的治疗效果,还可以促进循证医学的研究。Nikfarjam等[17]提出一种结合机器学习与基于图推理的系统,针对不同时间关系设计独立的抽取组件。Yang等[18]提出结合患者病情记录特定规则和CRF模型来识别病情记录中的时间关系。Lee等[19]提出一种基于卷积神经网络来提取科学概念之间同义词或下位词关系的模型,该模型由预处理、卷积神经网络和基于规则的后处理三部分组成,在ScienceIE数据集上完成实验。Seol等[20]提出一种利用CRF模型识别与患者相关的临床事件,利用SVM提取事件之间关系,之后利用定义的语义单元整理事件之间的因果关系。其中,临床事件包括症状、目的、检查结果、诊断等九类,事件之间的关系有TAP、问题、行动、问题-行动4种。
1.3 深度学习方法的分类相比于前文所提及的用于命名实体识别和关系抽取的传统方法,深度学习方法[21-23]的优势在于无须领域专家制定复杂的抽取特征,神经网络模型自身就可学习到句子中隐藏的语义表示。图 2为基于深度学习的实体关系抽取框架。
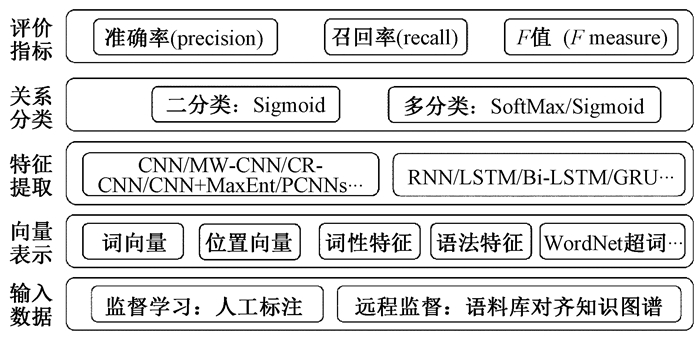
|
图 2 基于深度学习的实体关系抽取框架 Fig. 2 Entity relation extraction framework based on deep learning |
本文重点探讨深度学习方法在生物医学领域实体关系抽取任务上的发展历程。深度学习的实体关系抽取方法可以从不同角度进行分类。例如,根据给定的非结构化医学文本中实体是否已知,可以将其划分为关系分类和实体关系抽取;根据所采用数据集的构建方式,可以将其划分为监督学习和远程监督的多实例学习;根据一个关系事实下的实体数目,可以将其划分为二元关系抽取和多元关系抽取;根据一条医学文本句子中需要抽取的关系事实的数目,可以将其划分为单关系抽取和多关系抽取。
1.3.1 关系分类和实体关系抽取在关系抽取之前,若给定文本中的实体信息,则将关系抽取称为关系分类问题。如前文所述,如果在给出句子的同时,提供句子的医学实体“小细胞肺癌”“老年吸烟者”“咳嗽”“胸痛”等,那么研究问题就变为识别两两实体之间存在的语义关系。这些语义关系根据专业领域不同,通常是预先定义好的,因此研究问题等价于分类任务。大多数基于深度学习的方法仅解决关系分类任务。如果仅给出句子,没有额外的实体信息,此时就是实体关系抽取任务。部分学者将关系分类称为关系抽取,但实体和关系联合抽取任务也常常简称为关系抽取,具体含义可由上下文进行推断。
根据设计思想的差异,实体关系抽取方法也可以分为流水线方法和联合抽取方法。流水线方法是指将实体关系抽取任务分成两步来完成,分别为命名实体识别和关系分类。若在第1步的命名实体识别部分存在错误,则第2步的关系分类不可避免地会受到第1步识别错误的影响,因此流水线方法通常存在误差传递问题。为解决这一问题,学者们提出联合抽取模型。联合抽取模型的解决办法通常是采用参数共享[24-25]或者统一标注方案[26]来实现联合编码。
1.3.2 监督学习和远程监督的多实例学习利用人工标注数据集进行模型训练的方式称为监督学习。i2b2/VA-2010关系抽取数据集[14]、BioCreative V化学药品与疾病关系抽取数据集[15]、ACE数据集[27]和SemEval-2010 task 8数据集[28]是关系抽取任务常用的监督数据集。这些数据集的每条语料中实体对信息已经标注完成,任务目标是正确预测实体对之间的语义关系。数据集由专业标注团队构建而成,拥有高质量的三元组和极少的噪声。但是,由于构建成本过高,使得构建的数据集规模较小。
利用远程监督方式构建的数据集进行模型训练的方式称为远程监督的多实例学习。Mintz等[29]提出一种用于自动生成大量训练数据的远程监督方法,假设当知识库中实体对存在某种关系时,文档中所有该实体对都表达这种关系。显然,远程监督方法的这种假设过于绝对,包含实体对的每个文档不可能都表达这种关系。比如知识库中的三元组〈麻疹, 并发症, 咳嗽〉,文档“【流行病学】麻疹患者是唯一的传染源。感染早期,病毒在患者呼吸道大量繁殖,含有病毒的分泌物经过患者的呼吸、咳嗽或喷嚏排出体外并悬浮于空气中,通过呼吸道进行传播,与患者密切接触或直接接触患者的鼻咽分泌物亦可传播。”文档中“麻疹”与“咳嗽”属于“传播途径”关系。
远程监督常使用的数据集有《纽约时报》(New York Times,NYT)数据集、WebNLG数据集和FewRel数据集等。为解决上述问题和降低数据集中噪声,Riedel等[30]提出宽松的远程监督假设,将问题建模为多实例学习问题,同时构建出NYT数据集。NYT数据集是以《纽约时报》为语料库,Freebase作为远程监督的知识图谱构建而成。WebNLG数据集是由Gardent等[31]针对自然语言生成(natural language generation,NLG)任务所提出的,以2015年10月发布的英文DBpedia作为远程监督的知识图谱,通过众包方式要求人工编写出准确包含给定三元组内容的句子。大规模精标注关系抽取数据集FewRel[32]是以Wikipedia作为语料库,Wikidata作为知识图谱构建而成。
1.3.3 二元关系抽取和多元关系抽取二元关系抽取中每个关系对由关系P、主实体S和客实体O三个部分组成。实体类型和关系根据不同的专业领域会预先定义。三个部分统称为关系三元组,简称三元组。主实体S和客实体O称为一个实体对。例如“脑炎@对单纯疱疹病毒性脑炎患者进行早期治疗(用阿昔洛韦),并对重症患者进行充分的支持性治疗可降低患者死亡率。”从中可以抽取三元组〈单纯疱疹病毒性脑炎,药物治疗,阿昔洛韦〉,其中主实体是“单纯疱疹病毒性脑炎”,客实体是“阿昔洛韦”,实体对之间的关系是“药物治疗”。而多元关系抽取是指三个或三个以上实体之间的关系。从句子中抽取的三个实体为“单纯疱疹病毒性脑炎”“早期治疗”“阿昔洛韦”,关系是“药物治疗”。多元关系可以用多个二元关系表示,本文研究的关系抽取均为二元关系抽取。
1.3.4 单关系抽取和多关系抽取单关系抽取是指需要抽取的句子中只包含一个关系三元组。多关系抽取则是抽取的句子中可能包含多个关系三元组,但是数目不定,可能是一个实体对存在多种关系,也可能是多个三元组存在实体重叠。三元组实体重叠问题示例如图 3所示,实体重叠类型可分为普通型、实体对重叠型和单个实体重叠型。关系分类和远程监督的多实例学习基本属于单关系抽取任务,但也有部分学者考虑到多关系抽取问题。
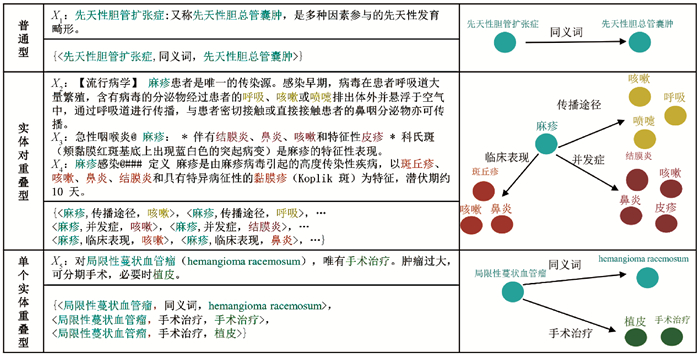
|
图 3 三元组实体重叠问题示例 Fig. 3 Examples of triplets overlapping problem |
利用深度学习技术进行关系抽取的早期工作,是在人工标注语料库的监督训练范式下进行的。基于有监督的实体关系抽取框架的演化过程如图 4所示。通过将问题建模为多分类问题,模型会尝试为句子中每个实体对预测相应的关系类型。
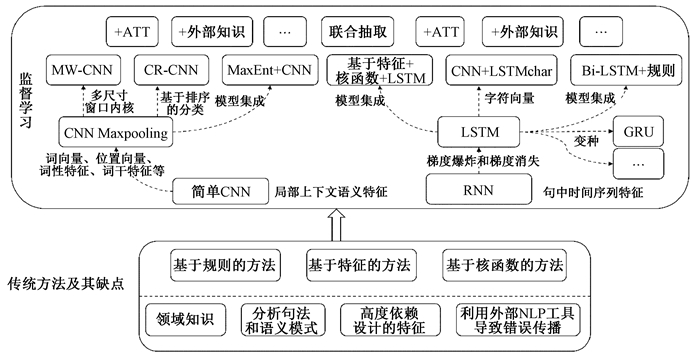
|
图 4 基于有监督的实体关系抽取框架的演化过程 Fig. 4 Evolution process of entity relation extraction framework based on supervision |
Liu等[33]用CNN网络取代手工构建文本特征,从而实现自动提取特征,并构造了一个端到端的网络,用词向量和词法特征对输入的句子进行编码,然后接卷积层、全连接层、SoftMax层,给出了所有关系类别的概率分布。该模型使用同义词向量代替预训练词向量,还尝试使用单词列表、词性列表和实体类型列表来合并一些词法特征。Zeng等[34]同样使用CNN来编码句子级别特征,不同之处在于模型使用预训练词向量,其提出使用的位置嵌入向量成为了深度学习关系抽取模型的标准,该模型还利用了句子中名词的信息和名词在WordNet中上位词信息。
生物医学领域的文本来源具有多样性,如科研文章、临床试验、电子病历等。传统的基于特征和核函数的方法,在性能上很大程度依赖于特征选择,而精心挑选出来的特征由于文章类型的差异往往不具有通用性,需要根据数据源重新进行调整。例如:科研文章往往格式规范、表述清晰,且使用规范的专业术语;而临床出院记录中的内容更具碎片化和不完整性,掺杂缩写表述和专业术语表达。Sahu等[35]首次提出将CNN用于提取临床文本中医学实体之间的关系,除了词向量和位置向量,模型的输入还增加词性特征、词干特征、实体类型信息来丰富句子的表示形式,该模型在i2b2/VA临床关系抽取数据集上的实验结果超过了以往的最优方法。
2.2 基于CNN模型的改进在文献[33-34]基础上,Nguyen等[36]在关系抽取的监督学习研究领域提出了拥有多尺寸窗口内核的MW-CNN模型。MW-CNN模型完全摆脱了利用外部词汇特征来丰富输入句子的表示形式,而是让CNN自己学习需要的特征。该模型结构与文献[33]类似,输入层由词向量和位置向量组成,上层为卷积层和最大池化层,利用不同窗口大小的卷积核来捕获更广泛的n-gram信息。迭代实验表明,使用2、3、4、5窗口长度的内核可以提供最佳的性能。通过Word2vec[37]训练的词嵌入来初始化词嵌入矩阵,实验结果相比于随机嵌入和静态Word2vec向量有显著提升。
在文献[34]基础上,Santos等[38]利用排序思想进行关系分类,提出了基于排序的CR-CNN模型。实验结果显示,该模型使用两两排序的损失函数,可以更容易区分开一些易于分错的类别,而SoftMax却只能增加正确类别的概率。此外,在训练时忽略其他关系类型的噪声,可以让模型更加专注于学习数据集所关注的九类关系类型,这里的其他关系类型是指当两个实体不属于九类关系类型时所分配的关系。最后,仅使用两个实体之间的短语可以在一定程度上替代位置嵌入的作用,且实现更为简单。
理解化学药品和疾病之间的联系(chemical-disease relation, CDR),关系到生物医学研究和卫生保健等多个领域,如新药发现和食品药品安全监督等。已发表的生物医学领域的科研文章中包含大量的化学药品与疾病的作用关系,人工提取这些关系代价很高且无法保证时效性。Gu等[39]采用ME模型和CNN模型分别抽取跨句关系和句间关系,将任务简化为句子级关系抽取,以解决篇章级医学实体间关系抽取任务。由〈化学药品,疾病〉实体对将每个文件分割成用于训练和测试的单句语料或跨句语料,如果实体对存在于同一个句子中则为单句抽取语料,否则为跨句抽取语料[40]。针对单句抽取语料和跨句抽取语料分别使用CNN模型和ME模型进行抽取,最后将抽取结果合并为篇章级格式,相比于单独的ME模型,在BioCreative V数据集CID任务上取得有效提升。
2.3 基于RNN模型虽然很多学者将基于CNN结构模型用于关系抽取任务中,但是基于CNN框架的模型无法学习到时序特征,特别是实体对之间长距离依赖关系。因此,Zhang等[41]尝试基于RNN建模长距离关系抽取模式,达到了与文献[34](CNN+PF)同样的实验效果。实验结果显示,循环模型结构不仅在SemEval-2010 task 8数据集上表现出不错的效果,还在KBT37数据集上获得更明显的提升。同时,模型使用更加简单的位置指示器(position indicator,PI),而非文献[34]使用的位置特征。PI直接使用标签来表示两个实体的位置,将〈e1〉, 〈\e1〉, 〈e2〉, 〈\e2〉作为4个指示器,比如“ 〈e1〉 people 〈\e1〉 have been moving back into 〈e2〉 downtown 〈\e2〉”。在训练时直接将这4个标签作为普通的单词即可,无须特殊处理,通过这样的方式来突出两个实体。此外,通过后续的一些附加实验验证了RNN的记忆优势适合对长文本进行建模。
Zhou等[42]提出集成基于特征的模型、基于核函数的模型和神经网络模型的抽取框架,应用于化学致病关系抽取任务,基于特征的模型获取表层词汇特征,基于核函数的模型捕捉结构化句法特征,神经网络模型利用语义表示信息。在BioCreative V数据集CID任务上的实验结果显示,该集成框架显著提高了模型的抽取性能。
生物医学文本中经常出现新的专业术语,因此关系抽取模型需要具有识别此类短语的能力。近期研究证明,字符向量的使用有助于在众多自然语言处理任务中识别未知术语。Nguyen等[43]提出利用CNN和LSTM分别训练字符向量表示,之后拼接词向量、位置向量和字符向量输入至CNN模型,在BioCreative V数据集CID任务上的实验结果显示,CNN+CNNchar模型结果和CNN+LSTMchar模型结果均超越所有的基线模型。
在生物医学领域,挖掘治疗药物与疾病的关系是至关重要的。Chikka等[44]提出融合双向长短期记忆网络(Bi-LSTM)和基于规则的方法解决i2b2-2010数据集中抽取疾病和治疗药物关系子任务。i2b2-2010数据集中疾病与治疗药物之间的关系可以分为TrAP、TrIP、TrWP、TrCP和TrNAP五类。该模型将单词级别特征(字向量、词向量、词性和位置特征)拼接后输入Bi-LSTM,之后将Bi-LSTM输出结果与句子级别特征拼接后输入至线性层判断关系类型。基于规则的方法在识别样本数目较少的关系类型时有较好表现。最终结果显示,融合深度学习和基于规则的模型在i2b2-2010数据集上取得当前最优。
2.4 基于注意力机制的模型深度学习方法的使用为减少手工制定特征提供可能,但是模型不可避免地会使用一些词汇资源(如WordNet)和NLP系统(如依赖解析器和命名实体识别)来获取高维特征。另外,重要信息可能出现在句中的任何位置。因此,Zhou等[45]提出基于神经注意力机制的Bi-LSTM框架,在不使用额外知识和自然语言处理系统的情况下,自动聚焦于对分类有决定性影响的词,捕捉句子中最重要的语义信息,该模型与基于排序的CR-CNN模型效果一致。
目前基于注意力机制的双向LSTM模型已经成为自然语言处理任务的标配,在关系分类任务上也取得了不错的效果,数据集中同种关系可以用多种不同的形式进行表述。Wang等[46]提出基于两层注意力机制(实体级注意力机制和关系级池化注意力机制)的卷积神经网络框架,用于学习不同结构的句子中与目标分类最相关的元素。实体级注意力(第1层注意力)机制用于输入层,通过计算句中单词与目标实体的余弦相似度,突出句中与目标实体相关的部分;关系级池化注意力(第2层注意力)机制用于卷积后池化阶段,确定与目标关系相关的部分。该模型在SemEval-2010 task 8数据集上的F1值达到88.0%,优于依赖丰富先验知识的方法。
药物引起的不良反应是一个极其危险的因素,可能会导致患者生病甚至死亡。因此,在生物医学研究中抽取药物不良反应事件(adverse drug event,ADE)是一个重要课题。药物不良反应事件涵盖药物引发的不良反应、非预期的副作用、停药或过量使用特定药物所带来的影响。Ramamoorthy等[47]受到机器阅读理解任务的启发,将药物不良反应抽取建模为问答形式。模型使用自注意力机制促进文本序列内交互,挖掘临床文本中局部上下文语义特征,实现药物识别和疾病实体同时抽取不良反应语义关系。实验数据集是抽取PubMed摘要由人工标注构建,语料数为6 821,以8:1:1的方式划分训练集、测试集和验证集,实验结果显示,F1值比基线模型提升9%。
除了文本上下文语义特征外,化学药物和疾病相关先验知识对关系抽取也很有帮助,生物医学领域中被大家所熟知的化学药物与基因组对比数据库(comparative toxicogenomic database,CTD)里包含了大量的结构化三元组。为充分利用数据库中知识表示,Li等[48]提出结合领域知识、注意力机制和分段策略的卷积神经网络模型,在BioCreative V数据集CID任务上F1值达到69.1%。Zhou等[49]提出知识指导下的卷积网络(knowledge-guided convolutional network, KCN),利用TransE模型[50]学习CTD中知识表示,之后在知识表示的指导下进行候选实例上的KCN训练KCN,最后将句内抽取和句间抽取结果合并为文档级结果,在BioCreative V数据集CID任务上F1值达到71.3%,超过以往最优模型。
2.5 实体关系联合抽取上述模型大部分是针对单独的关系分类任务。在早期的实体关系联合抽取任务上,Mintz等[29]和Gormley等[51]以管道方式处理任务,将任务分割为两个子任务:命名实体识别和关系分类。然而,管道方案忽略了两个子任务之间的联系和跨任务之间的依赖关系,性能也受到上游错误传递的影响。为解决这些问题,学者们提出许多实体关系联合抽取模型,传统的联合抽取模型[52-55]严重依赖于特征工程。Miwa等[56]提出采取端到端神经网络模型用于实体关系联合抽取任务,通过将双向序列和双向树状结构的LSTM-RNNs堆叠,来捕获单词序列信息和依存句法树结构信息,实现了实体识别和关系抽取的参数共享。
在生物医学自然语言处理中,细菌及其生存环境关系抽取研究受到广泛关注。生物医学自然语言处理共享任务(BioNLP-ST)2016年发布细菌群落(bacteria biotope,BB)子任务。以“在海洋中,弧菌无处不在”为例,任务目标为抽取细菌实体“弧菌”、生存环境位置“海洋”及判断两实体之间的“生存环境”关系。在文献[56]模型基础上,Li等[57]提出两点改进策略:第1,为避免误差传递导致关系分类错误,在关系分类任务中引入“无效实体”关系,用于纠正命名实体识别错误;第2,为解决原模型贪婪搜索算法带来的标签之间错误传播,使用集束搜索代替贪婪搜索。实验结果显示,在细菌群落数据集上,增加“无效实体”关系后F1值提升约2%,使用集束搜索后F1值提升近6%。
3 远程监督的多实例学习Riedel等[30]为解决远程监督的局限性,放宽远程监督假设的限制,建模假设“如果实体对存在某种关系,那么包中至少有一个句子反映该关系”,将任务建模为多实例学习问题。
基于远程监督的实体关系抽取框架的演化过程如图 5所示。这样就可以利用远程监督创建大规模的训练数据,同时对标签中的噪声具有更好的鲁棒性。多实例学习是有监督学习的一种形式,将一组句子规定为一个包,对包进行标注,而不再需要标注每一个句子实例。在关系抽取中,每个实体对定义为一个包,包中包含着存在该实体对的所有句子。
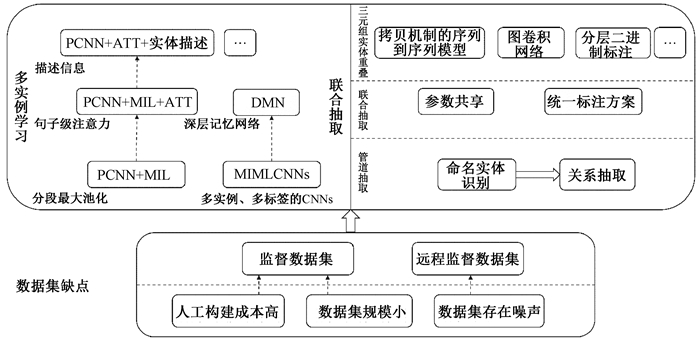
|
图 5 基于远程监督的实体关系抽取框架的演化过程 Fig. 5 Evolution process of entity relation extraction framework based on distant supervision |
Zeng等[58]提出了分段卷积神经网络(piecewise convolutional neural networks,PCNNs)模型,使用多实例学习的模式,借助神经网络模型[34, 36]建立一个远程监督数据的关系抽取器,其重要贡献是提出了跨越整个句子的分段最大池化。这样的最大池化层虽然大大减小了隐藏层的大小,但是不足以捕获句子中实体之间的结构。因此,可以通过对句子的不同段池化而不是整个句子的最大池化来加以避免。每个句子可以很自然地根据两个实体分为三部分,通过在每个段内执行分段最大池化以获得更丰富的表示,同时仍然保留与输入句子长度无关的向量。由于该方法假设“包中至少有一条语句表达实体对之间的关系”,因此在训练和测试阶段仅使用最大概率的语句,这意味着模型忽略了包中由其他句子提供的大量有用数据信息。即使包中并非所有句子都表达实体对之间的正确关系,但仅使用单个句子是异常严格的约束。
借助多实例学习的PCNN模型表现出优于传统非深度学习模型的性能,如Mintz等[29]于2009年提出的基于远程监督的模型;Hoffmann等[59]于2011年提出的多实例学习方法MultiR;Surdeanu等[60]于2012年提出的多实例学习方法MIML。同时,消融实验下PCNN模型也优于CNN模型,证明了多实例学习优于传统学习方法。为解决模型只考虑包中最相关句子的问题,Lin等[61]针对多实例问题,对单个包中所有的实例使用注意力机制。当使用包中所有实例的加权注意力机制表示损失时,模型能够从噪声中识别重要句子,并且利用包中的所有信息来进行关系类别预测。可以观察到,“只有一句最有可能的句子”的模型是句子级注意力机制的一个特例,即最有可能的句子的权重为1,而其他句子的权重都为0。结果表明,使用句子级注意力机制模型可以显著提高CNN和PCNN模型的准确率与召回率。由于模型学习到散落在多实例中的信息,因此能够以较高的置信度预测正确的关系类型。
3.2 多实例、多标签的CNNs模型Jiang等[62]提出了多实例、多标签的CNNs(multi-instance multi-label CNNs,MIMLCNNs)模型,使用跨文档的最大池化层解决信息损失问题[30]。类似于前文所述的注意力机制,首先对包i中的每个句子dij确定一个向量表示rij,然后采用句子向量维数的最大值来确定包i的最终向量表示。这使得最终特征向量的每个特征都来自于最相关的文档,而不是整个特征向量来自于包中最相关的一个文档。同时,也解决了关系抽取的多标签问题。到目前为止,已有模型对一个实体对仅预测单个关系类型,但是相同的实体对可能从不同文档抽取多种关系(称为重叠关系),例如〈麻疹, 传播途径, 咳嗽〉和〈麻疹, 临床表现, 咳嗽〉,对于相同的实体对〈麻疹, 咳嗽〉来说都是有效的关系。于是在最终的分类层将SoftMax改为Sigmoid,这意味着网络独立地预测每个关系类别的概率,而不是预测所有关系上的概率分布。由于MIMLCNNs模型能利用包中多个文档的信息,因此能够像句子级注意力机制一样提升PCNN和CNN模型的性能。
3.3 深层记忆网络模型Feng等[63]提出,不同的单词在不同关系类型下以及对不同的实体对重要程度不同,这一点类似于监督学习中的多层注意力机制。同时,关系类型之间并不是独立的,会有重叠依赖现象,所提出的多标签本质上是因为标签之间有相互依赖关系,即如果〈A, capital, B〉成立,〈A, contains, B〉也会成立。对于第一个问题,借鉴输入层注意力机制的思路,即根据单词与实体对的相似度来分配权重,但是这里不使用传统的注意力, 而是基于记忆网络的思想。对于第二个问题,使用多层关系的注意力机制来引入关系类型之间的相关度。利用单词级别的思路[46]计算单词与目标实体的相关性,并且利用多层来挖掘更深层次的关系。关系级的动机则是考虑到数据中的关系依赖性,使用注意力机制来考虑关系之间的相关性。
3.4 引入外部知识模型Ji等[64]引入额外的知识图谱信息,即实体描述信息。例如,NYT数据集是通过与Freebase做实体链接等来链接句子中的实体,而其实每个实体在Freebase都有一段文字描述。该研究认为现有工作集中在NYT和Freebase数据本身上,忽略了数据集背后的知识图谱信息,因此引入实体描述信息加强对实体嵌入的学习。此外,在处理多实例学习方面,同样使用了句子级注意力机制[61]。模型分为输入模块、注意力模块和实体描述模块三部分,其中输入模块中每个句子的词由词向量与位置向量连接表示,接着是卷积层与分段最大池化层,最终得到每个句子的向量。多实例学习的注意力模块,通过计算包内每个实例与关系类型的相关性确定权重。该研究的贡献主要在于从知识图谱中引入额外的实体描述信息,加强嵌入向量的学习。不过两部分的融合在本质上相当于在原有基础上加一个范式约束而已,或者说一个先验的惩罚项。
3.5 实体关系联合抽取大多数联合抽取的神经模型[65-66]采用参数共享的方式来实现联合抽取。为获取关系三元组,模型需要将检测到的实体对输入到关系分类器,以识别实体之间的关系。单独解码设计导致实体识别与关系抽取的训练目标分离,使得实体识别任务和关系预测任务之间的联系被切断。Zheng等[67]通过引入一种全新的统一标注方案来实现联合编码,将三元组关系抽取任务转化为不需要NER或RC的端到端序列标注问题。由于实体和关系的信息被集成到统一的标注方案中,因此模型可以将关系三元组作为一个整体来学习。然而,句子中可能会包含多个三元组,且存在前文所描述的三元组实体重叠现象。由于设计时每种实体仅能属于一种类型,传统的序列标注方案不能在实体重叠时将同一实体标注为多种类型。重叠现象也给关系分类带来了很大的挑战,因为实验假设一个实体对最多只存在一种关系[66]。针对此现象,Zeng等[24]提出一种具有拷贝机制的序列到序列模型;Fu等[25]提出基于图卷积网络(graph convolutional network, GCN)的方法;Wei等[26]使用预训练模型BERT进行编码,并设计一种分层二进制标注策略,将实体对的语义关系视为主体到客体的映射函数。
4 未来研究方向根据所采用数据集的构建方式,前文分别回顾了监督学习和远程监督的多实例学习模型。表 1记录了监督学习模型在BioCreative V数据集CID任务上的结果,以及远程监督的多实例学习模型在NYT数据集和WebNLG数据集上的结果。从表 1可以看出,监督学习模型和远程监督的多实例学习模型在数据集上的性能呈持续提升趋势。深度学习模型所使用的数据集中,预先定义的关系类别分布均匀,且提供丰富的训练样例,单个样例表述相对简短、关系密度低,需要抽取三元组重叠现象并不严重。
|
|
表 1 监督学习模型和多实例学习模型的结果 Tab. 1 Results of supervised learning models and multi-instance learning models |
BioCreative V数据集、NYT数据集以及WebNLG数据集等实体关系抽取任务的常用数据集大多为英文数据集,目前尚未发现公开的面向中文医学领域实体关系抽取的基准数据集。正在构建的中文医学信息抽取数据集CMeIE,通过收集多来源的医疗文本,参考医学命名实体和实体关系的标注体系及规范[5, 68-69],利用张坤丽等[70]开发的标注工具完成语料标注工作。将目前多种抽取框架和不同预训练模型应用于医学实体关系的联合抽取,并在CMeIE数据集上进行统计分析,探讨中文医学实体关系抽取任务所面临的多种挑战和未来研究方向。
4.1 不平衡关系抽取数据集中关系分布往往并不平衡,以CMeIE数据集为例,该数据集包含44种关系子类型,其中12种子关系的语料数目占语料总数的80%,而23种子关系的语料数目不足700条。即使通过远程监督等办法能够自动获取大量的训练数据,但由于真实场景中关系和实体对的长尾分布特点,绝大部分的关系和实体对的可用样例仍然较少,而且对于医疗领域的特有关系,受限于数据规模的问题可用样例也很有限。而神经网络模型作为典型的需要大量训练数据支撑的技术,在训练样例过少时性能会受到极大影响。因此,研究者们希望探索有效提升模型学习能力的方法,以更好地利用有限训练样例取得满意的抽取性能。借鉴计算机视觉领域的少次学习研究,通过设计少次学习机制,如元网络[71]、原型网络[72]、GNN[73]和SNAIL[74]等,模型能够利用从过往数据中学到的泛化知识,结合新类型数据的少量训练样本,实现快速迁移学习。
4.2 篇章级关系抽取现有关系抽取工作主要聚焦于句子级关系抽取,即根据句内信息进行关系抽取,各类神经网络模型也擅长编码句子级语义信息,在很多公开评测数据集上取得最佳效果。针对CMeIE数据集的统计结果显示,目前该数据集有28 008条语料,其中13 364条语料属于两个句子拼接而成,占比48%;数据集中每条语料的平均文本长度为85.53字符,限定语料最长为300字符。篇章级三元组抽取即三元组中两个医学实体属于不同的句子,实体间关系也是通过两个句子语义关系表达的。为实现多个实体间的跨句关系抽取,需要对文档中的多个句子进行阅读推理,这显然超出了目前句子级关系抽取模型的能力范围,是未来需要解决的问题之一。
4.3 开放关系抽取BioCreative V数据集和CMeIE数据集是由专家制定标注方案,在实体和实体关系的标注体系及规范指导下标注完成的;NYT数据集和WebNLG数据集则分别是以Freebase知识图谱和DBpedia知识图谱监督方式构建的。然而在医学领域的实体关系抽取任务中,可能会涉及复杂的文本类型和众多的实体关系类型,远远超过人为定义的关系种类数目。在这种情况下,传统的关系分类模型无法有效获取文本中蕴含的实体间的新型关系。如何利用深度学习模型自动发现实体间的新型关系并实现开放关系抽取,具有重要的现实应用价值。
5 结束语为准确高效地扩展知识图谱,从海量非结构化医学文本中自动获取新的世界知识已成为必由之路。以实体关系抽取为代表的知识获取技术已经取得一些成果,特别是近年来深度学习模型极大地推动了关系抽取研究的发展。本文依据数据集的构建方式,详细阐述了监督学习和远程监督的多实例学习,对相关模型的优点和不足进行分析,并探讨了面向医学文本的实体关系抽取任务所面临的多种挑战和未来发展方向。
| [1] |
VRANDEČICĆ D, KRÖTZSCH M. Wikidata: a free collaborative knowledgebase[J]. Communications of the ACM, 2014, 57(10): 78-85. DOI:10.1145/2629489 (  0) 0) |
| [2] |
LEHMANN J, ISELE R, JAKOB M, et al. DBpedia:a large-scale, multilingual knowledge base extracted from Wikipedia[J]. Semantic web, 2015, 6(2): 167-195. DOI:10.3233/SW-140134 (  0) 0) |
| [3] |
BOLLACKER K, EVANS C, PARITOSH P, et al. Freebase: a collaboratively created graph database for structuring human knowledge[C]//Proceedings of the 2008 ACM SIGMOD International Conference on Management of Data. New York: Association for Computing Machinery, 2008: 1247-1250.
(  0) 0) |
| [4] |
DAVIS A P, GRONDIN C J, JOHNSON R J, et al. The comparative toxicogenomics database: update 2017[J]. Nucleic acids research, 2017, 45(1): 972-978. (  0) 0) |
| [5] |
奥德玛, 杨云飞, 穗志方, 等. 中文医学知识图谱CMeKG构建初探[J]. 中文信息学报, 2019, 33(10): 1-9. BYAMBASUREN O, YANG Y F, SUI Z F, et al. Preliminary study on the construction of Chinese medical knowledge graph[J]. Journal of Chinese information processing, 2019, 33(10): 1-9. (  0) 0) |
| [6] |
GRISHMAN R, SUNDHEIM B. Message understanding conference-6: a brief history[C]//Proceedings of the 16th Conference on Computational Linguistics. Morristown: Association for Computational Linguistics, 1996: 466-471.
(  0) 0) |
| [7] |
REBHOLZ-SCHUHMANN D, YEPES A, LI C, et al. Assessment of NER solutions against the first and second CALBC silver standard corpus[J]. Journal of biomedical semantics, 2011, 2(S5): 63-71. (  0) 0) |
| [8] |
HANISCH D, FUNDEL K, MEVISSEN H T, et al. ProMiner: rule-based protein and gene entity recognition[J]. BMC bioinformatics, 2005, 6(S1): 1-9. (  0) 0) |
| [9] |
SAVOVA G K, MASANZ J J, OGREN P V, et al. Mayo clinical Text Analysis and Knowledge Extraction System (cTAKES): architecture, component evaluation and applications[J]. Journal of the American medical informatics association, 2010, 17(5): 507-513. DOI:10.1136/jamia.2009.001560 (  0) 0) |
| [10] |
YANG Z H, LIN H F, LI Y P. Exploiting the performance of dictionary-based bio-entity name recognition in biomedical literature[J]. Computational biology and chemistry, 2008, 32(4): 287-291. (  0) 0) |
| [11] |
LAFFERTY J, MCCALLUM A, PEREIRA F C N. Conditional random fields: probabilistic models for segmenting and labeling sequence data[C]//Proceedings of the 18th International Conference on Machine Learning. San Francisco: Morgan Kaufmann Publishers Inc, 2001: 282-289.
(  0) 0) |
| [12] |
TANG B Z, CAO H X, WU Y H, et al. Recognizing clinical entities in hospital discharge summaries using structural support vector machines with word representation features[J]. BMC medical informatics and decision making, 2013, 13(S1): 1-10. (  0) 0) |
| [13] |
CHANG Y C, DAI H J, WU J Y, et al. TEMPTING system: a hybrid method of rule and machine learning for temporal relation extraction in patient discharge summaries[J]. Journal of biomedical informatics, 2013, 46: 54-62. DOI:10.1016/j.jbi.2013.09.007 (  0) 0) |
| [14] |
UZUNER Ö, SOUTH B R, SHEN S Y, et al. 2010 i2b2/VA challenge on concepts, assertions, and relations in clinical text[J]. Journal of the American medical informatics association, 2011, 18(5): 552-556. DOI:10.1136/amiajnl-2011-000203 (  0) 0) |
| [15] |
WEI C H, PENG Y F, LEAMAN R, et al. Overview of the BioCreative V chemical disease relation (CDR) task[C]//Proceedings of the 5th BioCreative Challenge Evaluation Workshop. Oxford: Oxford University Press, 2015: 154-166.
(  0) 0) |
| [16] |
JELIER R, JENSTER G, DORSSERS L C J, et al. Co-occurrence based meta-analysis of scientific texts: retrieving biological relationships between genes[J]. Bioinformatics, 2005, 21(9): 2049-2058. DOI:10.1093/bioinformatics/bti268 (  0) 0) |
| [17] |
NIKFARJAM A, EMADZADEH E, GONZALEZ G. Towards generating a patient's timeline: extracting temporal relationships from clinical notes[J]. Journal of biomedical informatics, 2013, 46: 40-47. DOI:10.1016/j.jbi.2012.08.002 (  0) 0) |
| [18] |
YANG Y L, LAI P T, TSAI R H. A hybrid system for temporal relation extraction from discharge summaries[C]//International Conference on Technologies and Applications of Artificial Intelligence. Cham: Springer, 2014: 379-386.
(  0) 0) |
| [19] |
LEE J Y, DERNONCOURT F, SZOLOVITS P. MIT at SemEval-2017 task 10: relation extraction with convolutional neural networks[C]//Proceedings of the 11th International Workshop on Semantic Evaluation.Stroudsburg: Association for Computational Linguistics, 2017: 978-984.
(  0) 0) |
| [20] |
SEOL J W, YI W J, CHOI J, et al. Causality patterns and machine learning for the extraction of problem-action relations in discharge summaries[J]. International journal of medical informatics, 2017, 98: 1-12. DOI:10.1016/j.ijmedinf.2016.10.021 (  0) 0) |
| [21] |
杨锦锋, 于秋滨, 关毅, 等. 电子病历命名实体识别和实体关系抽取研究综述[J]. 自动化学报, 2014, 40(8): 1537-1562. YANG J F, YU Q B, GUAN Y, et al. An overview of research on electronic medical record oriented named entity recognition and entity relation extraction[J]. Acta automatica sinica, 2014, 40(8): 1537-1562. (  0) 0) |
| [22] |
鄂海红, 张文静, 肖思琪, 等. 深度学习实体关系抽取研究综述[J]. 软件学报, 2019, 30(6): 1793-1818. E H H, ZHANG W J, XIAO S Q, et al. Survey of entity relationship extraction based on deep learning[J]. Journal of software, 2019, 30(6): 1793-1818. (  0) 0) |
| [23] |
庄传志, 靳小龙, 朱伟建, 等. 基于深度学习的关系抽取研究综述[J]. 中文信息学报, 2019, 33(12): 1-18. ZHUANG C Z, JIN X L, ZHU W J, et al. Deep learning based relation extraction: a survey[J]. Journal of Chinese information processing, 2019, 33(12): 1-18. (  0) 0) |
| [24] |
ZENG X R, ZENG D J, HE S Z, et al. Extracting relational facts by an end-to-end neural model with copy mechanism[C]//Proceedings of the 56th Annual Meeting of the Association for Computational Linguistics.Stroudsburg: Association for Computational Linguistics, 2018: 506-514.
(  0) 0) |
| [25] |
FU T J, LI P H, MA W Y. GraphRel: modeling text as relational graphs for joint entity and relation extraction[C]//Proceedings of the 57th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2019: 1409-1418.
(  0) 0) |
| [26] |
WEI Z, SU J, WANG Y, et al. A novel cascade binary tagging framework for relational triple extraction[C]//Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2020: 1476-1488.
(  0) 0) |
| [27] |
DODDINGTON G. Automatic content extraction (ACE) program-task definitions and performance measures[C]//Proceedings of the 3rd International Conference on Language Resources and Evaluation. Lisbon: European Language Resources Association, 2004: 837-840.
(  0) 0) |
| [28] |
HENDRICKX I, KIM S N, KOZAREVA Z, et al. SemEval-2010 task 8: multi-way classification of semantic relations between pairs of nominals[C]//Proceedings of the 5th International Workshop on Semantic Evaluation. Uppsala: Association for Computational Linguistics, 2010: 33-38.
(  0) 0) |
| [29] |
MINTZ M, BILLS S, SNOW R, et al. Distant supervision for relation extraction without labeled data[C]//Proceedings of the Joint Conference of the 47th Annual Meeting of the Association for Computational Linguistics and the 4th International Joint Conference on Natural Language Processing. Morristown: Association for Computational Linguistics, 2009: 1003-1011.
(  0) 0) |
| [30] |
RIEDEL S, YAO L M, MCCALLUM A. Modeling relations and their mentions without labeled text[M]//Machine Learning and Knowledge Discovery in Databases. Berlin: Springer, 2010: 148-163.
(  0) 0) |
| [31] |
GARDENT C, SHIMORINA A, NARAYAN S, et al. Creating training corpora for NLG micro-planners[C]//Proceedings of the 55th Annual Meeting of the Association for Computational Linguistics.Stroudsburg: Association for Computational Linguistics, 2017: 179-188.
(  0) 0) |
| [32] |
HAN X, ZHU H, YU P F, et al. FewRel: a large-scale supervised few-shot relation classification dataset with state-of-the-art evaluation[C]//Proceedings of the Conference on Empirical Methods in Natural Language Processing.Stroudsburg: Association for Computational Linguistics, 2018: 4803-4809.
(  0) 0) |
| [33] |
LIU C Y, SUN W B, CHAO W H, et al. Convolution neural network for relation extraction[M]//Advanced Data Mining and Applications. Berlin: Springer, 2013: 231-242.
(  0) 0) |
| [34] |
ZENG D, LIU K, LAI S, et al. Relation classification via convolutional deep neural network[C]//Proceedings of the 25th International Conference on Computational Linguistics. Dublin: Dublin City University and Association for Computational Linguistics, 2014: 2335-2344.
(  0) 0) |
| [35] |
SAHU S, ANAND A, ORUGANTY K, et al. Relation extraction from clinical texts using domain invariant convolutional neural network[C]//Proceedings of the 15th Workshop on Biomedical Natural Language Processing.Stroudsburg: Association for Computational Linguistics, 2016: 206-215.
(  0) 0) |
| [36] |
NGUYEN T H, GRISHMAN R. Relation extraction: perspective from convolutional neural networks[C]//Proceedings of the 1st Workshop on Vector Space Modeling for Natural Language Processing.Stroudsburg: Association for Computational Linguistics, 2015: 39-48.
(  0) 0) |
| [37] |
MIKOLOV T, SUTSKEVER I, CHEN K, et al. Distributed representations of words and phrases and their compositionality[C]//Conference and Workshop on Neural Information Processing Systems. La Jolla: Neural Information Processing Systems Foundation Inc, 2013: 3111-3119.
(  0) 0) |
| [38] |
SANTOS C, XIANG B, ZHOU B W. Classifying relations by ranking with convolutional neural networks[C]//Proceedings of the 53rd Annual Meeting of the Association for Computational Linguistics and the 7th International Joint Conference on Natural Language Processing.Stroudsburg: Association for Computational Linguistics, 2015: 626-634.
(  0) 0) |
| [39] |
GU J H, SUN F Q, QIAN L H, et al. Chemical-induced disease relation extraction via convolutional neural network[EB/OL]. [2020-05-01]. https://academic.oup.com/database/article/doi/10.1093/database/bax024/3098440.
(  0) 0) |
| [40] |
GU J H, QIAN L H, ZHOU G D. Chemical-induced disease relation extraction with various linguistic features[EB/OL].[2020-05-12]. https://academic.oup.com/database/article/doi/10.1093/database/baw042/2630319.
(  0) 0) |
| [41] |
ZHANG D X, WANG D. Relation classification via recurrent neural network[EB/OL].[2020-05-08]. https://arxiv.org/abs/1508.01006.
(  0) 0) |
| [42] |
ZHOU H W, DENG H J, CHEN L, et al. Exploiting syntactic and semantics information for chemical-disease relation extraction[EB/OL].[2020-05-12]. https://academic.oup.com/database/article/doi/10.1093/database/baw048/2630338.
(  0) 0) |
| [43] |
NGUYEN D Q, VERSPOOR K. Convolutional neural networks for chemical-disease relation extraction are improved with character-based word embeddings[C]//Proceedings of the Workshop on Biomedical Natural Language Processing.Stroudsburg: Association for Computational Linguistics, 2018: 129-136.
(  0) 0) |
| [44] |
CHIKKA V R, KARLAPALEM K. A hybrid deep learning approach for medical relation extraction[EB/OL]. [2020-05-08]. https://arxiv.org/abs/1806.11189.
(  0) 0) |
| [45] |
ZHOU P, SHI W, TIAN J, et al. Attention-based bidirectional long short-term memory networks for relation classification[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2016: 207-212.
(  0) 0) |
| [46] |
WANG L L, CAO Z, DE MELO G, et al. Relation classification via multi-level attention CNNs[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2016: 1298-1307.
(  0) 0) |
| [47] |
RAMAMOORTHY S, MURUGAN S. An attentive sequence model for adverse drug event extraction from biomedical text[EB/OL].[2020-05-01]. https://arxiv.org/abs/1801.00625.
(  0) 0) |
| [48] |
LI H D, CHEN Q C, TANG B Z, et al. Chemical-induced disease extraction via convolutional neural networks with attention[C]//IEEE International Conference on Bioinformatics and Biomedicine. Washington: IEEE Computer Society, 2017: 1276-1279.
(  0) 0) |
| [49] |
ZHOU H W, LANG C K, LIU Z, et al. Knowledge-guided convolutional networks for chemical-disease relation extraction[J]. BMC bioinformatics, 2019, 20(1): 260-273. (  0) 0) |
| [50] |
BORDES A, USUNIER N, GARCIA-DURAN A, et al. Translating embeddings for modeling multi-relational data[C]//Conference and Workshop on Neural Information Processing Systems. La Jolla: Neural Information Processing Systems Foundation Inc, 2013: 2787-2795.
(  0) 0) |
| [51] |
GORMLEY M R, YU M, DREDZE M. Improved relation extraction with feature-rich compositional embedding models[C]//Proceedings of the Conference on Empirical Methods in Natural Language Processing. Stroudsburg: Association for Computational Linguistics, 2015: 1774-1784.
(  0) 0) |
| [52] |
YU X, LAM W. Jointly identifying entities and extracting relations in encyclopedia text via a graphical model approach[C]//Proceedings of the 23rd International Conference on Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2010: 1399-1407.
(  0) 0) |
| [53] |
LI Q, JI H. Incremental joint extraction of entity mentions and relations[C]//Proceedings of the 52nd Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2014: 402-412.
(  0) 0) |
| [54] |
MIWA M, SASAKI Y. Modeling joint entity and relation extraction with table representation[C]//Proceedings of the Conference on Empirical Methods in Natural Language Processing. Stroudsburg: Association for Computational Linguistics, 2014: 1858-1869.
(  0) 0) |
| [55] |
REN X, WU Z, HE W, et al. Cotype: joint extraction of typed entities and relations with knowledge bases[C]//Proceedings of the 26th International Conference on World Wide Web. Geneva: International World Wide Web Conferences Steering Committee, 2017: 1015-1024.
(  0) 0) |
| [56] |
MIWA M, BANSAL M. End-to-end relation extraction using LSTMs on sequences and tree structures[EB/OL]. [2020-05-01]. https://arxiv.org/abs/1601.00770.
(  0) 0) |
| [57] |
LI F, ZHANG M S, FU G H, et al. A neural joint model for extracting bacteria and their locations[M]//Advances in Knowledge Discovery and Data Mining. Cham: Springer, 2017: 15-26.
(  0) 0) |
| [58] |
ZENG D J, LIU K, CHEN Y B, et al. Distant supervision for relation extraction via piecewise convolutional neural networks[C]//Proceedings of the Conference on Empirical Methods in Natural Language Processing. Stroudsburg: Association for Computational Linguistics, 2015: 1753-1762.
(  0) 0) |
| [59] |
HOFFMANN R, ZHANG C, LING X, et al. Knowledge-based weak supervision for information extraction of overlapping relations[C]//Proceedings of the 49th Annual Meeting of the Association for Computational Linguistics: Human Language Technologies. Stroudsburg: Association for Computational Linguistics, 2011: 541-550.
(  0) 0) |
| [60] |
SURDEANU M, TIBSHIRANI J, NALLAPATI R, et al. Multi-instance multi-label learning for relation extraction[C]//Proceedings of the Joint Conference on Empirical Methods in Natural Language Processing and Computational Natural Language Learning. Stroudsburg: Association for Computational Linguistics, 2012: 455-465.
(  0) 0) |
| [61] |
LIN Y K, SHEN S Q, LIU Z Y, et al. Neural relation extraction with selective attention over instances[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2016: 2124-2133.
(  0) 0) |
| [62] |
JIANG X, WANG Q, LI P, et al. Relation extraction with multi-instance multi-label convolutional neural networks[C]//Proceedings of the 26th International Conference on Computational Linguistics. Osaka: the COLING Organizing Committee, 2016: 1471-1480.
(  0) 0) |
| [63] |
FENG X C, GUO J, QIN B, et al. Effective deep memory networks for distant supervised relation extraction[C]//Proceedings of the 26th International Joint Conference on Artificial Intelligence. Palo Alto: AAAI Press, 2017: 4002-4008.
(  0) 0) |
| [64] |
JI G, LIU K, HE S, et al. Distant supervision for relation extraction with sentence-level attention and entity descriptions[C]//Proceedings of the 26th International Joint Conference on Artificial Intelligence. Palo Alto: AAAI Press, 2017: 3060-3066.
(  0) 0) |
| [65] |
MIWA M, BANSAL M. End-to-end relation extraction using LSTMs on sequences and tree structures[C]//Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg: Association for Computational Linguistics, 2016: 1105-1116.
(  0) 0) |
| [66] |
KATIYAR A, CARDIE C. Going out on a limb: joint extraction of entity mentions and relations without dependency trees[C]//Proceedings of the 55th Annual Meeting of the Association for Computational Linguistics.Stroudsburg: Association for Computational Linguistics, 2017: 917-928.
(  0) 0) |
| [67] |
ZHENG S C, WANG F, BAO H Y, et al. Joint extraction of entities and relations based on a novel tagging scheme[C]//Proceedings of the 55th Annual Meeting of the Association for Computational Linguistics.Stroudsburg: Association for Computational Linguistics, 2017: 1227-1236.
(  0) 0) |
| [68] |
昝红英, 窦华溢, 贾玉祥, 等. 基于多来源文本的中文医学知识图谱的构建[J]. 郑州大学学报(理学版), 2020, 52(2): 45-51. ZAN H Y, DOU H Y, JIA Y X, et al. Construction of Chinese medical knowledge graph based on multi-source corpus[J]. Journal of Zhengzhou university (natural science edition), 2020, 52(2): 45-51. (  0) 0) |
| [69] |
昝红英, 刘涛, 牛常勇, 等. 面向儿科疾病的命名实体及实体关系标注语料库构建及应用[J]. 中文信息学报, 2020, 34(5): 19-26. ZAN H Y, LIU T, NIU C Y, et al. Construction and application of named entity and entity relations corpus for pediatric diseases[J]. Journal of Chinese information processing, 2020, 34(5): 19-26. (  0) 0) |
| [70] |
张坤丽, 赵旭, 关同峰, 等. 面向医疗文本的实体及关系标注平台的构建及应用[J]. 中文信息学报, 2020, 34(6): 36-44. ZHANG K L, ZHAO X, GUAN T F, et al. A platform for entity and entity relationship labeling in medical texts[J]. Journal of Chinese information processing, 2020, 34(6): 36-44. (  0) 0) |
| [71] |
MUNKHDALAI T, YU H. Meta networks[C]//Proceedings of the 34th International Conference on Machine Learning. Massachusetts: the Journal of Machine Learning Research, 2017: 2554-2563.
(  0) 0) |
| [72] |
SNELL J, SWERSKY K, ZEMEL R. Prototypical networks for few-shot learning[C]//Conference and Workshop on Neural Information Processing Systems. Los Angeles: Neural Information Processing Systems Foundation Inc, 2017: 4077-4087.
(  0) 0) |
| [73] |
GARCIA V, ESTRACH J B. Few-shot learning with graph neural networks[EB/OL]. [2020-05-01]. https://arxiv.org/pdf/1711.04043v3.pdf .
(  0) 0) |
| [74] |
MISHRA N, ROHANINEJAD M, CHEN X, et al. A simple neural attentive meta-learner[EB/OL]. [2020-05-01]. https://arxiv.org/pdf/1707.03141.pdf.
(  0) 0) |
 2020, Vol. 52
2020, Vol. 52


