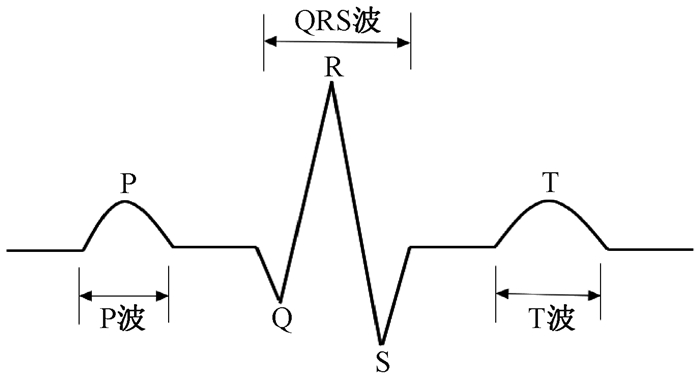
据世界卫生组织报告,每年死于心血管疾病的患者超过一千万人。心电图(ECG)是最常用的非侵入性诊断工具之一,对心电图进行分析,可用于心脏疾病的筛查和诊断[1]。正常的心电图中,每个心跳周期内包含三个主要的特征波:P波、QRS波和T波,如图 1所示。心电特征波能够反映心脏的生理状态信息,通过对其形状、幅值和持续时间的分析,可以用来辅助诊断心血管疾病[2]。对于常见的ECG异常,如心律失常、房颤等,诊断依赖专家和临床医生对ECG进行目视检查。然而,随着心电数据规模不断扩大,分析大量的数据非常耗时,且受到专业知识的限制,及心电专家个人主观判断和经验的影响[3]。
|
图 1 心跳周期示意图 Fig. 1 Schematic diagram of the heartbeat cycle |
近年来,从心电图这种生理时间序列中自动进行特征波分割已成为新的研究热点。传统的信号处理算法有小波变换[4]、信号包络[5]等,它们的缺点是只针对QRS波定位和分割。为了能够同时分割P波和T波,各种基于特征的机器学习方法,如神经网络(NN)[6]、朴素贝叶斯[7]、隐马尔可夫模型(HMM)[8]、高斯核函数模型[9]等已经被用来对ECG特征波进行分割。但基于机器学习的方法是高度特征驱动的[10],心电特征波分割的任务需要具有心电专业知识的专家或临床医生的配合,才能更好地进行特征建模。为了克服机器学习对特征依赖的局限性,基于深度学习(deep learning, DL)的技术被用于心电信号特征波的分割任务,其中卷积神经网络(以U-Net[11]语义分割网络为例)和长短期记忆(long-short-term memory, LSTM)[12]网络是最常用的心电特征波分割方法。
本文主要总结了心电信号中特征波分割方法的相关研究,包括传统信号处理算法、机器学习算法、深度学习方法。依据算法的不同对各种方法进行了梳理和分类,并对各个模型的特点及性能进行了评述。
1 特征波分割的数值准备 1.1 数据集的选取对心电信号中的特征波分割,首先要准备数据,主要包括数据集的选取和数据预处理。
目前,公开可用的心电信号数据库较少,常用的有特征波标注的数据集有QTDB[13]和LUDB[14]。QTDB由105条长度为15 min、采样频率为250 Hz的动态双导联记录组成;LUDB由200条长度为10 s、采样频率为500 Hz的12导联记录组成。这两个公开数据集均对心跳周期中的特征波(包含波的起止点和峰值)进行了注释。此外,MIT-BIH心律失常数据库[15]、中国心血管疾病数据库(Chinese cardiovascular disease database,CCDD)[16]等数据集也有部分特征波的标签信息。
1.2 数据的预处理心电信号的预处理主要包括去噪、归一化处理、数据增强等。
去噪主要是去除原始ECG信号中基线漂移噪声。基线漂移是一种常见的低频心电伪影噪声,由患者的呼吸或运动引起,不包含任何诊断信息。通常使用带有低通和高通滤波器的离散小波变换(DWT)[17]进行基线噪声的去除,这也是对ECG信号进行处理的第一步。
归一化处理主要是处理心电信号中可能存在奇异的样本数据从而增加模型训练时间、导致模型收敛失败的问题。通常心电信号使用的是最大最小归一化方法[18],可以使所有输入心电信号的均值接近0,该方法的公式为
| $ x^{\prime}=\frac{x-\min (x)}{\max (x)-\min (x)}, $ | (1) |
其中:x是输入的原始信号;x'是经过归一化处理后的输出信号。
数据增强策略常用来扩充心电数据的数量,这是由于公开可用数据集的数据量较少,并且记录内的心拍变异性较低。常用的心电信号数据增强方法有缩放电压、裁剪、延时、添加高斯噪声、添加基线漂移等[19],这些方法在扩充数据量的同时,也保证了心电信号的生理特性不被改变。
2 基于信号处理的特征波分割算法应用在心电信号特征波分割任务上的信号处理算法有小波变换[20-23]、信号包络[24-26]等。下面介绍这两种常见的信号处理算法。
2.1 小波变换在信号处理算法中,小波变换法已经成为一种非常成熟的心电信号特征波识别算法,该方法在处理心电信号这种时序数据上具有很大优势。
1995年Li等[20]开创性使用小波变换来检测心电的特征点,借助小波变换的多尺度功能,将QRS波与其他的高P波或T波区分出来,在MIT-BIH数据库上识别QRS波的准确率达到99.80%。Martínez等[21]使用小波二次样条作为原型,通过识别矩形脉冲卷积的导数来定位特征点,该算法提供了准确可靠的心电图信号分割结果,特别是对T波终点的定位有显著的改善。Di Marco等[22]使用基于导数的离散小波变换,提出在严格功率约束和有限计算资源下,在线检测QRS波和描绘心电图的合适候选算法,该算法采用线性代数的简化结构,成功用于长期非诊断性动态监测的可穿戴设备。Kalyakulina等[23]使用双正交小波变换,提出将基于导数的离散小波变换简化为构建一组高频和低频滤波器来处理心电信号,该算法允许使用多导联心电信号进行校正以提高分割质量。
2.2 信号包络将输入心电信号的高频峰值点连线,就可以得到上方一条线(正)和下方一条线(负),这两条线称为心电信号的包络线,包络线是反映高频信号振幅变化的曲线。使用信号包络方法检测心电特征波主要步骤有:带通滤波、R峰值检测、计算滤波后心电图的包络、加窗和后处理分割操作。
Illanes-Manriquez等[24]提出了一种用于QRS波起始和偏移检测的新算法,该算法的独创性在于它主要计算与QRS波包络覆盖区域的相关指标,该包络信号具有与Q波和S波有关的凸波特性,并在多个数据集上已经证明其稳健性和有效性。Park等[25]提出了一种基于小波变换(WT)和改进的香农能量包络的快速QRS波检测方法,通过小波变换来去除噪声,再从SEE中提取峰值能量包络并估计R峰值,并结合所提出的后处理方法有效验证每个检测峰值并提示漏检,进一步提高了峰值检测精度。Xu等[26]提出了一种基于香农能量包络和希尔伯特变换的QRS波检测算法,对包络信号进行希尔伯特变换,然后通过检测过零位置识别包络上的R峰,所提出的检测算法在复杂干扰下仍能正确检测出QRS波,具有较强的鲁棒性。
3 基于机器学习的特征波分割算法应用在心电特征波分割任务上的基于机器学习的算法有神经网络(NN)[27-29]、贝叶斯模型[30-32]、隐马尔可夫模型(HMM)[33-36]、高斯核函数模型[37-39]等。下面介绍这四种常见的机器学习算法。
3.1 神经网络神经网络(neural network,NN)是一种仿生学模型,仿照了生物神经网络的组成和功能,一般由输入层、隐藏层和输出层组成。
Celleret等[27]首次提出了将小波变换和神经网络结合用于ECG诊断的方法,使用Daubechies和Symlet小波将心电图信号分解为6个级别,然后从这6个级别中提取统计参数并作为神经网络下流任务的输入。模糊神经网络(fuzzy neural network,FNN)被证明比常规神经网络具有更高的抗噪声能力,Shyu等[28]将FNN加入检测系统中,在QRS波分割任务和室性早搏的分类任务中取得了很好的结果,该算法使用相同的小波基提取特征,有效地消除了高频和低频噪声的影响,在实现过程中计算量较小且复杂度较低。Andrysiak等[29]提出了一种基于基函数的信号稀疏表示方法,使用人工神经网络检测QRS波并分析ECG信号中的异常,将信号的稀疏特征与神经网络建模的分类属性相结合,获得了令人满意的QRS波检测效果,并具有较高的心电信号异常检测效率。
3.2 贝叶斯模型使用贝叶斯方法进行心电信号特征波的分割,首先要建立一个非QRS波间期的信号模型,用脉冲来近似代表P波和T波,然后定义似然函数、先验分布和后验分布,最后使用采样器进行心电信号特征提取。
2010年Lin等[30]引入部分坍塌的Gibbs采样器原理,采用贝叶斯算法和马尔可夫链蒙特卡罗算法同时进行特征波的分割,充分利用了心电信号的强局部依赖性。该算法不仅可以检测P波和T波的峰值和边界,还准确估计了每个分析窗口的波形。2014年Lin等[31]在原有基础上进一步改进模型,首次提出了一种块Gibbs采样器,利用心电信号的局部依赖性和序列性质,加快了采样器的收敛速度,还提出了利用边缘化的粒子滤波器来估计动态模型的未知参数的序列蒙特卡罗方法,使得该算法需要更小的内存需求和更低的计算复杂度。Chin等[32]提出了一种基于贝叶斯框架的新方法,融合了方差检测和最大似然估计,分别确定QRS波的起始点、持续时间和周期,能够对QRS波进行实时检测,该方法被验证易于迁移到不同的数据库中,因此具有良好的鲁棒性。
3.3 隐马尔可夫模型使用隐马尔可夫模型进行心电信号特征波的分割,首先利用小波变换提取的参数来表征不同特征波的振幅类别,然后选用特定的HMM对每个特征波分段进行建模,最终实现对心电特征波的分割。
Graja等[33]提出了一种基于隐马尔可夫树模型的ECG描绘方法,这种方法可以通过树结构将ECG特征波与小波系数连接起来,从而检测波的变化,该方法与基于上、下文概念的量表间融合方法相关联,成功应用在QT数据集上的特征波分割任务。Krimi等[34]将小波变换与HMM框架相结合,利用HMM解决了ECG特征波分割和分类如何正确选择小波变换的问题,并证明每个小波函数都有其自身的特殊性,多尺度小波组合结果一般更优。Akhbari等[35]提出多隐马尔可夫模型(MultiHMM),该方法为每个ECG段训练一个HMM模型,在训练步骤中执行一个粗略的分割来定义每个HMM的训练数据,此模型的优势在于能够成功分割病理性搏动(双相波),并能解决双拍分割问题。Thomas等[36]提出了一种时间无约束的隐马尔可夫模型,用于对非均匀的ECG特征波进行分割,每个特征波使用一个HMM表示,该模型即使在低平均采样率的条件下也能够显示出较高的分割准确性。
3.4 高斯核函数模型高斯核函数是一种参数化函数,用于模拟波形的变化,高斯函数的拟合优度可以作为心电特征波发生的可能性,并可以结合波形的先验知识来实现精确描绘。
Dubois等[37]提出使用广义正交回归(GOFR)和特定的参数化高斯mesa函数(GMF)结合的方法,GOFR将心电信号分解为高斯mesa函数,每个波都由一个GMF建模,该模型具有更高的可解释性,并在QT数据集上评估了QRS波的检测效率。González等[38]提出了一种基于信号微分的自适应P波分割方法,引入了已检测P波形态的自适应斜率阈值并创建每个P波的高斯模型来辅助分割,该算法可用于识别心房内电特性的渐进性变化,这有助于预测心律失常(如心房颤动)的发生。Rao等[39]提出了一种基于双混合高斯函数(GF)和动态规划的对P波和T波进行检测和分割的方法,使用峰值位置的先验知识,对P波和T波在相邻心电周期中的位置使用平滑度约束,实验证明该算法在ECG信号的不同波形形态中准确描绘了P波和T波的峰值。
表 1是对机器学习算法应用在特征波分割任务的总结。
|
|
表 1 基于机器学习的特征波分割算法总结 Tab. 1 Summary of feature-wave segmentation algorithm based on machine learning |
深度学习算法最大的特点是需要大量数据训练,根据预定义的标签标记每个心电信号,再用来训练,使计算机能够自动识别任何给定的心电信号。深度学习与人类专家相比,最大的优势在于能够无限期地学习,不断提高模型的性能和鲁棒性[40]。
近年来,深度学习在远程医疗和生物医学工程中的应用呈指数级增长。这些方法已被用于解决生物医学信号中的许多挑战,例如ECG心律失常分类[41]和脑电图(EEG)信号分类[42]。卷积神经网络(CNN)、循环神经网络(RNN),以及两者结合卷积循环神经网络(CRNN)可以进一步提高模型性能。下面介绍这三种常见的深度学习算法在心电特征波分割任务上的应用。
4.1 卷积神经网络卷积神经网络,简称CNN,是一类深度神经网络,由卷积层组成,其中每一层在训练过程中通过反向传播自动学习其核心系数[43]。心电信号是一维的时序数据,因此使用CNN的变体1-D-CNN来对时间序列信号进行操作。
Camps等[44]提出了一个特征波分割系统,该系统由一个分段模块和一个分割模块组成,每个模块由一个ConvNet和NN组成,第一个模块识别QRS波,第二个模块分别定位QRS波的起始点和偏移量。Chen等[45]提出了一种新的端到端深度学习特征波分割方案,称为区域聚合网络,可以取代传统的全连通层作为回归器用于检测心电信号的特征点,并使用数据增强方法弥补QT数据库中数据不足的问题,提高了特征波的检测精度。Habib等[46]提出了一种新的基于两级注意力机制的CNN模型结构,用于QRS波的检测,并在公开的ECG数据库上对基于数据库内和数据库间的泛化能力进行测试,以探索其对不同数据集的依赖性。Jia等[47]提出了一种由多个CNN和后处理网络组成的基本条件和由序列条件随机场(CRF)组成的辅助处理网络,创造性地将ECG分割问题看作一维信号分割问题。Jimenez等[48]分析了U-Net架构对一维数据的适应性,旨在检测和分割心电图的P波、QRS波和T波,并在QT数据集上进行验证。Lee等[49]提出了一种FCN模型,直接训练从输入到真值数据的映射,然后通过FCN模型输出一个预测映射,其中包含关于输入的单个样本的分类信息,最终由FCN模型对输入的单个样本是否属于QRS波进行分类。Moskalenko等[50]提出了一种基于类U-Net全卷积神经网络的ECG分割算法模型,该算法接收任意采样率的ECG信号作为输入,使用的参数少,具有良好的泛化能力。Duraj等[51]提出一种基于压缩激励块(通过使网络能够执行通道特性校准来提高网络的代表性能力)的1-D残差U-Net模型,在12导联的ECG信号中提取特征波。Yu等[52]首次提出了基于CNN的QRS波检测方法,该方法使用更快的RCNN直接检测图像中的QRS波,然后通过CNN定位和测量R峰,最后在ST-ECG数据集上进行了分割效果检验。
4.2 循环神经网络循环神经网络(RNN)是一种旨在对时序数据建模的神经网络。相比CNN,RNN可以更好地学习心电时序信号中的长期依赖关系,因此更适用于心电特征波分割任务,但RNN在处理长期依赖关系时经常遇到梯度消失或梯度爆炸问题,因此GRU、LSTM和双向LSTM(Bi-LSTM)作为RNN变体,被用于解决经典RNN中存在的梯度消失等关键问题[53]。
Peimankar等[54]提出了一种端到端的深度学习方法来检测心电信号中的P波,在QTDB数据集上使用5倍交叉验证对四种不同的分类器(LSTM-CE、LSTM-SSE、BiLSTM-CE和BiLSTM-SSE)进行训练,最后使用集成学习理论对四种分类器进行融合,输出最优特征波分割精度。Malali等[55]提出了一种卷积长短时记忆(ConvLSTM)神经网络来分割心电波形,它由一个卷积层和一个双向LSTM架构组成,添加额外的特征(如心电波的导数以及平滑的心电波)并结合自注意力机制,该深度学习模型的性能明显优于现有的信号处理和基于HMM的方法。Abrishami等[56]提出一种基于长短时记忆层和循环神经网络(BiLSTM-RNN)的心电信号分割方法来检测特征波,用BiLSTM可以学习心电信号的长期相关性,能够根据QRS波之前的采样点(如P波)和之后的采样点(如S波)来定位QRS波,该方法适用于心电特征波分割任务,特别是在T波分割准确率上有很大的提升。
4.3 卷积循环神经网络卷积循环神经网络(CRNN)将CNN与RNN相结合,在空间和时间上同时对心电信号进行特征提取,进一步提升了特征波的分割效果[57]。
He等[58]提出了一种基于U-Net和Bi-LSTM的双通道自动检测QRS波的新算法,采用“平滑标签”方法改善R波峰标注的不平衡性,该算法不仅在大型ECG数据库的QRS波检测中表现良好,而且可以扩展到其他医学信号研究领域。Peimankar等[59]提出了一种实时对心拍进行分割的深度学习模型,称为DENS-ECG,该模型使用两层Bi-LSTM并结合集成学习来检测心电信号的特征波,实验证明该方法具有良好的灵活性和准确性,可用于实时远程健康监测,帮助心脏科医生居家分析心电信号。Londhe等[60]提出了一种更加鲁棒的心电特征波分割模型,由通道混合卷积层和Bi-LSTM层组成,该混合模型能够提取前向和后向时间戳中的时间依赖以及短时和长时依赖,能够在连续原始的ECG信号中对特征波进行分割。
表 2是对基于深度学习算法应用在特征波分割任务上的总结。
|
|
表 2 基于深度学习的特征波分割算法总结 Tab. 2 Summary of feature wave segmentation algorithm based on deep learning |
尽管深度学习方法在各项指标已经取得很高的评分,但是缺乏大规模、高质量的心电信号数据库,缺乏数字支持(许多医院仍然使用打印纸质心电图),缺乏详细的节拍级标记的数据,限制了心电信号的进一步研究[61]。
因此另一个研究方向是通过对信号域中的相关问题进行迁移学习,迁移学习能够将知识从标记的源域转移到目标域, 用来自旧域的标记图像学习精确分类新域的图像[62]。通过执行半监督学习或自监督预训练,来获得更好的潜在表示,而自监督对比学习[63]是目前应用在生理信号上的一个最新技术,该技术减少了对标记数据的过度依赖,增强了训练模型的鲁棒性。
5 自监督学习算法深度监督学习在过去十年中取得了巨大的成功,但它严重依赖高质量数据标签和易受攻击的缺陷促使人们在ECG分割方面寻找其他的解决模式,促进自监督学习算法在心电特征波分割领域的发展。
5.1 自监督学习概述总体来说,自监督学习方法分为两个阶段:自监督预训练阶段(特征提取)和下流任务定义(监督微调)阶段。第一阶段通过编码器从大规模的无标签数据中挖掘自身的监督信息,进行特征的提取,对模型进行预训练;第二阶段通过定义具体的下流任务(特征波分割或异常分类任务),使用少量的有标签数据对前一阶段的预训练模型进行监督微调和优化[64]。常见的自监督模型架构有:MoCo[65]、SimCLR[66]等。
5.2 在医学数据上的应用近年来,人们将自监督学习应用于医学成像任务,在影像学、病理学和皮肤病学等各种领域中取得了很好的效果。目前,自监督学习已经成功应用于心电信号的异常分类[67]和情绪分类[68]、脑电图的睡眠分期和病理学检测[69]、分类胸部X光的数据[70],成功实现细胞、乳腺肿瘤、皮肤病变和脑肿瘤的分割[71]。
自监督学习应用在心电特征波分割任务的研究还较少,但从自监督学习本身的优势可以看出,该任务在未来一定会有更多的探索和实现。
6 总结与展望本文对心电信号特征波的分割任务进行了全面回顾和总结,重点关注了不同的算法模型。对传统信号处理算法、机器学习算法、深度学习算法应用于该任务的模型和性能进行了总结,并介绍了自监督学习算法及其应用在心电特征波分割任务上的可行性。
研究表明,主流深度学习已经达到了较高的水平。在性能上,它们可以取代传统的人工特征提取和特定自动分类器相结合的机器学习方法,但是目前主流心电特征波分割方法也存在着诸多的问题与挑战。
(1) 缺乏大规模、高质量的心电信号特征波标注的公开数据库,并且标签信息具有不确定性,可能由于心电专家的误判造成数据信息错误。
(2) 目前已有算法模型的迁移性和鲁棒性较差。心电波形因人而异,甚至同一人在不同时期的心电波形也会有很大差别,这些差别可能是病理条件甚至是情绪变化导致。因此需要开发一个鲁棒性更强的心电特征波分割模型。
(3) 目前理论研究仅考虑敏感性、特异性和阳性预测率等评测指标,不足以支持其临床应用,今后应考虑对网络结构进行优化,并提升模型的鲁棒性。
(4) 随着便携式可穿戴医疗系统的发展,在未来的研究中需要考虑平台化、用户易用性和实用性。
针对上述问题与挑战,应加强与医院的合作,在保护患者隐私的前提下,利用医院存储的大量心电数据进行研究应用。另外,通过建立数据处理规则,提高模型鲁棒性。最终实现良好的可用性和用户可操作性的系统。
| [1] |
KAUR G, SINGH G, KUMAR V. A review on biometric recognition[J]. International journal of bio-science and bio-technology, 2014, 6(4): 69-76. DOI:10.14257/ijbsbt.2014.6.4.07 (  0) 0) |
| [2] |
COUCEIRO R, CARVALHO P, HENRIQUES J, et al. Detection of atrial fibrillation using model-based ECG analysis[C]//The 19th International Conference on Pattern Recognition. Piscataway: IEEE Press, 2008: 1-5.
(  0) 0) |
| [3] |
IWASAKI Y K, NISHIDA K, KATO T, et al. Atrial fibrillation pathophysiology: implications for management[J]. The Journal of innovations in cardiac rhythm management, 2011, 124(20): 2264-2274. (  0) 0) |
| [4] |
CESARI M, MEHLSEN J, MEHLSEN A B, et al. A new wavelet-based ECG delineator for the evaluation of the ventricular innervation[J]. IEEE journal of translational engineering in health and medicine, 2017, 5: 2000215. (  0) 0) |
| [5] |
MANIKANDAN M S, SOMAN K P. A novel method for detecting R-peaks in electrocardiogram (ECG) signal[J]. Biomedical signal processing and control, 2012, 7(2): 118-128. DOI:10.1016/j.bspc.2011.03.004 (  0) 0) |
| [6] |
GUPTA V, MITTAL M. Blood pressure and ECG signal interpretation using neural network[J]. International journal applied engine research, 2018, 13(6): 127-132. (  0) 0) |
| [7] |
AKHBARI M, SHAMSOLLAHI M B, JUTTEN C. Comparison of ECG fiducial point extraction methods based on dynamic Bayesian network[C]//2017 Iranian Conference on Electrical Engineering. Piscataway: IEEE Press, 2017: 95-100.
(  0) 0) |
| [8] |
NIEDERHAUSER T, HAEBERLIN A, JESACHER B, et al. Model-based delineation of non-uniformly sampled ECG signals[C]//Computing in Cardiology. Piscataway: IEEE Press, 2017: 1-4.
(  0) 0) |
| [9] |
SPICHER N, KUKUK M. ECG delineation using a piecewise Gaussian derivative model with parameters estimated from scale-dependent algebraic expressions[C]//The 41st Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Piscataway: IEEE Press, 2019: 5633-5637.
(  0) 0) |
| [10] |
PEIMANKAR A, PUTHUSSERYPADY S. Ensemble learning for detection of short episodes of atrial fibrillation[C]//The 26th European Signal Processing Conference. Piscataway: IEEE Press, 2018: 66-70.
(  0) 0) |
| [11] |
RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-assisted Intervention. Cham: SpringerInternational Publishing, 2015: 234-241.
(  0) 0) |
| [12] |
GRAVES A. Supervised sequence labelling with recurrent neural networks[J]. Nural computation, 1997, 9(8): 1735-1780. DOI:10.1162/neco.1997.9.8.1735 (  0) 0) |
| [13] |
LAGUNA P, MARK R G, GOLDBERG A, et al. Database for evaluation of algorithms for measurement of QT and other waveform intervals in the ECG[J]. Computers in cardiology, 1997, 24: 673-676. (  0) 0) |
| [14] |
KALYAKULINA A I, YUSIPOV I I, MOSKALENKO V A, et al. LUDB: a new open-access validation tool for electrocardiogram delineation algorithms[J]. IEEE access, 2020, 8: 186181-186190. DOI:10.1109/ACCESS.2020.3029211 (  0) 0) |
| [15] |
MOODY G B, MARK R G. The impact of the MIT-BIH arrhythmia database[J]. IEEE engineering in medicine and biology magazine, 2001, 20(3): 45-50. DOI:10.1109/51.932724 (  0) 0) |
| [16] |
ZHANG J W, LIU X, DONG J. CCDD: an enhanced standard ECG database with its management and annotation tools[J]. International journal on artificial intelligence tools, 2012, 21(5): 1-26. (  0) 0) |
| [17] |
AQIL M, JBARI A, BOUROUHOU A. ECG signal denoising by discrete wavelet transform[J]. International journal of online engineering, 2017, 13(9): 51-68. DOI:10.3991/ijoe.v13i09.7159 (  0) 0) |
| [18] |
SUN P, WU Q H, WEINDLING A M, et al. An improved morphological approach to background normalization of ECG signals[J]. IEEE transactions on biomedical engineering, 2003, 50(1): 117-121. DOI:10.1109/TBME.2002.805486 (  0) 0) |
| [19] |
STABENAU H F, BRIDGE C P, WAKS J W. ECGAug: a novel method of generating augmented annotated electrocardiogram QRST complexes and rhythm strips[J]. Computers in biology and medicine, 2021, 134: 104408. DOI:10.1016/j.compbiomed.2021.104408 (  0) 0) |
| [20] |
LI C, ZHENG C, TAI C. Detection of ECG characteristic points using wavelet transforms[J]. Malaria journal, 1995, 42(1): 21-28. (  0) 0) |
| [21] |
MARTÍNEZ J P, ALMEIDA R, OLMOS S, et al. A wavelet-based ECG delineator: evaluation on standard databases[J]. IEEE transactions on bio-medical engineering, 2004, 51(4): 570-581. DOI:10.1109/TBME.2003.821031 (  0) 0) |
| [22] |
DI MARCO L Y, CHIARI L. A wavelet-based ECG delineation algorithm for 32-bit integer online processing[J]. Biomedical engineering online, 2011, 10: 23. DOI:10.1186/1475-925X-10-23 (  0) 0) |
| [23] |
KALYAKULINA A I, YUSIPOV I I, MOSKALENKO V A, et al. Finding morphology points of electrocardiographic-signal waves using wavelet analysis[J]. Radiophysics and quantum electronics, 2019, 61(8): 689-703. (  0) 0) |
| [24] |
ILLANES-MANRIQUEZ A, ZHANG Q. An algorithm for robust detection of QRS onset and offset in ECG signals[J]. Computers in cardiology, 2008, 35: 857-860. (  0) 0) |
| [25] |
PARK J S, LEE S W, PARK U. R peak detection method using wavelet transform and modified Shannon energy envelope[J]. Journal of healthcare engineering, 2017, 2017: 4901017. (  0) 0) |
| [26] |
XU W S, DU F Y. A robust QRS complex detection method based on Shannon energy envelope and Hilbert transform[J]. Journal of mechanics in medicine and biology, 2022, 22(3): 2240013. DOI:10.1142/S0219519422400139 (  0) 0) |
| [27] |
CELLERET B G, CHAZAL P. Low computational cost classifiers for ECG diagnosis using neural networks[C]//Proceedings of the 20th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Piscataway: IEEE Press, 1998: 1337-1340.
(  0) 0) |
| [28] |
SHYU L Y, WU Y H, HU W. Using wavelet transform and fuzzy neural network for VPC detection from the Holter ECG[J]. IEEE transactions on bio-medical engineering, 2004, 51(7): 1269-1273. DOI:10.1109/TBME.2004.824131 (  0) 0) |
| [29] |
ANDRYSIAK T. Machine learning techniques applied to data analysis and anomaly detection in ECG signals[J]. Applied artificial intelligence, 2016, 30(6): 610-634. DOI:10.1080/08839514.2016.1193720 (  0) 0) |
| [30] |
LIN C, MAILHES C, TOURNERET J Y. P-and T-wave delineation in ECG signals using a Bayesian approach and a partially collapsed Gibbs sampler[J]. IEEE transactions on bio-medical engineering, 2010, 57(12): 2840-2849. DOI:10.1109/TBME.2010.2076809 (  0) 0) |
| [31] |
LIN C, KAIL G, GIREMUS A, et al. Sequential beat-to-beat P and T wave delineation and waveform estimation in ECG signals: block Gibbs sampler and marginalized particle filter[J]. Signal processing, 2014, 104: 174-187. DOI:10.1016/j.sigpro.2014.03.011 (  0) 0) |
| [32] |
CHIN W L, CHANG C C, TSENG C L, et al. Bayesian real-time QRS complex detector for healthcare system[J]. IEEE internet of things journal, 2019, 6(3): 5540-5549. DOI:10.1109/JIOT.2019.2903530 (  0) 0) |
| [33] |
GRAJA S, BOUCHER J M. Hidden Markov tree model applied to ECG delineation[J]. IEEE transactions on instrumentation and measurement, 2005, 54(6): 2163-2168. DOI:10.1109/TIM.2005.858568 (  0) 0) |
| [34] |
KRIMI S, OUNI K, ELLOUZE N. An approach combining wavelet transform and hidden Markov models for ECG segmentation[C]//The 3rd International Conference on Information and Communication Technologies: From Theory to Applications. Piscataway: IEEE Press, 2008: 1-6.
(  0) 0) |
| [35] |
AKHBARI M, SHAMSOLLAHI M B, SAYADI O, et al. ECG segmentation and fiducial point extraction using multi hidden Markov model[J]. Computers in biology and medicine, 2016, 79: 21-29. DOI:10.1016/j.compbiomed.2016.09.004 (  0) 0) |
| [36] |
THOMAS J, ROSE C, CHARPILLET F. A multi-HMM approach to ECG segmentation[C]//The 18th IEEE International Conference on Tools with Artificial Intelligence. Piscataway: IEEE Press, 2006: 609-616.
(  0) 0) |
| [37] |
DUBOIS R, MAISON-BLANCHE P, QUENET B, et al. Automatic ECG wave extraction in long-term recordings using Gaussian mesa function models and nonlinear probability estimators[J]. Computer methods and programs in biomedicine, 2007, 88(3): 217-233. DOI:10.1016/j.cmpb.2007.09.005 (  0) 0) |
| [38] |
GONZÁLEZ F, ALCARAZ R, RIETA J J. Electrocardiographic P-wave delineation based on adaptive slope Gaussian detection[C]//Computing in Cardiology Conference. Piscataway: IEEE Press, 2017: 1-4.
(  0) 0) |
| [39] |
RAO A, GUPTA P, GHOSH P K. P-and T-wave delineation in ECG signals using parametric mixture Gaussian and dynamic programming[J]. Biomedical signal processing and control, 2019, 51: 328-337. DOI:10.1016/j.bspc.2019.03.001 (  0) 0) |
| [40] |
TANG J J, LI X X, HUA J, et al. A review of classification of ECG detection based on neural network[J]. Computer applications and software, 2021, 38(5): 1-9. (  0) 0) |
| [41] |
KIYASSEH D, ZHU T, CLIFTON D A. CLOCS: contrastive learning of cardiac signals across space, time, and patients[C]//International Conference on Machine Learning. New York: ACM Press, 2021: 5606-5615.
(  0) 0) |
| [42] |
BANVILLE H, CHEHAB O, HYVÄRINEN A, et al. Uncovering the structure of clinical EEG signals with self-supervised learning[J]. Journal of neural engineering, 2021, 18(4): 046020. DOI:10.1088/1741-2552/abca18 (  0) 0) |
| [43] |
WANG Y H, TIE Y, WANG F, et al. Tumor type prediction based on Hilbert curve and convolutional neural network[J]. Journal of Zhengzhou university (natural science edition), 2021, 53(4): 89-94. (  0) 0) |
| [44] |
CAMPS J, RODRíGUEZ B, MINCHOLÉ A. Deep learning based QRS multilead delineator in electrocardiogram signals[C]//Computing in Cardiology Conference. Piscataway: IEEE Press, 2018, 1-4.
(  0) 0) |
| [45] |
CHEN M, WANG G J, XIE P W, et al. Region aggregation network: improving convolutional neural network for ecg characteristic detection[C]//The 40th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Piscataway: IEEE Press, 2018: 2559-2562.
(  0) 0) |
| [46] |
HABIB A, KARMAKAR C, YEARWOOD J. Impact of ECG dataset diversity on generalization of CNN model for detecting QRS complex[J]. IEEE access, 2019, 7: 93275-93285. DOI:10.1109/ACCESS.2019.2927726 (  0) 0) |
| [47] |
JIA D, ZHAO W, LI Z, et al. An electrocardiogram delineator via deep segmentation network[C]//The 41st Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Piscataway: IEEE Press, 2019: 1913-1916.
(  0) 0) |
| [48] |
JIMENEZ G, ALCAINE A, CAMARA O. U-net architecture for the automatic detection and delineation of the electrocardiogram[C]//2019 Computing in Cardiology. Piscataway: IEEE Press, 2019: 1-4.
(  0) 0) |
| [49] |
LEE J S, LEE S J, CHOI M, et al. QRS detection method based on fully convolutional networks for capacitive electrocardiogram[J]. Expert systems with applications, 2019, 134: 66-78. DOI:10.1016/j.eswa.2019.05.033 (  0) 0) |
| [50] |
MOSKALENKO V, ZOLOTYKH N, OSIPOV G. Deep learning for ECG segmentation[C]//International Conference on Neuroinformatics. Cham: Springer International Publishing, 2019: 246-254.
(  0) 0) |
| [51] |
DURAJ K, PIASECZNA N, KOSTKA P, et al. Semantic segmentation of 12-lead ECG using 1D residual U-net with squeeze-excitation blocks[J]. Applied sciences, 2022, 12(7): 3332. (  0) 0) |
| [52] |
YU R, GAO Y, DUAN X, et al. QRS detection and measurement method of ECG paper based on convolutional neural networks[C]//The 40th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Piscataway: IEEE Press, 2018: 4636-4639.
(  0) 0) |
| [53] |
XU X, LIU H. ECG heartbeat classification using convolutional neural networks[J]. IEEE access, 2020, 8: 8614-8619. (  0) 0) |
| [54] |
PEIMANKAR A, PUTHUSSERYPADY S. An ensemble of deep recurrent neural networks for P-wave detection in electrocardiogram[C]//IEEE International Conference on Acoustics, Speech and Signal Processing. Piscataway: IEEE Press, 2019: 1284-1288.
(  0) 0) |
| [55] |
MALALI A, HIRIYANNAIAH S, SIDDESH G M, et al. Supervised ECG wave segmentation using convolutional LSTM[J]. ICT express, 2020, 6(3): 166-169. (  0) 0) |
| [56] |
ABRISHAMI H, HAN C, ZHOU X, et al. Supervised ecg interval segmentation using LSTM neural network[C]//Proceedings of the International Conference on Bioinformatics & Computational Biology. Piscataway: IEEE Press, 2018: 71-77.
(  0) 0) |
| [57] |
SODMANN P, VOLLMER M, NATH N, et al. A convolutional neural network for ECG annotation as the basis for classification of cardiac rhythms[J]. Physiological measurement, 2018, 39(10): 104005. (  0) 0) |
| [58] |
HE R, LIU Y, WANG K, et al. Automatic detection of QRS complexes using dual channels based on U-net and bidirectional long short-term memory[J]. IEEE journal of biomedical and health informatics, 2021, 25(4): 1052-1061. (  0) 0) |
| [59] |
PEIMANKAR A, PUTHUSSERYPADY S. DENS-ECG: A deep learning approach for ECG signal delineation[J]. Expert systems with applications, 2021, 165: 113911. (  0) 0) |
| [60] |
LONDHE A N, ATULKAR M. Semantic segmentation of ECG waves using hybrid channel-mix convolutional and bidirectional LSTM[J]. Biomedical signal processing and control, 2021, 63: 102162. (  0) 0) |
| [61] |
CHEPLYGINA V, BRUIJNE M, PLUIM J P W. Not-so-supervised: a survey of semi-supervised, multi-instance, and transfer learning in medical image analysis[J]. Medical image analysis, 2019, 54: 280-296. (  0) 0) |
| [62] |
黄露, 曾庆山. 基于平衡概率分布和实例的迁移学习算法[J]. 郑州大学学报(理学版), 2020(3): 55-61. HUANG L, ZENG Q S. Balanced distribution adaptation and instance based transfer learning algorithm[J]. Journal of Zhengzhou university (natural science edition), 2020(3): 55-61. (  0) 0) |
| [63] |
JAISWAL A, BABU A R, ZADEH M Z, et al. A survey on contrastive self-supervised learning[J]. Technologies, 2020, 9(1): 2. (  0) 0) |
| [64] |
LIU X, ZHANG F J, HOU Z Y, et al. Self-supervised learning: Generative or contrastive[J]. IEEE transactions on knowledge and data engineering, 2021, 99: 1. (  0) 0) |
| [65] |
HE K, FAN H, WU Y, et al. Momentum contrast for unsupervised visual representation learning[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE Press, 2020: 9729-9738.
(  0) 0) |
| [66] |
CHEN T, KORNBLITH S, NOROUZI M, et al. A simple framework for contrastive learning of visual representations[C]//International Conference on Machine Learning. New York: ACM Press, 2020: 1597-1607.
(  0) 0) |
| [67] |
MEHARI T, STRODTHOFF N. Self-supervised representation learning from 12-lead ECG data[J]. Computers in biology and medicine, 2022, 141: 105114. (  0) 0) |
| [68] |
SARKAR P, ETEMAD A. Self-supervised ECG representation learning for emotion recognition[J]. IEEE transactions on affective computing, 2020, 11(3): 1541-1554. (  0) 0) |
| [69] |
BANVILLE H, ALBUQUERQUE I, HYVÄRINEN A, et al. Self-supervised representation learning from electroencephalography signals[C]//IEEE 29th International Workshop on Machine Learning for Signal Processing. Piscataway: IEEE Press, 2019: 1-6.
(  0) 0) |
| [70] |
AZIZI S, MUSTAFA B, RYAN F, et al. Big self-supervised models advance medical image classification[C]//Proceedings of the IEEE/CVF International Conference on Computer Vision. Piscataway: IEEE Press, 2021: 3478-3488.
(  0) 0) |
| [71] |
PUNN N S, AGARWAL S. BT-Unet: a self-supervised learning framework for biomedical image segmentation using Barlow twins with U-net models[EB/OL]. (2021-12-07)[2022-03-15]. https://arxiv.org/abs/2112.03916.
(  0) 0) |
 2023, Vol. 55
2023, Vol. 55


