2. 河南省离子束生物工程重点实验室 河南 郑州 450001;
3. 郑州大学 农学院 河南 郑州 450001
2. Henan Provincial Key Laboratory of Ion Beam Bio-engineering, Zhengzhou 450001, China;
3. School of Agricultural Sciences, Zhengzhou University, Zhengzhou 450001, China
随着畜牧业的快速发展,人们对于畜产品的需求也逐渐增加。缺乏充足、优质的绿色饲料是制约畜产品快速发展的主要因素之一[1]。苜蓿(Medicago sativa)具有较高的营养价值和蛋白质含量,已成为奶牛日粮中使用最为广泛的饲料成分之一[2],但苜蓿的生产多为季节性,无法持续为畜牧业提供优质饲料。青贮是一种保持饲料作物常绿的方法,通过厌氧发酵延长饲料的储藏时间,提高饲料适口性。进行苜蓿青贮的理想干物质条件为30%~35%[3],但在该条件下,苜蓿的可溶性碳水化合物含量低,缓冲能较高,相较于其他牧草,自然青贮品质较差。此外,苜蓿的收获季节大多为雨季,导致苜蓿自然晾晒和干燥困难,同样增加了自然青贮的难度。植物乳杆菌作为青贮添加剂能够有效提高苜蓿的青贮品质,加速乳酸发酵进程,快速降低青贮环境的pH值,提高发酵品质[1, 4-6]。细菌菌群在青贮过程中具有重要作用,同时也受到多种因素的影响,包括青贮添加剂、青贮条件以及材料附生微生物等[7-8]。因此,研究添加植物乳杆菌对苜蓿青贮发酵过程中细菌菌群组成的影响具有一定的意义。高通量测序技术[9]已经广泛应用于测定各种环境中细菌菌群的组成,包括青贮环境。目前,利用高通量测序评估干物质含量低于理想状态下,添加或不添加植物乳杆菌的苜蓿青贮效果,以及细菌菌群与pH值、发酵代谢产物之间的相关性报道较少。为此,本文通过高通量测序技术探究添加植物乳杆菌对紫花苜蓿青贮60 d的细菌菌群以及发酵品质的影响。
1 材料与方法 1.1 苜蓿青贮饲料的制备实验所用紫花苜蓿是在河南省郑州市(34.76°N,113.65°E;海拔110.4 m)收割的初花期紫花苜蓿。自然状态下萎蔫至含水量为26.60%,用铡刀将其铡为1~2 cm的小段。使用植物乳杆菌YX (台湾亚新生物技术有限公司)或植物乳杆菌ZZU A341 (山西初花期紫花苜蓿附生菌株)作为青贮添加剂。将切碎的紫花苜蓿混合均匀,分为3个处理组:①无添加剂对照组(CK);②植物乳杆菌YX (LPI,1×106 cfu/g);③植物乳杆菌ZZU A341 (LPA,1×106 cfu/g)。将约500 g苜蓿碎饲料和5 mL OD600为0.8的细菌悬液(1×106 cfu/g)混合均匀(CK组中添加5 mL无菌水),抽真空后在室温(25 ℃)下保存,每个处理组设置三个重复样本。
1.2 微生物组成、化学成分、发酵常规指标分析青贮前测定苜蓿原料的微生物组成、化学成分和发酵常规指标,缓冲能(BC)采用盐酸-氢氧化钠法[10]测定。青贮60 d后在超净工作台进行拆袋处理,每个样本混合均匀后共分为三份样品,依次分析其细菌菌群、化学成分以及发酵常规指标。第一份样品在拆袋后储藏于-20 ℃,用来获取青贮饲料60 d的细菌DNA。使用细菌DNA试剂盒D3350-02 (Omega Biotek, Norcross, GA, USA)提取细菌总DNA,用1%琼脂糖凝胶电泳检测所提取DNA的浓度,量化后的DNA样本保存在-20 ℃以备后续分析,利用正向引物515 F和反向引物806 R扩增不同区域(16S V4)的16S rDNA基因[11]。PCR产物从2%琼脂糖凝胶中提取,使用GeneJET凝胶提取试剂盒(Thermo Fisher Scientific Inc., Carlsbad, CA, USA)进一步纯化。16S rDNA扩增子测序使用Thermo Fisher’s Ion S5TMXL (北京百迈客生物科技有限公司)进行。利用FLASH(版本1.2.11)组装reads,根据QIIME质量控制程序(版本1.9.1)删除质量较差的reads,通过Uparse软件(v7.0.1001)进行序列分析。第二份样品将10 g青贮苜蓿与90 mL无菌水混合,用pH计(上海梅特勒-托莱多仪器有限公司)测量pH值。使用高效液相色谱仪(Waters Inc., Massachusetts, USA)测定样品中有机酸浓度,色谱柱规格为Carbomix H-NP10: 8%(7.8 m×300 mm×10 μm),流动相为0.025 4% H2SO4,流速为0.6 mL/min,柱温为55 ℃。采用苯酚-次氯酸钠比色法[12]测定氨态氮(AN)含量。第三份样品在60 ℃下烘干48 h至恒重,测定其干物质(DM)含量。将干燥后的样品用打粉机粉碎后使用40目筛子过滤,采用蒽酮-硫酸法[13]测定样品中可溶性碳水化合物(WSC)的含量。
1.3 统计分析使用IBM SPSS(版本21.0)对实验数据进行统计分析,利用R软件(版本2.15.3)进行细菌群落的α多样性分析、主成分分析、Spearman相关性分析。使用Uparse软件对所有样品的Clean read进行聚类,用Mothur方法与SILVA132(http://www.arb-silva.de/)的SSUrRNA数据库进行细菌菌群注释分析。
2 实验结果与分析 2.1 苜蓿原料的特性青贮前紫花苜蓿原料的pH值为6.06,DM含量为26.60%,WSC含量为17.56 g/kg DM,BC含量为460.00 mE/kg DM,在新鲜苜蓿样品中未检测到有机酸和AN。
2.2 苜蓿青贮60 d的化学成分与发酵常规指标苜蓿青贮60 d的化学成分与发酵常规指标分别如表 1、表 2所示,其中CK代表无添加剂自然青贮,LPI、LPA分别代表添加植物乳杆菌YX或ZZU A341青贮。青贮60 d后,CK组的DM含量显著降低(P < 0.05),DM损失率高于LPI或LPA组;LPI或LPA组的pH值和乳酸含量与CK组差异显著(P < 0.05),AN含量低于CK组,且仅在CK组中检测到丁酸。
|
|
表 1 苜蓿青贮60 d的化学成分 Tab. 1 The chemical composition of alfalfa silage for 60 d |
|
|
表 2 苜蓿青贮60 d的发酵指标 Tab. 2 The fermentation index of alfalfa silage for 60 d |
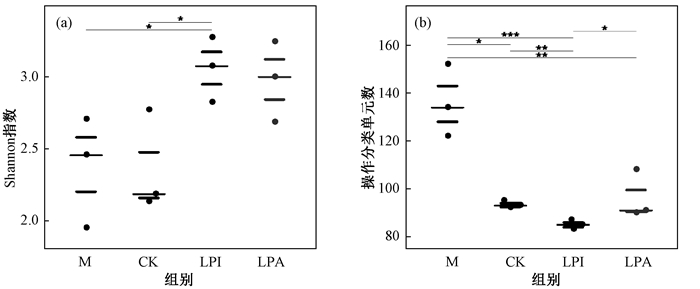
青贮60 d时,利用高通量测序技术分析青贮苜蓿微生物菌群的均一性与多样性。图 1为青贮饲料不同组别间Shannon指数和Shannon指数组间差异蜜蜂群图(*P < 0.05,**P < 0.01,***P < 0.001),反映了不同样本间微生物物种均一性和多样性的差别。如图 1所示,与青贮前相比,青贮后样品中微生物菌群的多样性降低。
|
图 1 Shannon指数(a)和Shannon指数组间差异(b)蜜蜂群图 Fig. 1 Beeswarm plots of Shannon indices (a) and difference of Shannon indices between groups (b) M,新鲜苜蓿;CK,自然青贮;LPI,添加植物乳杆菌YX青贮;LPA,添加植物乳杆菌ZZU A341青贮。 |
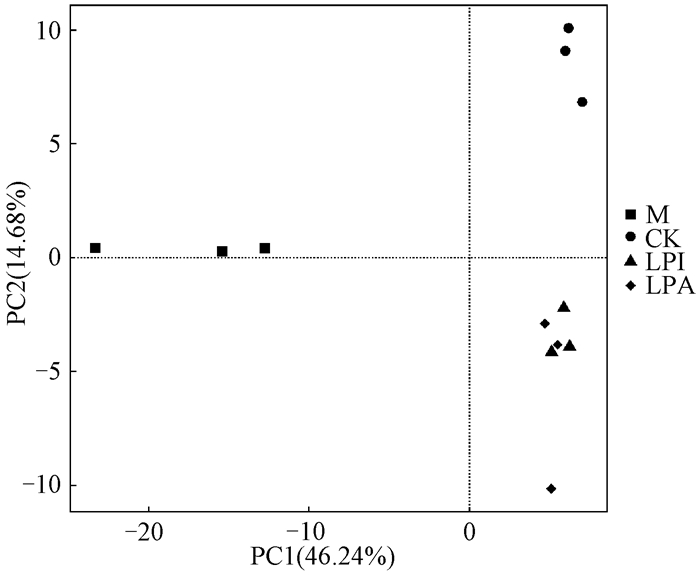
基于发酵产物与微生物信息对青贮60 d的结果进行主成分分析(PCA)。在主成分分析图中,距离越接近,表明微生物组成越相似。图 2为基于青贮苜蓿发酵产物与微生物信息的主成分分析。两个主成分PC1和PC2可以分别解释46.24%和14.68%的差异。结果显示,CK组与LPI或LPA组的微生物菌群聚类均明显分离。
|
图 2 基于青贮苜蓿发酵产物与微生物信息的主成分分析 Fig. 2 Principal component analysis of alfalfa silage fermentation products and microbial information M,新鲜苜蓿;CK,自然青贮;LPI,添加植物乳杆菌YX青贮;LPA,添加植物乳杆菌ZZU A341青贮。 |
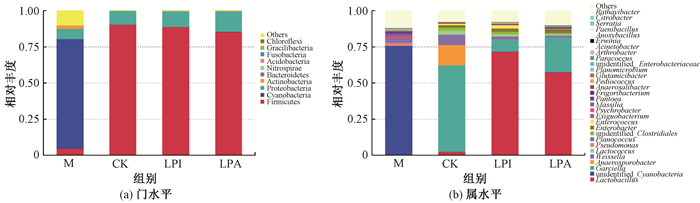
图 3为门水平和属水平上细菌菌群相对丰度柱形图。图 3(a)为青贮60 d门水平上各细菌菌群的相对丰度,青贮后Firmicutes门和Proteobacteria门相对丰度增加。图 3(b)为属水平上各细菌菌群的相对丰度,CK组中的优势细菌菌群为Garciella属(59.75%),其次为Weissella属(7.18%)和Lactobacillus属(2.45%)。LPI或LPA组中的优势菌群为Lactobacillus属,相对丰度分别为71.73%和57.44%;Garciella属的相对丰度分别为8.29%和24.22%,Weissella属的相对丰度分别为2.03%和1.18%。
|
图 3 门水平和属水平上细菌菌群相对丰度柱形图 Fig. 3 Column chart of relative abundance of bacterial flora at the phylum level and genus level M,新鲜苜蓿;CK,自然青贮;LPI,添加植物乳杆菌YX青贮;LPA,添加植物乳杆菌ZZU A341青贮。 |
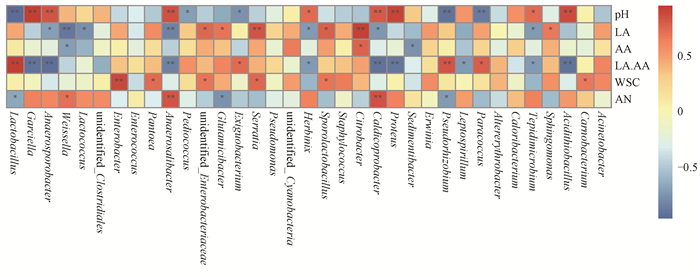
利用Spearman相关系数研究青贮苜蓿细菌菌群相对丰度与pH值、发酵代谢产物之间的相关性,得到两两之间的相关性系数(r)和显著性P值。r介于-1和1之间,r < 0时为负相关,r>0时为正相关。Spearman发酵代谢产物与细菌菌群相关性分析热图如图 4所示(*P < 0.05,**P < 0.01,***P < 0.001)。结果表明,Lactobacillus属与pH值和AN含量呈显著负相关(r=-0.97,r=-0.91;r=-0.67,r=-0.73;P < 0.05),与乳酸和乙酸的比值呈显著正相关(r=0.93,r=0.82;P < 0.05)。Garciella属和Weissella属与pH值和AN含量呈正相关(r=0.95,r=0.40;r=0.63,r=0.72);Weissella属与乳酸和乙酸的比值呈显著负相关(r=-0.90,r=-0.72;P < 0.05)。
|
图 4 Spearman发酵代谢产物与细菌菌群相关性分析热图 Fig. 4 Heat map of correlation analysis between Spearman fermentation metabolites and bacterial flora LA,乳酸;AA,乙酸;LA.AA,乳酸和乙酸的比值;WSC,可溶性碳水化合物;AN,氨态氮。 |
苜蓿的WSC含量低、BC含量高,与其他牧草相比较难青贮,且收获时的雨季会增加苜蓿材料的湿度。苜蓿原料的含水量会对青贮过程中的发酵速率和细菌菌群产生影响,含水量过高容易引起梭菌发酵,不利于乳酸菌繁殖[14]。本文旨在研究苜蓿DM含量低于理想状态下,添加植物乳杆菌青贮对发酵过程中细菌菌群及发酵品质的影响。青贮60 d后,与LPI或LPA组相比,CK组乳酸积累量较低,pH值、AN含量和DM损失率较高,表明在较高含水量条件下自然青贮发酵品质较差。乳酸菌作为青贮添加剂已经广泛应用于牧草青贮实验中[15]。在本研究中,LPI或LPA组乳酸发酵充足,降低了青贮环境的pH值,有效抑制了青贮过程中的丁酸发酵。AN被认为是氨基酸脱氨、脱酸的结果[16],会降低青贮饲料的营养价值,本实验中LPI或LPA组的AN含量均低于CK组,青贮品质较好。
Ni等[1]认为,高含水量的苜蓿青贮中,乳酸积累进程较为缓慢,无法快速降低青贮环境的pH值以及细菌菌群多样性。LPI或LPA组的细菌菌群多样性低于CK组,与其具有较低的pH值和AN含量相关。与CK组相比,LPI或LPA组的细菌菌群组成发生明显改变,这可能是其发酵品质优于CK组的原因之一[17]。Eikmeyer等[5]利用NGS技术分析牧草青贮14 d和58 d的细菌菌群,Firmicutes门为优势菌门;Keshri等[18]认为,青贮环境中较低的pH值和厌氧条件有利于Firmicutes门的生长繁殖,均与本文的研究结果一致。LPI或LPA组中Garciella属和Weissella属相对丰度较低,Lactobacillus属相对丰度较高,表明植物乳杆菌作为青贮添加剂可以改变细菌菌群组成。一般认为AN与青贮过程中Clostridial发酵有关,Garciella是一种厌氧嗜热菌,属于Clostridiales (门)、Clostridia (属),存在于青贮环境中将会增加青贮难度[19-21]。在本研究中与LPI或LPA组相比,CK组中Garciella属的相对丰度较高,还具有较高的AN含量和pH值,与环境因子分析中Garciella属与pH值和AN含量呈正相关相符。牧草自然青贮中的优势菌属一般为Lactococcus属、Leuconostoc属和Weissella属[5];燕麦进行自然青贮的优势菌属为Weissella属、Leuconostoc属和Pediococcus属[22]。本研究中,CK组中Weissella属相对丰度高于LPI或LPA组,Weissella属发酵产酸能力可能低于植物乳杆菌,导致乳酸发酵不足以延迟pH值下降,无法有效抑制青贮环境中不良微生物的活动,这一结果与本实验中Weissella属与pH值和AN含量呈正相关一致。有研究报道,在牧草青贮过程中参与乳酸发酵的细菌菌群主要有Lactobacillus属、Pedicoccus属、Weissella属和Leuconostoc属[17, 23]。青贮过程中Lactobacillus属相对丰度较高,代表青贮实验较为成功[7],本研究中Lactobacillus属在LPI或LPA组中相对丰度较高,且青贮组中细菌菌群多样性、AN含量和pH值均较低,该现象和Lactobacillus属与pH值及AN含量呈显著负相关,以及与乳酸和乙酸的比值呈显著正相关相符。
| [1] |
NI K K, WANG F F, ZHU B G, et al. Effects of lactic acid bacteria and molasses additives on the microbial community and fermentation quality of soybean silage[J]. Bioresource technology, 2017, 238: 706-715. DOI:10.1016/j.biortech.2017.04.055 (  0) 0) |
| [2] |
DUNIÈRE L, SINDOU J, CHAUCHEYRAS-DURAND F, et al. Silage processing and strategies to prevent persistence of undesirable microorganisms[J]. Animal feed science and technology, 2013, 182(1/2/3/4): 1-15. (  0) 0) |
| [3] |
HE L W, CHEN N, LV H J, et al. Gallic acid influencing fermentation quality, nitrogen distribution and bacterial community of high-moisture mulberry leaves and stylo silage[J]. Bioresource technology, 2020, 295: 1-7. (  0) 0) |
| [4] |
FILYA I, SUCU E, KARABULUT A. The effect of Lactobacillus buchneri on the fermentation, aerobic stability and ruminal degradability of maize silage[J]. Journal of applied microbiology, 2006, 101(6): 1216-1223. DOI:10.1111/j.1365-2672.2006.03038.x (  0) 0) |
| [5] |
EIKMEYER F G, KÖFINGER P, POSCHENEL A, et al. Metagenome analyses reveal the influence of the inoculant Lactobacillus buchneri CD034 on the microbial community involved in grass ensiling[J]. Journal of biotechnology, 2013, 167(3): 334-343. DOI:10.1016/j.jbiotec.2013.07.021 (  0) 0) |
| [6] |
OGUNADE I M, JIANG Y, PECH CERVANTES A A, et al. Bacterial diversity and composition of alfalfa silage as analyzed by Illumina MiSeq sequencing: effects of Escherichia coli O157: H7 and silage additives[J]. Journal of dairy science, 2018, 101(3): 2048-2059. DOI:10.3168/jds.2017-12876 (  0) 0) |
| [7] |
GUAN H, YAN Y H, LI X L, et al. Microbial communities and natural fermentation of corn silages prepared with farm bunker-silo in Southwest China[J]. Bioresource technology, 2018, 265: 282-290. DOI:10.1016/j.biortech.2018.06.018 (  0) 0) |
| [8] |
KASMAEI M K, DICKSVED J, SPÖRNDLY R, et al. Separating the effects of forage source and field microbiota on silage fermentation quality and aerobic stability[J]. Grass and forage science, 2017, 72(2): 281-289. DOI:10.1111/gfs.12238 (  0) 0) |
| [9] |
ADAMS I P, GLOVER R H, MONGER W A, et al. Next-generation sequencing and metagenomic analysis: a universal diagnostic tool in plant virology[J]. Molecular plant pathology, 2009, 10(4): 537-545. DOI:10.1111/j.1364-3703.2009.00545.x (  0) 0) |
| [10] |
PLAYNE M J, MCDONALD P. The buffering constituents of herbage and of silage[J]. Journal of the science of food and agriculture, 1966, 17(6): 264-268. DOI:10.1002/jsfa.2740170609 (  0) 0) |
| [11] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 (  0) 0) |
| [12] |
BRODERICK G A, KANG J H. Automated simultaneous determination of ammonia and total amino acids in ruminal fluid and in vitro media[J]. Journal of dairy science, 1980, 63(1): 64-75. DOI:10.3168/jds.S0022-0302(80)82888-8 (  0) 0) |
| [13] |
MURPHY R P. A method for the extraction of plant samples and the determination of total soluble carbohydrates[J]. Journal of the science of food and agriculture, 1958, 9(11): 714-717. DOI:10.1002/jsfa.2740091104 (  0) 0) |
| [14] |
韩吉雨. 青贮发酵体系中乳酸菌多样性的研究[D]. 呼和浩特: 内蒙古农业大学, 2009. HAN J Y. Study on lactic acid bacteria diversity in silage fermentation system[D]. Hohhot: Inner Mongolia Agricultural University, 2009. (  0) 0) |
| [15] |
LIU Q H, SHAO T, BAI Y F. The effect of fibrolytic enzyme, Lactobacillus plantarum and two food antioxidants on the fermentation quality, alpha-tocopherol and beta-carotene of high moisture Napier grass silage ensiled at different temperatures[J]. Animal feed science and technology, 2016, 221: 1-11. DOI:10.1016/j.anifeedsci.2016.08.020 (  0) 0) |
| [16] |
SCHERER R, GERLACH K, SVDEKUM K H. Biogenic amines and gamma-amino butyric acid in silages: formation, occurrence and influence on dry matter intake and ruminant production[J]. Animal feed science and technology, 2015, 210: 1-16. DOI:10.1016/j.anifeedsci.2015.10.001 (  0) 0) |
| [17] |
NI K K, ZHAO J Y, ZHU B G, et al. Assessing the fermentation quality and microbial community of the mixed silage of forage soybean with crop corn or sorghum[J]. Bioresource technology, 2018, 265: 563-567. DOI:10.1016/j.biortech.2018.05.097 (  0) 0) |
| [18] |
KESHRI J, CHEN Y, PINTO R, et al. Microbiome dynamics during ensiling of corn with and without Lactobacillus plantarum inoculant[J]. Applied microbiology and biotechnology, 2018, 102(9): 4025-4037. DOI:10.1007/s00253-018-8903-y (  0) 0) |
| [19] |
KUNG L, SHAVER R. Interpretation and use of silage fermentation analysis reports[J]. Focus on forage, 2001, 3(13): 1-5. (  0) 0) |
| [20] |
MIRANDA-TELLO E, FARDEAU M L, SEPULVEDA J, et al. Garciella nitratireducens gen. nov., sp. nov., an anaerobic, thermophilic, nitrate- and thiosulfate-reducing bacterium isolated from an oilfield separator in the Gulf of Mexico[J]. International journal of systematic and evolutionary microbiology, 2003, 53(5): 1509-1514. DOI:10.1099/ijs.0.02662-0 (  0) 0) |
| [21] |
ZHANG Q, YU Z, WANG X G, et al. Effects of inoculants and environmental temperature on fermentation quality and bacterial diversity of alfalfa silage[J]. Animal science journal, 2018, 89(8): 1085-1092. DOI:10.1111/asj.12961 (  0) 0) |
| [22] |
ROMERO J J, ZHAO Y, BALSECA-PAREDES M A, et al. Laboratory silo type and inoculation effects on nutritional composition, fermentation, and bacterial and fungal communities of oat silage[J]. Journal of dairy science, 2017, 100(3): 1812-1828. DOI:10.3168/jds.2016-11642 (  0) 0) |
| [23] |
PANG H L, QIN G Y, TAN Z F, et al. Natural populations of lactic acid bacteria associated with silage fermentation as determined by phenotype, 16S ribosomal RNA and recA gene analysis[J]. Systematic and applied microbiology, 2011, 34(3): 235-241. DOI:10.1016/j.syapm.2010.10.003 (  0) 0) |
 2021, Vol. 53
2021, Vol. 53


