2. 郑州大学 医学工程技术与数据挖掘研究所 河南 郑州450001
2. Biomedical Engineering Technology and Data Mining Research Institution of Zhengzhou University, Zhengzhou 450001, China
生物学上一般采用BLAST(basic local alignment search tool)工具来获取蛋白质氨基酸序列的相似度[1].随着相关研究工作的进展,大量学者设计开发了其他序列匹配算法以改进BLAST算法的不足[2].然而,氨基酸序列的相似性只能提示两个蛋白是否具有足够的同源性,并不能满足学者对于功能表达相似性的研究.因此,现有研究中已出现了许多关于蛋白质结构相似度比对的工具.CE(combinatorial extension)和FATCAT(flexible structure alignment by chaining AFPs (aligned fragment pairs) with twists)算法是较早开始应用的蛋白质结构比对方法[3],其中CE是采用增量式组合扩展的方法逐段比较对齐的两个蛋白结构片段,最后将其组合起来评价蛋白质相似度.FATCAT算法是CE算法的进一步改进.DALI是L Holm等设计开发的蛋白质对结构相似度在线工具,其主要计算方法是计算一对蛋白中原子的均方根误差(root-mean-square deviation, RMSD),但用户在使用时需要上传处理过的PDB文件(该方法只能计算ATOM/HETATM部分).文献[4]结合TM得分旋转矩阵和动态调整方法设计出TM-align算法,该算法的计算速度大约是DALI和SAL方法的20倍,CE算法的4倍.鉴于各算法的不同,比对结果往往也不一致.RCSB PDB比对工具是RCSB PDB(RCSB protein data bank)数据库自主开发的一种用于匹配蛋白质结构相似度的Java web start应用程序,操作简便,可以实时在线精确匹配PDB数据库中的蛋白质结构文件,应用相对较为广泛.
现有蛋白质相似度比较方法中,基本都是从蛋白质三级结构出发,将其比对结果应用于蛋白质功能的相似性评价上.本文拟从多参数的角度评价蛋白质的相似度,建立相似度和各参数之间的数学关系模型,并依此对蛋白质功能的相似性进行提示.并采用该算法计算并筛选出与新发现的胃癌蛋白p42.3相似的蛋白,成功找出了p42.3的功能调控路径,从而证明了该算法的可用性.
1 材料和方法 1.1 数据收集 1.1.1 总体相似度和参数选择从PDB(http://www.rcsb.org/pdb/home/home.do)数据库中先行收集相似蛋白质共1 005对,进而下载其结构数据PDB文件.然后通过RCSB PDB结构比对工具(http://www.rcsb.org/pdb/workbench/workbench.do?action=menu)获取每一对蛋白质的结构相似度作为标准相似度.在PDB文件中,只取ATOM及HETATM部分的数据进行9个参数相似度的计算,分别为空间密度、原子个数、氨基酸个数、氨基酸种类、C元素比例、N元素比例、O元素比例、P元素位置、S元素位置[5-6],并分别标记为S1~S9.参数的选择标准均以与蛋白质功能表达相关为出发点.
1.1.2 密度相似度(S1)首先在蛋白质内部以该蛋白中心原子为原点建立空间坐标系,将其余原子的坐标按统一位移向量变化.然后,计算每一个原子距原点的距离,根据距离将蛋白质划分为一层层的球壳.统计每一层球壳的原子数目,并比较两个蛋白在每一层的原子个数相似度,而后加权求和.当层数取得无穷大时,每一层球壳的厚度便无穷小,此时所计算的参数便可视为蛋白质的密度相似度.假设将蛋白质平均划分为n层,每一层相似度计算公式simi,每一层的相似度权值计算公式wi,n1i为其中为第一个蛋白第i层的原子个数,n2i为第二个蛋白第i层的原子个数,n1为第一蛋白质的原子总数,n2为另一个蛋白质的原子总数.
| $ si{m_i} = 1-\frac{{\left| {{n_{1i}}-{n_{2i}}} \right|}}{{\left| {{n_{1i}} + {n_{2i}}} \right|}}, i = 1, 2, \ldots, n, \;\;{w_i} = (\frac{{{n_{1i}}}}{{{n_1}}} + \frac{{{n_{2i}}}}{{{n_2}}})/2, i = 1, 2, \ldots, n. $ |
因此,两个蛋白质的密度相似度可以表示为
每一个蛋白分子所包含的原子数目决定了分子的大小和质量,而氨基酸的数目和种类影响着蛋白质的功能.
| $ S2 = 1-\frac{{\left| {{n_1}-{n_2}} \right|}}{{\left| {{n_1} + {n_2}} \right|}};S3 = 1-\frac{{\left| {{m_1} - {m_2}} \right|}}{{\left| {{m_1} + {m_2}} \right|}};S4 = 1 - \frac{{\left| {{k_1} - {k_2}} \right|}}{{\left| {{k_1} + {k_2}} \right|}}, $ | (1) |
其中:n1、m1、k1分别为第一个蛋白质的原子总数、氨基酸数目和氨基酸种类;n2、m2、k2分别为第二个蛋白质的原子总数、氨基酸数目和氨基酸种类.
1.1.4 C、N、O元素比例相似度(S5、S6、S7)S5~S7计算方法相同,均按照公式(2) 进行计算,其中:ne1是第一个蛋白质中的C/N/O元素个数; ne2是第二个蛋白质中的C/N/O元素个数; n1和n2分别为两个蛋白质的原子总数.
| $ S = 1-\frac{{\left| {\frac{{{n_{e1}}}}{{{n_1}}}-\frac{{{n_{e2}}}}{{{n_2}}}} \right|}}{{\left| {\frac{{{n_{e1}}}}{{{n_1}}} + \frac{{{n_{e2}}}}{{{n_2}}}} \right|}}. $ | (2) |
P和S元素在蛋白质中的含量相对较偏少,但其对蛋白质作用的发挥起着关键的作用,因此,P、S元素的相似度也是评价两个同源蛋白相似度的一个重要因素.其中,S8为P元素位置相似度,计算定义为:若两个蛋白均不含P元素,则该相似度为1;若其中一个含有而另一个不含有,则相似度为0.如果两个蛋白质均含有P元素,查找P元素位置和其距原点原子的距离,并按照距离获取其所在的层数(S1计算过程中的分层),如果两个蛋白所含P原子位于相同层,则相似度为1;在相邻层,则相似度为0.5,除此之外的情况则相似度为0.S9为S元素的位置相似度,计算方法同P元素.
因此,按照上述方法可以计算出每一对蛋白的9个参数的相似度,将其与总相似度S一起组成样本数据用于之后的建模分析.所收集部分数据如表 1所示.
|
|
表 1 所收集数据S及S1~S9的均值和标准差 Table 1 Mean and standard deviation of S1~S9 and collected data S |
神经网络为采用单层感知器结构时,所建立模型可视为线性模型[7].作为一种线性分类器,单层感知器尽管结构简单,却能够学习并解决相当复杂的问题.假设S和S1~S9之间的关系是线性的,因此单层感知器可以对其建模一个方程.神经网络学习的过程就是一个权值调节过程,最终会输出一个权值矩阵[8].在本方法中,随机选取所有数据集中80%的数据用于对模型进行训练,将其余的20%数据用于对模型验证,所设置训练误差阈值为0.001,最大训练次数为5 000,输入层节点数为9,输出层节点数为1,因此,训练结束后会得到一个1*9的系数向量w和一个常数项B.此时,相似度S按
选取数据集中剩余的20%的数据对模型进行误差验证.将数据按照相似度S的公式进行计算,并与已知结果进行比较,误差计算方法
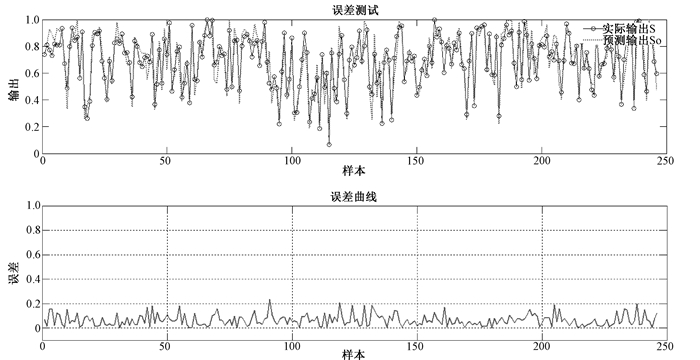
所建立神经网络为单层感知器线性神经网络,训练过程如图 1所示.该训练过程结束后,输出各参数系数如表 2所示.由此可得,所建立的数学模型为:S=0.319 8S1+0.034 3S2+0.027 9S3+0.061 8S4+0.065 3S5+0.106 2S6+0.103 2S7+0.147 7S8+0.148 0S9-0.014 2.仿真误差如图 2所示,平均误差ME计算结果为8.67%.分别用该算法模型和RCSB PDB比对工具及BLAST进行相似度的计算,并比较其结果如表 3所示.
|
图 1 训练过程 Figure 1 Training process |
|
|
表 2 各参数系数 Table 2 Coefficient of every parameter |
|
图 2 误差曲线 Figure 2 Error curve |
|
|
表 3 所用模型与已有工具比较 Table 3 Comparison between models and existing tools |
本文提出一种新的基于多参数和线性神经网络的蛋白质相似度算法,建立了蛋白质相似度和其9个参数之间的数学模型.该算法是从分析蛋白质结构相似度出发,旨在对蛋白质功能的相似性进行提示,所选用参数也均与蛋白质功能的表达相关.
已有一些学者的研究证明,多参数评价蛋白质相似度较单一参数更为合适[9-10, 13],文献[11]通过比较两个蛋白的骨架碳原子曲线参数比较蛋白质的相似度,例如曲率、扭力和翻转变体等.文献[12]通过对氨基酸以及蛋白质多肽链中的特殊结构的分析,综合考虑了蛋白质结构中骨架碳原子数、突变原子数、亲水微粒数和螺旋数4个参数,并依托模糊数学等价矩阵理论,提出一种新的相似度算法,证明其性能比考虑单一参数更好.神经网络是近代应用逐渐广泛的人工智能算法[14],并且对神经网络采用单层感知器即可建立线性模型,在数据量较大时,对数据进行特征提取后,再利用神经网络进行分类会节省时间.因此,本文所采用的简单线性神经网络模型,对其进行的误差分析及结果验证都表明了其良好的性能.从表 3可以看出,本算法的计算结果同RCSB PDB比对工具的结果基本相同,但个别具有一定差异.如1AAX(酪氨酸激酶)和101M(抹香鲸肌红蛋白)的相似度,3B94(人GITRL蛋白)和4DB5(家兔GITRL蛋白)的相似度相比较,RCSB PDB结构比对工具的结果较高,而3WD5(人TNFα与阿达木抗体结合蛋白)和2TNF(小鼠TNFα蛋白)的相似度相对其较低.具体分析可知,1AAX和101M以及3B94和4DB5均含有S元素且所在位置非常接近,而S元素在蛋白质中的作用一般是形成二硫键,以此来影响蛋白质高级结构的生物活性和蛋白质的复性等功能特点[15].而3WD5和2TNF虽然也含有S元素,但其所在位置较为不同(一个在蛋白分子表面, 而另一个在内部靠近中心位置), 另外,BLAST同源性也表明了二者的相似程度.
采用本文的算法,可以初步计算并筛选出与p42.3具有相同结构域且总体相似度在80%以上的蛋白质集,推测出p42.3的生物学功能和调控路径与这些蛋白相似.在后期进行的Weston Blot及PCR生物学实验结果验证了这一预测.本文中所采用的9个参数主要提取自蛋白质的空间结构PDB文件,在参数的选择和计算方法方面尚需要进一步改进.随着研究的深入和样本量的增多,算法将会得到进一步的优化.
| [1] |
JOHNSON M, ZARETSKAYA I, RAYTSELIS Y, et al. NCBI blast: a better web interface[J]. Nucleic acids research, 2008, 36(S2): 5-9. (  0) 0) |
| [2] |
YE Y, CHOI J H, TANG H. RAP rearch: a fast protein similarity search tool for short reads[J]. BMC bioinformatics, 2011, 12(1): 159. DOI:10.1186/1471-2105-12-159 (  0) 0) |
| [3] |
SHINDYALOV I N, BOURNE P E. Protein structure alignment by incremental combinatorial extension (CE) of the optimal path[J]. Protein engineering, 1998, 11(9): 739-747. DOI:10.1093/protein/11.9.739 (  0) 0) |
| [4] |
ZHANG J H, LU C L, SHANG Z G, et al. P42.3 gene expression in gastric cancer cell and its protein regulatory network analysis[J]. Theoretical biology and medical modelling, 2012, 11(9): 53. (  0) 0) |
| [5] |
王昕, 毛炳蔚, 王福伟, 等. 蛋白质空间结构的统计分析[J]. 山西大同大学学报(自然科学版), 2008, 24(5): 3-8. (  0) 0) |
| [6] |
章社生, 何康, 范宁, 等. 蛋白质空间结构数字特性统计分析及应用[J]. 武汉工程大学学报, 2010, 32(5): 45-48. (  0) 0) |
| [7] |
何立群, 占永平. 感知器神经网络模型研究[J]. 九江学院学报(自然科学版), 2014(4): 37-43. (  0) 0) |
| [8] |
ATLAS K. 神经网络的优化与用于优化的神经网络[D]. 大连: 大连理工大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10141-1013197371.htm
(  0) 0) |
| [9] |
高华龙. 蛋白质空间结构相似性比较方法研究[D]. 大连: 大连交通大学, 2012. http://d.wanfangdata.com.cn/Thesis/Y1583884
(  0) 0) |
| [10] |
HAO Y, FAN T, NAN K. Optimization and corroboration of the regulatory pathway of p42.3 protein in the pathogenesis of gastric carcinoma[J]. Computational and mathematical methods in medicine, 2015, 5(28): 1-9. (  0) 0) |
| [11] |
KOTLOVYI V, NICHOLS W L, TEN EYCK LF. Protein structural alignment for detection of maximally conserved regions[J]. Biophysical chemistry, 2003, 105(2/3): 595-608. (  0) 0) |
| [12] |
徐占, 董洪伟. 多特征框架下的蛋白质相似性比较与分类[J]. 图学学报, 2010, 31(1): 191-196. (  0) 0) |
| [13] |
张萍萍, 张建华, 尹咪咪. 蛋白质空间结构相似度多参数算法模型的建立[J]. 郑州大学学报(理学版), 2016, 48(2): 105-109. (  0) 0) |
| [14] |
张睿. 计算智能方法及应用研究[J]. 电脑开发与应用, 2012, 25(10): 1-3. DOI:10.3969/j.issn.1003-5850.2012.10.001 (  0) 0) |
| [15] |
徐国恒. 二硫键与蛋白质的结构[J]. 生物学通报, 2010, 45(5): 5-6. (  0) 0) |
 2017, Vol. 49
2017, Vol. 49


