2. 南京农业大学 作物遗传与种质改良 国家重点实验室 江苏 南京 210095;
3. 河南科技学院 生命科技学院 河南 新乡 453003;
4. 郑州大学 农学院 河南 郑州 450001
2. State Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University, Nanjing 210095, China;
3. School of Life Science and Technology, Henan Institute of Science and Technology, Xinxiang 453003, China;
4. School of Agricultural Sciences, Zhengzhou University, Zhengzhou 450001, China
面包小麦(Triticum aestivum L., 2n=2x=14, AABBDD)是小麦属异源六倍体物种,是目前世界上种植最广、交易量最大的粮食作物之一,也是全球植物蛋白的主要来源[1]。因此,小麦的可持续生产对世界范围内的社会进步和稳定具有重要意义。自20世纪60年代绿色革命以来,世界小麦产量增加了2倍,预计在21世纪中叶将继续保持增长[2]。在此期间,小麦产量的增长主要是通过提高单位面积的作物产量来实现的。培育遗传背景优良、种子耐贮藏的新品种,提高肥料利用率,施用农药和杀虫剂,改善灌溉等都是小麦单产增加的因素。其中,选育和推广高产、适应性广、品质好、抗逆性强、肥料利用率高的小麦新品种对小麦产量的提高发挥了重要作用[3]。
小麦新品种选育的成功始于选择合适的亲本,优良亲本的杂交组合往往会选育出一批好的品种。例如,周8425B是20世纪70年代从国际玉米小麦改良中心引进的合成六倍体小黑麦(2n=6x=42,AABBRR)产生的小麦-黑麦自发罗伯逊易位(RT) 1BL.1RS系。以其为亲本,在河南省及周边地区培育出了100多个品种,其中周麦16号、矮抗58号等79个品种通过国家或省级审定[4]。小麦品种小偃6号是小麦与十倍体长穗偃麦草(Thinopyrum ponticum,2n=10x=70, JSJSJSJSJJJJJJ)杂交的后代,曾经是中国种植面积最大的小麦品种,以小偃6号为核心亲本,选育出了50多个大面积推广品种,累计推广面积达到2 000万hm2,增产1 500万t[5]。因此,解析大面积推广品种及其核心亲本的遗传组成,有助于了解如何培育优良品种,提高重要农艺性状和育种效率。近年来,不少研究人员利用简单序列重复(SSR)标记、高密度单核苷酸多态性(SNP)标记或高分辨率荧光原位杂交(FISH)对小麦主栽品种和亲本的遗传基础进行了解析[6-7],但尚未见到结合FISH和基因芯片杂交技术对小麦品种进行遗传解析的报道。
百农207是2014年通过国家农作物品种审定委员会审定的小麦新品种,自2016年起成为河南省和我国小麦主产区黄淮南片年种植面积最大的高产品种(135万hm2)。其亲本周麦16和百农64也是在该地区曾经大面积推广的品种,是目前育种家最常使用的核心亲本[8-9]。本研究结合高分辨率FISH和小麦15 K SNP芯片杂交,对百农207及其亲本和姊妹系进行了染色体和基因组构成2个层次的分析,旨在为我国黄淮河流域南部地区优良新品种的培育,以及为百农207及其亲本百农64和周麦16的更好利用提供依据。
1 材料与方法 1.1 植物材料本研究共选用13个小麦品种(系),其主要农艺性状如表 1所示。其中百农207是以周麦16为母本,与百农64杂交选育的,同时从该杂交组合中还选育出10个不同农艺性状的姊妹系。所有材料均由河南科技学院提供。
|
|
表 1 所用材料及其主要农艺性状 Tab. 1 Materials used and their key agronomic characteristics |
每个材料从20个幼苗中采集新鲜叶片,用CTAB (十六烷基三甲基溴化铵)法提取基因组DNA,并用1%琼脂糖凝胶电泳检测DNA提取液的完整性和浓度。然后将合格DNA样品与含有13 947个SNP标记的15 K基因芯片杂交,筛选SNPs标记。该芯片由中国农业科学院设计,杂交由中金玉生物技术有限公司实施完成。
1.3 SNP数据分析与基因型图谱构建利用得到的SNP基因型数据对百农207及其亲本和姊妹系的基因组构成进行分析。如果特定SNP位点的百农207基因型与特定亲本的基因型相同,则认为百农207从该特定亲本遗传了该基因座。存在于百农207中但双亲均不存在的基因座被视为新基因座,而在百农207中不存在的双亲基因座被视为缺失基因座。随后,计算每个亲本对百农207基因组贡献的SNP位点数。一个亲本对百农207的基因组贡献被定义为从一个亲本遗传的SNP位点数量与亲本间多态型SNP位点总数之比。根据SNP位点侧翼序列与中国春参考序列v.1.1 (IWGSC 2018)的序列比对,将不同染色体上的SNP位点从短臂到长臂进行排序。用TASSEL v5.0主成分分析法(PCA)对百农207的亲本和姊妹系进行聚类分析[10]。
1.4 根尖细胞染色体制备及荧光原位杂交按文献[7]所述进行根尖细胞染色体制备,使用BX51奥林巴斯相差显微镜(日本东京奥林巴斯公司)进行细胞学观察。
以8种单链寡核苷酸为探针进行双色非变性原位杂交(ND-FISH)。用6-羧四甲基罗丹明(TAMRA)标记的pAs1-1、pAs1-3、pAs1-4、pAs1-6、AFA-3、AFA-4探针产生红色信号,而6-羧富勒烯(FAM)标记的pSc119.2-1和(GAA)10重复序列产生绿色信号。所有寡核苷酸均由上海生工生物科技有限公司合成。杂交后,在BX51奥林巴斯荧光显微镜下观察染色体,并通过SPOT CCD (点冷彩色数码相机)拍摄图像,使用Adobe Photoshop (v6.0) (美国Adobe)进行分析。
1.5 抗穗发芽基因分子标记分析基于文献[11-14]设计抗穗发芽基因的PCR引物序列,由上海生工生物科技有限公司合成(表 2)。进行PCR扩增反应混合液体积为15 μL,分别含有7.5 μL 2×EasyTaq-PCR Supermix (北京全式金科技公司)、0.5 pmol正向和反向引物以及100 ng基因组DNA。PCR扩增使用F50SSR反应系统[15]。PCR产物经1.5%琼脂糖凝胶分离,溴化乙啶染色后用Tanon 2500凝胶成像系统(上海Tanon科技有限公司)进行观察。
|
|
表 2 抗穗发芽基因分子标记引物 Tab. 2 Primers for molecular markers of pre-harvest sprouting resistance genes |
百农207是以周麦16为母本、百农64为父本杂交选育的半矮秆小麦品种。周麦16和百农64都是国家审定的高产小麦品种,在河南和邻近省份常作为核心亲本使用。其中周麦16是高产半矮秆品种,大穗大粒,但抗倒春寒和抗穗发芽性差、千粒重低; 而百农64对条锈病和叶锈病的综合抗性强,品质好,籽粒饱满,但分蘖力低,成熟时小穗轴易断。百农207成功地整合了亲本的优势,弥补了亲本的不足,具有产量高、稳定性好、晚春耐倒春寒能力强、抗穗发芽、千粒重高等特点。根据黄淮南片麦区37个地点两年的区域试验和14个地点一年的生产试验(由国家农业技术推广服务中心和河南省农业科学院小麦研究中心联合进行),百农207在2010—2013年的年平均产量为7 542~8 761 kg/hm2,比商品对照品种周麦18增产3.8%~7.0%(表 3)。2014年,百农207通过国家农作物品种审定委员审定,自2016年起成为河南省推广速度最快的新品种。此外,从同一个杂交组合中还筛选出10个具有突出特点的百农207姊妹系(表 1),但在产量和其他农艺性状上均未超过百农207。
|
|
表 3 百农207的产量及主要农艺性状 Tab. 3 Yields and key agronomic traits of BN207 |
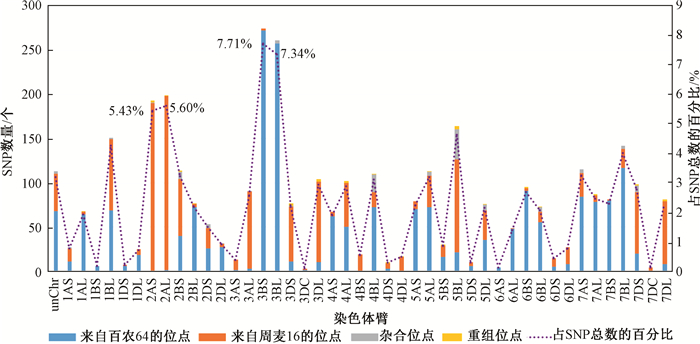
共检测了13 947个SNP位点,得到7 565个(54.2%)有效位点。其中百农64和周麦16相同的SNP位点有4 009个(52.9%),具有多态性的位点有3 556个(47%)。在亚基因组中,B基因组SNPs的数量最多,占总SNPs数量的41.3%,占多态性SNPs数量的51%,其次是亚基因组A(35.1%,45.4%),而SNP数量最少的是亚基因组D(20.2%,41.6%)。每个染色体上平均有360.2个SNP位点,但SNP位点数量在染色体之间变化很大,其中3B染色体上SNP位点最多,有694个,其次是2A染色体,有528个,而4D染色体上只有108个SNP位点。
基于多态性SNP位点对百农207的基因组构成进行分析,结果表明,父本百农64对百农207基因组的贡献率为55.3%(1 968/3 556),母本周麦16对百农207基因组的贡献率为40.7%。其余142个(3.2%)SNP位点包括111个(3.1%)杂合位点和31个(0.9%)仅百农207特有的新位点或重组位点。此外,在双亲相同的4 009个SNP位点中,检测到33个新SNP位点,使百农207新位点或重组位点总数增加到64个,占有效SNP总数(7 565个)的0.85%。此外,亲本对百农207的亚基因组贡献明显不同,亚基因组B中绝大多数SNP位点来自百农64,而亚基因组A和D中相对较多的位点则来自周麦16。
图 1为百农207中SNP位点的染色体分布及亲本贡献率。对百农207染色体SNP位点分布的分析结果表明,其SNPs来源(父本或母本)存在显著差异。用S代表染色体短臂,L代表长臂,其中3B、1BS、2BL、2DL、4AS、5AS、6A、6B、7AL和7BS染色体的SNP位点主要来自父本百农64,而2A、3A、3D、4BS、4D、5BL、6D和7D染色体的多数等位基因则来自母本周麦16。大多数杂合SNP位点位于5BL(34/111, 30.6%)、4BL(20/111, 18.0%)和2BS(8/111, 7.2%),而大多数重组位点位于5B(25/64, 39.1%),其次位于3D(7/64, 10.9%)。
|
图 1 百农207中SNP位点的染色体分布及亲本贡献率 Fig. 1 SNP distribution at chromosomes of BN207 and contribution ratios of its parents |
百农207与其双亲及10个姊妹系的SNP位点比较结果表明,百农207与其姊妹系的染色体构成存在显著差异。但总体而言,百农207及其姊妹系的1A、4A、6A、3B染色体上的SNPs绝大多数来自百农64,而2A、5A、1B、6D和7D染色体上的SNPs则大部分是从周麦16遗传来的。
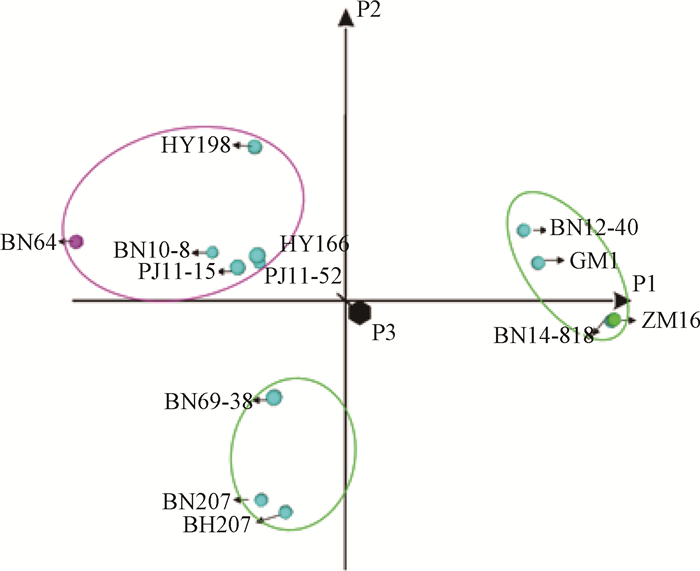
基于主成分分析法的百农207及其亲本和姊妹系间的亲缘关系如图 2所示。通过主成分分析法将百农207及其亲本和姊妹系分为3个集群。第1个集群包括周麦16和3个姊妹系(百农14-818、百农12-40和冠麦1号),第2个集群包括百农64、华育198、百农10-8、华育166、PJ11-52和PJ11-15,第3个集群有百农207以及2个姊妹系百旱207和百农69-38,进一步证实百农207和百旱207是所有姊妹系中结合双亲不同性状(SNP位点)最多的品种。
|
图 2 基于主成分分析法的百农207及其亲本和姊妹系间的亲缘关系 Fig. 2 Genetic relationship among BN207, its parents and sister lines based on PCA |
在姊妹系中,百农14-818与母本周麦16几乎完全相同,只有3.55%的SNP位点不同。在这些SNPs中,只有1.66%的SNPs是从百农64遗传来的,而且主要集中位于5B染色体上(57.6%)。因此,本品系可作为周麦16的近等基因系,用于分析百农14-818和周麦16之间性状差异的遗传基础。
与百农207相比,百旱207耐旱性更好,它与百农207的共有SNP位点占89.8%。这2个品种之间剩余的10.2%的SNP差异可能有助于研究与百农207的产量优势和百旱207的耐旱性相关的基因。此外,PJ11-15品系含有32.0%以上的杂合SNP位点,说明该品系在遗传上仍不稳定。
2.4 百农207及其双亲和姊妹系的染色体构成分析对33株小麦进行了FISH分析,百农207及其亲本和姊妹系的核型如图 3所示。图 3中所用材料自上而下依次为:周麦16、百农64、百农207、华育198、冠麦1号、百旱207、PJ11-52、PJ11-15、百农14-818、百农10-8、百农12-40、百农69-38和华育166。探针pAs1-1、pAs1-3、pAs1-4、pAs1-6、AFA-3和AFA-4用TAMRA标记并产生红色信号; pSc119.2-1和(GAA)10用FAM标记并产生绿色信号。白色箭头表示多态性,黄色箭头表示结构重排,白色和黄色星号表示重组。

|
图 3 百农207及其亲本和姊妹系的核型 Fig. 3 Karyotypes of BN207, its parents and sister lines |
从图 3可以看出,亲本百农64和周麦16及其衍生系存在9种染色体变异。其中在百农64中检测到6B染色体的臂间倒位(perInv),与文献[7]的鉴定结果一致。另外5种结构变异包括:来自周麦16由小麦长臂1B和黑麦1R染色体短臂组成的罗伯逊易位RT1BL.1RS; 位于5A长臂中部的AFA家族串联重复序列的红色信号区段(Trsb); 7BL终端区域的pSc119.2-1和(GAA)10重复序列的绿色信号区; 7A染色体短臂末端的绿色信号重复序列; 1DL末端的绿色信号重复序列。其余3种染色体变异包括百农64中3B染色体长臂的中段和4A染色体长臂上各有一个更强的绿色信号,而周麦16的6BS染色体上有一个更强的红色信号(AFA家族重复)。
染色体核型分析结果表明,百农207中的7A、1B、3B、perInv 6B和1D染色体来自百农64,4A和7B染色体来自周麦16,而5A和6B变异来自双亲染色体的重组。百农207中5A染色体与百农64相似,但在5A染色体长臂着丝粒附近有较弱的绿色信号; 6B染色体与百农64同样有臂间倒位,但在6B染色体短臂的末端区域有较多的红色AFA家族重复,与周麦16一致。在10个姊妹系中,有5个(华育198、冠麦1号、百农14-818、百农12-40和百农69-38)含有来自周麦16的RT1BL.1RS染色体,4个(冠麦1号、百旱207、百农12-40和百农69-38)含有来自周麦16的Trsb 5A,只有2个(百旱207和百农10-8)含有来自百农64的perInv 6B。有3个品系(冠麦1号、百农12-40和百农69-38)同时有来自周麦16的Trsb 5AL和RT1BL.1RS结构变异,百旱207则含有来自百农64的perInv 6B和周麦16的Trsb 5A。
2.5 染色体重组区间的物理定位将FISH分析结果与SNP位点的物理位置结合,对染色体重组区域进行了物理定位。利用3个含有perInv 6B的品系(百农207、百旱207和百农10-8),通过对6B染色体上不同亲本来源SNP位点的分析,发现6B染色体最靠着丝粒的位点重组,在短臂上发生在SNP标记AX-109464405(42.5 Mb)和AX-110448396(50.1 Mb)之间,而在长臂上发生在AX-109595550(475.5 Mb)和AX-111693296(476.2 Mb)之间。因此,6B染色体臂间倒位区段被定位在短臂50.1 Mb到长臂475.5 Mb之间,大小为425.4 Mb,占整个6B染色体长度的一半以上。
含有Trsb 5A的4个品系(冠麦1号、百旱207、百农12-40和百农69-38)的SNP位点分析结果表明,Trsb 5A有3个非重组区(重组抑制区)。第1个非重组区包括整个短臂,其余2个位于长臂,即包括从着丝粒(109.1 Mb)到SNP位点AX-108726870(422 Mb)的312.9 Mb区段,以及从SNP位点AX-110578543(480.4 Mb)到AX-94589715 (546.5 Mb)的66.1 Mb区段。综合双亲间非重组SNP位点的物理位置和FISH显示重复序列片段位置分析,5A染色体长臂中部的AFA家族串联重复序列区段总长为66.1 Mb。
在母本周麦16中发现了来自其父本周麦8425B的RT1BL.1RS染色体。在5个携带RT1BL.1RS的高代品系(华育198、冠麦1号、百农14-818、百农12-40和百农69-38)中,经过百农64来源SNP位点与来自周麦16位点的比较分析,发现黑麦1R短臂与小麦1B短臂之间存在严重的重组抑制现象,并且在1B染色体长臂上也检测到2个不重组区段。第1个不重组区段长198.93 Mb,从着丝粒(249.2 Mb)到SNP位点AX-110687238 (448.13 Mb)结束,第2个不重组区段长126.35 Mb,从位于451.58 Mb的SNP位点AX-108725476到位于577.93 Mb的AX-110091358之间。百农207及其一半姊妹系不含有RT1BL.1RS染色体,表明这种曾经存在于河南及邻省70%以上的小麦品种中的染色体正逐渐失去对现代小麦品种的主导地位。
2.6 百农207抗穗发芽基因的分子标记鉴定百农207是耐穗发芽的硬白小麦品种,与感病对照周麦18相比,萌动种子和发芽种子的比率都较低,表现出较好的抗穗发芽能力,而其姊妹系华育166则和对照周麦18一样,不抗穗发芽(表 4)。
|
|
表 4 百农207穗发芽抗性鉴定 Tab. 4 Identification of resistance to pre-harvest sprouting of BN207 |
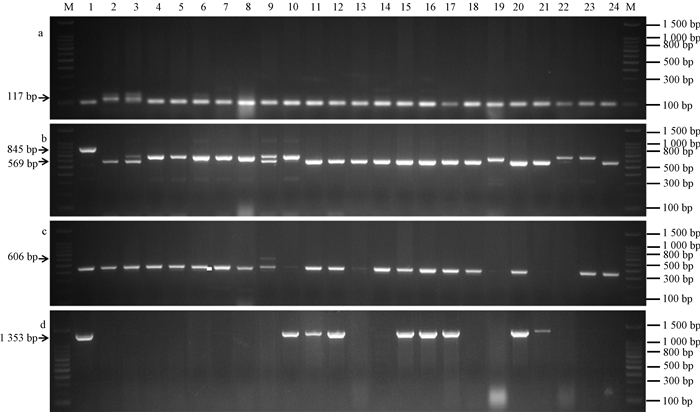
从小麦15 K SNP芯片检测到百农207及其双亲百农64和周麦16具有2个穗发芽抗性基因的主效QTL位点,但其单倍型对穗发芽抗性有负效应[16]。为了鉴定百农207中与抗穗发芽相关的基因,选用了4个与抗性基因(TaVp1B3、PM19-A1、Dorm-1和Tamyb10)紧密相关的PCR标记进行检测。百农207及其亲本等小麦品种抗穗发芽基因的PCR扩增如图 4所示(图中箭头表示穗发芽抗性基因的特异性标记)。结果表明,百农207及其父本百农64能扩增出与PM19-A1基因连锁的117 bp特异性片段,以及与TaVp1B3基因连锁的569 bp特异性标记,但母本周麦16未能扩增到这些特异性标记片段。因此,百农207至少含有2个穗发芽抗性基因,即位于4AL的PM19-A1基因和3BL的TaVp1B3基因,并且这2个基因均来自其父本百农64。进一步利用34株百农207单株进行抗穗发芽基因TaVp1B3鉴定,结果只有24株(70.6%)扩增到TaVp1B3的569 bp特异性片段。因此,百农207后代存在TaVp1B3基因分离,利用该基因特异分子标记继续选择,可以进一步提高百农207对穗发芽的整体抗性水平。
|
图 4 百农207及其亲本等小麦品种抗穗发芽基因的PCR扩增 Fig. 4 PCR patterns of pre-harvest sprouting resistance genes of BN207, its parents and other wheat varieties M: 100 bp DNA marker; 1:中国春; 2:百农207;3:百农64;4:周麦16;5~24:周麦16、周麦22、平安602、平安0518、扬麦5、宁麦9、扬麦13、扬麦14、扬麦16、扬麦17、扬麦23、扬麦158、NAU01、南农0686、NAU617、南农9918、望水白、生选6号、偃展4110、西农979; a~d: PM19-A1; TaVp1B3; Dorm-1; Tamyb10 |
本研究从染色体和基因组水平分析了百农207及其亲本百农64和周麦16,以及10个姊妹系的遗传组成。结果发现,百农207中父本百农64的SNP贡献率(55.3%)高于母本周麦16(40.7%),染色体间有显著差异。其中1BS、3BL、6AS、2AS、4BS、4DL染色体上的SNPs几乎均来源于双亲之一。解析百农207及其亲本的染色体和基因组构成,有助于育种家更好地了解这些品种的遗传贡献,快速、准确地鉴定品种的真伪,改进育种程序,保护种植户和育种者的合法权益。
6B染色体臂间倒位是我国小麦品种中最常见的染色体结构变异类型(15.3%)[8],但目前尚未见对倒位区段进行物理定位的报道。本研究通过FISH和SNP位点重组分析,将该臂间倒位定位在425.4 Mb区间,从短臂的50.1 Mb到长臂的475.5 Mb。此外,将来自周麦16染色体5A长臂上的AFA家族串联重复序列区段定位在SNP位点AX-110578543(480.4 Mb)到AX-94589715(546.5 Mb)的66.1 Mb区间,为perInv 6B和Trbs 5A结构变异提供了直接证据。
本研究利用TaVp1B3特异性标记对百农207单株进行穗发芽抗性基因筛选,在群体中仅鉴定出70.6%的阳性单株,表明百农207中存在TaVp1B3基因分离,筛选具有TaVp1B3基因的单株可以进一步提高百农207对穗发芽的抗性。总之,通过全基因组高密度SNP和FISH的联合分析,揭示了小麦品种百农207的染色体构成、基因组构成以及双亲的贡献,从染色体和分子2个层次证实百农207较大程度结合了双亲的遗传物质,并首次对染色体变异perInv 6B和Trsb 5A的重组区段进行了物理定位。结果表明,将高通量基因分型技术与FISH技术相结合,能够更准确地评价小麦品种的基因组来源,为新品种选育和优良育种亲本利用提供更全面的研究基础。
| [1] |
RASHEED A, MUJEEB-KAZI A, OGBONNAYA F C, et al. Wheat genetic resources in the post-genomics era: promise and challenges[J]. Annals of botany, 2018, 121(4): 603-616. (  0) 0) |
| [2] |
GODFRAY H C J, BEDDINGTON J R, CRUTE I R, et al. Food security: the challenge of feeding 9 billion people[J]. Science, 2010, 327(5967): 812-818. DOI:10.1126/science.1185383 (  0) 0) |
| [3] |
FEYERHERM A M, PAULSEN G M, SEBAUGH J L. Contribution of genetic improvement to recent wheat yield increases in the USA[J]. Agronomy journal, 1984, 76(6): 985-990. DOI:10.2134/agronj1984.00021962007600060027x (  0) 0) |
| [4] |
庄巧生. 中国小麦品种改良及系谱分析[M]. 北京: 中国农业出版社, 2003. ZHUANG Q S. Chinese wheat improvement and pedigree analysis[M]. Beijing: China Agriculture Press, 2003. (  0) 0) |
| [5] |
LI Z S, LI B, ZHENG Q, et al. Review and new progress in wheat wide hybridization for improving the resistance to biotic and abiotic stresses[M]//OGIHARA Y, TAKUMI S, HANDA H. Advances in wheat genetics: from genome to field. Tokyo: Springer, 2015: 377-385.
(  0) 0) |
| [6] |
JIANG P, ZHANG P P, ZHANG X, et al. Genetic contribution of Ningmai 9 wheat to its derivatives evaluated by using SNP markers[J]. International journal of genomics, 2016, 1-6. (  0) 0) |
| [7] |
HUANG X Y, ZHU M Q, ZHUANG L F, et al. Structural chromosome rearrangements and polymorphisms identified in Chinese wheat cultivars by 656 high-resolution multiplex oligonucleotide FISH[J]. Theoritical and applied genetics, 2018, 131(9): 1967-1986. DOI:10.1007/s00122-018-3126-2 (  0) 0) |
| [8] |
王竹林, 王德森, 何中虎, 等. 小麦品种百农64慢白粉病抗性QTL的定位[J]. 中国农业科学, 2006, 39(10): 1956-1961. WANG Z L, WANG D S, HE Z H, et al. QTL mapping for slow mildewing resistance in Chinese wheat cultivar Bainong 64[J]. Scientia agricultura sinica, 2006, 39(10): 1956-1961. DOI:10.3321/j.issn:0578-1752.2006.10.002 (  0) 0) |
| [9] |
JIN H, WEN W E, LIU J D, et al. Genome-wide QTL mapping for wheat processing quality parameters in a Gaocheng 8901/Zhoumai 16 recombinant inbred line population[J]. Frontiers in plant science, 2016, 7: 1-16. (  0) 0) |
| [10] |
BRADBURY P J, ZHANG Z, KROON D E, et al. TASSEL: software for association mapping of complex traits in diverse samples[J]. Bioinformatics, 2007, 23(19): 2633-2635. DOI:10.1093/bioinformatics/btm308 (  0) 0) |
| [11] |
YANG Y, MA Y Z, XU Z S, et al. Isolation and expression characterization of novel Vp-1 genes in wheat varieties with distinct PHS tolerance and ABA responsiveness[J]. Journal of experimental botany, 2007, 58(11): 2863-2871. DOI:10.1093/jxb/erm073 (  0) 0) |
| [12] |
曹雪连, 张衡, 姜昊, 等. 分子标记PM19-A1对1 015份小麦抗穗发芽基因型的筛选及其有效性验证[J]. 麦类作物学报, 2016, 36(10): 1283-1290. CAO X L, ZHANG H, JIANG H, et al. Detection and validation of molecular marker PM19-A1 associated with pre-harvest sprouting resistance in 1 015 wheat varieties[J]. Journal of triticeae crops, 2016, 36(10): 1283-1290. DOI:10.7606/j.issn.1009-1041.2016.10.03 (  0) 0) |
| [13] |
张春利.小麦抗穗发芽分子标记的发掘与验证[D].北京: 中国农业科学院, 2008. ZHANG C L. Development and validation of functional marker for resistance to pre-harvest sprouting[D]. Beijing: Chinese Academy of Agricultural Sciences, 2008. (  0) 0) |
| [14] |
HIMI E, MAEKAWA M, MIURA H, et al. Development of PCR markers for Tamyb10 related to R-1, red grain color gene in wheat[J]. Theoretical and applied genetics, 2011, 122(8): 1561-1576. DOI:10.1007/s00122-011-1555-2 (  0) 0) |
| [15] |
LIU W X, KOO D, XIA Q, et al. Homoeologous recombination-based transfer and molecular cytogenetic mapping of powdery mildew-resistant gene Pm57 from Aegilops searsii into wheat[J]. Theoretical and applied genetics, 2017, 130(4): 841-848. DOI:10.1007/s00122-017-2855-y (  0) 0) |
| [16] |
ZHOU Y, TANG H, CHENG M P, et al. Genome-wide association study for pre-harvest sprouting resistance in a large germplasm collection of Chinese wheat landraces[J]. Frontiers in plant science, 2017, 8: 1-13. (  0) 0) |
 2020, Vol. 52
2020, Vol. 52


