2. 四川大学 华西医院 四川 成都 610041;
3. 奥塔哥大学 计算机科学系 新西兰 达尼丁 9054
2. West China Hospital, Sichuan University, Chengdu 610041, China;
3. Department of Computer Science, University of Otago, Dunedin 9054, New Zealand
阻塞性睡眠呼吸暂停(obstructive sleep apnea,OSA)是影响人体睡眠健康的重要疾病之一。OSA的症状包括睡眠时气短、喘息、身体抽动、呼吸停止等[1-2]。然而,大多数OSA患者直到病情变得严重时才发现自己的病情,错过了早期治疗的机会。在这种情况下,OSA的早期预警是一个值得关注的问题。
尽管已经有许多方法来自动分类OSA事件[3],但较少研究关注预测睡眠呼吸暂停事件的发生。预测OSA事件对于OSA患者的治疗非常重要。例如,通过在预测的OSA事件之前增加自动气道压力装置的气压,可以防止OSA事件的发生,从而保持患者在睡眠期间的连续气流[4]。目前,在OSA事件预测任务中,主要借助传统机器学习算法和深度学习算法。
在传统机器学习算法中,Sharma等[5]使用Hermite基函数估计心电图的QRS波群,基于ECG特征,使用K-邻近、SVM和最小二乘支持向量机等机器学习分类器进行OSA的预测。Zarei等[6]通过分解ECG信号获得小波变换系数,进行非线性特征提取,然后基于SVM预测OSA事件。还有一些其他的机器学习方法用于OSA的预测,包括线性和非线性方法[7]等。基于传统机器学习的算法对特征的设计有很大的依赖性,只有当设计的特征质量较高时才能得到较好的OSA预测结果。
基于深度学习的方法利用神经网络自动学习特征[8],与传统机器学习相比,能够减少特征依赖,在OSA事件预测任务中取得了较好的成绩。Maali等[9]使用气流、腹部和胸部运动信号,应用多个人工神经网络通过事件发生的前30 s、60 s、90 s和120 s预测OSA。Le等[10]设计基于Dirichlet过程的混合高斯模型,从多感官套件记录的生理信号中预测OSA事件的发生,此模型已部署在便携式睡眠监测设备上。Haidar等[11]提出了基于卷积神经网络和马尔可夫链预测睡眠呼吸暂停事件的方法,通过前60 s呼吸信号,预测30 s内OSA事件。Taghizadegan等[12]提出了一种基于多导睡眠图信号动态行为表示的新模式,来预测30 s内的OSA事件。
虽然现有的研究在OSA事件预测中取得了不错的成绩,但是大多数研究忽略了呼吸信号间处于非欧氏空间[13]这个客观事实,对呼吸信号时空依赖特征的研究不足。
基于以上分析,提出了一种基于循环图神经网络(recurrent graph convolution network,RGCN)的OSA预测模型。该方法概述如下。
1) 本文重点研究OSA预测中一直被忽视的数据样本之间的关联性特征分析。利用已有的睡眠呼吸信号特征,构建一种可以表示呼吸信号样本关联性特征的睡眠呼吸图结构。
2) 本文基于图卷积和循环卷积构建时空循环卷积块。通过堆叠时空循环卷积块挖掘信号在时间和空间维度上的相关性,可以有效提高呼吸信号预测的有效性。
3) 基于真实数据,将提出的模型与现有模型进行对比。实验结果证明此方法不仅能有效预测呼吸信号,并且利用预测的呼吸信号进行OSA的分类,取得了准确度较高的结果。
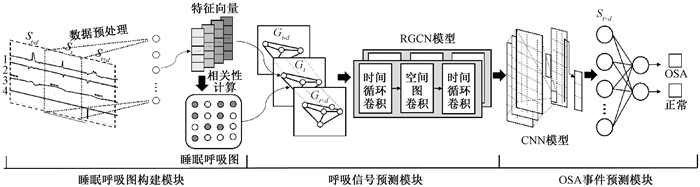
1 基于循环图卷积的OSA预测方法 1.1 基于循环图卷积的OSA预测模型如图 1所示,基于循环图卷积网络的OSA预测模型包含三个模块:睡眠呼吸图构建模块、呼吸信号预测模块、OSA事件预测模块。首先,四个通道的睡眠呼吸信号通过数据预处理操作得到各自的特征向量。然后通过相关性计算得到各个节点之间的特征矩阵,并构建睡眠呼吸图。其次,采用RGCN模型对呼吸信号间的时间和空间依赖进行学习和提取,通过两个时空循环卷积块得到ΔT时刻后的呼吸信号序列St+d。最后,使用卷积神经网络对预测的呼吸信号序列St+d进行特征提取并通过softmax分类器进行OSA预测分类。
|
图 1 阻塞性睡眠呼吸事件预测流程图 Fig. 1 Flow chart of obstructive sleep apnea event prediction |
首先定义睡眠呼吸网络为一个无向图G=(V, E, A),其中:V表示顶点集合,网络中的每个顶点分别代表鼻腔气流呼吸信号、胸部呼吸努力信号、腹部呼吸努力信号、血氧信号,|V|=N是睡眠呼吸网络中的顶点数;E表示边的集合,即顶点之间的连接;A表示睡眠呼吸网络G的邻接矩阵。
对于信号特征提取,本文将原始信号序列定义为S=(s1, s2, …, sl)∈RL×N×Ts,其中:L表示样本数;Ts表示每个样本的时间序列长度。对于每个样本si,si∈S(i=1, 2, 3,…, L),通过低通滤波和高通滤波对数据进行去噪声处理得到向量特征,并定义每个样本si的特征矩阵Xi=(x1i, x2i, …, xNi)T∈RN×F, xni∈RF(n∈{1, 2, …, N})表示样本i处节点n的向量特征。
对于待识别的每个睡眠呼吸信号的相关性,睡眠图学习模块利用了皮尔森相关系数计算图卷积的邻接矩阵A,计算为
| $ \boldsymbol{A}_{m n}=r\left(x_{m}, x_{n}\right)=\frac{\sum\limits_{i=1}^{n}\left(x_{i}^{m}-\bar{x}_{m}\right)\left(x_{i}^{n}-\bar{x}_{n}\right)}{\sqrt{\sum\limits_{i=1}^{n}\left(x_{i}^{m}-\bar{x}_{m}\right)^{2}} \sqrt{\sum\limits_{i=1}^{n}\left(x_{i}^{n}-\bar{x}_{n}\right)^{2}}}, $ | (1) |
其中:Amn=r(xm, xn),m, n∈{1, 2, …, N}表示节点xm和xn之间基于输入特征矩阵Xi=(x1i, x2i, …, xNi)T∈RN×F的连接关系;r代表变量X和变量Y的皮尔逊相关系数;xm和xn表示两者的样本均值。
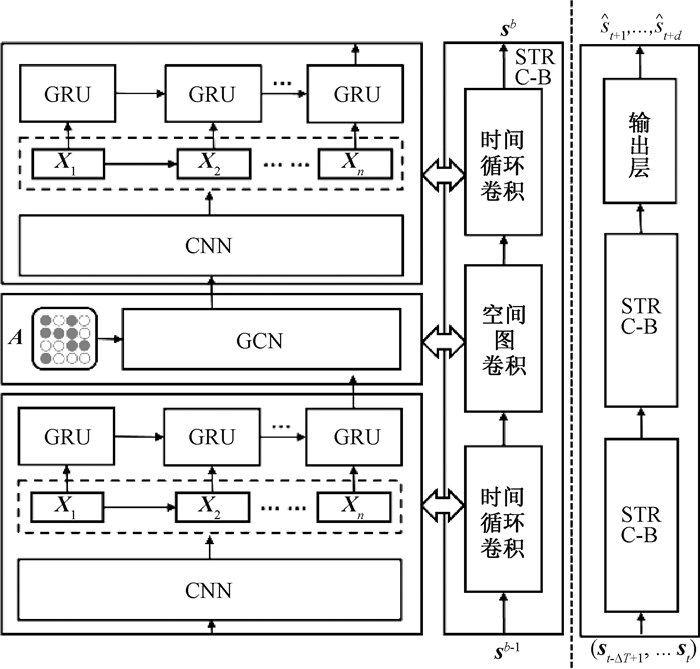
1.1.2 呼吸信号预测模块呼吸信号预测模块基于RGCN模型实现,用于提取空间和时间特征。将睡眠呼吸图中邻近节点的信息进行汇总,提取每个睡眠呼吸图的空间特征,并利用来自相邻呼吸阶段的时间依赖性来捕获时间特征。如图 2所示。RGCN由2个时空循环卷积块组成,每个时空循环卷积块又包括了2个时间循环卷积层和1个空间图卷积层。每个模块的细节描述如下。
|
图 2 RGCN模型结构图 Fig. 2 The overall structure of RGCN |
1) 空间特征提取
本文从谱图理论出发,通过图卷积的方法从空间维度中提取相应的特征。对于要预测的每段呼吸信号,之前已经通过相关性计算得到了用于图卷积的邻接矩阵A,并且将邻接矩阵A和特征矩阵X共同构成睡眠呼吸图,每个睡眠呼吸图使用GCN模型提取遥远区域的空间依赖关系。
本文直接使用图结构数据在空间域上进行高阶特征提取,并且利用图拉普拉斯算子中的切比雪夫展开来减少模型的计算量。
2) 时间特征提取
专家通过结合相邻呼吸信号对当前呼吸信号进行预测,为了捕捉这样的呼吸信号转换规则,本文使用CNN+GRU结构在时间维度上执行卷积操作。相比LSTM来说,GRU模型具有参数量更少、模型训练能力更快等特点。因此,本文采用CNN+GRU的结构对呼吸信号数据进行时间相关性的分析,通过单元间的信息传递捕捉呼吸信号的时间动态特征。
3) 动态时空图卷积块
最后本文通过构造时空循环卷积块(spatial-temporal recurrent convolution block,STRC-B)对时间域和空间域进行联合处理,以实现时空域特征的融合,并且使用2块时空循环卷积块进行堆叠。
如图 2所示,中间的空间层能够实现图卷积和时间循环卷积之间的过渡,有利于在空间状态中实现快速传播。该架构充分应用瓶颈策略,利用图卷积层对通道c进行维度的升降,实现数据的特征压缩和尺度压缩。另外,在每个时空循环卷积块中,为了避免过度拟合,在每一层后都采用了dropout处理。对于块b-1来说,时空循环卷积块的输入sb-1和输出sb都是3维向量。
本文在网络的末尾添加了一个输出层(如图 2最右侧),该输出层为增添了全连接层的时间循环卷积层。该输出层将最后一个STRC-B的输出与单步预测之间进行映射,所以可以利用该模型得到最后的输出Z∈Rn×c,再利用c通道上的线性转换
通过预测得到St+d的呼吸信号以后,再通过CNN层对呼吸信号进行特征提取,最后输出St+d这段序列的阻塞性睡眠呼吸事件预测结果。在之前的研究[14]中,CNN可以成功地学习从多导睡眠图中检测呼吸暂停事件所需要的特征。本文使用上文预测得到的鼻腔气流信号、腹部和胸部信号的30 s片段,基于CNN将其分为正常事件和阻塞性睡眠呼吸暂停事件,以达到阻塞性睡眠呼吸暂停预测的目的。
2 实验结果 2.1 数据集与数据预处理 2.1.1 数据集MESA数据集[15]中包括2 056名不同种族和性别的参与者睡眠时的多导睡眠图记录。每个记录都有一个信号文件(.EDF)以及事件注释文件(.XML)。本实验采用其中36条多导睡眠图(polysomnography,PSG)记录,每条数据包含7~9 h的PSG记录。PSG记录中的信号包括鼻腔气流信号、血氧信号、胸部呼吸努力信号、腹部呼吸努力信号。本实验包含9 597条训练集、3 199条测试集以及3 199条验证集。
2.1.2 数据预处理从MESA数据库中读取的原始数据中含有大量噪声,因此本文通过滤波处理对所有鼻腔气流信号、胸部呼吸努力信号和腹部呼吸努力信号进行噪声去除。将鼻腔气流信号进行0.24 Hz的低通滤波和0.008 Hz的高通滤波,胸部呼吸信号进行0.1 Hz的低通滤波和0.009 Hz的高通滤波,腹部呼吸信号进行0.35 Hz的低通滤波。通过对滤波前、后的波形比较,滤波后的信号更加平滑,说明该方法能有效减小原始信号中的噪声,降低了后续模型预测的误差。
2.2 实验设置 2.2.1 对照模型设置本文选择SVR模型[16]、LSTM模型[17]、GCN模型[18]、T-GCN模型[19]、STGCN模型[20]进行对比实验,并将模型相关工作分为三部分:空间拓扑构建、空间依赖建模和时间依赖建模,如表 1所示。
|
|
表 1 不同模型的相关工作 Tab. 1 Related work on different models |
1) 基于RGCN模型的呼吸信号预测模型的参数设置。实验经超参数调优后,四通道呼吸信号的频率设置为5 Hz,RGCN中CNN模块卷积核大小设置为3,初始学习率为0.000 1,批大小为128,共训练100个周期。本文使用平均平方误差函数作为损失函数(MSEloss),使用Adam作为优化算法计算,
| $ {MSEloss}\left(x_{i}, y_{i}\right)=\left(x_{i}-y_{i}\right)^{2} \text {, } $ |
其中: yi代表第i个样本的真实值; xi代表第i个样本的预测值。
2) 基于CNN模型的OSA事件预测模型参数设置。CNN模块由3个卷积层、3个最大池化层和1个具有两个节点的softmax层组成,分别输出下一个30 s的正常呼吸和OSA类的概率。最终的预测类别是与概率最高的节点对应的类别。每个卷积层后面都有一个最大池化层,池大小为2。三个卷积层的过滤器数量设置为32,并使用ReLU激活函数。三层中每一层的内核为:Conv1的卷积核大小为3×10,步长为10;Conv2的卷积核大小为1×6,步长为6;Conv3的卷积核大小为1×4,步长为4。第一层内核高度设置为3,因此滤波器同时通过三个信号并组合特征。在训练期间,使用0.5的dropout来避免过度拟合。Adam优化器用于最小化分类交叉熵函数。训练epoch的数量设置为300,批量大小为100。
2.2.3 模型性能评估指标1) 呼吸信号预测实验的评估指标。本文的信号预测模型评估同样采用国际通用的连续变量预测评测指标,即平均绝对误差(mean absolute error,MAE)和均方根误差(root mean squared error,RMSE)来衡量变化精度,分别计算为
| $ \begin{gathered} \operatorname{MAE}(X, h)=\frac{1}{m} \sum\limits_{i=1}^{m}\left|h\left(x_{i}\right)-y_{i}\right|, \\ \operatorname{RMSE}(X, h)=\sqrt{\frac{1}{m} \sum\limits_{i=1}^{m}\left(h\left(x_{i}\right)-y_{i}\right)^{2}} 。\end{gathered} $ |
2) OSA预测实验的评估指标。本文的OSA事件预测模型采用经典的预测评测指标,即准确率(Accuracy)、召回率(Recall)、精确率(Precision)和F1值(F1)。以呼吸事件Rm为例,把信号的标注结果视为真实标签,计算公式为
| $ { Accuracy }_{m}=\frac{T P_{m}+T N_{m}}{T P_{m}+T N_{m}+F P_{m}+F N_{m}}, $ |
| $ { Recall }_{m}=\frac{T P_{m}}{T P_{m}+F N_{m}}, $ |
| $ { Precision }_{m}=\frac{T P_{m}}{T P_{m}+F P_{m}}, $ |
| $ F 1_{m}=\frac{2 * P_{m} * R_{m}}{P_{m}+R_{m}}, $ |
其中:TPm代表测试样本中预测类别为Rm且真实类别也为Rm的样本数;FPm代表预测类别为Rm而真实类别不为Rm的样本数;TPm+FPm代表预测类别为Rm的样本数;TNm代表预测类别为Rm但真实类别不为Rm的样本数;FNm代表预测类别不为Rm且真实类别不为Rm的样本数。
2.3 实验结果为了验证本文设计的基于RGCN呼吸信号预测模型的有效性,本文通过前30 s、60 s和90 s的信号预测后30 s的睡眠呼吸信号。不同模型在MASE数据集上根据前ΔT时间预测后30 s呼吸信号的效果如表 2所示。
|
|
表 2 不同模型的呼吸信号预测性能对比 Tab. 2 Comparison of respiratory signal prediction performance based on different models |
从表 2可知,本文提出的基于时空图循环卷积网络在ΔT=60 s时,RGCN模型的性能达到最好,MAE达到1.061 3,RMSE达到2.994 1,比基线模型STGCN降低了0.231 7和0.832 2,该结果表明运用循环时间网络比单纯用一维卷积能更好地提取时间信息,验证了该模型的有效性。
由于呼吸信号采集口鼻、胸部、腹部等部位处于非欧氏空间中,并且每个部位间相互联系,因此图是最适合用来表示节点连接的结构。将GCN和LSTM进行对比发现,GCN模型的MAE和RMSE分别下降了0.314 9和1.234 0,也验证了在睡眠呼吸信号预测任务中,使用图卷积神经网络学习空间特征更能有助于信号拟合。而将T-GCN和GCN对比,T-GCN模型的MAE和RMSE分别下降了0.249和0.107,T-GCN性能比GCN性能更好,说明学习信号的时间特征依赖在呼吸信号预测任务中效果更好。
在不同模型加入了卷积层后的阻塞性睡眠呼吸暂停事件预测整体效果如表 3所示。
|
|
表 3 不同模型的OSA事件预测性能对比 Tab. 3 Comparison of OSA event prediction performance based on different models |
由表 3可以看出,同样在MESA数据集上,与其他模型相比,本文采用的时空图循环卷积RGCN模型的Accuracy、Recall、Precision和F1取得了更高的值,验证了本文基于CNN模型的OSA事件预测方法的有效性。与图神经网络模型相比,LSTM的F1值最低,表明该模型只关注单个变体的时间序列,无法有效捕捉变体之间的相关性,这也严重限制了面对多个变体的时间序列时的效率。此外,与GCN相比,T-GCN、STGCN和RGCN的F1值更高,说明基于不同的睡眠呼吸信号使用时空卷积捕捉信号间的相关性使得OSA预测效果更好。STGCN与RGCN相比,F1提高了3.5%,说明同样是使用图卷积提取空间信息,使用循环卷积网络提取时间信息比使用单纯的卷积网络预测效果更好。
3 结束语OSA作为影响人体睡眠健康的重要疾病之一,如何实现OSA早筛对医学信息技术领域的发展具有重大意义。因此,本文设计了基于RGCN模型的OSA事件预测方法。通过对比实验验证了本文提出的模型不仅能有效预测呼吸信号,并且利用预测出的呼吸信号进行OSA的分类也能取得准确度较高的结果。为睡眠呼吸监测中OSA的实时预警研究奠定了基础。在未来的工作中,将考虑把二分类任务拓展到多分类任务,进一步研究基于少样本的OSA预测问题,从而增加预测OSA事件的细度。
| [1] |
LÉGER D, STEPNOWSKY C. The economic and societal burden of excessive daytime sleepiness in patients with obstructive sleep apnea[J]. Sleep medicine reviews, 2020, 51: 101275. (  0) 0) |
| [2] |
BENJAFIELD A V, AYAS N T, EASTWOOD P R, et al. Estimation of the global prevalence and burden of obstructive sleep apnoea: a literature-based analysis[J]. The lancet respiratory medicine, 2019, 7(8): 687-698. DOI:10.1016/S2213-2600(19)30198-5 (  0) 0) |
| [3] |
FAIZAL W M, GHAZALI N N N, BADRUDDIN I A, et al. A review of fluid-structure interaction simulation for patients with sleep related breathing disorders with obstructive sleep[J]. Computer methods and programs in biomedicine, 2019, 180: 105036. DOI:10.1016/j.cmpb.2019.105036 (  0) 0) |
| [4] |
POMBO N, GARCIA N, BOUSSON K. Classification techniques on computerized systems to predict and/or to detect apnea: a systematic review[J]. Computer methods and programs in biomedicine, 2017, 140: 265-274. DOI:10.1016/j.cmpb.2017.01.001 (  0) 0) |
| [5] |
SHARMA H. An algorithm for sleep apnea detection from single-lead ECG using Hermite basis functions[J]. Computers in biology and medicine, 2016, 77: 116-124. DOI:10.1016/j.compbiomed.2016.08.012 (  0) 0) |
| [6] |
ZAREI A, ASL B M. Automatic detection of obstructive sleep apnea using wavelet transform and entropy-based features from single-lead ECG signal[J]. IEEE journal of biomedical and health informatics, 2019, 23(3): 1011-1021. (  0) 0) |
| [7] |
SHOKOUEINEJAD M, FERNANDEZ C, CARROLL E, et al. Sleep apnea: a review of diagnostic sensors, algorithms, and therapies[J]. Physiological measurement, 2017, 38(9): R204-252. DOI:10.1088/1361-6579/aa6ec6 (  0) 0) |
| [8] |
TUNCER A S, AKILOTU B N, Toraman S, et al. A deep learning-based decision support system for diagnosis of OSAS using PTT signals[J]. Medical hypotheses, 2019, 127: 15-22. DOI:10.1016/j.mehy.2019.03.026 (  0) 0) |
| [9] |
MAALI Y, AL-JUMAILY A. Multi neural networks investigation based sleep apnea prediction[J]. Procedia computer science, 2013, 24: 97-102. DOI:10.1016/j.procs.2013.10.031 (  0) 0) |
| [10] |
LE T Q, CHENG C Q, SANGASOONGSONG A, et al. Wireless wearable multisensory suite and real-time prediction of obstructive sleep apnea episodes[J]. IEEE journal of translational engineering in health and medicine, 2013, 1: 2700109. (  0) 0) |
| [11] |
HAIDAR R, KOPRINSKA I, JEFFRIES B. Sleep apnea event prediction using convolutional neural networks and Markov chains[C]//International Joint Conference on Neural Networks. Piscataway: IEEE Press, 2020: 1-8.
(  0) 0) |
| [12] |
TAGHIZADEGAN Y, DABANLOO N J, MAGHOOLI K, et al. Prediction of obstructive sleep apnea using ensemble of recurrence plot convolutional neural networks (RPCNNs) from polysomnography signals[J]. Medical hypotheses, 2021, 154: 110659. (  0) 0) |
| [13] |
HOSSEN A, QASIM S. Identification of obstructive sleep apnea using artificial neural networks and wavelet packet decomposition of the hrv signal[J]. The journal of engineering research, 2020, 17(1): 24. (  0) 0) |
| [14] |
HAIDAR R, MCCLOSKEY S, KOPRINSKA I, et al. Convolutional neural networks on multiple respiratory channels to detect hypopnea and obstructive apnea events[C]//International Joint Conference on Neural Networks. Piscataway: IEEE Press, 2018: 1-7.
(  0) 0) |
| [15] |
ZHANG G Q, CUI L C, MUELLER R, et al. The national sleep research resource: towards a sleep data commons[J]. Journal of the American medical informatics association, 2018, 25(10): 1351-1358. (  0) 0) |
| [16] |
WANG G Y, LI Z B, LI G J, et al. Real-time liver tracking algorithm based on LSTM and SVR networks for use in surface-guided radiation therapy[J]. Radiation oncology, 2021, 16(1): 13. (  0) 0) |
| [17] |
GREFF K, SRIVASTAVA R K, KOUTNÍK J, et al. LSTM: a search space odyssey[J]. IEEE transactions on neural networks and learning systems, 2017, 28(10): 2222-2232. (  0) 0) |
| [18] |
KIPF T N, WELLING M. Semi-supervised classification with graph convolutional networks[EB/OL]. (2017-02-22)[2022-03-20]. https://doi.org/10.48550/arXiv.1609.02907.
(  0) 0) |
| [19] |
ZHAO L, SONG Y J, ZHANG C, et al. T-GCN: a temporal graph convolutional network for traffic prediction[J]. IEEE transactions on intelligent transportation systems, 2020, 21(9): 3848-3858. (  0) 0) |
| [20] |
YU B, YIN H T, ZHU Z X. Spatio-temporal graph convolutional networks: a deep learning framework for traffic forecasting[C]//Proceedings of the 27th International Joint Conference on Artificial Intelligence. Palo Alto: AAAI Press, 2018: 3634-3640. .
(  0) 0) |
 2023, Vol. 55
2023, Vol. 55


