| 基于薄层CT的影像组学模型对非小细胞肺癌EGFR DEL19和L858R敏感性突变的预测价值 |
2. 浙江省影像诊断与介入微创研究重点实验室, 浙江 丽水 323000;
3. 浙江省丽水市中医院放射科, 浙江 丽水 323000
肺癌为全球发病率最高的恶性肿瘤,且绝大多数为非小细胞肺癌(non-small cell lung cancer, NSCLC)[1]。NSCLC患者除手术治疗外,放、化疗等手段也可显著提高患者预后,但不良反应损伤较大,限制了其在临床中的应用。随着基因学的发展,靶向治疗逐渐显现出对肿瘤治疗的优势,相对于传统放化疗,其可针对性杀灭肿瘤细胞,减少对机体正常细胞的损害,从而减少不良反应。肺癌靶向药物发展迅速,最具代表性的有表皮生长因子受体酪氨酸激酶抑制剂(EGFR-TKIs),其可有效延长EGFR敏感性突变NSCLC患者的无进展生存期及总生存期,还可提高患者的生活质量[2]。而EGFR敏感性突变中,以19号外显子缺失(DEL19)及21号外显子替代(L858R)最常见,且DEL19突变稍多于L858R突变[3];两者对TKIs治疗的反应性不同,TKIs治疗后DEL19突变患者的无进展生存期及总生存期明显优于L858R突变患者[3-4]。研究表明,L858R突变患者的化疗效果更好[5]。因此,准确识别DEL19突变及L858R突变对临床决策尤为重要。但目前基因测序成本较高,且为侵入性检查,检测结果也会受取样影响造成偏差。
近年来,各种组学研究发展迅速,利用组学的方法可从影像中挖掘出更多肉眼难以识别的特征,为疾病的诊疗提供依据[6]。影像组学已应用于各个系统,帮助临床进行精准诊断及预后评估[7-12]。Kim等[11-12]运用影像组学成功预测脑胶质瘤患者的异柠檬酸脱氢酶(IDH)及O6-甲基鸟嘌呤DNA甲基转移酶(MGMT)突变类型。Weng等[13-14]将影像组学应用于肺癌患者的诊断及EGFR突变类型预测。这些研究均表明影像组学不仅能够对疾病的宏观表现进行分析,还可对微观的基因分型进行甄别。本研究基于NSCLC患者的CT平扫图像构建可预测EGFR DEL19及L858R突变的影像组学模型,并评估其预测效能。
1 资料与方法 1.1 一般资料收集丽水市中心医院2015年7月至2019年1月经病理证实的NSCLC患者共590例,经纳入及排除标准筛选,最终纳入149例,其中DEL19突变63例,L858R突变86例。149例患者按照6∶4的比例随机分为训练集(104例)和验证集(45例)。训练集DEL19突变44例,L858R突变60例;验证集DEL19突变19例,L858R突变26例。
纳入标准:(1)CT平扫前未接受任何针对肿瘤的治疗;(2)经组织病理学诊断为NSCLC; (3)基因检测证实存在EGFR DEL19或L858R突变。排除标准:(1)非原发肿瘤;(2)获取CT图像前已行放化疗等姑息性治疗;(3)图像存在伪影,影响病灶观察;(4)存在其他类型突变者。
本研究经医院伦理委员会批准[科研伦审(2022)第(122)号]。
1.2 仪器与方法使用Philips Brilliance 64排CT扫描仪行CT平扫。扫描参数:200 m A, 120 kV,层厚0.9 mm;运用iDose3混合迭代重建算法进行重建,层距0.45 mm,层厚2 mm;视野(15 cm×15 cm)~(20 cm×20 cm),螺距1.2,旋转时间350 ms,矩阵1 024×1 024。图像在Philips Extended Brilliance工作站(EBW)进行处理。
1.3 纹理特征提取将所有患者的胸部薄层CT肺窗DICOM格式图像导入ITK-SNAP软件(http://www.itksnap.org/)。由2名具有10年以上胸部影像诊断经验的医师,分别独立勾画病灶ROI,勾画时沿高密度病灶轮廓边缘手动逐层分割,最后融合生成容积感兴趣区(volume of interest, VOI),如有不同意见,则与第3位具有20年胸部影像诊断经验的医师共同讨论决定。将分割好的VOI文件及原始图DICOM文件导入GE A.K.软件(版本3.0.1.A)进行影像组学分析,共得到396个不同的纹理特征。
1.4 降维及特征筛选基于薄层CT平扫图像,利用放射组学A.K.软件共提取到396个纹理特征进行预处理,用中位数替代参数中的异常值。首先行Z-score标准化处理,以去除不同数据的单位限制。符合正态分布的数据采用方差分析进行筛选,不符合正态分布数据则采用Kruskal-Wallis检验降维。采用Spearman秩相关检验去冗余(去除相关性 > 0.9的参数)。最后采用最小绝对收缩与选择算子算法(LASSO)回归分析及10倍交叉验证法对数据降维,筛选出高度相关的纹理特征。对得到的纹理参数行多因素logistic回归分析。
1.5 统计学分析与模型构建采用组内相关系数(intraclass correlation coefficient, ICC)对2名医师提取的特征行一致性评估,ICC≥0.75表示一致好。使用R语言行统计分析(版本3.4.4, https://www.r-project.org/)。利用多因素logistic回归分析构建训练集和验证集的预测模型,即基于CT图像的影像组学模型、基于临床特点的临床模型,以及两者的联合模型,并用ROC曲线及列线图评价各模型的预测效能。以P < 0.05为差异有统计学意义。
2 结果 2.1 临床资料的单因素、多因素分析(表 1)| 表 1 149例非小细胞肺癌患者临床资料的单因素、多因素分析 |
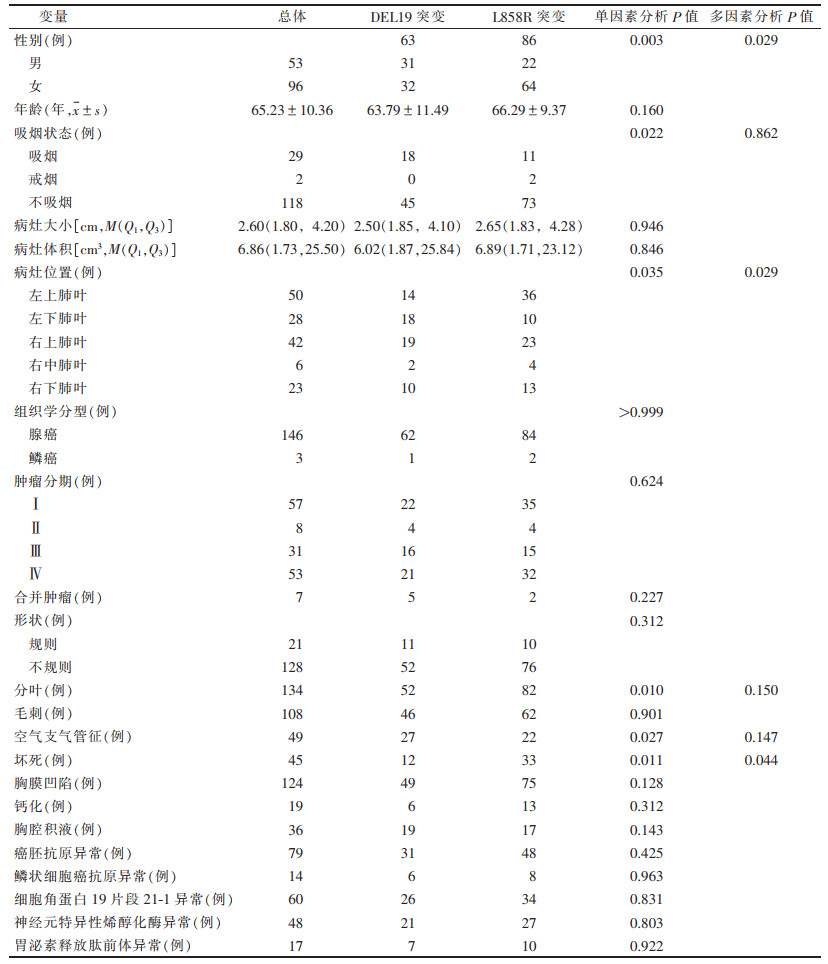 |
单因素分析显示,性别、吸烟状态、病灶位置、分叶、空气支气管征、坏死与2个突变亚型均有相关性(均P < 0.05)。多因素分析显示,性别、病灶位置、坏死与2个突变亚型均有相关性(均P < 0.05)。
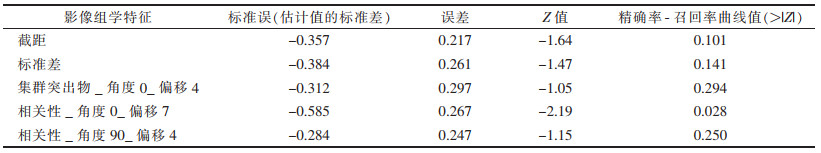
2.2 影像组学特征2名医师分割的病灶VOI所提取的影像组学特征一致性好(ICC=0.834~0.892)。在病灶VOI中提取396个影像组学特征。经过降维筛选出4个显著性最强的纹理特征,分别为标准差(stdDeviation)、集群突出物_角度0_偏移4(ClusterProminence_angle0_offset4)、相关性_角度0_偏移7(Correlation_angle0_offset7)和相关性_角度90_偏移4(Correlation_angle90_offset4)。基于以上4个纹理参数(表 2),计算出相应的影像组学评分纳入模型构建。2种突变亚型患者的小提琴图显示,两者影像组学评分差异有统计学意义(图 1)。
| 表 2 筛选出4个组学特征的模型系数 |
 |
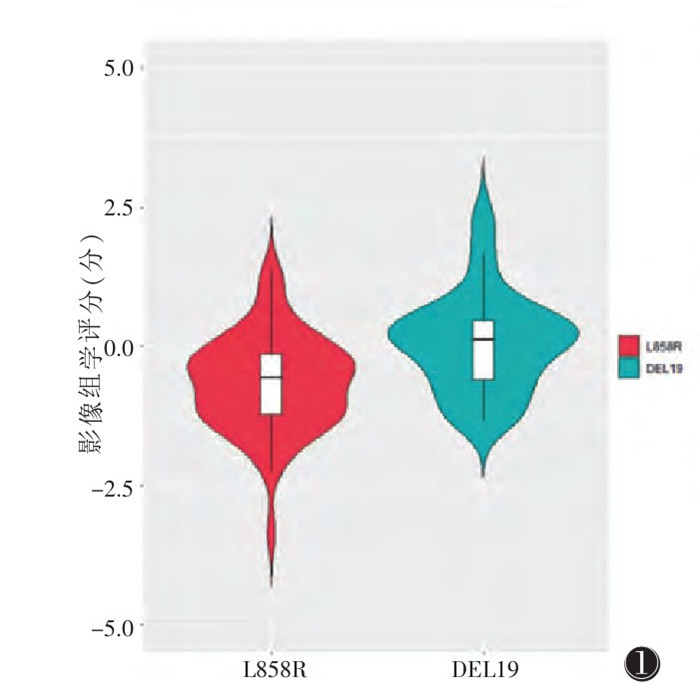 |
| 图 1 表皮生长因子受体基因(EGFR) L858R突变和DEL19突变影像组学评分的小提琴图 |
2.3 模型预测效能
临床特征模型显示,训练集AUC为0.643(95%CI0.543~0.735),敏感度和特异度分别为62.2%和64.4%;验证集AUC为0.778(95%CI 0.630~0.888),敏感度为84.2%,特异度为73.1%。影像组学模型显示,训练集AUC为0.713(95%CI 0.616~0.798),敏感度和特异度分别为64.4%和76.3%;验证集AUC为0.698(95%CI0.543~0.826),敏感度为73.7%,特异度为61.5%。联合模型显示,训练集AUC为0.744(95%CI 0.649~0.824),敏感度和特异度分别为75.6%和71.2%;验证集AUC为0.874(95%CI 0.742~0.954),敏感度为84.2%,特异度为88.5%(图 2)。验证集中联合模型的预测效能优于影像组学模型(P=0.004)。列线图与校正曲线显示,联合模型在训练集和验证集中的预测概率与实际概率相近,与对角线拟合效果优于影像组学模型(图 3, 4),对EGFR有较好的诊断能力。
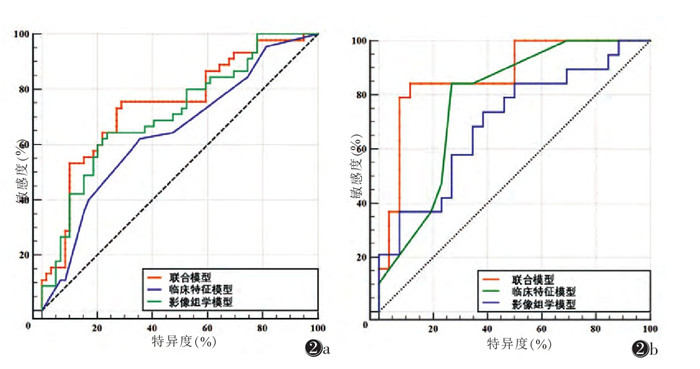 |
| 注:图 2a为训练集(AUC=0.744, 95%CI 0.649~0.824);图 2b为验证集(AUC=0.874, 95%CI 0.742~0.954) 图 2 基于训练集与验证集构建3种模型的ROC曲线 |
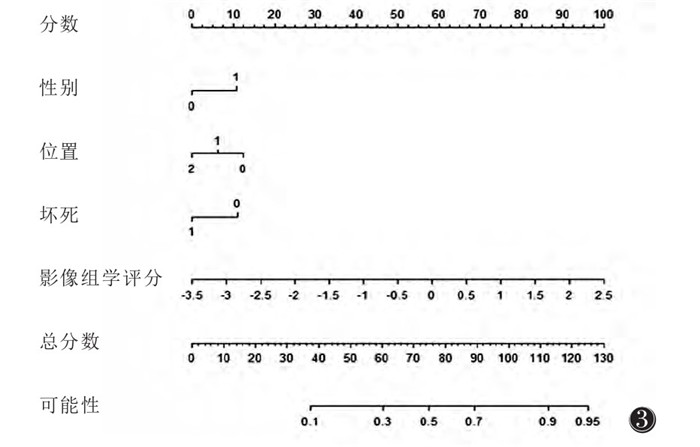 |
| 图 3 联合模型列线图 |
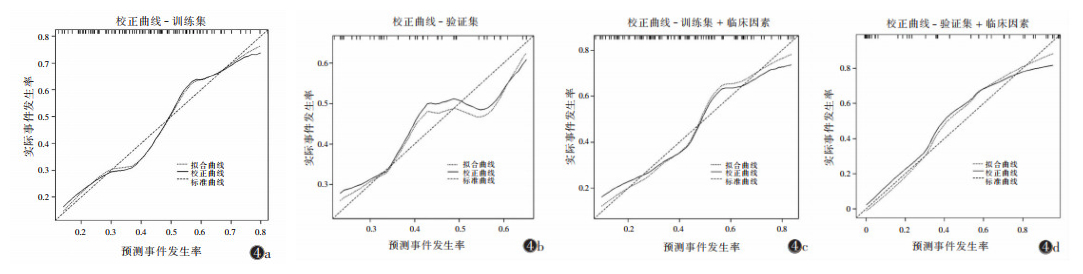 |
| 注:图 4a, 4b分别为影像组学模型训练集与验证集校正曲线;图 4c, 4d分别为纳入临床因素的联合模型训练集与验证集校正曲线 图 4 影像组学模型及联合模型校正曲线 |
3 讨论
随着分子生物学的发展,分子靶向药物更多的应用于肿瘤的治疗之中。NSCLC患者治疗不仅基于临床特点及肿瘤形态学,更多地依赖于个体的基因突变特征[15],尤其是EGFR突变特点。DEL19和L858R突变约占NSCLC患者EGFR敏感性突变的90%[16]。也有研究表明,与传统化疗相比,二代TKIs类药物阿法替尼治疗可有效提高NSCLC EGFR敏感性突变患者的无进展生存期[17-18]。而随着EGFR靶向药物研究的深入,部分研究表明,NSCLC EGFR DEL19突变患者对TKIs治疗反应性优于L858R突变患者,且L858R突变的NSCLC患者化疗效果更好[3-5]。Yang等[5]的研究表明,对于L858R突变患者,相比于TKIs,含铂类化疗药物治疗的中位总生存期更长。而Kato等[19]的研究也表明,TKIs相较于化疗能明显改善DEL19患者的总生存期,而L858R患者效果较差。因此,对于NSCLC患者的TKIs靶向治疗,临床不仅需明确是否存在EGFR敏感性突变,也应区分敏感性突变亚型,这更有利于患者的精准及个体化治疗。目前,临床最常用的基因检测方法为活检,但其为侵入性检查,存在由于取样误差造成样本无法代表病灶的整体生物学特征的可能。临床研究表明,由于缺乏足够的肿瘤细胞或可扩增的DNA,导致10%~20%的NSCLC活检组织不足以进行分子分析[6],对临床工作造成困扰。
CT扫描2 mm的薄层图像已普及,且平扫较增强扫描适用范围更广,具有便捷、廉价、无创、可重复等优点。本研究采用影像组学对CT图像进行数据挖掘,提取人眼难以识别的图像纹理特征,构建影像组学模型,探讨基于影像组学的预测模型对NSCLC患者EGFR敏感性突变亚型的预测能力。
既往文献报道多是基于临床或宏观影像因素区分EGFR突变状态。Rizzo等[20-21]认为,空气支气管征、胸膜凹陷征、肺气肿和吸烟与NSCLC患者EGFR敏感性突变相关。肖磊等[22]的研究显示,EGFR敏感突变与非敏感突变组(包括野生和非敏感突变)的空气支气管征、胸膜凹陷征、空泡征、磨玻璃密度影、毛刺征、分叶征及胸腔积液差异均无统计学意义。以上研究表明,空气支气管征、胸膜凹陷征等影像学征象有助于区分EGFR突变与否,但未发现其对识别敏感性突变有帮助。本研究纳入149例发生EGFR敏感性突变(包括DEL19及L858R)的NSCLC患者,筛选出3个临床(包括性别、病灶部位、坏死)及影像特征与上述突变亚型相关,但基于临床特点及影像学所能选取的特征有限。影像组学则可充分挖掘医学影像深部数据,探索其与EGFR突变的关系,用于疾病诊断、预后评估和指导临床决策[23]。多项研究报道影像组学在预测EGFR突变中的应用,但鲜有报道运用影像组学预测DEL19或L858R突变[24-25]。Liu等[26]利用影像组学所构建模型预测DEL19、L858R突变的特异度分别为86.7%(69.3%~96.2%)和70.4%(49.8%~86.3%)。该研究采用CT增强扫描图像,优点是可提供更多可供挖掘的数据,缺点是检查成本升高,同时部分患者存在对比剂使用禁忌证,使该模型适用范围受限。Li等[27]利用影像组学识别DEL19和L858R突变与野生型及其他突变,预测效能较好,其在验证集中的AUC达0.775,该研究识别了DEL19和L858R突变与其他突变或野生型的基因型,但并未进一步区分DEL19突变和L858R突变。本研究选择NSCLC患者薄层CT平扫图像,层厚2 mm,检查成本相对较低,可重复性好,无明确禁忌证,优势明确,所构建的模型适用范围更广泛,能达到理想效果;影像组学模型在验证集中预测DEL19和L858R突变的AUC达0.698;联合临床因素所构建的联合模型在验证集中AUC显著提高,达0.874;验证集中联合模型的预测效能优于影像组学模型(P=0.004);列线图也表明联合模型对DEL19突变和L858R突变的预测效能更佳。
本研究的局限性:(1)入组患者有限,因部分患者仅行EGFR突变的检测,未行DEL19和L858R位点的检测,后期将扩大样本量进一步分析。(2)采用2 mm层厚平扫图像,优点是构建模型适用范围更广,缺点是厚层图像可供挖掘的数据不如增强扫描图像。(3)纳入的临床因素相对较少,可能是由于样本量小、未筛选出其他相关的临床因素。后续研究应增大样本量,将病灶按大小分层,并纳入纵隔窗可显示的薄层增强扫描图像进行研究,这样可获取多一个维度的纹理特征。
综上所述,以2 mm薄层平扫图像为基础构建的影组学模型应用范围广,且能够较准确预测NSCLC患者敏感性突变亚型,联合相关临床因素所构建的联合模型对DEL19和L858R突变的预测效能最优。
| [1] |
HERBST R S, MORGENSZTERN D, BOSHOFF C. The biology and management of non-small cell lung cancer[J]. Nature, 2018, 553(7689): 446-454. DOI:10.1038/nature25183 |
| [2] |
SUDA K, MITSUDOMI T. Role of EGFR mutations in lung cancers: prognosis and tumor chemosensitivity[J]. Arch Toxicol, 2015, 89(8): 1227-1240. DOI:10.1007/s00204-015-1524-7 |
| [3] |
ROSELL R, MORAN T, QUERALT C, et al. Screening for epidermal growth factor receptor mutations in lung cancer[J]. N Engl J Med, 2009, 361(10): 958-967. DOI:10.1056/NEJMoa0904554 |
| [4] |
KANEDA T, YOSHIOKA H, TAMIYA M, et al. Differential efficacy of cisplatin plus pemetrexed between L858R and DEL-19 in advanced EGFR-mutant non-squamous non-small cell lung cancer[J]. BMC Cancer, 2018, 18(1): 6. DOI:10.1186/s12885-017-3952-7 |
| [5] |
YANG J C, WU Y L, SCHULER M, et al. Afatinib versus cisplatin-based chemotherapy for EGFR mutation-positive lung adenocarcinoma(LUX-Lung 3 and LUX-Lung 6): analysis of overall survival data from two randomised, phase 3 trials[J]. Lancet Oncol, 2015, 16(2): 141-151. DOI:10.1016/S1470-2045(14)71173-8 |
| [6] |
GILLIES R J, KINAHAN P E, HRICAK H. Radiomics: images are more than pictures, they are data[J]. Radiology, 2016, 278(2): 563-577. DOI:10.1148/radiol.2015151169 |
| [7] |
蔡一奇, 施明明, 许利斌, 等. 影像组学在胃肿瘤诊疗中的应用进展[J]. 温州医科大学学报, 2020, 50(12): 1025-1029. |
| [8] |
蒋乐真, 郭艺帆, 苏洁惠, 等. 基于CT平扫的影像组学模型在预测老年人腰椎骨质疏松中的价值[J]. 温州医科大学学报, 2021, 51(11): 885-890. |
| [9] |
夏水伟, 周永进, 陈春妙, 等. 基于MRI增强T1WI影像组学预测模型鉴别高级别胶质瘤IDH 1突变型与野生型的价值[J]. 温州医科大学学报, 2021, 51(10): 800-805. |
| [10] |
周永进, 钟屹, 赵雪妙, 等. 磁共振扩散加权成像联合T2WI图纹理分析定量评估乳腺良恶性结节的价值[J]. 温州医科大学学报, 2019, 49(9): 667-672. |
| [11] |
KIM M, JUNG S Y, PARK J E, et al. Diffusion-and perfusion-weighted MRI radiomics model may predict isocitrate dehydrogenase(IDH)mutation and tumor aggressiveness in diffuse lower grade glioma[J]. Eur Radiol, 2020, 30(4): 2142-2151. DOI:10.1007/s00330-019-06548-3 |
| [12] |
TAN Y, MU W, WANG X C, et al. Wang, et al. Wholetumor radiomics analysis of DKI and DTI may improve the prediction of genotypes for astrocytomas: a preliminary study[J]. Eur J Radiol, 2020, 124: 108785. DOI:10.1016/j.ejrad.2019.108785 |
| [13] |
WENG Q, HUI J, WANG H, et al. Radiomic feature-based nomogram: a novel technique to predict EGFR-activating mutations for EGFR tyrosin kinase inhibitor therapy[J]. Front Oncol, 2021, 11: 590937. DOI:10.3389/fonc.2021.590937 |
| [14] |
HU X, YE W, LI Z, et al. Non-invasive evaluation for benign and malignant subcentimeter pulmonary groundglass nodules (≤1 cm) based on CT texture analysis[J]. Br J Radiol, 2020, 93(1114): 20190762. DOI:10.1259/bjr.20190762 |
| [15] |
CHEN S, FENG S, WEI J, et al. Pretreatment prediction of immunoscore in hepatocellular cancer: a radiomics-based clinical model based on Gd-EOB-DTPA-enhanced MRI imaging[J]. Eur Radiol, 2019, 29(8): 4177-4187. DOI:10.1007/s00330-018-5986-x |
| [16] |
CHO A, HUR J, MOON Y W, et al. Correlation between EGFR gene mutation, cytologic tumor markers, 18F-FDG uptake in non-small cell lung cancer[J]. BMC Cancer, 2016, 16(224): 1-8. |
| [17] |
GARCIA-FIGUEIRAS R, BALEATO-GONZALEZ S, PADHANI A R, et al. How clinical imaging can assess cancer biology[J]. Insights Imaging, 2019, 10(1): 28. DOI:10.1186/s13244-019-0703-0 |
| [18] |
GAO Y, SONG P, LI H, et al. Elevated serum CEA levels are associated with the explosive progression of lung adenocarcinoma harboring EGFR mutations[J]. BMC Cancer, 2017, 17(1): 484. DOI:10.1186/s12885-017-3474-3 |
| [19] |
KATO T, YOSHIOKA H, OKAMOTO I, et al. Afatinib versus cisplatin plus pemetrexed in Japanese patients with advanced non-small cell lung cancer harboring activating EGFR mutations: subgroup analysis of LUXLung 3[J]. Cancer Sci, 2015, 106(9): 1202-1211. DOI:10.1111/cas.12723 |
| [20] |
RIZZO S, RAIMONDI S, DE JONG EEC, et al. Genomics of non-small cell lung cancer(NSCLC): association between CT-based imaging features and EGFR and K-RAS mutations in 122 patients-an external validation[J]. Eur J Radiol, 2019, 110: 148-155. DOI:10.1016/j.ejrad.2018.11.032 |
| [21] |
RIZZO S, PETRELLA F, BUSCARINO V, et al. CT radiogenomic characterization of EGFR, K-RAS, and ALK mutations in non-small cell lung cancer[J]. Eur Radiol, 2016, 26(1): 32-42. DOI:10.1007/s00330-015-3814-0 |
| [22] |
肖磊, 顾潜彪, 张堃, 等. CT影像组学标签预测肺腺癌表皮生长因子受体基因敏感突变[J]. 中国介入影像与治疗学, 2019, 16(4): 220-224. |
| [23] |
LIU Y, KIM J, BALAGURUNATHAN Y, et al. Radiomic features are associated with EGFR mutation status in lung adenocarcinomas[J]. Clin Lung Cancer, 2016, 17(5): 441-448. |
| [24] |
LU L, SUN S H, YANG H, et al. Radiomics prediction of EGFR status in lung cancer-our experience in using multiple feature extractors and the cancer imaging archive data[J]. Tomography, 2020, 6(2): 223-230. |
| [25] |
TU W, SUN G, FAN L, et al. Radiomics signature: a potential and incremental predictor for EGFR mutation status in NSCLC patients, comparison with CT morphology[J]. Lung Cancer, 2019, 132(5): 28-35. |
| [26] |
LIU G, XU Z, GE Y, et al. 3D radiomics predicts EGFR mutation, exon-19 deletion and exon-21 L858R mutation in lung adenocarcinoma[J]. Transl Lung Cancer Res, 2020, 9(4): 1212-1224. |
| [27] |
LI S, DING C, ZHANG H, et al. Radiomics for the prediction of EGFR mutation subtypes in non-small cell lung cancer[J]. Med Phys, 2019, 46(10): 4545-4552. |
 2023, Vol. 21
2023, Vol. 21


