| 肺癌影像的人工智能研究 |
2. 浙江中医药大学附属第一医院,浙江 杭州 310006
肺癌是最常见的恶性肿瘤之一,据美国癌症学会2020年1月公布的数据,肺癌发病率和死亡率均居恶性肿瘤首位[1],早期诊断与治疗可有效提高患者生存率[2]。CT是肺癌筛查和临床诊断最常用的检查方法,在肺癌早期检出和随访治疗中有非常重要的作用。随着低剂量CT肺筛查技术日益普及,检出的肺结节越来越多[3],放射科医师面临工作量增加、人力补充不足、诊断要求提高等多方面挑战。近年来,随着计算机技术的发展与进步,人工智能技术引起了广泛关注。运用人工智能技术帮助放射科医师解决上述挑战已成为趋势。本文就人工智能技术简介和肺癌影像的人工智能研究应用等综述如下。
1 人工智能技术简介人工智能亦称机器智能,指由人类制造的机器所表现出来的智能,通常指通过普通计算机程序手段实现的人类智能技术[4],以人工神经网络、支持向量机及卷积神经网络等为代表。人工智能是当前兴起的革命性新技术,结合医学影像,通过大样本病例的回顾性机器学习训练,从而达到诊断和预测疾病的目的[5]。人工智能在临床实践领域是对各种医学图像的诊断和预测分析,包括视网膜和皮肤病变的照片、病理显微图像和放射图像等[6-9],可提高诊断速度、准确率和报告质量等方面的影像医学综合效能[9]。人工智能包括机器学习,深度学习作为机器学习中的先进技术,是当前肺癌影像研究最常用的方法(图 1)。深度学习由多个工作层组成,如视觉皮层,工作流程从初始层到最高层[10]。而人工智能运用的深度学习技术可使模型性能不断提升,以更好匹配实际需要,目前在研究中得到了广泛应用。
 |
| 图 1 人工智能与机器学习、深度学习之间的范畴关系简图 |
影像组学主要通过高通量提取医学图像的特征,采用经过处理的特征建立模型来诊断和预测临床疾病[11-12],其流程主要包括图像获取、病灶勾画、特征提取、特征选择及机器建模(图 2)。深度学习模型是一个不断学习提高的人工智能方法,与之相比,影像组学模型一经建立,参数不会自动修改,不具有自主学习能力。因此,影像组学不是单纯的人工智能方法,而是综合运用人工智能的部分技术与统计学分析,其对研究对象的样本量要求不如机器学习和深度学习高。从近期文献[13-16]报道看,其在影像医学与核医学中的研究应用较广泛。
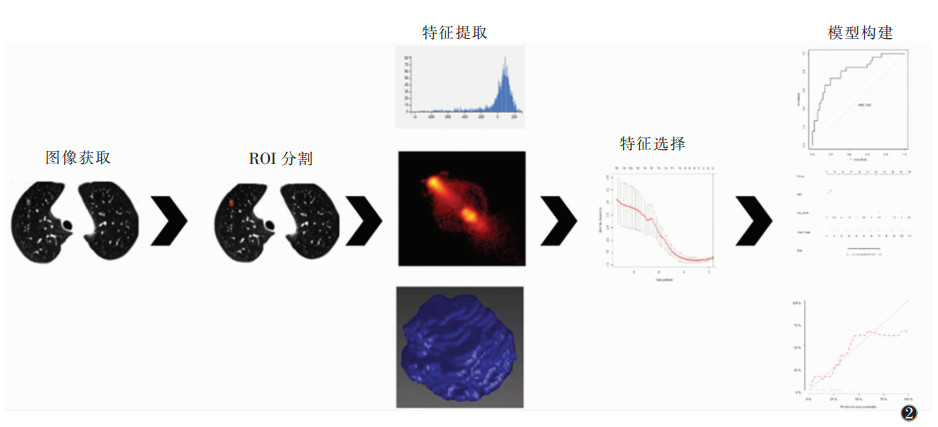 |
| 图 2 影像组学工作流程 |
2 肺癌影像的人工智能研究应用
肺癌因其高发病率和死亡率一直是基础和临床研究的热点。近年来,人工智能技术发展迅速,其对肺癌研究的内容十分广泛,包括病因分析、各种诊断(如活检优选、组织病理学、基因分类等)、预后评估及治疗方案优选等[17-32],主要集中在肺部病灶的检测(图 3)、诊断和预测,相关文献报道见表 1。
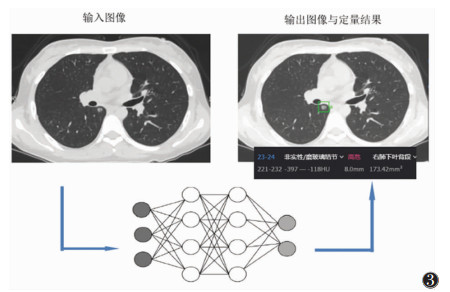 |
| 图 3 人工智能肺结节检测工作流程 |
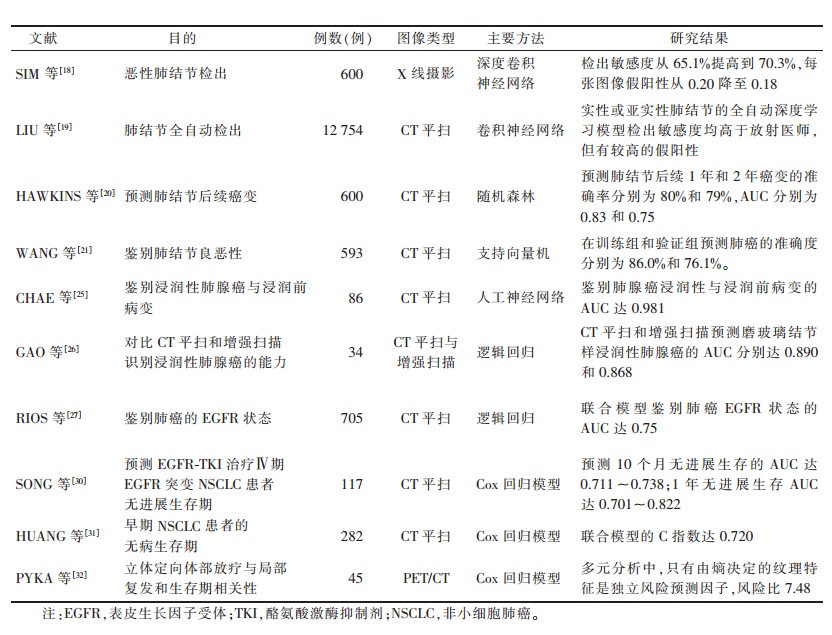
| 表 1 肺癌影像人工智能研究报告摘录 |
 |
2.1 检测
运用人工智能技术可提高肺癌的检测效率,加强肺结节患者的管理[17-18]。SIM等[18]的多中心研究表明,结合深度卷积神经网络,放射科医师对胸部X线图像的阅片敏感度从65.1%提高至70.3%,每张图像假阳性从0.20降至0.18。LIU等[19]的研究采用卷积神经网络建立深度学习模型,对12 754例的CT扫描图像行全自动检测,不论是不同大小及部位的实性结节,还是亚实性结节,全自动检测模型的检出敏感度均高于2位放射科医师,模型总阳性率为70.1%,高于2位放射科医师(39.4%和56.6%),但模型存在较高的假阳性(48.4%)。HAWKINS等[20]的研究探讨基线低剂量CT图像的影像组学分析检出和预测肺结节后续癌变的能力,研究利用美国国家肺癌筛查试验的数据检出肺结节,并通过随机森林分类器获取23个稳定的特征,建立的预测1年和2年癌变的肺结节模型,准确率分别为80%和79%(AUC分别为0.83和0.75)。
2.2 诊断传统影像学特征在鉴别肺癌的病理类型和基因突变方面虽有进展,但仍存在较大挑战[21-22]。而借助影像组学和深度学习等人工智能方法,可提取肉眼无法辨识的图像信息特征,在鉴别肺癌病理类型和基因突变方面具有较好的表现[23-24]。
WANG等[21]回顾性分析了影像组学技术鉴别肺结节良恶性的预测能力,该研究在国际开放性数据库———肺部影像数据库联盟与图像数据资源计划中获取593例肺部肿瘤患者的CT图像,定量分析提取150个影像组学特征,经过数据处理并用支持向量机建模,最后得出实验组和验证组敏感度、特异度、准确率分别为82.5%、89.5%、86.0%和74.6%、78.9%、76.1%。CHAE等[25]利用影像组学特征回顾性分析58例浸润性肺腺癌(含微浸润性腺癌7例)和28例浸润前病变(不典型腺瘤样增生4例、原位腺癌24例),通过平均CT值、CT值标准差、质量、峰度和熵值建立的人工神经网络模型,鉴别浸润性与浸润前病变的AUC达0.981。GAO等[26]的研究显示,CT平扫薄层图像的影像组学纹理特征识别表现为磨玻璃结节样浸润性肺腺癌的AUC为0.890,与增强扫描(AUC为0.868)均有较高的价值,且两者差异无统计学意义(P=0.1 897)。ROIS等[27]利用影像组学技术鉴别生长因子受体(epidermal growth factor receptor,EGFR)阳性和阴性的病例,将影像组学特征(AUC为0.69)与EGFR相关的临床特征(AUC为0.70)结合后,联合模型AUC达0.75,且利用影像组学技术可鉴别EGFR阳性和KRAS阳性的肿瘤患者(AUC为0.80)。
2.3 预测人工智能技术不仅在肺癌的检出和诊断中有较多的研究和应用,在预测肺癌患者预后生存期、放疗及免疫治疗反应等方面也有较大进展[28-29]。SONG等[30]回顾性分析非小细胞肺癌(non small cell lung cancer,NSCLC)Ⅳ期患者EGFR突变的肺癌CT影像组学特征(1 032个),并建模预测该类患者接受EGFR-TKI治疗的无进展生存期,结果表明,与临床病理特征模型相比,联合影像组学的综合模型可显著提高临床收益(P<0.000 1)。
HUANG等[31]运用影像组学特征回顾性分析282例早期(ⅠA~ⅡB)NSCLC患者的无病生存期(disease free survival,DFS),揭示影像组学特征与DFS显著相关,是独立于临床病理风险因素的重要预测指标,而列线图预测DFS的C指数为0.72(95%CI 0.71~ 0.73),高于临床病理风险因素C指数0.69(95%CI 0.68~0.70)(P<0.000 1)。PYKA等[32]回顾性分析45例NSCLC患者立体定向放疗资料,从中提取PET/CT图像数据,研究立体定向放疗与局部复发及生存期的相关性,结果显示熵值和相关性在NSCLC患者T1期(肿瘤直径≤3 cm)和T2期(肿瘤直径>3 cm)两亚组中均与肿瘤局部复发有关系,AUC分别为0.801和0.776,且在多元回归分析中,熵值被认为是疾病相关存活率的独立预测因素(HR=7.48,P=0.016)。
3 肺癌影像人工智能研究的局限及挑战肺癌影像人工智能研究虽取得了一定成果,但其仍处在发展的初级阶段。因此,需认识其研究应用的局限性和面临的挑战:①样本量问题[33-35],大部分研究基于单中心且例数较少,可能会导致研究结果偏倚。病例相对缺乏也导致大部分研究采用影像组学方法,而无法采用更智能化的深度学习或机器学习方法。因此,有必要建立多中心大样本的数据库,在此基础上进行相关人工智能的肺癌研究,可获得更客观、科学的研究结果。②勾画或标记问题[36],由于技术原因,很多情况下肺癌病灶无法实现自动勾画或标记,而人工手动勾画、半自动勾画或标记会导致可重复性问题。由于目前尚无大样本支持深度学习训练自动勾画或标记模型,自动勾画或标记软件尚未得到广泛应用。随着计算机技术的发展及多中心大样本数据库的建成,自动勾画或标记技术将得到广泛验证。③模型的标准化与规范化问题,因处于发展初级阶段,各种模型、软件各具特色和专用性,缺乏标准,较难规范、统一,这将导致研究结果的可重复性和可比性问题。因此,需相关研究团队尽早合作建立起统一标准的特征计算技术和方法,有利于影像组学特征的研究和深度学习、机器学习等的高质量模型构建,以便进一步实现目标疾病的高效、精准诊断和预测。
4 小结目前常用的肺癌影像人工智能研究方法是在机器学习技术上发展起来的深度学习,以及与之相关的影像组学方法。人工智能在肿瘤检出、高效诊断和疗效预测等方面均有较高价值,但其尚处于发展初期,存在样本量不大、病灶勾画与标记以手动或半自动为主,以及特征或模型计算方法缺乏标准和规范等局限,在肺癌影像中的研究有待进一步深入。
| [1] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer Statistics, 2020[J]. CA Cancer J Clin, 2020, 70(1): 7-30. DOI:10.3322/caac.21590 |
| [2] |
KOVALCHIK S A, TAMMEMMAGI M, BERG C D, et al. Targeting of low-dose CT screening according to the risk of lung-cancer death[J]. N Engl J Med, 2013, 369(3): 245-254. DOI:10.1056/NEJMoa1301851 |
| [3] |
KAKINUMA R, OHMATSU H, KANWKO M, et al. Progression of focal pure ground-glass opacity detected by low-dose helical computed tomography screening for lung cancer[J]. J Comput Assist Tomogr, 2004, 28(1): 17-23. DOI:10.1097/00004728-200401000-00003 |
| [4] |
KAHN C E. From images to actions:opportunities for artificial intelligence in radiology[J]. Radiology, 2017, 285(3): 719-720. |
| [5] |
PARK S H, HAN K. Methodologic guide for evaluating clinical performance and effect of artificial intelligence technology for medical diagnosis and prediction[J]. Radiology, 2018, 286(3): 800-809. DOI:10.1148/radiol.2017171920 |
| [6] |
GULSHAN V, PENG L, CORAM M, et al. Development and validation of a deep learning algorithm for detection of diabetic retinop athy in retinal fundus photographs[J]. JAMA, 2016, 316(22): 2402-2410. DOI:10.1001/jama.2016.17216 |
| [7] |
ESTEVA A, KUPREL B, NOVOA R A, et al. Dermatologist-level classification of skin cancer with deep neural networks[J]. Nature, 2017, 542(7639): 115-118. DOI:10.1038/nature21056 |
| [8] |
EHTESHAMI B, VETA M, JOHANNES D P, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer[J]. JAMA, 2017, 318(22): 2199-2210. DOI:10.1001/jama.2017.14585 |
| [9] |
RECHT M P, DEWEY M, DREYER K, et al. Integrating artificial intelligence into the clinical practice of radiology: challenges and recommen dations[J/OL]. Eur Radiol, 2020.[2020-02-17].https: //www.ncbi.nlm.nih.gov/pubmed/?term=Integrating+artificial+intellig ence+into+the+clinical+practice+of+radiology%3A+challenges+and +recommendations .DOI: 10.1007/s00330-020-06672-5.
|
| [10] |
CHEN J H, ASCH S M. Machine learning and prediction in medicine-beyond the peak of inflated expectations[J]. N Engl J Med, 2017, 376(26): 2507-2509. DOI:10.1056/NEJMp1702071 |
| [11] |
GILLIES R J, KINAHAN P E, HRICAK H. Radiomics:images are more than pictures, they are data[J]. Radiology, 2016, 278(2): 563-577. |
| [12] |
WILSON R, DEVARA J A. Radiomics of pulmonary nodules and lung cancer[J]. Transl Lung Cancer Res, 2017, 6(1): 86-91. |
| [13] |
THAWANI R, MCLANE M, BEIG N, et al. Radiomics and radiogenomics in lung cancer :a review for the clinician[J]. Lung Cancer, 2018, 115: 34-41. DOI:10.1016/j.lungcan.2017.10.015 |
| [14] |
LAMBIN P, LEI T H, DEIST T M, et al. Radiomics:the bridge between medical imaging and personalized medicine[J]. Nat Rev Clin Oncol, 2017, 14(12): 749-762. DOI:10.1038/nrclinonc.2017.141 |
| [15] |
吉蘅山, 张龙江, 朱虹. 肺癌PET/CT影像组学研究进展[J]. 中华放射学杂志, 2019, 53(2): 154-157. DOI:10.3760/cma.j.issn.1005-1201.2019.02.017 |
| [16] |
WANG B, TANG Y, CHEN Y, et al. Joint use of the radiomics method and frozen sections should be considered in the prediction of the final classification of peripheral lung adenocarcinoma manifesting as ground-glass nodules[J]. Lung Cancer, 2020, 139: 103-110. DOI:10.1016/j.lungcan.2019.10.031 |
| [17] |
HASSANI C, VARGHESE B A, NIEVA J, et al. Radiomics in pulmonary lesion imaging[J]. AJR Am J Roentgenol, 2019, 212(3): 497-504. DOI:10.2214/AJR.18.20623 |
| [18] |
SIM Y, CHUNG M J, KOTTER E, et al. Deep convolutional neural network-based software improves radiologist detection of malignant lung nodules on chest radiographs[J]. Radiology, 2020, 294: 199-209. DOI:10.1148/radiol.2019182465 |
| [19] |
LIU K, LI Q, MA J, et al. Evaluating a fully automated pulmonary nodule detection approach and its impact on radiologist performance[J]. Radiology:Artificial Intelligence, 2019, 1(3): e180084. DOI:10.1148/ryai.2019180084 |
| [20] |
HAWKINS S, WANG H, LIU Y, et al. Predicting malignant nodules from screening CTs[J]. J Thorac Oncol, 2016, 11(12): 2120-2128. DOI:10.1016/j.jtho.2016.07.002 |
| [21] |
WANG J, LIU X, DONG D, et al. Prediction of malignant and benign of lung tumor using a quantitative radiomic method[J]. Conf Proc IEEE Eng Med Biol Soc, 2016, 2016: 1272-1275. |
| [22] |
IKEDA K, AWAI K, MORI T, et al. Differential diagnosis of ground-glass opacity nodules :CT number analysis by threedimensional computerized quantification[J]. Chest, 2007, 132(3): 984-990. DOI:10.1378/chest.07-0793 |
| [23] |
COUDRAY N, OCAMPO P S, SAKELLAROPOULOS T, et al. Classification and mutation prediction from non-small cell lung cancer histopathology images using deep learning[J]. Nat Med, 2018, 24(10): 1559-1567. DOI:10.1038/s41591-018-0177-5 |
| [24] |
宋兰, 朱振宸, 姜蕾, 等. CT影像组学在预测肺腺癌ALK融合基因表达中的价值初探[J]. 中华放射学杂志, 2019, 53(11): 963-967. |
| [25] |
CHAE H D, PARK C M, PARK S J, et al. Computerized texture analysis of persistent part-solid ground-glass nodules:differentiation of preinvasive lesions from invasive pulmonary adenocarcinomas[J]. Radiology, 2014, 273(1): 285-293. DOI:10.1148/radiol.14132187 |
| [26] |
GAO C, XIANG P, YE J, et al. Can texture features improve the differentiation of infiltrative lung adenocarcinoma appearing as ground glass nodules in contrast-enhanced CT?[J]. Eur J Radiol, 2019, 117: 126-131. DOI:10.1016/j.ejrad.2019.06.010 |
| [27] |
RIOS V E, PARMAR C, LIU Y, et al. Somatic mutations drive distinct imaging phenotypes in lung cancer[J]. Cancer Res, 2017, 77(14): 3922-3930. DOI:10.1158/0008-5472.CAN-17-0122 |
| [28] |
何兰, 黄燕琪, 马泽兰, 等. CT影像组学在非小细胞肺癌临床分期中的价值[J]. 中华放射学杂志, 2017, 51(12): 906-911. DOI:10.3760/cma.j.issn.1005-1201.2017.12.004 |
| [29] |
BANNA G L, OLIVIER T, RUNDO F, et al. The promise of digital biopsy for the prediction of tumor molecular features and clinical outcomes associated with immunotherapy[J]. Front Med, 2019, 6: 172. DOI:10.3389/fmed.2019.00172 |
| [30] |
SONG J, SHI J, DONG D, et al. A new approach to predict progression-free survival in stage Ⅳ EGFR-mutant NSCLC patients with EGFR-TKI therapy[J]. Clin Cancer Res, 2018, 24(15): 3583-3592. DOI:10.1158/1078-0432.CCR-17-2507 |
| [31] |
HUANG Y, LIU Z, HE L, et al. Radiomics signature:a potential biomarker for the prediction of disease-free survival in early-stage (Ⅰ or Ⅱ) non-small cell lung cancer[J]. Radiology, 2016, 281(3): 947-957. DOI:10.1148/radiol.2016152234 |
| [32] |
PYKA T, BUNDSCHUH R A, ANDRATSCHKE N, et al. Textural features in pre-treatment[F18 ]-FDG-PET/CT are correlated with risk of local recurrence and disease-specific survival in early stage NSCLC patients receiving primary stereotactic radiation therapy[J]. Radiat Oncol, 2015, 10: 100. DOI:10.1186/s13014-015-0407-7 |
| [33] |
TAM V H, KABBARA S, YEH R F, et al. Impact of sample size on the performance of multiple-model pharmacokinetic simulations[J]. Antimicrob Agents Chemother, 2006, 50(11): 3950-3952. DOI:10.1128/AAC.00337-06 |
| [34] |
ZHANG Y, OIKONOMOU A, WONG A, et al. Radiomics-based prognosis analysis for non-small cell lung cancer[J]. Sci Rep, 2017, 7: 46349. DOI:10.1038/srep46349 |
| [35] |
FIGUEROA R L, ZENG T Q, KANDULA S, et al. Predicting sample size required for classification performance[J]. BMC Med Inform Decis Mak, 2012, 12: 8. DOI:10.1186/1472-6947-12-8 |
| [36] |
KUMAR V, GU Y, BASU S, et al. Radiomics:the process and the challenges[J]. Magn Reson Imaging, 2012, 30(9): 1234-1248. DOI:10.1016/j.mri.2012.06.010 |
 2020, Vol. 18
2020, Vol. 18


