2. 首都师范大学生命科学学院, 北京 100048;
3. 中国科学院水生生物研究所水生生物多样性与保护重点实验室, 湖北 武汉 430072;
4. 西安交通大学生命科学与技术学院, 陕西 西安 710049
在超过10亿年漫长的动物演化中,处于真核生物起源阶段的原生动物历经一次次分化和演化,形成了多样性极高的一类极具代表性的单细胞真核生物。其中的纤毛虫类群,在生殖方式(独特的接合生殖)、双态核型(分司不同功能的大、小核)、极端多样化的细胞发生模式等方面都代表了真核生物细胞分化与进化的最高阶段,已成为细胞学、生理学、遗传学等多个领域研究的模型生物。因此,围绕纤毛虫基础生物学的研究,已成为当今国际原生动物学领域中最活跃的一个分支。纤毛虫作为水体微食物网的核心组分,在物质循环、能量流动和水环境健康维护中发挥着重要作用[1-5]。此外,很多兼性和专性寄生、栖生类群又常因构成养殖动物的病害等成为产业的防范对象[6]。因此,以纤毛虫为对象的研究一直是国际上基础生物学和相关应用学科所关注的重要热点之一。
然而,由于诸多原因,在中国以海洋纤毛虫为核心的基础生物学研究长期以来十分欠缺,特别是有关物种-基因水平的多样性等基本资料构建、与生态学、环境生物学、病害学、围绕模式种所开展的适应与进化、共生机制形成、与环境耦合的群落结构与动态变化等研究,普遍处于程度不同的缺位状态。而基于分子技术等现代手段的纤毛虫学研究起于20世纪后叶,先天不足加上缺乏系统的投入和有传承的推进,使得在我国以纤毛虫学为核心的现代原生动物学研究与国际水平曾长期处于距离越拉越大的趋势。
这一现象直到21世纪初才有了根本改观:借助于国家自然科学基金、多项重大基金以及国际合作项目的连续支持,在以作者所在团队为核心的研究队伍以及合作者们的不懈努力下,中国的纤毛虫学研究(特别在海洋领域)在多个分支中实现了从追赶、并跑、再到领跑的变化。在这个研究探索、队伍建设和发展过程中,以OUC(中国海洋大学)为符号的原生动物学研究室已发展成为国际上本领域内为数不多的旗舰级研究中心之一,形成了我们的突出特色和集团优势,并在多样性、细胞学等传统强项健康发展的同时,以纤毛虫为模式的表观遗传学、组学、纤毛虫大小核间的基因进化等领域也逐渐形成了我们的新拓展和新增长点,为未来纤毛虫基础生物学领域的研究奠定了重要基础、开启了新篇章。
1 纤毛虫的多样性传统的纤毛虫物种多样性研究主要限于欧洲与北美地区,沿海则包括大西洋两岸、地中海及太平洋的东岸。而在整个西太平洋和东亚地区,有关纤毛虫的系统、专业级的研究普遍空缺或粗浅,大多数类群均缺乏了解、不明或不详,因此,基本的家底信息长期不明[1]。
在过去的30年中,作者团队及合作者先后开展了下列多样性工作:(1)围绕我国的黄渤海和南海近岸水域以及以红树林为核心的湿地纤毛虫,完成了对其区系、物种多样性的系统、全面的调查研究,形成了中国首份海洋纤毛虫原生动物的专业级家底信息(见图 1)[1, 6-13];(2)作为基因多样性研究的重要内容,我们构建了全球最大的海洋纤毛虫的遗传资料(包括1 700余种-种群的DNA)信息库,同步完成了对相关种类18S rRNA等基因的测序,进而构成了GenBank中有关资料的重要组成(见表 1);(3)完成了对海水养殖水体中以危害性纤毛虫(及鱼类寄生粘孢子虫)为主的病原原生动物分类、病原鉴定、检索文献等资料性工作[14];(4)系统开展了有关胶州湾纤毛虫群落结构与变化、与环境耦合的生态响应及生物环境监测技术的探索[15]。
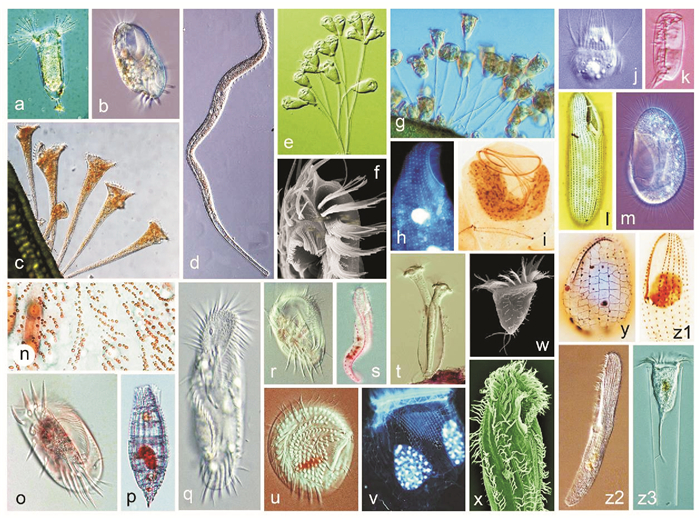
|
(a粗壮壳吸管虫,自陈相瑞等[7];b寡毛双眉虫,自宋微波等[1];c喇叭虫(未发表);d旋口虫(未发表);e, i聚缩虫(未发表); f卡尔半列虫,自Luo et al.[10];g钟虫(未发表);h海喙虫(未发表);j中缢虫(未发表);k瘦小偏体虫,自宋微波等[14];l中华嗜污虫,自Pan et al.[11];m帆口虫(未发表);n拟黄色黄色新尾柱虫,自Zhang et al.[13];o扇形游仆虫(未发表);p纺锤披巾虫,自Chen et al.[8];q缩颈半腹柱虫(未发表);r,y游仆虫(未发表);s美丽异列虫(未发表);t杯状靴纤虫(未发表);u乳突盘毛虫,自Fan et al.[9];v拟铃虫(未发表);w夏威夷拟稀毛虫,自Luo et al.[10];x多变拟双列虫(未发表);y游仆虫(未发表);z1伪康纤虫(未发表);z2清亮阿内格虫,自Yan et al.[12];z3真铃虫(未发表)。a Acineta foetida, from Chen et al.[7]; b Diophrys oligothrix, from Song et al.[1]; c Stentor sp. (original); d Spirostomum sp. (original); e, i Zoothamnium sp. (original); f Hemiholosticha kahli, from Luo et al.[10]; g Vorticella sp. (original); h Remanella sp. (original); j Mesodinium sp. (original); k Dysteria pusilla, from Song et al.[14]; l Philaster sinensis, from Pan et al.[11]; m Pleuronema sp. (original); n Neourostylopsis flava paraflava, from Zhang et al.[13]; o Euplotes vannus (original); p Tiarina fusa, from Chen et al.[8]; q Hemigastrostyla enigmatica (original); r, y Euplotes sp. (original); s Anteholosticha pulchra (original); t Cothurnia cyathiforme (original); u Discotricha papillifera, from Fan et al.[9]; v Tintinnopsis sp. (original); w Psilotrichides hawaiiensis, from Luo et al.[10]; x Paraistichella variabilis (original); z1 Pseudocohnilembus sp. (original); z2 Anigsteinia clarissima, from Yan et al.[12]; z3 Eutintinnus sp. (original). ) 图 1 纤毛虫原生动物的形态学与物种多样性 Fig. 1 Species diversity and morphology of ciliated protozoans |
|
|
表 1 作者团队所完成的主要纤毛虫类群DNA库、标记基因及组学数据库情况 Table 1 DNA bank, genome/transcriptome data, and contribution to the GenBank rRNA gene database by authors' group |
这些工作的核心成果包括全面开展了中国温带、热带海区资料高度缺乏的40余目级类群自由生纤毛虫之形态分类学研究,首次给出了包括纤毛图式等现代物种鉴定的专业级资料,完成了对国际间长期不详、鉴定混乱的大量“半知种类”的重描述、新定义及新模建立、异名清理等大量的各类历史遗留问题的修订。成果还包括研究建立了新目、新科、新属、新种等大量新阶元,填补了国际间包括温带、热带沿岸富营养区、离岸水体、热带红树林、河口低盐区、砂隙潮间带等生境中纤毛虫区系研究的空白[16-29]。
这是国际范围内迄今原生动物多样性领域中堪称浩大的一项工程:作者所在团队连续投入了30年、全视野地覆盖了温带-热带沿海海洋纤毛虫各类群,描绘了多种生境中包括物种、基因、生态系多样性的完整版图,为研究相关类群的起源演化、近岸水体生态学、微型生物的协同进化与适应、大尺度下的水环境变化与原生动物生物地理格局等建立了重要档案资料和理论依据,也为生物资源及环境评估与保护、为水产养殖中的病害防治等应用领域提供了一份重要的手册性资料。
我们在此领域的近期新目标将重点关注迄今尚缺乏研究、兼具“水”“陆”特征和微气象条件的淡水湿地生态系统。即围绕湿地生态系中纤毛虫的多样性、群落结构与时空分布格局等,聚焦该生境中纤毛虫的物种-基因-生境多样性、生态系资料的建立、遗传信息库构建以及包括细胞学、基因进化等在内的基础生物学探讨。这项工作的目标和意义在于将完成涉及环境健康维持、资源开发与利用、特别是大时空尺度上湿地环境变迁的跟踪以及历史档案资料的构建,这些要素均为国内外急需面对的科学问题。预期成果将建立和呈现中国淡水湿地生态系统内纤毛虫多样性与生态学等家底信息的全面、专业级资讯,填补国内外空白,体现科学研究服务于国家需要这一重要诉求,并有望在新物种发现、纤毛虫遗传信息库的完善、分子生态学探索、生态学时空信息的构建等领域形成一批创新成果,巩固和扩大我们的学科优势及在领域内的旗舰地位。
2 纤毛虫细胞发生模式纤毛虫的重要特征之一即为体表的纤毛器发生了结构和功能上的高度分化和特化。在细胞的分裂过程中通过不同的模式和过程经由原基而逐步发育为新生细胞的纤毛器,该过程被称为“细胞发生”。这一过程本质上反映了细胞结构的分化与去分化,并由此构成了纤毛虫学中有关“细胞分化”这一重要的科学问题。
鉴于这一背景,纤毛虫的细胞发生学研究长期以来是纤毛虫学和细胞生物学研究的重要分支。该领域研究的主要意义在于:为探讨原生动物这一多源发生体系的系统关系与演化提供理论支持;为亲缘种、隐存种的廓清提供发生学的鉴别特征;为在分子水平上揭示基因调控及相关的机制研究提供背景信息。
近20年来,作者团队以高度特化的腹毛类纤毛虫为模式,所完成的个体发生学研究覆盖了目前所知的几乎所有发生类型,涉及隶属37科、88属的150余种的细胞分化模式和过程(见图 2)。这些工作详细跟踪报道了无性繁殖行为中的细胞器、皮膜结构及核器的构建过程,刻画了不同类群的皮层/胞器起源-分化-命运、重要结构的近祖-衍化分析[30]。
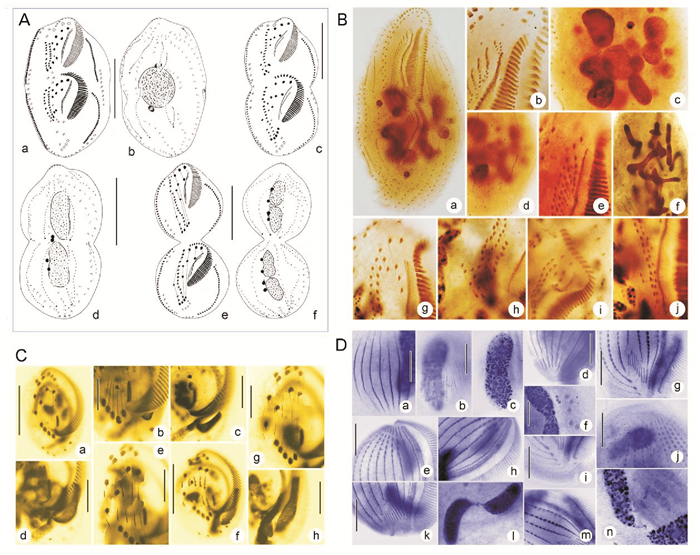
|
(A自Luo et al.[33];B自Zhang et al.[13];C自Jiang et al.[31];D自Shao et al.[35]。A from Luo et al.[33]; B from Zhang et al.[13]; C from Jiang et al.[31]; D from Shao et al.[35]. ) 图 2 纤毛虫原生动物的细胞发生模式与结构分化 Fig. 2 Patterns of morphogenesis in ciliated protozoans |
代表性工作包括:(1)对信息残缺类群完成了发生模式的修正和补足,进而完善了对游仆虫属内发生模式的刻画;(2)完整描述了具大-小口型多态性的瞬膜虫的生活史;(3)构建了散毛目内背面纤毛器结构的第6种分型:半腹柱虫型;(4)阐释了原本系统位置相距甚远的伪瘦尾虫属和圆纤虫属同源,二者位于进化末端且发生背面结构的二次退化;(5)对比尾列虫属与瘦尾虫属发生模式的高相似度,界定其均为尾柱目和高等类群散毛目的居间类群,即尾柱目向散毛目进化的过渡态及关键节点;(6)揭示了伪小双虫属属内发生的高度不保守性;(7)通过比对伪角毛虫属、趋角虫属、偏角毛虫属和类角毛虫属的多元分析,为揭示伪角毛虫科的系统演化提供了重要依据[30-36]。
上述成果揭示和描述了包括原腹毛类祖先型“原基滞后分化”、瘦尾虫“次级背触毛原基片段化”、独角虫背触毛独特的“两组发生式”、针毛虫缘棘毛的“尾棘毛发育模式”和类角毛虫波动膜原基“多次片段化”等一系列发生学新现象[30]。
我们同时利用细胞发生学信息解答了若干长期存疑的系统进化问题,包括证明了凯毛虫代表了广义腹毛纲中的1个原始、低等的类群,为确定3个纲-亚纲级类群间真实的演化关系提供了重要的依据,同时综合发生学信息建立了1新亚纲-原腹毛亚纲,解答了由上世纪后半叶以来长期存疑的凯毛虫科系统定位及其与腹毛类和异毛类的系统关系等问题。其次,明确了盘头类作为一个腹毛类祖先型,代表了介于游仆亚纲和腹毛亚纲之间的居间类群,证明其与腹毛亚纲具有更为接近的亲缘关系并应为腹毛亚纲中独立的侧支,并且建立了1个目级新阶元-盘头目,该成果揭示了近40年来悬疑的盘头类的系统定位及其与腹毛亚纲和游仆亚纲的系统关系等问题[30-36]。
3 纤毛虫标记基因测序和分子系统演化由于纤毛虫具有极高的物种多样性、复杂的生活史和十分久远的进化史,许多“相近阶元”的分类学特征或许经历了趋同、趋异或起源不详的演化过程。因此,建立在形态学基础上的类群间的系统演化关系在诸多类群中均存在着悬疑、不明和争议,特别是在一些演化邻接阶元信息不明的类群。近年来,这些悬疑和争议随着以标记基因序列分析为代表的分子系统学技术的引入而不断得到澄清或形成新的观点。
但一个突出的问题构成了新的瓶颈:在国际GenBank信息库中,太多的纤毛虫类群因缺乏标记基因信息而导致系统发育树构建中形成新的盲区。此外,信息库中许多缺乏分类学依据的序列(错误鉴定的素材)为系统发育研究引入了新干扰和新混乱。
在此背景下,我们在开展上述纤毛虫多样性研究的过程中,完成了涵盖了纤毛虫常见类群的1 400余条核糖体标记基因的测序工作,累计向GenBank数据库贡献近20%纤毛虫的标记基因序列(见表 1)。迄今为止,基于标记基因信息对门下13个纲55个目级阶元的系统进化关系进行了深层次的梳理和探讨,揭示了大量类群的系统进化地位(见图 3)[37-52]。代表性成果包括:(1)建立了两新纲(Mesodiniea,Protocruziea)[36-37]、一新亚纲(Protohypotrichia)、两新目(Lynnellida,Discotrichida)[45-47]以及一新亚目(Pseudoamphisiellina)[48];(2)系统阐述了五个亚纲级类群(腹毛类、游仆类、寡毛类、缘毛类、盾纤类)内部的系统关系:明确了游仆亚纲及其辖属的2目7科均为单源发生系,证实了盘头目与游仆目具有更密切的系统关系;新建立的Lynnella属代表了一个介于寡毛亚纲和环毛亚纲之间的一个目级新阶元[44];(3)同步探讨了有关物种选择、建树方法、环境未知序列和二级结构的使用、旁系同源基因、数据缺失和异质性等一系列因素对系统发育关系的影响[49-52]。
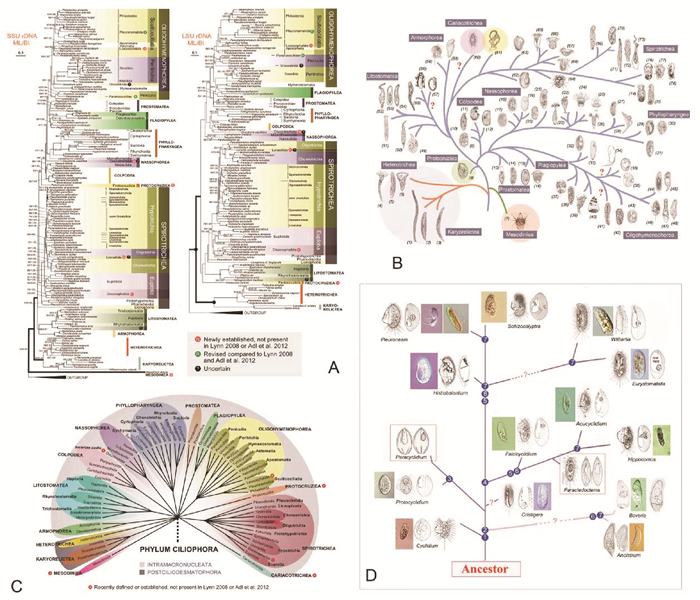
|
(A~C自Gao et al.[44];D自Gao et al.[37]。A~C from Gao et al.[44]; D from Gao et al.[37]. ) 图 3 纤毛虫原生动物的分子系统发育 Fig. 3 Molecular phylogeny of ciliated protozoans |
作为一份新成果,我们结合单细胞组学测序技术,构建了一种高效自动化序列分析方法(Guided Phylogenomic Search in trees, GPSit),其具有一站式、高度流程化的特点并集成了全部的必需和可选操作,大大减少人工操作时间。该方法支持同时使用超级数据集与超级树方法进行系统发育基因组学分析,能够很好地重构深层系统发育关系并揭示浅层系统发育关系中的相关类群的系统进化地位。该方法很大程度上纾解了(不可培养类群)基因组数据难以获取的困难[53]。
迄今为止的工作显示,单基因和少数基因为基础的纤毛虫分子系统学研究依然不能回答一些包括隐存种厘定、关系混乱的形态属的界定、形态特征-细胞发生学信息与分子信息相悖等问题。这些工作需要在系统基因组学(Phylogenomics)研究的水平上去探讨和寻求解答。因此,作为新的关注点之一,我们将高通量测序技术、DNA信息库利用与组学资讯开发整合成新的工作平台,在组学和大数据背景下去解析包括物种鉴定、适应与进化相关的系统学科学问题。作者预期,新设想的实施将引领和推动纤毛虫分子系统发育学的发展并将构成系统学领域新的研究热点。
4 纤毛虫基因条形码技术探索作为近几十年来新形成的一项革命性成果,基因条码技术成为衔接(以形态学为标志的)传统分类学、进化生物学、生态学、遗传学及系统分类学等学科间交叉研究的桥梁。其衍生出的相关分析方法对阐释涉及传统分类学、系统发生学、进化生态学、生物地理学、生物信息学等相关学科的一系列重要科学问题均产生了积极的促进作用[54-55]。
在信息化时代,通用性、客观性和可操作性等生物多样性工作的基本需求,函需在纤毛虫物种鉴定、分类、进化及生态学等研究中引入辅助性的分子标记,而这一客观需求推动了纤毛虫领域分子技术的革新和多学科的交叉发展[2, 56-57]。到本世纪初,得益于DNA条形码技术和基于高通量测序技术的各种组学技术的快速发展,纤毛虫分子标记的研究再次迎来新的机遇和挑战。其中,适于纤毛虫类群的DNA条形码候选基因在不同层次、不同领域的应用和推广对当今纤毛虫生物多样性的研究提出了新的要求,也带来了新的认知。
该技术自21世纪初建立以来,在动物、植物、真菌等类群的生物多样性研究中大放异彩,但在真核微生物上的应用却严重滞后。作为为数不多的代表性工作之一,作者团队在过去几年中围绕纤毛虫DNA条形码候选基因的筛选工作开展了建设性的探索[58-60]。
研究进展可以概括为如下几点:(1)证实了常规barcode条码标记基因——线粒体细胞色素c氧化酶I(COI)基因5’端序列不仅可(例如在草履虫属内)进行近缘种的物种界定,而且具有鉴别隐存种的优势;(2)比较探讨了COI基因不同层面的差异对种类鉴定和种群结构分析的影响,明确了线粒体基因的异质性,揭示了COI基因在同一细胞内部以及个体间、种群间的遗传差异,但该差异不影响其物种界定的能力;(3)以前口虫为材料,我们综合比较了7个核基因变异区和线粒体COI基因作为DNA条形码的区分效力,揭示了COI、ITS1、ITS2、28S rRNA基因D2区具有明显的barcode gap区,特别是COI有发现隐存种的潜力;(4)我们随后的研究发现,虽COI基因有较大优势,但其在不同类群间遗传分化较大且存在长度各异的插入区,不利于通用引物设计。此外,高变的ITS1/ITS2区及18S rRNA基因V2/V9区虽在一定程度上可进行物种界定但却无法较好的反映物种间的演化关系。鉴于此,我们提出纤毛虫物种多样性研究适宜于用18S rRNA基因V4区对非近缘物种进行判读,再借助进化速率较快的28S rRNA基因D1~D2区或COI基因进行近缘种或隐存种刻画的策略(见图 4)[61]。

|
(A,D自Zhao et al.[58];B自Zhao et al.[60];C自Zhao et al.[59]。A, D from Zhao et al.[58]; B from Zhao et al.[60]; C from Zhao et al.[59]. ) 图 4 纤毛虫原生动物的DNA条形码研究 Fig. 4 DNA barcoding of ciliated protozoans |
而随着高通量测序技术的推进,应运而生的宏条形码技术对纤毛虫物种多样性分析的分子标记又提出了新的要求:合适的通用引物、合适的片段长度和丰富准确的参考数据库。因此,与前期的基因标记筛选工作相比,更为迫切的任务是物种参考数据库的构建。目前制约建库的首要因素是素材(物种、种群)的采集与正确鉴定,这仍需寄希望于分类学家专业的形态分类学知识。得益于作者及合作团队过去二十年研究工作的积累和由此构建的内容丰富的纤毛虫DNA库提供的素材,基于18S rRNA基因V4区的宏基因组学分析初步完成了淡水环境样本较大规模的摸底分析,实现了从类群水平到群落水平的推进。可以预见的前景是,基因条形码技术的完善和应有将进一步助力于揭示纤毛虫各种生态系统中重要生态学功能、地位及其与环境间的相互关系。
5 纤毛虫的组学与基因进化随着高通量测序技术和生物信息学技术的迅猛发展,基因组和转录组等组学大数据的解析为研究生命的起源与演化、发育、遗传、代谢等一系列生物学基本问题提供了全新的视野和有力的工具,已经成为当前生物科学领域中最具影响力的前沿领域[62-64]。与此领域大背景相对照,纤毛虫组学的研究目前尚处于方兴未艾的起步阶段,大量未知等待解答。近年来,作者依托所在团队的分类学背景,聚焦进化地位特殊种、环境极端种等类群,目前已经完成了对包括大弹跳虫、玫瑰喇叭虫、扇形游仆虫、具沟急游虫等十余种基因组和转录组的深度测序。此外,我们利用单细胞测序技术,累计完成了对80余代表物种单细胞水平的基因组/转录组测序(见图 5)。

|
(A~D, K自Chen et al.[65];E, F自Chen et al.[53];G~J自Zheng et al.[66];L自Gao et al.[70];M自Gao et al.[71]。A~D, K from Chen et al.[65]; E, F from Chen et al.[53]; G~J from Zheng et al.[66]; L from Gao et al.[70]; M from Gao et al.[71].) 图 5 纤毛虫原生动物的基因组与进化 Fig. 5 Genome and evolution of ciliated protozoans |
作为代表性成果之一,我们以扇形游仆虫为模式,分析了高质量的大核基因组、部分小核基因组及不同环境胁迫下的转录组数据[65],结果包括:(1)其大核基因组中含有超过25 000个完整的单基因的微型染色体(单倍体基因组大小约85MB);(2)小核基因组中丰度最高的内部删除序列的边界都存在保守的序列基序5’-TA-3’,且染色体断裂位点(CBS)被复制并保留在大核基因组中;(3)通过分析不同环境胁迫(营养缺乏、极端温度、极端盐度等)下转录组的差异表达,揭示了对外部物理或化学刺激作出反应的基因簇;(4)在盐度和化学应激下,HSP70基因的转录显著增加,但是对温度刺激没有反应;推测这种耐温性与调节区温度压力敏感元件的演化缺失有关。这些数据的解析为理解具有高度适应能力的微生物的独特生物学提供了分子证据。
我们对大弹跳虫的基因组和转录组的工作显示:(1)其大核染色体呈现高度的片段化,微型染色体的拷贝数不均匀,与基因表达水平相关;(2)DNA和RNA水平存在明显的动态变化,包括大核形成过程中内部删除序列的可变剪切和转录过程中内含子的大规模可变剪切;(3)大多数微型染色体的副端粒区存在一个稳定的TATAbox结构,其转录终止机制高度简洁;(4)对不同温度下的转录组测序数据进行差异表达分析可以从细胞膜结构和跨膜转运、细胞内吞和溶酶体、细胞周期控制、核小体组蛋白等层面解读了大弹跳虫对低温胁迫的适应机制。
结合基因组与转录组数据,我们对玫瑰喇叭虫的大核基因组(内含子与非编码基因间区)和种群遗传学研究表明:(1)与同属种天蓝喇叭虫相比较显示,喇叭虫的进化历程中存在一次全基因组的倍增事件,玫瑰喇叭虫的基因组呈现为未发生全基因组倍增的祖先形态;(2)两种喇叭虫均具有极短的内含子和非编码基因间区,且非编码区域能够有效地起始转录。
以柠檬类瘦尾虫为材料,我们利用端粒引物PCR扩增和高通量测序相结合的方法,快捷地获取了其部分大核基因组数据[66],确认该物种的端粒序列为(C4A4)2C4,主要发现包括:(1)大核基因组基本为“一条基因一条染色体”的模式,有少量染色体包含两或三个基因;(2)密码子偏好性分析显示其存在终止密码子重分配现象;(3)染色体分析显示出明显不对称的GC偏移和正义链亚端粒区高AT偏好,同时在亚端粒区域的3’端发现11 bp高AT含量的结构单元及存在一个40 bp不敏感的GC振荡区,推测可能与端粒的添加、转录和复制的启动等有关;(4)发现潜在的TATA-box存在于编码链的5’亚端粒区,集中在30~50 nt区段。
我们对具沟急游虫转录组的深度测序和分析[67],完成了超级树和超级数据集两种系统发育关系重构方法的比较,结果表明两种方法重构的纤毛虫进化关系在纲级水平上是一致的;基于包括具沟急游虫在内的13种纤毛虫的形态学和基因表达差异信息比较,在组学水平上探究了表层胞质蛋白基因的表达水平与其细胞形态的相关性并揭示了Alveolin和EPC两个表层胞质蛋白基因家族的表达量与围口动基列、射出体、口区等形态学特征具有显著相关性。
基因重组作为一重要的产生基因多样性及驱动进化的机制,在许多真核细胞中都有发生。这一过程可以发生在某些特定基因上,也可以发生在全基因组层面。最剧烈和普遍的基因重组发生在纤毛虫的大核基因组形成过程中。由于不同纤毛虫的大小核数目不尽相同以及高度的物种多样性,其细胞大小核演化过程中序列的删除、乱序基因的重排、染色体片段化及基因多倍化程度等基因重组的模式和机制也因此呈现出显著的差异[68-69]。围绕此过程和机理的研究对于了解物种形成和分化、真核生物基因组进化等都具有重要的理论意义。
作者及合作者利用已完成转录组分析的钩刺斜管虫为材料,对其基因乱序和不同基因家族的选择性拼接模式等进行了探讨和揭示[70-71],主要成果包括:(1)对高通量转录组数据的分析,从中筛选出112个候选基因家族,发现选择性拼接在基因家族中广泛存在且这些基因可能是该纤毛虫所特有;(2)首次测定钩刺斜管虫中三个选择性拼接基因家族,发现不同基因家族具有不同的选择性拼接模式;(3)提出了乱序基因进化的理论模型:基因乱序由基因复制及之后的分化来演绎,选择性拼接可能是基因乱序进化过程的中间形式;(4)钩刺斜管虫隐匿种间大小核基因的重组模式较为保守,但是小核特异序列的进化速率较高,且接头序列存在移位现象,揭示了基因重排在物种分化中的作用[72]。以上工作为基因组结构对基因家族进化的影响给出了新观点和新诠释,为纤毛虫物种分化-形成机理提供了重要资讯。
在国际范围内,围绕纤毛虫大小核间的基因组重组模式和机制的研究目前尚处于探索阶段。与现有纤毛虫的庞大系统相比,迄今有关两型核间基因组重组的认知均来自对四膜虫、草履虫及个别“一个基因一条染色体”淡水模式种的研究,而对于具有更高多样性的海洋类群仍一无所知。我们的目标是建立一海洋纤毛虫新型模式动物:扇形游仆虫。以此为材料,通过高通量测序技术及生物信息学手段,揭示其大小核基因组的重组模式;在此基础上,探讨大小核基因组的重组机制,为后续更深入的机理研究建立第一手的数据资料;同时,优化扇形游仆虫接合生殖的诱导条件,揭示其有性生殖过程中大小核演化的完整过程、界定性别决定相关的调控基因。这项工作的目的是将该物种构建为一新的海洋模式生物,从而为拓展更大空间的基因进化研究奠定基础。
6 以四膜虫为模式的表观遗传学在过去近半个世纪中,以四膜虫为模式动物的研究曾先后取得了包括核酶和端粒酶在内的重大发现。伴随着全基因组测序及基因操作技术的建立,特别是自人类获得其无菌培养体系以来,四膜虫已发展成为细胞生物学、进化生物学、遗传学、发育生物学等领域中理想的模式材料[73]。
与大部分纤毛虫一样,四膜虫的大小核具有(几乎)相同的DNA序列却有着不同的表观遗传调控因素,二者在基因组大小、基因表达、基因拷贝数等各方面具有巨大的差异。迄今所知,这些差异在很大程度上是由表观遗传因素(如组蛋白修饰、DNA甲基化等)进行调控的。
利用四膜虫为模式材料,我们及合作者团队近年来聚焦于组蛋白修饰和DNA甲基化(见图 6),陆续开展和完成了以下工作:
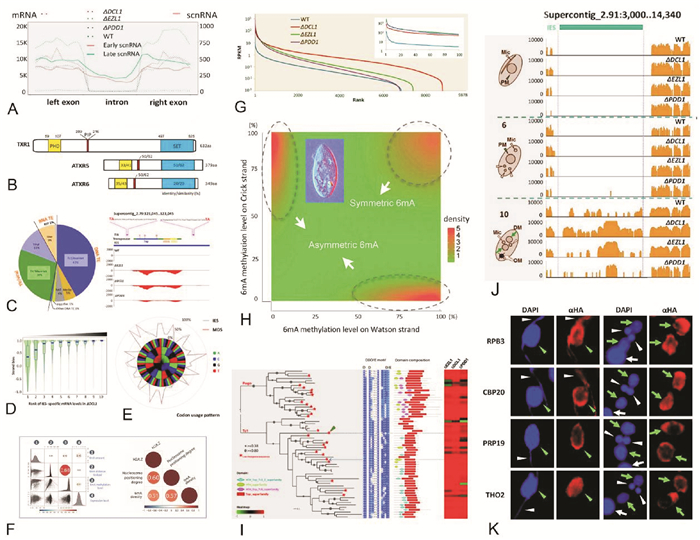
|
(A, C, D, E, G, I, J, K自Zhao et al.[81];F, G改自Wang et al.[78];H改自Gao et al.[74]。A, C, D, E, G, I, J, K was adapted from Zhao et al.[81; F, G was adapted from Wang et al.[78]; H was adapted from Gao et al.[74]. ) 图 6 四膜虫的表观遗传学 Fig. 6 Epigenetic studies using Tetrahymena as model organism |
(1) 首次鉴定了组蛋白单甲基化酶TXR1(Tetrahymena Trithorax Related-1),可特异性地催化组蛋白第27位赖氨酸的甲基化(H3K27me1)。我们还证实了TXR1通过与PCNA的结合定位到复制叉上,催化形成H3K27me1,以维持正常的染色体构象或影响下游作用因子的结合。本工作为研究表观遗传信息和遗传信息的协同复制提供了重要的模式平台[74-77]。
(2) 绘制了首份单碱基分辨率的四膜虫N6-腺嘌呤甲基化(6mA)全基因组分布图谱,并在此基础上系统刻画了其序列特征和染色质环境:发现6mA位点绝大部分位于5’-AT-3’序列上,偏向性的分布在RNA聚合酶II转录基因的5’端,但与转录活性仅存在弱相关性。另外,6mA聚集在连接DNA上,且该区域两侧富集占位稳定性高、含有组蛋白变体H2A.Z的核小体。上述发现揭示了6mA的分布由序列特征和染色质环境共同决定,并由此提出6mA与基因转录、染色质重塑等同为染色质调控的重要组成,为后续研究6mA的功能和调控机制提供了重要数据[78]。
(3) 改进了分离条件从而获得了大量高纯度的大核和小核并利用优化的酶切和离心方案将核小体分辨率提高至单核小体水平。在此基础上,通过分别对大核和小核的单核小体样品进行高通量测序及分析,我们发现顺式元件主要决定核小体占位,而反式元件主要决定核小体定位。同时,顺式和反式元件对于核小体的全局分布具有协同调控效应。此工作充分利用了四膜虫的双元核对照系统,为顺式(序列特征)和反式(转录活性)作用元件共同决定核小体分布的理论提供了直接证据[79-80]。
(4) 发现了转座子的编码RNA(表达)和非编码RNA(沉默)利用同一套转录系统进行转录,两种RNA之间的平衡由Polycomb抑制通路和RNA干扰通路共同调控。新发现的现象包括:Polycomb抑制通路蛋白和RNA干扰通路蛋白的敲除株系中,通常状况下应为沉默状态的转座子的表达被大规模激活,某些被激活的转座子可能会编码有活性的转座酶并控制自身的转座。该工作系首次针对四膜虫中转座子的调控机制进行的探讨,解释了其沉默与重新激活这一看似矛盾的现象,为深入研究转座子与宿主的协同进化提供了重要理论支持[81]。
我们进展中的工作仍将继续围绕甲基化这一主流方向,特别聚焦在DNA 6mA催化酶学系统和表观遗传识别信号系统的鉴定和刻画、组蛋白TXR1蛋白调控复制的分子机制的解析、转座子对基因组稳定性的影响等问题。此外,还将尝试构建新的模式研究体系,即以海洋纤毛虫为模式的动物模型。这项探索的潜在意义在于:将极大地补充现有纤毛虫模式生物紧缺的现状,为未来深入挖掘真核生物表观遗传调控中的新现象和新机制奠定基础。
致谢: 借此感谢(中国海洋大学)原生动物学研究室的历届毕业生以及在读生们,在过去的20余年中,他们为实验室的建设和发展、为该文中所涉成果做成了巨大的贡献。特别谢意送给我们的国际合作者们,他们的加盟与合作,共同成就了团队的今天。
| [1] |
宋微波, A沃伦, 胡晓钟. 中国黄渤海的自由生纤毛虫[M]. 北京: 科学出版社, 2009: 518. Song W B, Warren A, Hu X Z. Free-living Ciliates in the Bohai and Yellow Seas, China[M]. Beijing: Science Press, 2009: 518. (  0) 0) |
| [2] |
伊珍珍, 苗苗, 高珊, 等. 纤毛虫原生动物的分子生物学研究:若干热点领域及新进展[J]. 科学通报, 2016, 61: 2227-2238. Yi Z Z, Miao M, Gao S, et al. On molecular biology of ciliated protozoa: Frontier areas and progresses[J]. China Science Bulletin, 2016, 61: 2227-2238. (  0) 0) |
| [3] |
Azam F, Fenchel T, Field J G, et al. The ecological role of water column microbes in the sea[J]. Marine Ecology Progress Series, 1983, 10: 257-263. DOI:10.3354/meps010257
(  0) 0) |
| [4] |
Lynn D H. The Ciliated Protozoa: Characterization, Classification, and Guide to the Literature: 3rd Ed[M]. Berlin: Springer, 2008: 605.
(  0) 0) |
| [5] |
Dolan J R, Montagnes D J S, Agatha S, et al. The Biology and Ecology of the Tintinnid Ciliates: Models for Marine Plankton[M]. Oxford: Wiley-Blackwell, 2013: 296.
(  0) 0) |
| [6] |
Hu X Z, Lin X F, Song W B. Ciliate Atlas: Species Found in South China Sea[M]. Beijing: Science Press, 2019: 631.
(  0) 0) |
| [7] |
陈相瑞, 宋微波, 胡晓钟. 青岛沿岸吸管虫(原生动物, 纤毛门)一新属、新种及九纪录种记述[J]. 动物分类学报, 2005, 30(3): 493-500. Chen X R, Song W B, Hu X Z. Morphology of suctorid ciliates from coastal waters off Qingdao, with description of a new genus and a new species (Protozoa, Ciliophora)[J]. Acta Zootaxonomica Sinica, 2005, 30: 493-500. DOI:10.3969/j.issn.1000-0739.2005.03.008 (  0) 0) |
| [8] |
Chen X R, Gao S, Liu W W, et al. Taxonomic descriptions of three marine colepid ciliates, Nolandia sinica spec. nov., Apocoleps caoi spec. nov. and Tiarina fusa (Claparède & Lachmann, 1858) Bergh, 1881 (Ciliophora, Prorodontida)[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 735-744. DOI:10.1099/ijs.0.026302-0
(  0) 0) |
| [9] |
Fan X P, Pan H B, Li L F, et al. Phylogeny of the poorly known ciliates, Microthoracida, a systematically confused taxon (Ciliophora), with morphological reports of three species[J]. Journal of Eukaryotic Microbiology, 2014, 61: 227-237. DOI:10.1111/jeu.12099
(  0) 0) |
| [10] |
Luo X T, Huang J, Li L F, et al. Phylogeny of the ciliate family Psilotrichidae (Protista, Ciliophora), a curious and poorly-known taxon, with notes on two algae-bearing psilotrichids from Guam, USA[J]. BMC Evolutionary Biology, 2019, 19: 125. DOI:10.1186/s12862-019-1450-z
(  0) 0) |
| [11] |
Pan X M, Yi Z Z, Li J Q, et al. Biodiversity of marine scuticociliates (Protozoa, Ciliophora) from China: Description of seven morphotypes including a new species, Philiaster sinensis spec. nov.[J]. European Journal of Protistology, 2015, 51: 142-157. DOI:10.1016/j.ejop.2015.02.005
(  0) 0) |
| [12] |
Yan Y, Fan Y B, Chen X R, et al. Taxonomy and phylogeny of three heterotrich ciliates (Protozoa, Ciliophora), with description of a new Blepharisma species[J]. Zoological Journal of the Linnean Society, 2016, 177: 320-334. DOI:10.1111/zoj.12369
(  0) 0) |
| [13] |
Zhang T Y, Qi H L, Zhang T T, et al. Morphology, morphogenesis and molecular phylogeny of a new brackish water subspecies, Neourostylopsis flava paraflava nov. subsp. (Ciliophora, Hypotrichia, Urostylidae), with redefinition of the genus Noeurostylopsis[J]. European Journal of Protistology, 2018, 66: 48-62. DOI:10.1016/j.ejop.2018.07.004
(  0) 0) |
| [14] |
宋微波, 赵元莙, 徐奎栋, 等. 海水养殖中的危害性原生动物[M]. 北京: 科学出版社, 2003: 483. Song W B, Zhao Y J, Xu K D, et al. Pathogenc Protozoa in Mariculture[M]. Beijing: Science Press, 2003: 483. (  0) 0) |
| [15] |
Jiang Y, Xu H L, Hu X Z, et al. Functional groups of marine ciliated protozoa and their relationships to water quality[J]. Environmental Sciecne and Pollution Research, 2013, 20: 5272-5280. DOI:10.1007/s11356-013-1525-0
(  0) 0) |
| [16] |
Xu Y, Li J M, Song W, et al. Phylogeny and establishment of a new ciliate family, Wilbertomorphidae fam. nov. (Ciliophora, Karyorelictea), a highly specialized taxon represented by Wilbertomorpha colpoda gen. nov., spec. nov[J]. Journal of Eukaryotic Microbiology, 2013, 60: 480-489. DOI:10.1111/jeu.12055
(  0) 0) |
| [17] |
Xu Y, Li J, Gao F, et al. Apotrachelocerca arenicola (Kahl, 1933) gen. n., comb. n. (Protozoa, Ciliophora, Trachelocercidae): Phylogeny and morphology[J]. Journal of Eukaryotic Microbiology, 2011, 58: 504-510. DOI:10.1111/j.1550-7408.2011.00578.x
(  0) 0) |
| [18] |
Yan Y, Xu Y, Al-Farraj S A, et al. Morphology and phylogeny of three trachelocercids(Protozoa, Ciliophora, Karyorelictea), with description of two new species and insight to the evolution of the family Trachelocercidae[J]. Zoological Journal of the Linnean Society, 2016, 177: 306-319. DOI:10.1111/zoj.12364
(  0) 0) |
| [19] |
Wu L, Clamp, J C, Yi Z, et al. Phylogeneticand taxonomic revision of an enigmatic group of haptorianciliates, with establishment of the Kentrophyllidae fam n. (Protozoa, Ciliophora, Litostomatea, Pleurostomatida)[J]. PLoS One, 2015, 10: 123720.
(  0) 0) |
| [20] |
Pan H B, Lin X F, Gong J, et al. Taxonomy of five species of cyrtophorids (Protozoa: Ciliophora) including consideration of the phylogeny of two new genera[J]. Zoological Journal of the Linnean Society, 2012, 164: 1-17. DOI:10.1111/j.1096-3642.2011.00751.x
(  0) 0) |
| [21] |
Qu Z S, Li L F, Lin X F, et al. Diversity of the cyrtophorid genus Chlamydodon (Protista, Ciliophora): Its systematics and geographic distribution, with taxonomic descriptions of three species[J]. Systematics and Biodiversity, 2018, 16: 497-511. DOI:10.1080/14772000.2018.1456493
(  0) 0) |
| [22] |
Miao M, Wang Y, Song W B, et al. Description of Eurystomatella sinica n. gen., n. sp., with establishment of a new family Eurystomatellidae n. fam. (Protista, Ciliophora, Scuticociliatia) and analyses of its phylogeny inferred from sequences of the small-subunit rRNA gene[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60: 460-468. DOI:10.1099/ijs.0.009621-0
(  0) 0) |
| [23] |
Fan X P, Hu X Z, Al-Farraj S A, et al. Morphological description of three marine ciliates (Ciliophora, Scuticociliatia), with establishment of a new genus and two new species[J]. European Journal of Protistology, 2011, 47: 186-196. DOI:10.1016/j.ejop.2011.04.001
(  0) 0) |
| [24] |
Liu W W, Yi Z Z, Xu D, et al. Two new genera of planktonic ciliates and insights into the evolution of the family Strombidiidae (Protista, Ciliophora, Oligotrichia)[J]. PloS One, 2015, 10: 131726.
(  0) 0) |
| [25] |
Luo X T, Gao F, Yi Z Z, et al. Taxonomy and molecular phylogeny of two new brackish hypotrichous ciliates, with the establishment of a new genus (Protozoa, Ciliophora)[J]. Zoological Journal of the Linnean Society, 2017, 179: 475-491.
(  0) 0) |
| [26] |
Fan Y B, Chen X M, Hu X Z, et al. Morphology and morphogenesis of Apoholosticha sinica n. g., n. sp. (Ciliophora, Hypotrichia), with consideration of its systematic position among urostylids[J]. European Journal of Protistology, 2014, 50: 78-88. DOI:10.1016/j.ejop.2013.06.003
(  0) 0) |
| [27] |
Pan Y, Li J M, Li L F, et al. Ontogeny and molecular phylogeny of a new marine ciliate genus, Heterokeronopsis g. n. (Protozoa, Ciliophora, Hypotricha), with description of a new species[J]. European Journal of Protistology, 2013, 49: 298-311. DOI:10.1016/j.ejop.2012.08.008
(  0) 0) |
| [28] |
Liu W W, Shao C, Gong J, et al. Morphology, morphogenesis and molecular phylogeny of a new marine urostylid ciliate (Ciliophora, Stichotrichia) from the South China Sea, and an overview of the convergent evolution of midventral complex within the Spirotrichea[J]. Zoological Journal of the Linnean Society, 2010, 158: 697-710. DOI:10.1111/j.1096-3642.2009.00565.x
(  0) 0) |
| [29] |
Jiang J M, Song W B. Two new Diophrys-like genera and their type species, Apodiophrys ovalis n. gen., n. sp. and Heterodiophrys zhui n. gen., n. sp. (Ciliophora: Euplotida), with notes on their molecular phylogeny[J]. Journal of Eukaryotic Microbiology, 2010, 57: 354-361. DOI:10.1111/j.1550-7408.2010.00482.x
(  0) 0) |
| [30] |
宋微波, 邵晨. 腹毛类纤毛虫的细胞发生模式[M]. 北京: 科学出版社, 2017: 498. Song W B, Shao C. Ontogenetic Patterns of Hypotrich Ciliates[M]. Beijing: Science Press, 2017: 498. (  0) 0) |
| [31] |
Jiang J M, Shao C, Xu H Z, et al. Morphogenetic observations on the marine ciliate Euplotes vannus (Müller, 1786) Diesing, 1850 during cell division (Protozoa, Ciliophora)[J]. Journal of the Marine Biological Association of the United Kingdom, 2010, 90: 683-689. DOI:10.1017/S0025315409991342
(  0) 0) |
| [32] |
Lu X T, Huang J, Shao C, et al. Morphology, cell-division, and phylogeny of Schmidingerothrix elongata spec. nov. (Ciliophora, Hypotricha), and brief guide to hypotrichs with Gonostomum-like oral apparatus[J]. European Journal of Protistology, 2018, 62: 24-42. DOI:10.1016/j.ejop.2017.11.001
(  0) 0) |
| [33] |
Luo X T, Li L F, Wang C D, et al. Morphologic and phylogenetic studies of two hypotrichous ciliates, with notes on morphogenesis in Gastrostyla steinii Engelmann, 1862 (Ciliophora, Hypotrichia)[J]. European Journal of Protistology, 2017, 60: 119-133. DOI:10.1016/j.ejop.2017.05.001
(  0) 0) |
| [34] |
Shao C, Song W B, Li L F, et al. Systematic position of Discocephalus-like ciliates (Ciliophora: Spirotrichea) inferred from SS rRNA gene and ontogenetic information[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58: 2962-2972. DOI:10.1099/ijs.0.65781-0
(  0) 0) |
| [35] |
Shao C, Song W B, Warren A, et al. Morphogenesis of Kiitricha marina Nozawa, 1941 (Ciliophora, Spirotrichea), a possible model for the ancestor of hypotrichs s. l.[J]. European Journal of Protistology, 2009, 45: 292-304. DOI:10.1016/j.ejop.2009.07.002
(  0) 0) |
| [36] |
Bai Y, Li S, Li Y, et al. Morphogenesis and molecular characterization of a little known soil ciliate, Oxytricha nauplia Berger et Foissner, 1987 (Ciliophora, Sporadotrichida)[J]. Acta Protozoologica, 2018, 57: 79-94.
(  0) 0) |
| [37] |
Gao F, Gao S, Wang P, et al. Phylogenetic analyses of cyclidiids (Protista, Ciliophora, Scuticociliatia) based on multiple genes suggest their close relationship with thigmotrichids[J]. Molecular Phylogenetics and Evolution, 2014, 75: 219-226. DOI:10.1016/j.ympev.2014.01.032
(  0) 0) |
| [38] |
Gao F, Katz L A, Song W B. Multigene-based analyses on evolutionary phylogeny of two controversial ciliate orders: Pleuronematida and Loxocephalida (Protista, Ciliophora, Oligohymenophorea)[J]. Molecular Phylogenetics and Evolution, 2013, 68: 55-63. DOI:10.1016/j.ympev.2013.03.018
(  0) 0) |
| [39] |
Gao F, Katz L A, Song W B. Insights into the phylogenetic and taxonomy of philasterid ciliates (Protozoa, Ciliophora, Scuticociliatia) based on analyses of multiple molecular markers[J]. Molecular Phylogenetics and Evolution, 2012, 64: 308-317. DOI:10.1016/j.ympev.2012.04.008
(  0) 0) |
| [40] |
Gao F, Li J M, Song W B, et al. Multi-gene-based phylogenetic analysis of oligotrich ciliates with emphasis on two dominant groups: Cyrtostrombidiids and strombidiids (Protozoa, Ciliophora)[J]. Molecular Phylogenetics and Evolution, 2016, 105: 141-150.
(  0) 0) |
| [41] |
Gao S, Huang J, Li J M, et al. Molecular phylogeny of the cyrtophorid ciliates (Protozoa, Ciliophora, Phyllopharyngea)[J]. PloS One, 2012, 7: e33198. DOI:10.1371/journal.pone.0033198
(  0) 0) |
| [42] |
Wang P, Wang Y R, Wang C D, et al. Further consideration on the phylogeny of ciliated protists: Analyses using both mitochondrial and nuclear data with focus on the extremely confused class Phyllopharyngea (Protista, Ciliophora)[J]. Molecular Phylogenetics and Evolution, 2017, 112: 96-106. DOI:10.1016/j.ympev.2017.04.018
(  0) 0) |
| [43] |
Chen X, Ma H G, Al-Rasheid K A S, et al. The molecular data suggests that the ciliate Mesodinium (Protista, Ciliophora) might represent an undescribed taxon at class level[J]. Zoological Systematics, 2015, 40: 31-40.
(  0) 0) |
| [44] |
Gao F, Warren A, Zhang Q Q, et al. The all-data-based evolutionary hypothesis of ciliated protists with a revised classification of the phylum Ciliophora (Eukaryota, Alveolata)[J]. Scientific Reports, 2016, 6: 24874. DOI:10.1038/srep24874
(  0) 0) |
| [45] |
Li L, Shao C, Song W B, et al. Is Kiitricha (Ciliophora, Spirotrichea) a member of euplotids or does it represent a low branch of spirotrichs?[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59: 439-446. DOI:10.1099/ijs.0.65801-0
(  0) 0) |
| [46] |
Liu W W, Yi Z Z, Lin X F, et al. Phylogeny of three choreotrich genera (Protozoa, Ciliophora, Spirotrichea), with morphological and morphogenetic investigations on three strobilidiid species[J]. Zoologica Scripta, 2012, 41: 417-434. DOI:10.1111/j.1463-6409.2012.00542.x
(  0) 0) |
| [47] |
Fan X P, Pan H B, Li L F, et al. Phylogeny of the poorly known ciliates, Microthoracida, a systematically confused taxon (Ciliophora), with morphological reports of three species[J]. Journal of Eukaryotic Microbiology, 2014, 61: 227-237. DOI:10.1111/jeu.12099
(  0) 0) |
| [48] |
Miao M, Shao C, Chen X, et al. Evolution of discocephalid ciliates: Molecular, morphological and ontogenetic data support a sister group of discocephalids and pseudoamphisiellids (Protozoa, Ciliophora) with establishment of a new suborder Pseudoamphisiellina subord. n.[J]. Science China Life Sciences, 2011, 54: 634-641. DOI:10.1007/s11427-011-4192-8
(  0) 0) |
| [49] |
Miao M, Warren A, Song W B, et al. Analysis of the internal transcribed spacer 2 (ITS2) region of scuticociliates and related taxa (Ciliophora, Oligohymenophorea) to infer their evolution and phylogeny[J]. Protist, 2008, 159: 519-533. DOI:10.1016/j.protis.2008.05.002
(  0) 0) |
| [50] |
Wang P, Gao F, Huang J, et al. A case study to estimate the applicability of secondary structures of SSU-rRNA gene in taxonomy and phylogenetic analyses of ciliates[J]. Zoologica Scripta, 2015, 44: 574-585. DOI:10.1111/zsc.12122
(  0) 0) |
| [51] |
Yi Z Z, Dunthorn M, Song W B, et al. Increased taxon sampling using both unidentified environmental sequences and identified cultures for microbial eukaryotes improves phylogenetic inference[J]. Molecular Phylogenetics and Evolution, 2010, 57: 937-941. DOI:10.1016/j.ympev.2010.08.001
(  0) 0) |
| [52] |
Yi Z Z, Strüder-Kypke M, Hu X Z, et al. Sampling strategies for improving tree accuracy and phylogenetic analyses: A case study in ciliate protists, with notes on the genus Paramecium[J]. Molecular Phylogenetics and Evolution, 2014, 71: 142-148. DOI:10.1016/j.ympev.2013.11.013
(  0) 0) |
| [53] |
Chen X, Wang Y R, Sheng Y L, et al. GPSit: An automated method for evolutionary analysis of nonculturable ciliated microeukaryotes[J]. Molecular Ecology Resources, 2018, 18: 700-713. DOI:10.1111/1755-0998.12750
(  0) 0) |
| [54] |
Kress W J, Erickson D L. DNA barcodes: Genes, genomics, and bioinformatics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(8): 2761-2762. DOI:10.1073/pnas.0800476105
(  0) 0) |
| [55] |
Kress W J, García-Robledo C, Uriarte M, et al. DNA barcodes for ecology, evolution, and conservation[J]. Trends in Ecology & Evolution, 2015, 30: 25-35.
(  0) 0) |
| [56] |
Allen S L, Adams J, Rushford C L. Interspecies relationships in the Paramecium aurelia complex: Acid phosphatase variation[J]. Journal of Eukaryotic Microbiology, 1983, 30: 143-147.
(  0) 0) |
| [57] |
Schlegel M. Comparative study of allozyme variation in eight species of hypotrichous ciliates (Polyhymenophora, Ciliophora)[J]. Journal of Zoological Systematics and Evolutionary Research, 1985, 23: 171-183.
(  0) 0) |
| [58] |
Zhao Y, Gentekaki E, Yi Z Z, et al. Genetic differentiation of the mitochondrial cytochrome oxidase c subunit I gene in genus Paramecium (Protista, Ciliophora)[J]. PLos One, 2013, 8: 77044. DOI:10.1371/journal.pone.0077044
(  0) 0) |
| [59] |
Zhao Y, Yi Z Z, Gentekaki E, et al. Utility of combining morphological characters, nuclear and mitochondrial genes: An attempt to resolve the conflicts of species identification for ciliated protists[J]. Molecular Phylogenetics and Evolution, 2016, 94: 718-729. DOI:10.1016/j.ympev.2015.10.017
(  0) 0) |
| [60] |
Zhao Y, Yi Z Z, Warren A, et al. Species delimitation for the molecular taxonomy and ecology of a widely distributed microbial eukaryotes genus Euplotes (Alveolata, Ciliophora)[J]. Proceedings of the Royal Society B: Biological, 2018, 285: 20172159. DOI:10.1098/rspb.2017.2159
(  0) 0) |
| [61] |
Pawlowski J, Audic S, Adl S, et al. CBOL protist working group: barcoding eukaryotic richness beyond the animal, plant, and fungal kingdoms[J]. PLoS Biology, 2012, 10: 1001419. DOI:10.1371/journal.pbio.1001419
(  0) 0) |
| [62] |
Chen X, Bracht J R, Goldman A D, et al. The architecture of a scrambled genome reveals massive levels of genomic rearrangement during development[J]. Cell, 2014, 158: 1187-1198. DOI:10.1016/j.cell.2014.07.034
(  0) 0) |
| [63] |
He M, Wang J, Fan X, et al. Genetic basis for the establishment of endosymbiosis in Paramecium[J]. The ISME Journal, 2019. DOI:10.1038/s41396-018-0341-4
(  0) 0) |
| [64] |
Singh D P, Saudemont B, Guglielmi G, et al. Genome-defence small RNAs exapted for epigenetic mating-type inheritance[J]. Nature, 2014, 509: 447-452. DOI:10.1038/nature13318
(  0) 0) |
| [65] |
Chen X, Jiang Y H, Gao F, et al. Genome analyses of the new model protist Euplotes vannus focusing on genome rearrangement and resistance to environmental stressors[J]. Molecular Ecology Resources, 2019. DOI:10.1111/1755-0998.13023
(  0) 0) |
| [66] |
Zheng W B, Wang C D, Yan Y, et al. Insights into an extensively fragmented eukaryotic genome: de novo genome sequencing of the multi-nuclear ciliate Uroleptopsis citrina[J]. Genome Biology and Evolution, 2018, 10: 883-894. DOI:10.1093/gbe/evy055
(  0) 0) |
| [67] |
Chen X, Zhao X L, Liu X H, et al. Phylogenomics of non-model ciliates based on transcriptomic analyses[J]. Protein Cell, 2015.
(  0) 0) |
| [68] |
Maurer-Alcalá X X, Knight R, Katz L A. Exploration of the germline genome of the ciliate Chilodonella uncinata through single-cell omics (Transcriptomics and genomics)[J]. mBio, 2018, 9: 1817-1836.
(  0) 0) |
| [69] |
Mochizuki K, Fine N A, Fujisawa T, et al. Analysis of a piwi-related gene implicates small RNAs in genome rearrangement in Tetrahymena[J]. Cell, 2002, 110: 689-699. DOI:10.1016/S0092-8674(02)00909-1
(  0) 0) |
| [70] |
Gao F, Katz L A. Phylogenomic analyses support the bifurcation of ciliates into two major clades that differ in properties of nuclear division[J]. Molecular Phylogenetics and Evolution, 2014, 70: 240-243. DOI:10.1016/j.ympev.2013.10.001
(  0) 0) |
| [71] |
Gao F, Roy S W, Katz L A. Analyses of alternatively processed genes in ciliates provide insights into the origins of scrambled genomes and may provide a mechanism for speciation[J]. MBio, 2015, 6: 1-8. DOI:10.3391/mbi.2015.6.1.01
(  0) 0) |
| [72] |
Zhang T T, Wang C D, Katz L A, et al. A paradox: Rapid evolution rates of germline-limited sequences are associated with conserved patterns of rearrangements in cryptic species of Chilodonella uncinata (Protist, Ciliophora)[J]. Science China Life Sciences, 2018, 61: 1071-1078. DOI:10.1007/s11427-018-9333-1
(  0) 0) |
| [73] |
Orias E, Cervantes M D, Hamilton E P. Tetrahymena thermophila, a unicellular eukaryote with separate germline and somatic genomes[J]. Research in Microbiology, 2010, 162: 578-586.
(  0) 0) |
| [74] |
Gao S, Xiong J, Zhang C C, et al. Impaired replication elongation in Tetrahymena mutants deficient in histone H3 Lys 27 monomethylation[J]. Genes & Development, 2013, 27: 1662-1679.
(  0) 0) |
| [75] |
Zhang C, Molascon A J, Gao S, et al. Quantitative proteomics reveals that the specific methyltransferases Txr1p and Ezl2p differentially affect the mono-, di-and trimethylation states of histone H3 lysine 27 (H3K27)[J]. Molecular & Cellular Proteomics, 2013, 12: 1678-1688.
(  0) 0) |
| [76] |
Zhang C, Gao S, Molascon AJ, et al. Quantitative proteomics reveals histone modifications in crosstalk with H3 lysine 27 methylation[J]. Molecular & Cellular Proteomics, 2014, 13: 749-759.
(  0) 0) |
| [77] |
Zhao X L, Wang Y Y, Wang Y R, et al. Histone methyltransferase TXR1 is required for both H3 and H3. 3 lysine 27 methylation in the well-known ciliated protist Tetrahymena thermophila[J]. Science China Life Sciences, 2017, 60: 264-270. DOI:10.1007/s11427-016-0183-1
(  0) 0) |
| [78] |
Wang Y, Sheng Y, Liu Y, et al. N6-methyladenine DNA modification in the unicellular eukaryotic organism Tetrahymena thermophila[J]. European Journal of Protistology, 2017, 58: 94-102. DOI:10.1016/j.ejop.2016.12.003
(  0) 0) |
| [79] |
Chen X, Gao S, Liu Y, et al. Enzymatic and chemical mapping of nucleosome distribution in purified micro-and macronuclei of the ciliated model organism, Tetrahymena thermophila[J]. Science China Life Sciences, 2016, 59: 909-919. DOI:10.1007/s11427-016-5102-x
(  0) 0) |
| [80] |
Xiong J, Gao S, Dui W, et al. Dissecting relative contributions of cis-and trans-determinants to nucleosome distribution by comparing Tetrahymena macronuclear and micronuclear chromatin[J]. Nucleic Acids Research, 2016, 44: 10091-10105.
(  0) 0) |
| [81] |
Zhao X, Xiong J, Mao F, et al. RNAi-dependent Polycomb repression controls transposable elements in Tetrahymena[J]. Genes & Development, 2019, 33: 348-364.
(  0) 0) |
2. College of Life Sciences, Capital Normal University, Beijing 100048, China;
3. The Key Laboratory of Aquatic Biodiversity and Conservation of Chinese Academy of Sciences, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China;
4. The Key Laboratory of Biomedical Information Engineering, Ministry of Education, School of Life Science and Technology, Xi'an Jiaotong University, Xi'an 710049, China
 2019, Vol. 49
2019, Vol. 49


