2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室,山东 青岛 266237
在深海热液生态系统中,来自海底的高温还原性热液与冷的氧化性海水相结合,形成了地球上独特的极端环境,具有黑暗、静水压力高、温度变化大、有毒物质(例如重金属、甲烷和硫化物)浓度高等特点[1-2]。在如此恶劣的环境下生存着大量的贻贝、管状蠕虫、虾蟹等大型底栖动物[3],不同于大部分依赖于光合作用的生态系统,深海热液生态系统依赖于化能合成,化能自养微生物为生态系统提供初级生产力[4],有研究提出参与硫化氢氧化的化能自养微生物在热液生态系统中维持着主要生产力[5],氢气、甲烷和还原性的金属离子也可以作为化能自养微生物的能量来源[6-7]。
已有研究通过分子生物学的手段以及显微镜技术发现在热液区动物的体内和体表存在大量的微生物,并与生物形成共附生关系,几乎所有热液区的动物都依赖于与其共生或者附生的化能自养微生物提供的初级生产力[8],这些共附生微生物在宿主的物质和能量循环中发挥着重要作用[9],参与热液区的硫代谢,甲烷代谢和氨代谢等过程。热液动物共附生细菌的多样性,能量利用,共生机制方面已经有了一些研究,但对于共附生细菌促进宿主适应热液极端环境的贡献方面研究较少。
目前主要在分子和细胞水平上对于热液微生物的极端环境适应机制进行了研究,有研究发现微生物通过群体感应(Quorum sensing, QS)来调控自身生理活性以适应环境压力[10]。群体感应是一种通过产生化学信号分子进行细胞间交流的机制,具有细菌密度依赖性,根据细胞密度的变化来调控基因表达,调节生理代谢[11-12]。当细胞密度达到一定阙值时,信号分子合成酶基因就会被诱导表达,产生更多信号分子与受体蛋白结合,启动下游基因表达,使细菌产生多种多样的群体行为[13],诸如形成生物膜、产生抗生素、发光等[14-15]。
2005年Johnson等[16]首次在深海热液环境中发现群体感应(QS)机制,他们发现海栖热孢菌(Thermogoga maritima)与詹氏甲烷球菌(Methanococus jannaschii)共培养时,产生的胞外多糖具有密度依赖性。通过共培养发现上调的多肽基因中包含QS系统的一个多肽,作者基于肽蛋白的QS系统调控胞外多糖的产生,推测这可能是热液生境微生物的生存策略。之后到2015年才从基因层面,在2株深海热液喷口来源的细菌和以ε-变形菌纲为主的生物膜中发现luxS类型的QS系统[17]。然而这些报道对深海热液环境中细菌的群体感应机制研究并不全面,作为自然界普遍存在的由酰基高丝氨酸内酯(N-Acyl homoserine lactons,AHLs)介导的luxI/R群体感应系统是否也存在于热液环境中还未见报道,若存在,QS系统调控的功能,以及它对细菌在热液环境的适应性上所发挥的作用也不清楚。
本研究利用传统的分离培养法对西南印度洋天成热液区贻贝可培养共附生细菌的多样性进行了初步研究,并从中筛选出群体感应细菌,对群体感应细菌的生理生化功能进行研究,包括产生的AHLs种类、成膜能力和利用DMSP(β-二甲基巯基丙酸内盐)的能力,以探讨贻贝共附生细菌在宿主适应热液区环境方面的贡献。
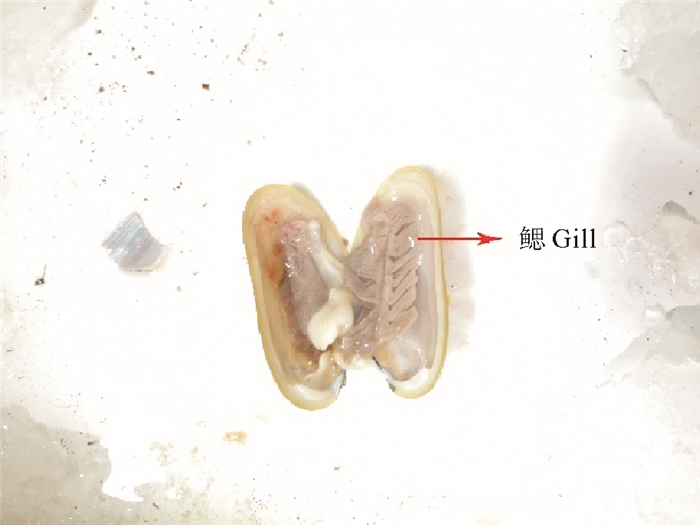
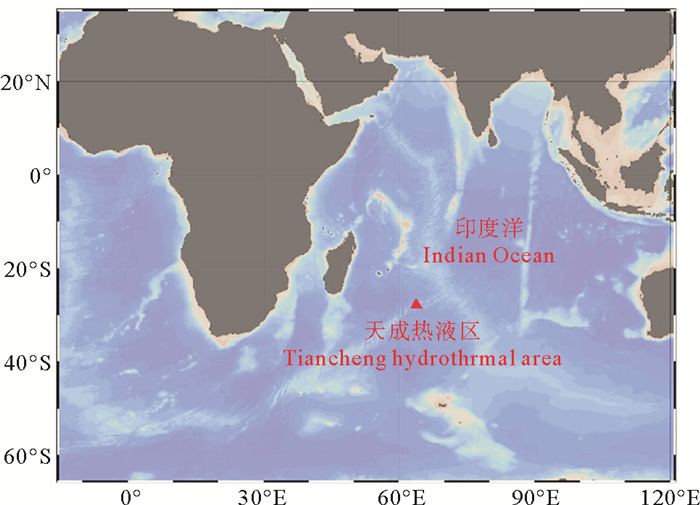
1 材料与方法 1.1 样品的采集本实验所用深海偏顶蛤(Bathymodiolus marisindicus,见图 1)于2019年4月16日采集自大洋科考52航次,西南印度洋第三航段的天成(TC)热液区,DY52-III-TC01ROV站位,水深2 700 m,经、纬度分别为63.923 2°E、27.850 8°S,见图 2。
|
图 1 采集自西南印度洋天成热液区的深海偏顶蛤 Fig. 1 Bathymodiolus marisindicus collected from the hydrothermal area of the Southwest Indian Ocean |
|
图 2 样品采集站位图 Fig. 2 Location map of sample collection station |
本研究采用2种常规的培养基2216E和R2A,这2种培养基含有丰富的C源和N源,有利于从可培养细菌中筛选群体感应细菌,培养基配方参照《海洋微生物学》(第二版)配制,分离细菌时采用10%浓度的培养基组分。
其他实验试剂和培养基配制方法如下:
(1) 20×Salt溶液:200 mL灭菌蒸馏水中加入(NH4)2SO4 8 g,MnSO4·H2O 4.8 mg,MgSO4 0.32 g,CaCl2 30.2 mg,过滤、除菌,使用孔径0.22 μm的无菌滤膜和注射器过滤加入灭菌的蓝盖瓶中。
(2) 20×Buffer溶液:蒸馏水中加入KH2PO442.8 g,NaOH 7 g,定容至200 mL,高压灭菌,待冷却后调节pH到7.0,使用孔径0.22 μm的无菌滤膜和注射器过滤加入灭菌的蓝盖瓶中。
(3) AHLs信号分子检测培养基[18]:在100 mL灭菌后的软琼脂(0.8%)中加入5 mL 20×Buffer、5 mL 20×Salt、1 mL葡萄糖(50%)、200 μL X-Gal,待冷却至45 ℃时加入1 mL Agrobacterium tumefaciens KYC55指示菌悬液(OD600nm =12)混匀。
(4) DMSP细菌培养基各成份含量(g/L)[19]:DMSP 8.051 8、NH4Cl 21.396 0、NaH2PO40.003 6、EDTA-Fe 0.042 1、ZnCl2 0.013 6、MnCl2 0.197 8、CoCl22.379 3×10-4、维生素B1 0.400 0、维生素B12 0.002 0、维生素H 0.002 0。
1.3 菌株的分离与培养在船上实验室取深海偏顶蛤鳃组织块(见图 1)5 g,加入10 mL无菌海水震荡,清洗未附着生长的细菌,再加入10 mL无菌海水,转移至无菌组织研磨器中,均质后转移至无菌试管中,按1∶1、1∶10、1∶100稀释后,各取200 μL涂布在10%浓度的2216E和10%浓度的R2A固体平板培养基上,于25 ℃培养14~20 d,观察长出的菌落,挑取不同的单菌落接种至含有2216E和R2A固体斜面培养基上,放入4 ℃冰箱保存。
航次结束后将试管中的菌落带回实验室,接种到对应的2216E和R2A固体平板培养基上划线培养,于25 ℃培养3~5 d,根据不同菌落形态,包括颜色、形状、大小和透明度等,挑取不同的单菌落划线纯化,使用30%甘油于-80 ℃超低温冰箱保种。
1.4 细菌16S rDNA基因测序及系统发育分析纯化细菌16S rDNA基因测序由青岛擎科生物公司完成,采用裂解法提取细菌基因组DNA,使用通用引物7F(5’-CAGAGTTTGATCCTGGCT-3’)和1 540R(5’-AGGAGGTGATCCAGCCGCA-3’)进行16S rDNA扩增,PCR反应体系(50 μL): PCR mix 46 μL;引物7F、1 540R各1 μL;Tamplate 2 μL。PCR反应程序:预变性98 ℃ 3 min;扩增(35个循环):变性98 ℃ 10 s,退火55 ℃ 15 s,延伸72 ℃ 20 s;最后72 ℃ 5 min。PCR产物经琼脂糖凝胶电泳检测后进行16S rDNA基因序列测定,将得到的16S rDNA基因序列在EZBioCloud(https://eztaxon-e.ezbiocloud.net/)上比对,并将测序结果上传至GenBank数据库得到序列号(MT914193-MT914217)。应用MEGA7.0软件,采取邻位相接法,Bootstrap方式,Replications设定为1 000,以Acidianus ambivalens DSM 3772作为外缘菌株,构建系统发育树。
1.5 群体感应细菌的筛选与信号分子解析 1.5.1 群体感应细菌的筛选群体感应细菌检测的报告菌株为Agrobacterium tumefaciens KYC55(由马里兰大学海洋环境技术研究所Hill实验室惠赠)。KYC55有着对中链AHLs较敏感的特性, 当它接触到外源的AHLs时,会产生β-半乳糖苷酶,X-gal遇到β-半乳糖苷酶会被分解,且生成的产物具有蓝色。采用琼脂扩散法,将热液贻贝可培养共附生细菌用灭菌牙签接种到对应的固体平板培养基上,于25 ℃培养1~2 d,待形成菌落后,将AHLs信号分子检测培养基(见1.2)倒入长有待检测单菌落的平板,于25 ℃培养12 h,若菌落周围产生蓝色斑点则为群体感应细菌,说明该菌株能够产生AHLs信号分子。
1.5.2 TLC生物自显影方法分析群体感应细菌AHLs的种类首先提取AHLs信号分子,将筛选出的群体感应细菌接种到200 mL的液体培养基中,震荡培养(25 ℃,180 r/min)至OD600nm为0.6~0.8,离心菌液(4 ℃,8 000 r/min,10 min),取上清液,调pH范围为6.9~7.0,使用300 mL乙酸乙酯分2次萃取,将上层萃取液旋蒸浓缩至1 mL,并转移到2.5 mL气相小瓶中,用氮气吹干后溶解在200 μL的色谱纯乙酸乙酯中,放于-20 ℃冰箱保存。
将群体感应细菌的乙酸乙酯提取物在反向TLC硅胶板(TLC aluminium sheets, 20 cm×20 cm, silica Gel 60F254, Merck, USA)上每隔2 cm点样,以60%甲醇溶液层析4 h,无菌操作台上吹干后放置在含有已凝固的软琼脂(0.8%)的方形容器内,上层倒入AHLs信号分子检测培养基(见1.2),于25 ℃静置培养12 h,观察TLC板上蓝斑的数量和位置,通过计算样品蓝斑的比移值(Rf值)和标准品蓝斑的比移值判断AHLs种类。比移值(Rf值)是层析原点到蓝斑中心的距离与原点到甲醇溶液层析前沿的距离的比值。AHLs标准品种类包括N-hexanoyl-L-homoserine lactone (C6-HSL)、N-(β-ketocaproyl)-L-Homoserine lactone (OC6-HSL)、N-octanoyl-L-homoserine lactone (C8-HSL)、N-(3-Oxoctanoyl)-L-homoserine (OC8-HSL)、N-(3-Oxodecanoyl)-L-homoserine (OC10-HSL), 均购置于Cayman Chemical公司(Ann Arbor, USA)
1.5.3 GC-MS法分析群体感应细菌AHLs的种类参照文献[20-21],使用Agilent7890A/5975C型气相色谱-质谱联用仪,将群体感应细菌的乙酸乙酯提取物和AHLs标准品一起用于GC-MS检测。载气为高纯度氦,流速为1 mL/min,质谱条件设置为电子电离源70 eV、发射电流500 μA、质谱四级管150 ℃、质谱源230 ℃,质谱仪在全扫描模式下运行,色谱分析采用离子检测模式(SIM,m/z=143),并与标准品色谱峰进行对照。
1.6 群体感应细菌的功能 1.6.1 结晶紫染色法测定菌株的成膜能力参照Zhu等[22]的方法,将群体感应细菌培养至对数生长期(OD600nm=0.8左右),接种10 μL细菌培养液到装有1 mL液体培养基的玻璃试管(100 mm×750 mm)中,25 ℃,150 r/min,震荡培养24 h,弃去培养液,1×PBS溶液清洗试管,去除未被黏附的细菌,加入2 mL结晶紫溶液(质量浓度0.1%),静置30 min,对黏附在试管壁上的生物膜进行染色,弃去染色液,1×PBS溶液清洗试管,倒扣试管干燥,加入2 mL 75%酒精洗脱,洗脱液用紫外分光光度计测量OD570 nm处的吸光值,以E. coli DH5a为阴性对照,评估成膜能力,每个菌株设置3个平行。阴性对照吸光度为ODc,QS菌结晶紫染色后洗脱液的吸光度为OD,若OD < ODc表示没有成膜能力,ODc < OD < 2ODc表示具有弱成膜能力,2ODc < OD < 4ODc表示具有中等成膜能力,4ODc < OD表示具有强成膜能力。
1.6.2 菌株利用DMSP能力的测定参照文献[23-24]将群体感应细菌培养至对数生长期(OD600nm=0.8左右),取100 μL稀释的QS细菌悬液(OD600nm =0.3)加入到含有1.9 mL 5%浓度的DMSP培养基(见1.2)的顶空瓶中,锡箔纸包裹瓶身,每个菌株设置3个平行,设置加入100 μL灭菌海水为空白对照,在25 ℃以180 r/min培养12 h,在Agilent7697A型气相色谱仪中检测产生的DMS。以气相色谱检测的DMSP碱解成DMS的峰面积和DMSP物质的量的关系(见式(1))绘制成标准曲线,再通过换算菌株降解DMSP的量计算DMSP的降解率(见式(2)):
| $ \sqrt{\text { 峰面积 }}=11.75 \times \mathrm{DMSP}_{\text {降解量 }}, R^2=0.993 \text { 。} $ | (1) |
| $ \mathrm{DMSP}_{\text {降解率 }}=\mathrm{DMSP}_{\text {降解量 }} / \mathrm{DMSP}_{\text {洂加量 }} \times 100 \%。$ | (2) |
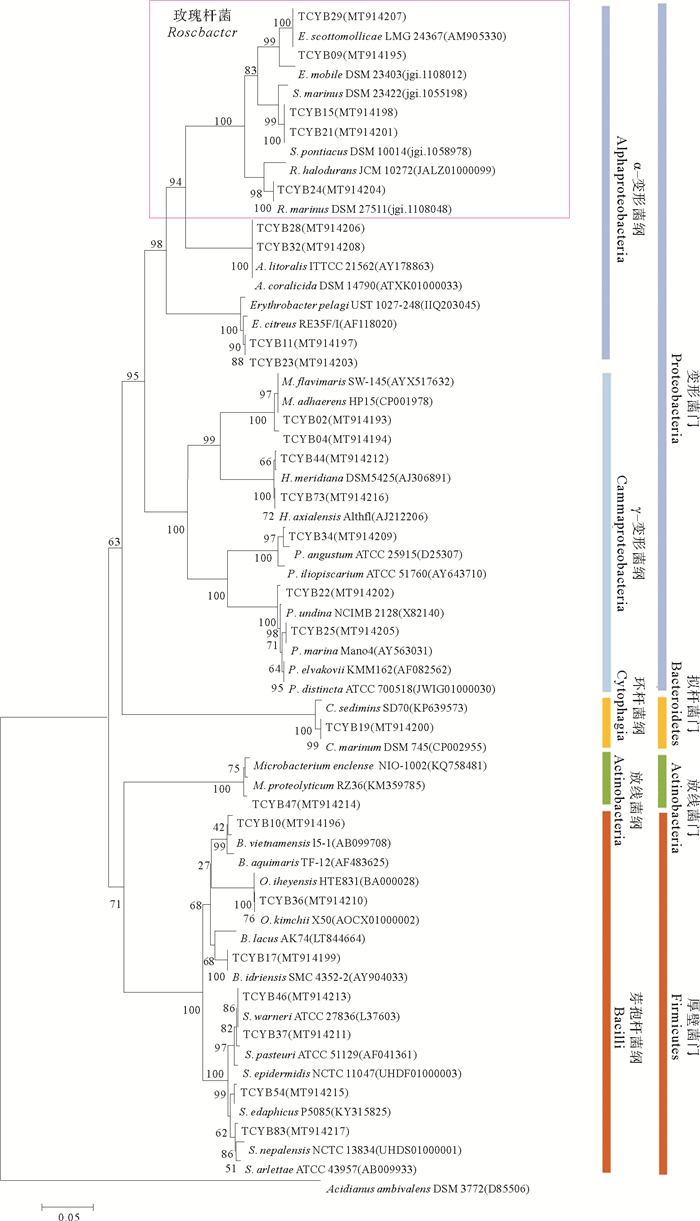
从热液贻贝中分离出可培养共附生细菌96株,根据不同菌落形态(大小、颜色、透明度等)及16S rDNA基因序列确定出25株不同菌株(见表 1),它们分属于4个门、5个纲、14个属,其中在2216E培养基上生长的有19株,在R2A培养基上生长的有6株。
|
|
表 1 天成热液区深海贻贝鳃共附生可培养细菌鉴定结果 Table 1 Identification results of culturable bacteria in the gill symbiosis of deep-sea mussels in Tiancheng hydrothermal area |
系统发育学分析如图 3所示,从门水平来看,这些菌株属于变形菌门(Proteobacteria,16株)、厚壁菌门(Firmicutes,7株)、放线菌门(Actinobacteria,1株)和拟杆菌门(Bacteroidetes,1株),分别占可培养共附生细菌的64%、28%、4%和4%,由此可见变形菌门(Proteobacteria)是优势菌群。从纲的水平来看,α-变形菌纲(Alphaproteobacteria)为优势菌群,其中有5株细菌属于玫瑰杆菌(Roseobacter)类群,占可培养共附生细菌的20%。从属水平来看,这些可培养细菌包括橙色单胞菌属(Aurantimonas)2株、小杆菌属(Epibacterium)2株、赤杆菌属(Erythrobacter)2株、玫瑰色鲜艳菌属(Roseivivax)1株、亚硫酸盐杆菌属(Sulfitobacter)2株、盐单胞菌属(Halomonas)2株、海杆菌属(Marinobacter)2株、发光杆菌属(Photobacterium)1株、假交替单胞菌属(Pseudoalteromonas)2株、微杆菌属(Marinobacter)1株、芽孢杆菌属(Bacillus)2株、大洋芽孢杆菌属(Oceanobacillus)1株、葡萄球菌属(Staphylococcus)4株和环杆菌属(Cyclobacterium)1株。由此可见热液贻贝共附生细菌有着较丰富的多样性。
|
图 3 热液贻贝可培养共附生细菌的系统发育树 Fig. 3 Phylogenetic tree of cultivate symbiotic bacteria of hydrothermal mussel |
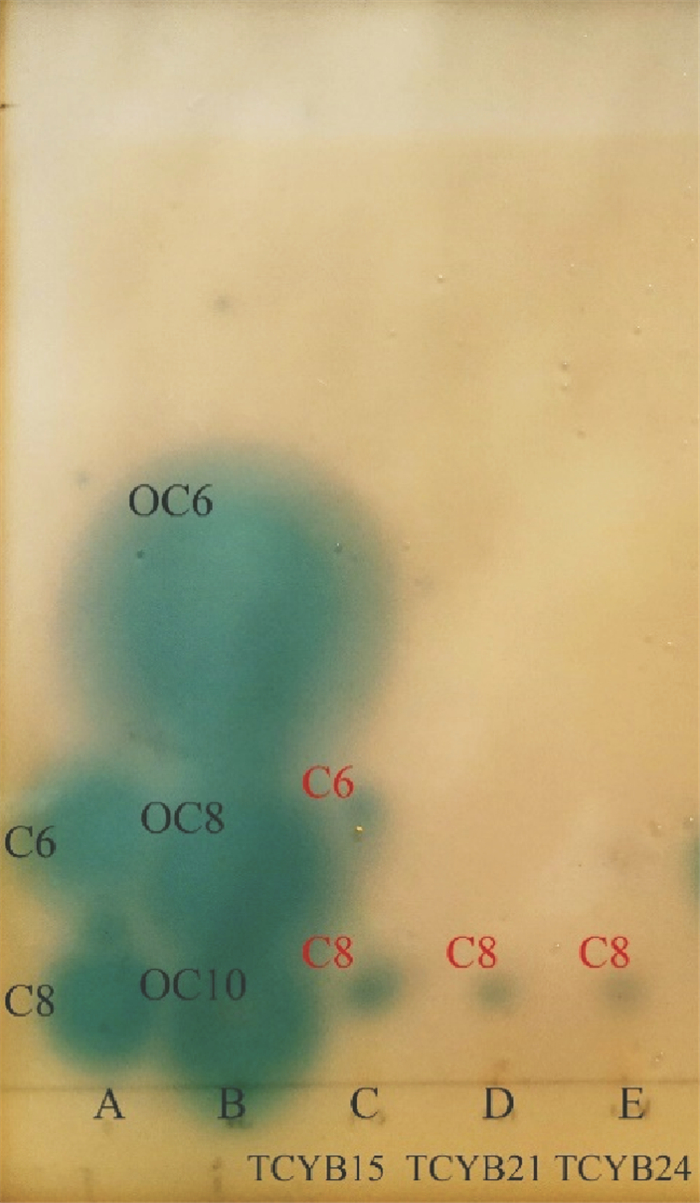
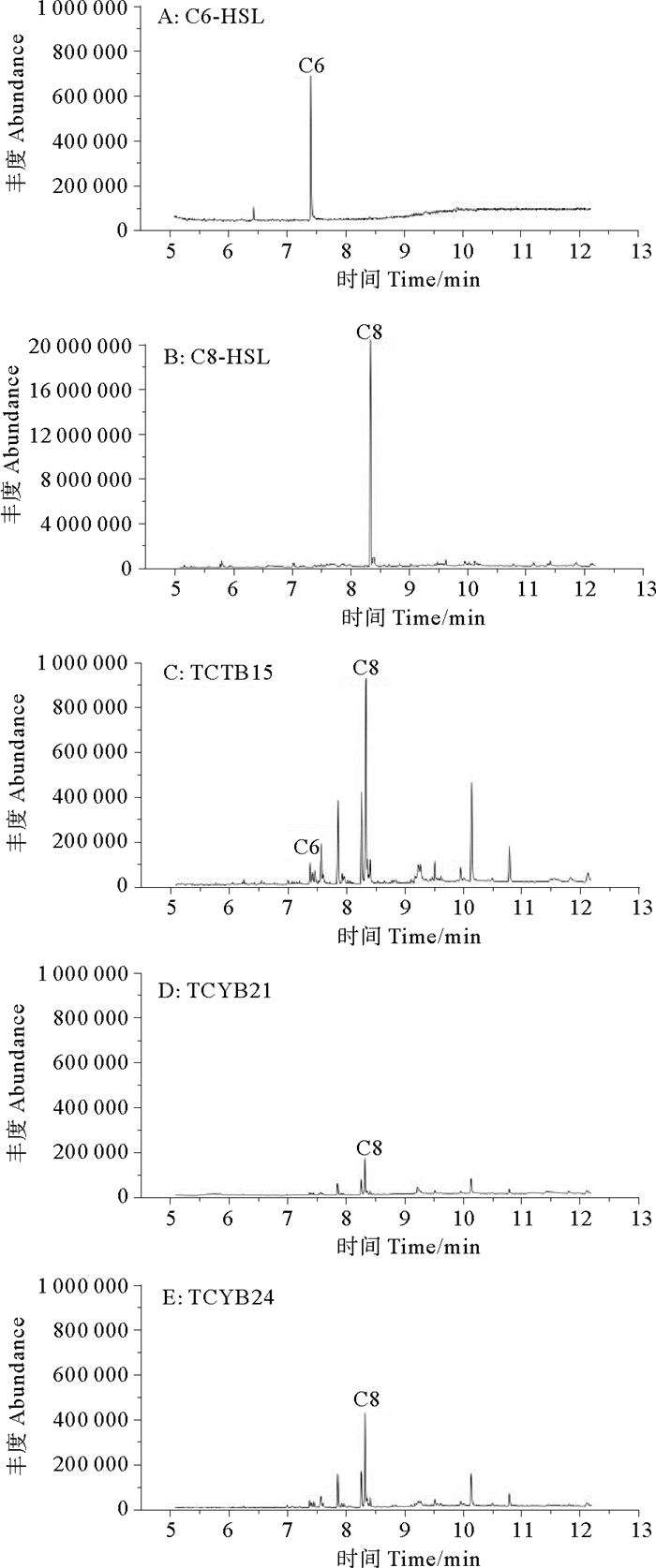
从热液贻贝共附生细菌中发现3株群体感应细菌,均生长在2216E培养基上,培养温度为25 ℃,分别是亚硫酸盐杆菌属的Sulfitobacter pontiacus TCYB15(MT914198)、S. pontiacus TCYB21(MT914201) 和海玫瑰色鲜艳菌属的Roseivivax marinus TCYB24 (MT914204),且全部属于玫瑰杆菌类群。TLC生物自显影结果表明,与标准品对比,通过计算Rf值,S. pontiacus TCYB15产生C8-HSL(Rf=0.073)和C6-HSL(Rf=0.248)2种信号分子,S. pontiacus TCYB21和R. marinus TCYB24只产生C8-HSL(Rf=0.073) 一种信号分子(见图 4)。GC-MS结果显示:TCYB15提取物中两个峰显示的保留时间分别为7.423和8.337,与标准品C6-HSL和C8-HSL一致,TCYB21和TCYB24提取物中一个峰显示的保留时间为8.357,与标准品C8-HSL一致(见图 5)。
|
(A:C系HSL混标,C6-HSL,C8-HSL,C12-HSL;B:OC系HSL混标,3-oxo-C6-HSL,3-oxo-C8-HSL,3-oxo-C10-HSL;C:菌株S. pontiacus TCYB15提取物;D:菌株S. pontiacus TCYB21提取物;E:菌株Roseivivax marinus TCYB24提取物。A: Mixture of synthetic HSL standards, C6-HSL, C8-HSL; B: Mixture of synthetic 3-oxo-HSL standards, OC6, OC8, OC10; C: Extracts of strain S. pontiacus TCYB15; D: Extracts of strain S. pontiacus TCYB21; E: Extracts of strain R. marinus TCYB24. ) 图 4 基于生物自显影方法检测群体感应(QS)菌产生AHLs的种类 Fig. 4 Profile picture of AHLs produced by Quorum Sensing(QS) bacteria based on bioautography |
|
(A:标准品,C6-HSL;B:标准品,C8-HSL;C:菌株S. pontiacus TCYB15提取物;D:菌株S. pontiacus TCYB21提取物;E:菌株R. marinus TCYB24提取物。A: HSL standard, C6-HSL; B: HSL standard, C8-HSL; C: Extracts of strain S. pontiacus TCYB15; D: Extracts of strain S. pontiacus TCYB21; E: Extracts of strain R. marinus TCYB24. ) 图 5 GC-MS方法检测AHLs信号分子种类 Fig. 5 Detection of AHLs signal molecule species by GC-MS method |
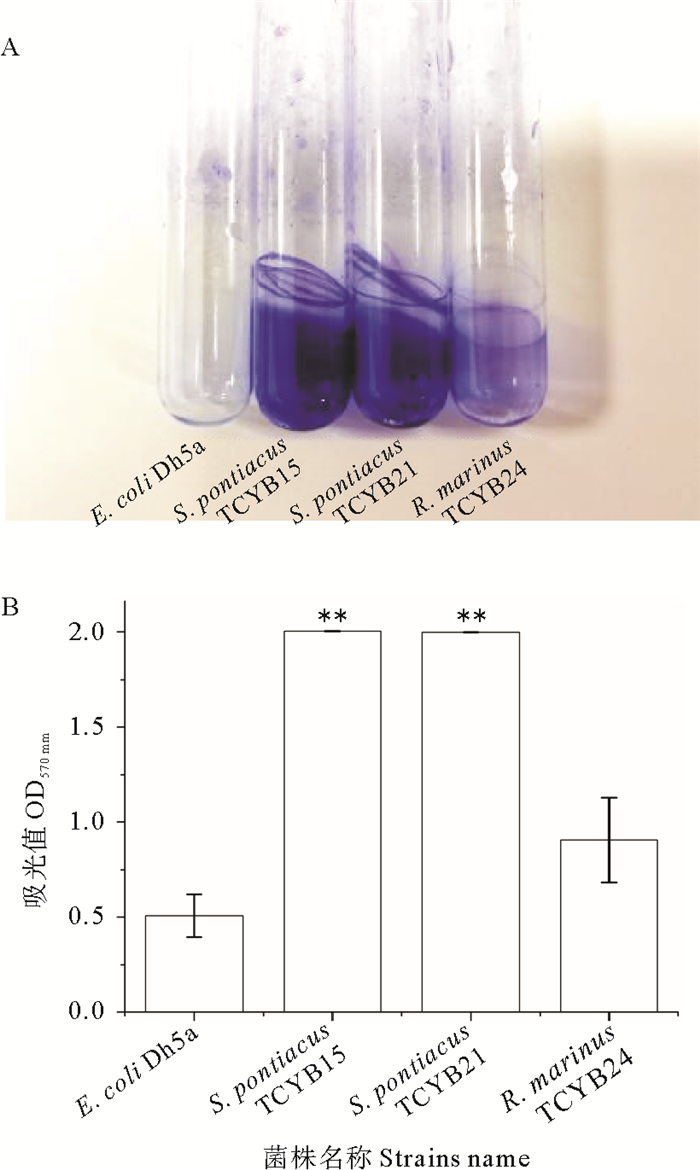
结晶紫染色结果表明,阴性对照菌株E.coli DH5a在试管壁上未留下紫色生物膜,洗脱液吸光值为0.507,菌株S. pontiacus TCYB15,S. pontiacus TCYB21和R. marinus TCYB24均在试管壁上形成紫色生物膜,洗脱液在OD570 nm处的吸光值分别为2.004,1.998和0.905,分别达到阴性对照吸光值的3.95倍,3.94倍,1.79倍,经过评估,有3株贻贝共附生群体感应细菌都具有成膜能力,其中亚硫酸盐杆菌属的3株S. pontiacus TCYB15和S. pontiacus TCYB21具有中等成膜能力,R. marinus TCYB24具有弱成膜能力(见图 6)。
|
(A:试管壁上形成的生物膜;B:570 nm处的吸光值;**:显著性差异分析中P值小于0.01,表示极显著差异。A: Absorbance at 570 nm; B: Biofilm formed on the wall of the test tube; ** : The P-value is less than 0.01 in the significant difference analysis, indicating a very significant difference. ) 图 6 细菌生物膜形成能力 Fig. 6 Bacterial biofilm formation ability |
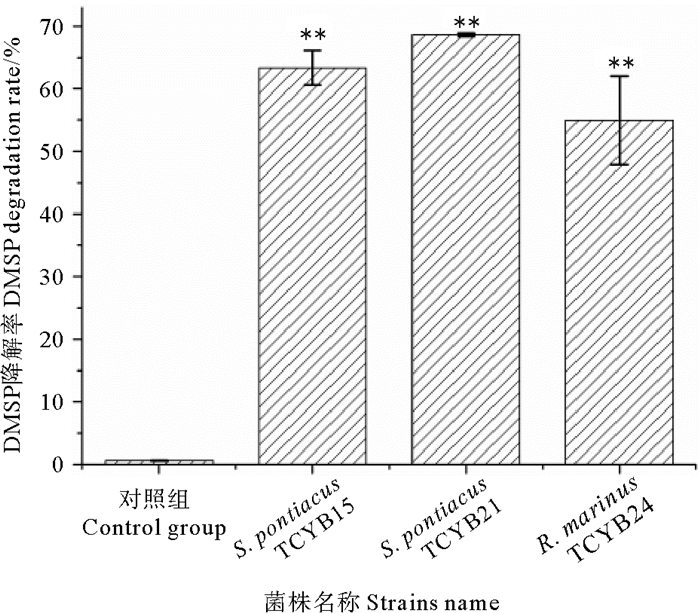
本研究以DMSP做为硫源,检测QS菌对其利用能力,结果显示,有3株QS细菌具有利用DMSP的能力。根据峰面积计算得出,空白对照组DMSP基本上没有降解,菌株S. pontiacus TCYB15的DMSP降解率为63.32%,菌株S. pontiacus TCYB21的DMSP降解率为68.63%,菌株R. marinus TCYB24的DMSP降解率为54.93%。由此可见,亚硫酸盐杆菌属的2株细菌S. pontiacus TCYB15和S. pontiacus TCYB21降解DMSP的能力较强(见图 7)。
|
(**:显著性差异分析中P值小于0.01,表示极显著差异。**: The P-value is less than 0.01 in the significant difference analysis, indicating a very significant difference. ) 图 7 QS菌的DMSP降解率 Fig. 7 DMSP degradation rate of QS bacteria |
由于生存环境的极端性,热液区的大多数微生物是难以培养的[25],然而依赖于培养的方法对于菌株生理功能的认识和评价是必要的[26],获得可培养菌株有助于了解热液区复杂的生命过程。因此,本研究对热液贻贝鳃中的共附生细菌进行了分离与鉴定,探究贻贝共附生细菌的种类和功能,并开展了热液贻贝共附生细菌中群体感应细菌的功能研究。
本研究以西南印度洋天成热液区深海偏顶蛤(Bathymodiolus marisindicus)为样品,在25 ℃温度下使用常规的2种培养基(2216E,R2A)对热液贻贝鳃中的共附生细菌进行了分离纯化,经16S rRNA基因测序和系统发育分析,得到25株不同的共附生细菌,它们由变形菌门、放线菌门、厚壁菌门和拟杆菌门组成,变形菌门数量最多,在纲的水平上,α-变形菌纲、γ-变形菌纲和芽孢杆菌纲为优势菌群。已有研究发现,从南海冷泉区分离得到的纯培养深海贻贝附生细菌分属于变形菌门、放线菌门、厚壁菌门和拟杆菌门,其中变形菌门占大部分[27]。Dick[1]在研究热液区各个生境中的主要微生物类型时发现,热液动物的内共生菌主要为γ-变形菌纲和α-变形菌纲,对海胆和珊瑚附生细菌的研究也发现γ-变形菌纲为优势菌群[28],由此可见,变形菌门是海洋无脊椎动物共附生细菌的优势门类,这些研究发现与本研究结果相一致。
多年的研究表明,海洋环境中存在一类群体感应细菌, 它们通过感应信号分子来调节群体生理现象,例如形成生物膜、产生毒素/色素/抗生素、生物发光、质粒转移和共生等[14-15]。尽管Johnson等[16]发现深海热液微生物中存在群体感应现象,但并未找到AHLs群体感应系统存在的直接证据,本研究证实深海热液微生物中确实存在群体感应(QS)细菌。从贻贝共附生细菌中筛选出了3株群体感应细菌:S. pontiacus TCYB15,S. pontiacus TCYB21和R. marinus TCYB24。Tait等[29]在研究AHLs调节石莼游动孢子的游泳行为时发现Sulfitobacter可以产生C4-HSL和C8-HSL两种信号分子,其中C8-HSL是海洋细菌中普遍存在的AHL[30],本实验中的3株群体感应细菌也均产生C8-HSL,另外在S. pontiacus TCYB15中同时检测到了C6-HSL,这说明Sulfitobacter能够产生多样化的AHLs信号分子。分子层面上,Ankrah对同一属的Sulfitobacter sp. strain CB2047基因组草图的研究发现,此菌株基因组存在酰基-高丝氨酸内酯(AHLs)合成酶基因,却没有发现AHLs结合蛋白基因,他认为单一的AHLs合成酶基因在Sulfitobacter中普遍存在[31]。Dai等[32]于2014年从深海环境中鉴定出了R. marinus这一新种,但是之后一直未见Roseivivax属的细菌可以产生AHLs的报道,本研究发现R. marinus TCYB24具有群体感应现象尚属首次报道。
在本研究中,发现这3株群体感应细菌均能形成生物膜,有学者认为生物膜的形成可以增强细菌抵抗环境压力的抗性并黏附周围海水中的营养物质,以此促进微生物的抗逆能力[33-34]。深海热液环境中物质匮乏、能量形式较为简单,且具有多种极端环境因子,所以从营养物质的获取和对不良环境的抵御方面来看,3株群体感应细菌的生物成膜特性很可能是微生物群落适应深海热液环境的主要机制之一。
海底热液反应区的海水-岩石相互作用产生了硫化氢和其他硫化物,例如多硫化物,单质硫和硫代硫酸盐等,硫化物和微生物之间的氧化还原反应在热液生态系统中建立起复杂的硫代谢网络[35]。玫瑰杆菌(Roseobacter)类群的细菌[36]在海洋硫循环中发挥着至关重要的作用,除了通过降解二甲基巯基丙酸内盐(DMSP)产生与气候调节相关的气体二甲基巯(DMS)之外[37],还可以利用其他有机硫(例如:甲基化硫、巯基甲醇和二甲基硫)作为营养物质,另外还具有代谢无机硫(例如:亚硫化物,硫化物,单质硫以及过硫化物)的能力,以降低这些物质对细菌的毒性[38-39]。深海热液环境中含有大量的硫化物,是海洋中硫元素的重要来源,这为海洋玫瑰杆菌的生存提供了丰富的硫源,值得关注的是该类群中的细菌大部分都具有群体感应现象,而本研究结果也表明,分离培养出的3株群体感应细菌均属于玫瑰杆菌类群,通过DMSP降解能力检测发现这3株群体感应细菌都能降解DMSP。林钰等[24]对马里亚纳海沟可培养细菌的研究也显示深海环境中存在大量DMSP降解菌,这说明深海环境中存在DMSP形式的硫化物,因此本文作者认为热液环境中的群体感应细菌,在与生物共附生的过程中,可以利用DMSP作为营养物质,在硫循环中发挥着重要作用。此外,DMSP是生物细胞内一种渗透压调节剂,可以起到在不利渗透压条件时保护生物的作用[40],群体感应细菌调节DMSP的吸收可能是细菌适应环境压力的保护机制之一。
本研究中筛选出的2株亚硫酸盐杆菌属的贻贝共附生群体感应细菌S. pontiacus TCYB15和S. pontiacus TCYB21,由于其来自于高温、高毒且硫化物和重金属含量都很高的深海热液环境,还具有成膜和利用DMSP的能力,所以下一步研究将对它们的基因组进行测序和功能注释,以便解析热液贻贝在特殊环境中的生存策略及与宿主和环境的相互作用关系。
| [1] |
Dick G J. The microbiomes of deep-sea hydrothermal vents: Distributed globally, shaped locally[J]. Nature Reviews Microbiology, 2019, 17(5): 271-283. DOI:10.1038/s41579-019-0160-2 (  0) 0) |
| [2] |
Yamamoto M, Takai K. Sulfur metabolisms in epsilon- and gamma-Proteobacteria in deep-sea hydrothermal fields[J]. Frontiers in Microbiology, 2011, 2: 192. (  0) 0) |
| [3] |
Sun Q l, Wang M Q, Sun L. Characteristics of the cultivable bacteria from sediments associated with two deep-sea hydrothermal vents in Okinawa trough[J]. World Journal of Microbiology & Biotechnology, 2015, 31(12): 2025-2037. (  0) 0) |
| [4] |
Karl D M, Wirsen C O, Jannasch H W. Deep-sea primary production at the galapagos hydrothermal vents[J]. Science, 1980, 207(4437): 1345-1347. DOI:10.1126/science.207.4437.1345 (  0) 0) |
| [5] |
Kvenvolden K A, Weliky K, Nelson H, et al. Sunmarine seep of carbon-dioxide in north sound, Alaska[J]. Science, 1979, 205(4412): 1264-1266. DOI:10.1126/science.205.4412.1264 (  0) 0) |
| [6] |
Jannasch H W, Mottl M J. Geomicrobiology of deep-sea hydrothermal vents[J]. Science, 1985, 229(4715): 717-725. DOI:10.1126/science.229.4715.717 (  0) 0) |
| [7] |
Petersen J M, Zielinski F U, Pape T, et al. Hydrogen is an energy source for hydrothermal vent symbioses[J]. Nature, 2011, 476(7359): 176-180. DOI:10.1038/nature10325 (  0) 0) |
| [8] |
Jeanthon C. Molecular ecology of hydrothermal vent microbial communities[J]. Antonie van Leeuwenhoek International Journal of General and Molecular Microbiology, 2000, 77(2): 117-133. DOI:10.1023/A:1002463825025 (  0) 0) |
| [9] |
Hentschel U, Schmid M, Wagner M, et al. Isolation and phylogenetic analysis of bacteria with antimicrobial activities from the Mediterranean sponges Aplysina aerophoba and Aplysina cavernicola[J]. FEMS Microbiology Ecology, 2001, 35(3): 305-312. DOI:10.1111/j.1574-6941.2001.tb00816.x (  0) 0) |
| [10] |
Montgomery K, Charlesworth J C, LeBard R, et al. Quorum sensing in extreme environments[J]. Life-Basel, 2013, 3(1): 131-148. DOI:10.3390/life3010131 (  0) 0) |
| [11] |
Williams P. Quorum sensing, communication and cross-kingdom signalling in the bacterial world[J]. Microbiology-Sgm, 2007, 153: 3923-3938. DOI:10.1099/mic.0.2007/012856-0 (  0) 0) |
| [12] |
Bassler B L. Small talk: Cell-to-cell communication in bacteria[J]. Cell, 2002, 109(4): 421-424. DOI:10.1016/S0092-8674(02)00749-3 (  0) 0) |
| [13] |
Rivas M, Seeger M, Holmes D S, et al. A lux-like quorum sensing system in the extreme acidophile Acidithiobacillus ferrooxidans[J]. Biological Research, 2005, 38(2-3): 283-297. (  0) 0) |
| [14] |
Vuong C, Gerke C, Somerville G A, et al. Quorum-sensing control of biofilm factors in Staphylococcus epidermidis[J]. Journal of Infectious Diseases, 2003, 188(5): 706-718. DOI:10.1086/377239 (  0) 0) |
| [15] |
Kannan R E, Saini S. Mathematical Modelling of Quorum Sensing in Bacteria[J]. INAE Letters, 2018, 3(3): 175-187. DOI:10.1007/s41403-018-0047-y (  0) 0) |
| [16] |
Johnson M R, Montero C I, Conners S B, et al. Population density-dependent regulation of exopolysaccharide formation in the hyper thermophilic bacterium Thermotoga maritima[J]. Molecular Microbiology, 2005, 55(3): 664-674. (  0) 0) |
| [17] |
Perez-Rodriguez I, Bolognini M, Ricci J, et al. From deep-sea volcanoes to human pathogens: A conserved quorum-sensing signal in Epsilonproteobacteria[J]. The ISME Journal, 2015, 9(5): 1222-1234. DOI:10.1038/ismej.2014.214 (  0) 0) |
| [18] |
Tempe J, Petit A, Holsters M, et al. Thermosensitive step association with transfer of Tl plasmid during conjugation-possible relation to transformation in grown gall[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(7): 2848-2849. DOI:10.1073/pnas.74.7.2848 (  0) 0) |
| [19] |
Johnson W M, Soule M C K, Kujawinski E B. Evidence for quorum sensing and differential metabolite production by a marine bacterium in response to DMSP[J]. The ISME Journal, 2016, 10(9): 2304-2316. DOI:10.1038/ismej.2016.6 (  0) 0) |
| [20] |
迟雯丹. 海洋细菌Ponticoccus sp. PD-2群体感应系统及对抑藻功能的调控探究[D]. 青岛: 国家海洋局第一海洋研究所, 2017. Chi W D. Study on the Quorum Sensing System and the Algicidal Function of Marine Bacterium Ponticoccus sp. PD-2[D]. Qingdao: First Institute of Oceanography, Ministry of Natural Resources, 2017. (  0) 0) |
| [21] |
Cataldi T R I, Blanco G, Palazzo L, et al. Occurrence of N-acyl-L-homoserine lactones in extracts of some Gram-negative bacteria evaluated by gas chromatography-mass spectrometry[J]. Analytical Biochemistry, 2007, 361(2): 226-235. DOI:10.1016/j.ab.2006.11.037 (  0) 0) |
| [22] |
Zhu J, Chai Y R, Zhong Z T, et al. Agrobacterium bioassay strain for ultrasensitive detection of N-acyl homoserine lactone-type quorum-sensing molecules: Detection of autoinducers in Mesorhizobium huakuii[J]. Applied and Environmental Microbiology, 2003, 69(11): 6949-6953. DOI:10.1128/AEM.69.11.6949-6953.2003 (  0) 0) |
| [23] |
Williams B T, Cowles K, Martinez A B, et al. Bacteria are important dimethyl sulfoniopropionate producers in coastal sediments[J]. Nature Microbiology, 2019, 4(11): 1815-1825. DOI:10.1038/s41564-019-0527-1 (  0) 0) |
| [24] |
林钰, 刘荣华, 周顺, 等. 马里亚纳海沟沉积物可培养异养细菌的多样性及其DMSP降解能力[J]. 微生物学报, 2021, 61(4): 828-844. Lin Y, Liu R H, Zhou S, et al. Diversity of culturable heterotrophic bacteria from sediments of the Mariana Trench and their ability to degrade dimethylsulfoniopropionate (DMSP)[J]. Acta Microbiologica Sinica, 2021, 61(4): 828-844. (  0) 0) |
| [25] |
Inagaki F, Kuypers M M M, Tsunogai U, et al. Microbial community in a sediment-hosted CO2 lake of the southern Okinawa Trough hydrothermal system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(38): 14164-14169. DOI:10.1073/pnas.0606083103 (  0) 0) |
| [26] |
Thornburg C C, Zabriskie T M, Mcphail K L. Deep-sea hydrothermal vents: Potential hot spots for natural products discovery?[J]. Journal of Natural Products, 2010, 73(3): 489-499. DOI:10.1021/np900662k (  0) 0) |
| [27] |
郭文捷, 赵瑾, 齐宏涛, 等. 南海冷泉区贻贝(Bathymodiolus platifrons)附生菌的分离培养与多样性分析[J]. 海洋科学, 2017, 41(2): 89-95. Guo W J, Zhao J, Qi H T, et al. Isolation and phylogenetic analysis of epiphytic bacteria from mussels collected from the cold seep[J]. Marine Sciences, 2017, 41(2): 89-95. (  0) 0) |
| [28] |
Lampert Y, Kelman D, Dubinsky Z, et al. Diversity of culturable bacteria in the mucus of the Red Sea coral Fungia scutaria[J]. Fems Microbiology Ecology, 2006, 58(1): 99-108. DOI:10.1111/j.1574-6941.2006.00136.x (  0) 0) |
| [29] |
Tait K, Williamson H, Atkinson S, et al. Turnover of quorum sensing signal molecules modulates cross-kingdom signalling[J]. Environmental Microbiology, 2009, 11(7): 1792-1802. DOI:10.1111/j.1462-2920.2009.01904.x (  0) 0) |
| [30] |
徐鲁燕, 郑立, 韩笑天, 等. 具有群体感应系统微藻共栖细菌的筛选及抑藻活性的研究[J]. 海洋与湖沼, 2012, 43(6): 1149-1155. Xu L Y, Zheng L, Han X T, et al. Screening of microalgae associated bacteria with quorum sensing system and thier algicidal activity[J]. Oceanologia et Limnologia Sinica, 2012, 43(6): 1149-1155. (  0) 0) |
| [31] |
Ankrah N Y D, Lane T, Budinoff C R, et al. Draft genome sequence of Sulfitobacter sp. CB2047, a member of the Roseobacter clade of marine bacteria, Isolated from an Emiliania huxleyi Bloom[J]. Genome announcements, 2014, 2(6): e01125-14. (  0) 0) |
| [32] |
Dai X, Shi X, Gao X, et al. Roseivivax marinus sp nov., isolated from deep water[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64: 2540-2544. DOI:10.1099/ijs.0.062760-0 (  0) 0) |
| [33] |
Zhang W P, Ding W, Li Y X, et al. Marine biofilms constitute a bank of hidden microbial diversity and functional potential[J]. Nature Communications, 2019, 10: 517. DOI:10.1038/s41467-019-08463-z (  0) 0) |
| [34] |
Flemming H C, Wingender J. The biofilm matrix[J]. Nature Reviews Microbiology, 2010, 8(9): 623-633. DOI:10.1038/nrmicro2415 (  0) 0) |
| [35] |
McCollom T M, Shock E L. Geochemical constraints on chemolithoautotrophic metabolism by microorganisms in seafloor hydrothermal systems[J]. Geochimica et Cosmochimica Acta, 1997, 61(20): 4375-4391. DOI:10.1016/S0016-7037(97)00241-X (  0) 0) |
| [36] |
陈正浩, 张永雨, 杨素萍. 海洋玫瑰杆菌类群研究进展[J]. 生态学报, 2015, 35(5): 1620-1629. Chen Z H, Zhang Y Y, Yang S P. Research progresses of marine Roseobacter lineage[J]. Acta Ecologica Sinica, 2015, 35(5): 1620-1629. (  0) 0) |
| [37] |
Dickschat J S, Zell C, Brock N L. Pathways and substrate specificity of DMSP catabolism in marine bacteria of the Roseobacter clade[J]. Chembiochem, 2010, 11(3): 417-425. DOI:10.1002/cbic.200900668 (  0) 0) |
| [38] |
Wagner-Doebler I, Biebl H. Environmental biology of the marine Roseobacter lineage[J]. Annual Review of Microbiology, 2006, 60: 255-280. DOI:10.1146/annurev.micro.60.080805.142115 (  0) 0) |
| [39] |
Buchan A, Gonzalez J M, Moran M A. Overview of the marine Roseobacter lineage[J]. Applied and Environmental Microbiology, 2005, 71(10): 5665-5677. DOI:10.1128/AEM.71.10.5665-5677.2005 (  0) 0) |
| [40] |
王艳, 齐雨藻, 沈萍萍, 等. 温度和盐度对球形棕囊藻细胞DMSP产量的影响[J]. 水生生物学报, 2003, 27(4): 367-371. Wang Y, Qi Y Z, Shen P P, et al. Effects of temperature and salinity on DMSP production in Phaeocystis globosa[J]. Acta Hydrobiologica Sinica, 2003, 27(4): 367-371. DOI:10.3321/j.issn:1000-3207.2003.04.007 (  0) 0) |
2. Laboratory for Marine Ecology and Environmental Science, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
 2022, Vol. 52
2022, Vol. 52


