SOX(SRY-related HMG-box)家族是一类具有HMG-box结构域的转录因子,主要参与脊椎动物的性别决定、组织器官形成、神经系统发生等生物学过程的调控[1-3]。该家族包含30多个成员,分别属于10个亚家族(A~J)[4],其中SOXB亚家族由一些具有Group B homology基序的成员组成,根据氨基酸序列特征及功能该亚族又可分为SOXB1亚族和SOXB2亚族。脊椎动物的SOXB1亚家族包含SOX1、SOX2、SOX3和SOX19,SOXB2亚家族包含SOX14和SOX21[5]。SOXB亚族成员主要在动物的神经系统和消化道中表达。Avilin等报道SOX2在小鼠受精后8.5 d胚胎的头褶、神经外胚层及神经管中表达,之后在脑、感觉基板和肠内胚层表达[6]; 在非洲爪蟾中,SOX2表达于中枢神经系统[7]; SOX14在鸡的后脑、内耳及视觉盖中表达[8]。在无脊椎动物中,soxb亚族基因主要在神经元细胞、腹神经索、感觉器官[9-14]及消化道(非洲海参Holothuria glaberrima的肠道[15]及紫色球海胆Strongylocentrotus purpuratus的前肠[14])中表达; 在果蝇(Drosophila melanogaster)中Dichaete(隶属于SOXB2亚族)的敲降会影响成神经细胞的分化并造成中枢神经系统的部分缺失[16-17]。
单环刺螠(Urechis unicinctus)隶属螠虫动物门(Echiuroidea),主要分布于俄罗斯、朝鲜半岛、我国的黄渤海和日本北海道、本洲岛等海区。消化道是动物进行食物消化和吸收的场所,对消化道发生、发育等的研究将有助于人们对动物消化道结构和消化生理等的认识[18-20]。目前,人们对单环刺螠消化道发生、成体消化道的组织学结构以及幼虫中的消化酶特性等已有了一些认识[21-22]。本实验前期获得的单环刺螠幼虫转录组数据显示,在消化道发生和发育的不同时期幼虫中SOXB亚族成员soxb1和soxb2的表达显著差异。为了揭示它们在单环刺螠消化道形成过程中的表达特征,本研究采用RACE技术克隆了二者的全长cDNA序列,并通过原位杂交技术确定其在胚胎及幼虫中的细胞学定位,旨在为探索该亚族基因在螠虫动物消化系统中的功能研究提供基础数据。
1 材料与方法 1.1 实验动物成体单环刺螠购自青岛市四方路海产品市场,产地为莱州海区。选择生殖期的雌、雄个体(体重为(28.7±8.6) g),解剖肾管以获取成熟的两性配子,体外人工授精(精卵比为10:1)后,将受精卵置于盛有50 L过滤海水的培养箱(50 cm×40 cm×40 cm)中进行孵化和培育。孵化期间水温为16.9 ℃,pH为7.8±0.06,盐度为29±1,微弱充气。受精后24 h,早期担轮幼虫孵出; 受精后36 h开始投喂小球藻(Chlorellavulgaris)及牟氏角毛藻(Cheatoceros muelleri),每天早晚各投喂1次并随着幼虫发育逐渐增加投饵量; 发育为体节幼虫后转入含泥沙底质的培养箱中,底质厚约6 cm。幼虫培养期间,水温17~20 ℃,每天换水2次,每次换水量不超过原体积的1/2。培养期间,定期于显微镜下观察胚胎和幼虫的发育,并分别取受精卵、二细胞、四细胞、八细胞、囊胚、原肠胚、担轮幼虫(早期、中期和晚期)、体节幼虫、蠕虫状幼虫等样本,于4%多聚甲醛中固定16 h(4 ℃),梯度甲醇脱水后,保存于无水甲醇中(-20 ℃),用于整体原位杂交。此外,取部分晚期担轮幼虫,于液氮中速冻后保存于-80 ℃冰箱,用于提取总RNA。
1.2 总RNA的提取和cDNA第一链的合成取上述冻存的单环刺螠晚期担轮幼虫样本,采用异硫氰酸胍法[23]提取幼虫总RNA,使用RNase-free的DNase I消除基因组DNA,使用紫外光分光光度计和1.2 %的琼脂糖凝胶电泳检测总RNA浓度和质量。使用SMARTer[TM]- RACE kit(Clontech,美国)并按照说明书合成RACE-ready cDNA。
1.3 靶基因全长cDNA克隆及序列分析从单环刺螠幼虫转录组中获得soxb1及soxb2 cDNA片段序列并设计RACE特异引物(见表 1),以单环刺螠晚期担轮幼虫的cDNA为模板,用SMARTerTM-RACE kit分别进行3’和5’扩增,扩增产物经琼脂糖凝胶电泳检测、胶回收、连接pMD19-T载体、转化大肠杆菌DH5α后,挑取单菌落进行PCR验证后送往华大基因测序。使用SeqMan软件分别将得到的soxb1及soxb2的3’ RACE和5’RACE序列进行拼接得到全长cDNA序列。通过NCBI在线BLAST程序(http://blast.ncbi.nlm.nih.gov/)进行同源性分析,分析目的蛋白序列与其他物种蛋白序列的一致性。使用MEGA7.0软件进行多序列比对并采用最大似然法(Maximum Likelihood,ML)构建进化树,设置Bootstrap为1 000次重复检测其置信度。
|
|
表 1 RACE引物序列 Table 1 Sequences of RACE primers |
根据Uu-soxb1及Uu-soxb2全长cDNA序列设计探针合成引物(见表 2),PCR扩增分别获得长度为424 bp(Uu-soxb1)和545 bp(Uu-soxb2)的序列。以该序列为模板,使用DIG RNA Labeling kit(SP6 /T7)并按照操作指南合成地高辛标记的正义和反义RNA探针。
|
|
表 2 原位杂交探针引物序列 Table 2 primer sequence of probes for in situ hybridization |
取无水甲醇中保存的各期胚胎和幼虫样品,梯度甲醇复水; 蛋白酶K(100 ng/mL)37 ℃下消化20 min(胚胎及担轮幼虫)或者30 min(体节幼虫及蠕虫状幼虫),其他步骤参考Fabioux等[24]的方法并使用地高辛RNA探针和DIG Nucleic Acid Detectionkit(Roche)进行整体原位杂交,于Nikon E80i显微镜下观察和拍照。此外,取4 mm蠕虫状幼虫按照常规石蜡切片方法,制备厚度为4 μm的组织切片并粘贴到涂有0.1%多聚赖氨酸的载玻片上,按照冯政夫等[25]的方法进行切片原位杂交,蛋白酶K(浓度为50 ng/mL)作用时间为15 min。
2 结果 2.1 靶基因全长cDNA序列的特征使用SeqMan软件,将RACE扩增获得的Uu-soxb1和Uu-soxb2的3’和5’cDNA序列片段拼接得到靶基因的全长cDNA序列。Uu-soxb1 cDNA全长1 871bp(GenBank acc. no.MG921310),开放阅读框(Open reading frame,ORF)长度为1 110 bp,编码369个氨基酸,5’非翻译区(Untranslated region,UTR)为114 bp,3’UTR为647 bp; Uu-soxb2 cDNA序列全长2 906 bp(GenBank acc. no.MG921311),ORF长度为897 bp,编码298个氨基酸,5’UTR为319 bp,3’UTR为1 690 bp。
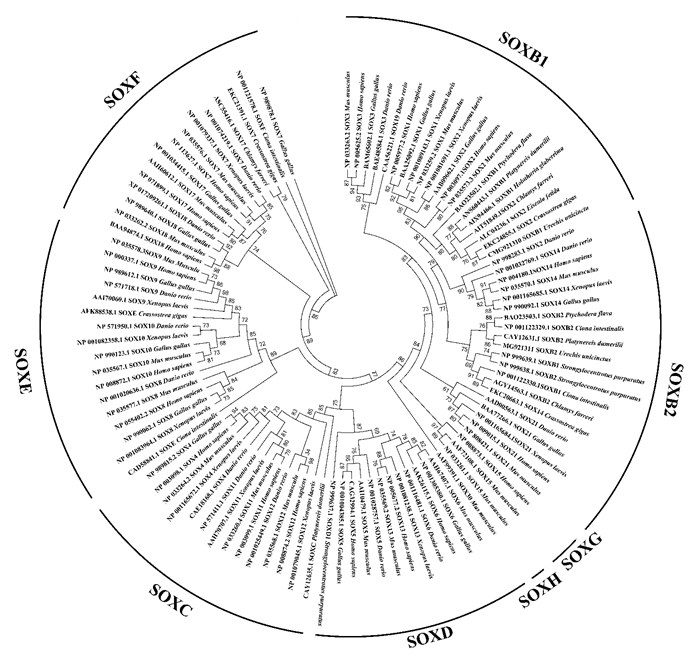
同源性分析表明,Uu-SOXB1蛋白与合浦珠母贝(Pinctada fucata)SOX2、沙蚕(Platynereis dumerilii)SOXB1的一致性分别为66%和59%。Uu-SOXB2与紫色球海胆和红色球海胆(Pseudocentrotus depressue)的SOXB2一致性均为69%,与褐家鼠(Rattus norvegicus)、大弹涂鱼(Boleophthalmus pectinirostris)和人(Homo sapiens)的SOX14一致性均为54%。多序列比对结果显示,本实验获得的两个单环刺螠预测氨基酸序列均具有SOX家族的HMG-box保守区和B亚族特有的同源区Group B homology; Uu-SOXB2具有脊椎动物SOX21蛋白羧基端特有的Poly-Ala区域,但单环刺螠两个SOXB亚族成员中均未见哺乳动物所特有的Poly-Gly、PRD repeat和Ser rich区域(见图 1)。系统进化分析发现,Uu-SOXB1首先与软体动物长牡蛎(Crassostrea gigas)及环节动物爱胜蚓(Eisenia fetida)的SOX2聚类,其次与半索动物聚类,最后与脊椎动物聚类; Uu-SOXB2首先与沙蚕聚类,然后与半索动物以及脊椎动物聚类(见图 2)。

|
(Ⅰ.HMG-box结构域; Ⅱ.Group B homology基序; Ⅲ.脊椎动物SOXB特有的Poly-Ala区域; Ⅳ.哺乳动物SOXB1特有的Poly-Gly区域; Ⅴ.哺乳动物SOX1特有的富含脯氨酸和组氨酸的PRD-repeat基序; Ⅵ.脊椎动物SOX2特有的Ser-rich区域; Ⅶ.脊椎动物SOX3特有的Pro-rich区域。Ⅰ. HMG-box; Ⅱ. Group B homology motif; Ⅲ. Poly-Ala region of vertebrate SOXB; Ⅳ. Poly-Gly region of mammal SOXB1; Ⅴ. PRD-repeat (rich of Pro and His) motif of mammal; Ⅵ. Ser-rich region of vertebrate SOX2; Ⅶ. Pro-rich region of vertebrate SOX3. ) 图 1 不同物种SOXB蛋白的多序列比对 Fig. 1 Multiple sequence alignment of the SOXBs from different species |
|
图 2 不同物种SOXB蛋白的系统进化分析 Fig. 2 Phylogenetic analysis among SOX proteins from different species |
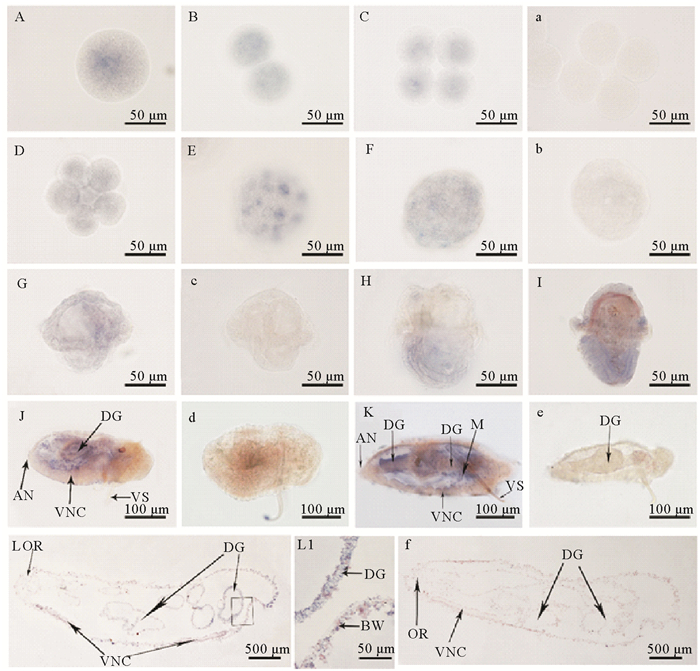
整体原位杂交结果显示,单环刺螠soxb1 mRNA阳性信号在受精卵、二细胞、四细胞、囊胚及原肠胚中均呈弥散状分布(见图 3 A~F)。至早期担轮幼虫时,Uu-soxb1 mRNA的阳性信号仍分散于整个虫体中,但幼虫上半球的信号强度似乎稍强于下半球(见图 3 G); 中期和晚期担轮幼虫中,阳性信号主要位于幼虫的体后部(口后纤毛环之后)(见图 3 H~I)。但至体节幼虫和蠕虫状幼虫(400 μm)时,明显的阳性信号转移至体壁及消化道处(见图 3 J~K)。当发育至4 mm蠕虫状幼虫时,阳性信号主要位于后部肠道及体壁处(见图 3L)。
|
(A~L: Uu-soxb1 mRNA反义探针的原位杂交,蓝色为阳性信号; a~f:Uu-soxb1 mRNA正义探针的原位杂交。A:受精卵; B:二细胞; C:四细胞; D:八细胞; E:囊胚; F:原肠胚; G:早期担轮幼虫; H:中期担轮幼虫; I:晚期担轮幼虫; J:体节幼虫; K:蠕虫状幼虫(400 μm); L:蠕虫状幼虫(4 mm)正中纵切; L1:L图的局部放大,示体壁和肠道细胞. AN:肛门; BW:体壁; DG:消化道; M:体壁乳突; OR:口部; VNC:腹神经索; VS:腹棘。A~L: in situ hybridization with anti-sense probe, the positive signals are in blue; a~f: negative controls are indicated with Uu-soxb1 mRNA sense probe. A: fertilized egg; B: 2-cells; C: 4-cells; D: 8-cells; E: blastula; F: gastrula; G: early-trochophore; H: mid-trochophore; I: late-trochophore; J: somite larva; K: worm-like larva (400 μm); L: median longitudinal section of worm-like larva at 4 mm; L1: amplification of the box in figure L, showing the cells of body wall and digestive gut. AN: anus; BW: body wall; DG:digestive gut; M: mastoid of body wall; OR: oral; VNC: ventral nerve cord; VS: ventral seta.) ) 图 3 Uu-soxb1 mRNA在单环刺螠胚胎和幼虫中的定位 Fig. 3 Localization of Uu-soxb1 mRNA in U. unicinctus embryos and larvae demonstrated by in situ hybridization |
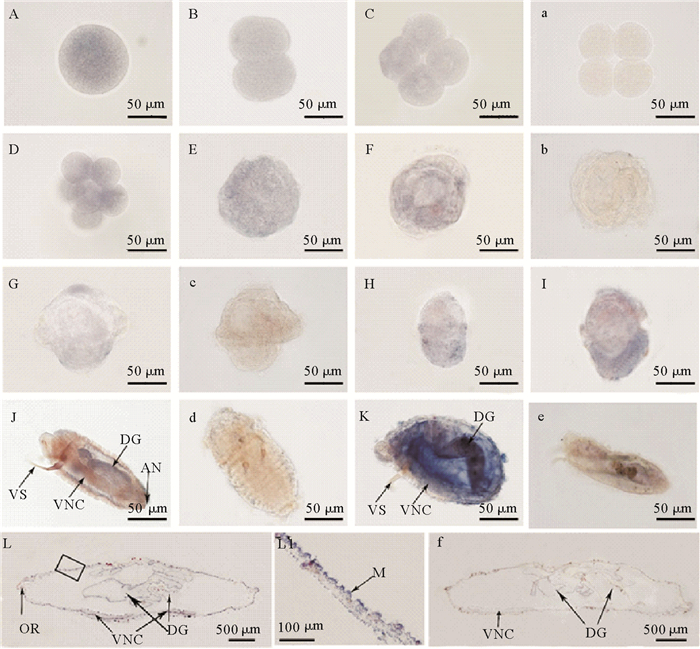
Uu-soxb2 mRNA原位杂交结果显示,阳性信号在受精卵、卵裂期胚胎、囊胚及原肠胚中均为弥散性分布(见图 4 A~F)。至早期担轮幼虫,阳性信号主要位于顶纤毛束的基部及幼虫体后部(口后纤毛环之后); 中期担轮幼虫中,阳性信号主要位于口前纤毛环及虫体的下半球; 晚期担轮幼虫时,阳性信号主要位于下半球(见图 4 G~I)。体节幼虫中的阳性信号集中在消化道和体壁内侧(见图 4 J); 蠕虫状幼虫中的靶基因mRNA则主要位于消化道、体壁及腹神经索(见图 4 K)。当发育至4 mm蠕虫状幼虫时,阳性信号主要位于消化道和体壁中(见图 4 L)。
|
(A~L: Uu-soxb2 mRNA反义探针的原位杂交,蓝色为的阳性信号; a~f:Uu-soxb2 mRNA正义探针的原位杂交. A:受精卵; B:二细胞; C:四细胞; D:八细胞; E:囊胚; F:原肠胚; G:早期担轮幼虫; H:中期担轮幼虫; I:晚期担轮幼虫; J:体节幼虫; K:蠕虫状幼虫(400 μm); L:蠕虫状幼虫(4 mm)正中纵切; L1:L图的局部放大,示体壁细胞。AN:肛门; DG:消化道; M:体壁乳突; OR:口部; VNC:腹神经索; VS:腹棘。A~L: in situ hybridization with anti-sense probe, the positive signals are in blue; a~f: negative controls are indicated with soxb2 mRNA sense probe. A: fertilized egg; B: 2-cell; C: 4-cell; D: 8-cell; E: blastula; F: gastrula; G: early-trochophore; H: mid-trochophore; I: late-trochophore; J: somite larva; K: worm-like larva (400 μm); L: median longitudinal section of juvenile at 4 mm; L1: amplification of the box in figure L. AN: anus; DG: digestive gut; M: mastoid of body wall; OR: oral; VNC: ventral nerve cord; VS: ventral seta. ) 图 4 Uu-soxb2 mRNA在单环刺螠胚胎和幼虫中的定位 Fig. 4 Localization of Uu-soxb2mRNA in U. unicinctus embryos and larvae demonstrated by in situ hybridization |
本研究获得了单环刺螠两个cDNA全长序列。分析两个预测的氨基酸序列,发现其与其他物种的SOXB蛋白具有高度的同源性,二者均具有HMG-box结构域和SOXB亚族蛋白特有的Group B homology基序,且与其他物种的HMG-box氨基酸序列一致性高达97%、Group B homology一致性高达87%以上。聚类分析结果显示,两个靶序列分别与其他物种的SOXB1和SOXB2聚类。一些学者认为,两侧对称动物中的SOXB1和SOXB2可以通过HMG-box序列第2位和第78位氨基酸残基的种类作为区分特征,其中SOXB1第2位是精氨酸(R)、第78位是苏氨酸(T); 而SOXB2则是第2位的组氨酸(H)和第78位的脯氨酸(P)[5, 26]。比较本研究获得的两个序列,我们发现一个SOXB序列符合SOXB1的HMG-box氨基酸特征,另一个符合SOXB2的序列特征,由此认为,本研究获得的两个全长cDNA序列分别是单环刺螠soxb1和soxb2,定名为Uu-soxb1和Uu-soxb2。
对NCBI数据库中已报道的SOXB进行多序列比对,发现SOXB特异的氨基酸序列在无脊椎动物和脊椎动物之间存在差异,脊椎动物SOXB亚族所具备的特有序列在多数无脊椎动物中是缺失的。在脊椎动物中,SOXB1除了含有SOXB亚族的Group B homology基序外,不同的SOXB1成员还含有各自独特的序列特征,例如:SOX1具有2个Poly-glycine、4个Poly-alanine、1个PRD repeat基序(富含组氨酸和脯氨酸); SOX2具有1个Poly-glycine、1个Ser-rich区; SOX3具有3个Poly-glycine、1个Pro-rich区; SOX19不具有特殊氨基酸序列。在SOXB2亚家族成员中,SOXB21具有3个Poly-alanine区,SOX14不具有特殊序列[4]。然而,在当前已报道的无脊椎动物SOXB氨基酸序列中,上述特有序列几乎都是缺失的,仅在沙蚕的SOXB2、紫海胆的SOX21、栉孔扇贝(Chlamys farreri)的SOXB2和仿刺参的SOX21氨基酸序列中具有脊椎动物SOX21特有的Poly-alanine区域[4]。本研究结果显示,单环刺螠SOXB1也不包含任何脊椎动物SOXB1亚家族成员所具有的特殊序列; SOXB2与沙蚕等的SOXB2特征一致,具有脊椎动物SOX21特有的Poly-alanine。由此,我们认为单环刺螠SOXB氨基酸序列与已报道的无脊椎动物序列特征基本一致。
Soxb在动物胚胎和幼虫中的表达特征已在几种无脊椎动物中报道,总体而言,它们的表达特征既有相同又存在一定的差异。首先,soxb是否是母源性表达存在物种间差异。本研究发现,Uu-soxb1和Uu-soxb2均在受精卵及早期胚胎中表达,显示其为母源性表达特征,与小头虫(Capitella teleta)的soxb1、soxb及紫色球海胆的soxb1表达特征一致[27-28],但与密西西比水母(Mnemiopsis leidyi)的sox1及栉水母(Pleurobrachia pileus)的sox3不同,后者最早在原肠胚的神经外胚层中检测到阳性信号[13, 29]; 此外,欧洲帽贝(Patella vulgata)的soxb mRNA最早在八细胞中检测到[30]。由此可见,soxb在动物早期胚胎中的起始表达时间存在多样性。
其次,soxb在动物幼虫发育过程中的表达图式存在差异。本研究我们揭示Uu-soxb1及Uu-soxb2 mRNA在单环刺螠幼虫发育过程中,其阳性信号多呈局域性分布,并在最后一个幼虫阶段——蠕虫状幼虫中定位于消化道和体壁中。这种由广布到区域性分布的表达图式在小头虫、紫色球海胆、栉孔扇贝及栉水母中亦有所报道[13, 27-28, 31]。本研究发现,在担轮幼虫中两基因的表达定位于不同的区域,Uu-soxb1 mRNA主要集中于幼虫的上半球,之后的幼虫时期该区域表达减弱,这与小头虫soxb1及紫色球海胆soxb1的表达特征类似,幼虫时期目的基因的表达亦主要集中在虫体的上半球[14, 27]; 而Uu-soxb2 mRNA则集中于顶纤毛环基部及后口纤毛环下部,Jager等对栉水母幼虫的研究中亦发现栉水母sox3在前感觉器官、顶器中检测到的表达[13],这一不同可能是由于形态学结构不同导致的。由此可见,soxb在无脊椎动物的幼虫发育过程中多呈现局域性分布的特征。
在担轮幼虫之后的发育中Uu-soxb1及Uu-soxb2 mRNA表现相似的表达特征,在体节幼虫及蠕虫状幼虫中,均主要集中于幼虫消化道及体壁的乳突处。已有研究显示,soxb1在非洲海参的肠道、紫色球海胆的前肠、文昌鱼的后肠中均有表达,soxb2的同源基因Dichaete也在果蝇后肠中检测到表达信号[14-15, 32-33],提示soxb亚族基因在动物消化道发育中的功能是保守的。然而,它们在消化道组织细胞中的表达存在时空差异性,在果蝇、海胆、海参及文昌鱼中soxb亚族基因mRNA表达于孵化前个体的消化道中,而在单环刺螠中Uu-soxb1及Uu-soxb2则在发育时期较晚的体节幼虫肠道中检测到阳性信号,Uu-soxb1在蠕虫状幼虫中仅局限于后部肠道。我们认为soxb亚族基因在不同物种之间的时空表达差异可能与物种各自的发育时空特异性相关,但尚需更多的研究数据证明。
| [1] |
Wegner M. From head to toes: The multiple facets of Sox proteins[J]. Nucleic Acids Research, 1999, 27(6): 1409-1420. DOI:10.1093/nar/27.6.1409
(  0) 0) |
| [2] |
Kamachi Y, Uchikawa M, Kondoh H. Pairing SOX off with partners in the regulation of embryonic development[J]. Trends in Genetics Tig, 2000, 16(4): 182-187. DOI:10.1016/S0168-9525(99)01955-1
(  0) 0) |
| [3] |
Schepers G E, Teasdale R D, Koopman P. Twenty pairs of sox: Extent, homology, and nomenclature of the mouse and human sox transcription factor gene families[J]. Developmental Cell, 2002, 3(2): 167-170. DOI:10.1016/S1534-5807(02)00223-X
(  0) 0) |
| [4] |
Bowles J, Schepers G, Koopman P. Phylogeny of the SOX family of developmental transcription factors based on sequence and structural indicators[J]. Developmental Biology, 2000, 227(2): 239-255. DOI:10.1006/dbio.2000.9883
(  0) 0) |
| [5] |
Uchikawa M, Kamachi Y, Kondoh H. Two distinct subgroups of Group B SOX genes for transcriptional activators and repressors: Their expression during embryonic organogenesis of the chicken[J]. Mechanisms of Development, 1999, 84(1-2): 103-120. DOI:10.1016/S0925-4773(99)00083-0
(  0) 0) |
| [6] |
Doerks T, Copley R R, Schultz J, et al. Systematic identification of novel protein domain families associated with nuclear functions[J]. Genome Research, 2002, 12(1): 47-56. DOI:10.1101/gr.203201
(  0) 0) |
| [7] |
Mizuseki K, Kishi M, Matsui M, et al. Xenopus Zic-related-1 and Sox-2, two factors induced by chordin, have distinct activities in the initiation of neural induction[J]. Development, 1998, 125(4): 579-586.
(  0) 0) |
| [8] |
Kamachi Y, Sockanathan S, Liu Q, et al. Involvement of SOX proteins in lens-specific activation of crystallin genes[J]. Embo Journal, 1995, 14(14): 3510-3519. DOI:10.1002/embj.1995.14.issue-14
(  0) 0) |
| [9] |
Lowe C J, Wu M, Salic A, et al. Anteroposterior patterning in hemichordates and the origins of the chordate nervous system[J]. Cell, 2003, 113(7): 853-865. DOI:10.1016/S0092-8674(03)00469-0
(  0) 0) |
| [10] |
Taguchi S, Tagawa K, Humphreys T, et al. Group B sox genes that contribute to specification of the vertebrate brain are expressed in the apical organ and ciliary bands of Hemichordate larvae[J]. Zoological Science, 2002, 19(1): 57-66. DOI:10.2108/zsj.19.57
(  0) 0) |
| [11] |
Detlev A, Valérie L, Le G M, et al. atonal- and achaete-scute-related genes in the annelid Platynereis dumerilii: Insights into the evolution of neural basic-Helix-Loop-Helix genes[J]. Bmc Evolutionary Biology, 2008, 8(1): 170-183. DOI:10.1186/1471-2148-8-170
(  0) 0) |
| [12] |
Vidal B, Santella A, Serrano-Saiz E, et al. C. elegans SoxB genes are dispensable for embryonic neurogenesis but required for terminal differentiation of specific neuron types[J]. Development, 2015, 142(14): 2464-2477. DOI:10.1242/dev.125740
(  0) 0) |
| [13] |
Jager M, Quéinnec E, Chiori R, et al. Insights into the early evolution of SOX genes from expression analyses in a ctenophore[J]. Journal of Experimental Zoology Part B Molecular and Developmental Evolution, 2008, 310B(8): 650-667. DOI:10.1002/jez.b.v310b:8
(  0) 0) |
| [14] |
Wei Z. Direct development of neurons within foregut endoderm of sea urchin embryos[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(22): 9143-9147. DOI:10.1073/pnas.1018513108
(  0) 0) |
| [15] |
Mashanov V S, Zueva O R, García-Arrarás J E. Expression of pluripotency factors in echinoderm regeneration[J]. Cell and Tissue Research, 2015, 359(2): 521-536. DOI:10.1007/s00441-014-2040-4
(  0) 0) |
| [16] |
Shen S P, Aleksic J, Russell S. Identifying targets of the Sox domain protein Dichaete in the Drosophila, CNS via targeted expression of dominant negative proteins[J]. Bmc Developmental Biology, 2013, 13(1): 1-15. DOI:10.1186/1471-213X-13-1
(  0) 0) |
| [17] |
Aleksic J, Ferrero E, Fischer B, et al. The role of Dichaete in transcriptional regulation during Drosophila embryonic development[J]. Bmc Genomics, 2013, 14(1): 861-878. DOI:10.1186/1471-2164-14-861
(  0) 0) |
| [18] |
方华华, 王波. 星斑川鲽仔稚鱼消化系统发育的组织学研究[J]. 中国农学通报, 2011, 27(14): 50-54. Fang H H, Wang B. Histological studies on the development of digestive system in larval and juvenile starry flounder[J]. Chinese Agricultural Science Bulletin, 2011, 27(14): 50-54. (  0) 0) |
| [19] |
Zambonino Infante J L, Cahu C L. Ontogeny of the gastrointestinal tract of marine fish larvae[J]. Comparative Biochemistry and Physiology Part C, 2001, 130(4): 477-487.
(  0) 0) |
| [20] |
姜俊, 冯琳, 胡凯, 等. 谷氨酰胺与鱼类消化系统生长发育以及肠上皮细胞蛋白质合成和抗氧化能力的关系[J]. 动物营养学报, 2011, 23(5): 735-739. Jang J, Feng L, Hu K, et al. Glutamine: The relationship with growth and development of digestive system, enterocyte protein synthesis and antioxidant ability in fish[J]. Chinese Journal of Animal Nutrition, 2011, 23(5): 735-739. DOI:10.3969/j.issn.1006-267x.2011.05.005 (  0) 0) |
| [21] |
陈宗涛, 张志峰, 康庆浩, 等. 单环刺螠消化道的发生和分化[J]. 中国水产科学, 2006, 13(5): 700-707. Chen Z T, Zhang Z F, Kang K H, et al. Ontogenesis and development of digestive canal in Urechis unicinctus[J]. Journal of Fishery Sciences of China, 2006, 13(5): 700-707. DOI:10.3321/j.issn:1005-8737.2006.05.002 (  0) 0) |
| [22] |
邵明瑜, 张志峰, 康庆浩, 等. 单环刺螠虫消化道组织学和细胞学[J]. 中国水产科学, 2003, 10(4): 265-270. Shao M Y, Zhang Z F, Kang K h, et al. Histology and cytology of digestive tract in Urechis unicinctus VonDrasche[J]. Journal of Fishery Sciences of China, 2003, 10(4): 265-270. DOI:10.3321/j.issn:1005-8737.2003.04.001 (  0) 0) |
| [23] |
萨姆布鲁克 J, 拉塞尔 D W. 分子克隆实验指南[M]. 北京: 化学工业出版社, 2008: 707-721. Sambrook J, Russell D W. Molecular Cloning Experiment Guide[M]. Beijing: Chemical Industry Press, 2008: 707-721. (  0) 0) |
| [24] |
Fabioux C, Huvet A, Lelong C, et al. Oyster vasa-like gene as a marker of the germline cell development in Crassostrea gigas[J]. Biochem Biophys Res Commun, 2004, 320(2): 592-598. DOI:10.1016/j.bbrc.2004.06.009
(  0) 0) |
| [25] |
冯政夫, 邵明瑜, 孙大鹏, 等. 栉孔扇贝Cf-dmrt4-like基因的克隆、序列特征及表达分析[J]. 中国水产科学, 2010, 17(5): 930-940. FENG Z F, SHAO M Y, SUN D P. Cloning, characterization and expression analysis of Cf-dmrt4-like gene in Chlamys farreri[J]. Journal of Fishery Sciences of China, 2010, 17(5): 930-940. (  0) 0) |
| [26] |
Zhong L, Wang D, Gan X, et al. Parallel expansions of sox transcription factor group B predating the diversifications of the arthropods and jawed vertebrates[J]. Plos One, 2011, 6(1): e16570. DOI:10.1371/journal.pone.0016570
(  0) 0) |
| [27] |
Sur A, Magie C R, Seaver E C, et al. Spatiotemporal regulation of nervous system development in the annelid Capitella teleta[J]. Evodevo, 2017, 8(1): 13-36. DOI:10.1186/s13227-017-0076-8
(  0) 0) |
| [28] |
Kenny A P, Kozlowski D, Oleksyn D W, et al. SpSoxB1, a maternally encoded transcription factor asymmetrically distributed among early sea urchin blastomeres[J]. Development, 1999, 126(23): 5473-5483.
(  0) 0) |
| [29] |
Schnitzler C E, Simmons D K, Pang K, et al. Expression of multiple Sox genes through embryonic development in the ctenophore Mnemiopsis leidyi, is spatially restricted to zones of cell proliferation[J]. Evodevo, 2014, 5(1): 15-32. DOI:10.1186/2041-9139-5-15
(  0) 0) |
| [30] |
Gouar M L, Guillou A, Vervoort M. Expression of a SoxB and a Wnt2/13 gene during the development of the mollusc Patella vulgata[J]. Development Genes and Evolution, 2004, 214(5): 250-256. DOI:10.1007/s00427-004-0399-z
(  0) 0) |
| [31] |
He Y, Bao Z M, Guo H H, et al. Molecular cloning and characterization of SoxB2 gene from Zhikong scallop Chlamys farreri[J]. Journal of Oceanology and Limnology, 2013, 31(6): 1216-1225. DOI:10.1007/s00343-013-3039-5
(  0) 0) |
| [32] |
Sánchezsoriano N, Russell S. Regulatory mutations of the Drosophila Sox gene Dichaete reveal new functions in embryonic brain and hindgut development[J]. Developmental Biology, 2000, 220(2): 307-321. DOI:10.1006/dbio.2000.9648
(  0) 0) |
| [33] |
Daniel M, Marianne B F. The amphioxus soxB Family: Implications for the evolution of vertebrate placodes[J]. International Journal of Biological Sciences, 2007, 3(6): 356-364.
(  0) 0) |
 2019, Vol. 49
2019, Vol. 49


