Gcm(Glial cells missing)基因首先于果蝇中被鉴定出来,其编码一段含Gcm-motif的转录因子,在神经元前体细胞分化为神经胶质细胞的过程中发挥重要作用[1-3]。目前,哺乳动物同源的Gcm也已经被分离鉴定出来[4-8],它们都拥有高度保守的可结合DNA的Gcm-motif[4]。哺乳动物的Gcm同源基因分为Gcm1和Gcm2两种类型,主要是在非神经组织中表达,功能多样[6-8]。例如:小鼠和人的Gcm1基因在胎盘中表达,参与胎盘中尿囊绒膜的形成[8-10];然而,Gcm1基因缺失的小鼠脑中星型胶质细胞数量并没有明显的减少[11],表明Gcm1的功能在小鼠与果蝇中可能存在差异。小鼠的Gcm2基因表达在第三咽囊和源自于第三咽囊的甲状旁腺[6, 12],参与甲状旁腺的形成[13];人GCM2基因纯合缺失可抑制甲状旁腺的正常形成及其功能[14]。相似地,鸡中的Gcm2基因也主要表达于咽囊和甲状旁腺[15]。
不同于陆生脊椎动物,鱼类没有甲状旁腺,但仍有Gcm2基因的表达。以硬骨鱼斑马鱼为例,Gcm2基因在咽上皮、咽囊(Pharyngeal pouches)、鳃丝和内鳃芽蕾(Internal gill buds)中高表达,是斑马鱼咽软骨、鳃丝及内鳃芽蕾形成所必需的[16-17]。相似地,软骨鱼狗鲨(Dogfish)的Gcm2基因也在咽囊及由该结构派生出的内鳃芽蕾中表达[15]。Gcm2作为哺乳动物甲状旁腺发育的标记基因,却在鱼类的鳃相关结构中高表达,暗示哺乳动物的甲状旁腺在进化上可能来源于鱼类的鳃[15]。另外,还发现Gcm2基因在斑马鱼的巨噬细胞中也有表达[16]。Gcm2在斑马鱼胞外钙离子平衡的调控中也发挥重要的作用[15, 18-20]。
以上这些研究表明,不同物种的Gcm基因很可能具有不同的生物学功能。头索动物文昌鱼是无脊椎动物进化到脊椎动物过程中的一个重要过渡类群,是研究脊椎动物起源与演化的理想的模式生物。本文通过生物信息学方法从文昌鱼基因组鉴定出Gcm同源基因(本文命名为Gcm2-like),分析了Gcm基因的系统进化,并利用RT-PCR和原位杂交技术对Gcm2-like基因在文昌鱼中的表达进行了检测,发现其在文昌鱼的鳃中高表达,为深入研究Gcm生物学功能与进化奠定了基础。
1 材料与方法 1.1 实验动物青岛文昌鱼(Branchiostoma japonicum,曾用名B. belcheri tsingtauense),在中国青岛市沙子口海域采集获取。在实验室每天以单细胞藻类喂食2次。在实验开始前,将其在消过毒的过滤海水中进行2 d的绝食,以去除肠道内的所有食物。
1.2 实验方法 1.2.1 总RNA提取、cDNA第一链合成使用OMEGA公司RNA提取试剂盒提取青岛文昌鱼不同组织的总RNA,通过Nanodrop仪和琼脂糖凝胶电泳检测RNA的浓度和质量。使用Takara公司cDNA反转录试剂盒获取各组织cDNA,通过β-actin检测得到的cDNA质量。β-actin上下游引物为:F:5′-TCTGGCATCATACCTTCTACAA-3′,R:5′- TCTGTGTCATCTTTTCCCTGTT-3′。
1.2.2 文昌鱼Gcm2-like基因的获取从NCBI和Ensembl网站获取斑马鱼、小鼠和人等物种的Gcm氨基酸序列,选择白氏文昌鱼或弗罗里达文昌鱼数据库,通过NCBI-BLAST-protein blast,找到文昌鱼的Gcm2-like蛋白的氨基酸序列,并进行反向BLAST确认。利用primer premier 5.0软件进行引物设计,以上述反转录出的cDNA为模板,进行PCR扩增。引物序列为:F:5’-AGGGACTGGGACATCAACGA-3’,R:5’-GGTCGTGCGTCCCTTTGG-3’;PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,35 cycles;72 ℃ 7 min;4 ℃长期保存。将PCR产物克隆至pGEM-TE载体并转化至DH5α中,对得到的阳性克隆菌进行测序,将测序结果与白氏文昌鱼基因组数据进行对比分析。
1.2.3 Gcm2-like基因序列、结构比较和系统进化分析对从NCBI中获取的代表物种Gcm序列的基因结构(外显子、内含子)进行对比,分析Gcm基因在进化过程中的保守性。用从NCBI和Ensembl网站获取的其他物种的Gcm氨基酸序列,同文昌鱼的Gcm2-like氨基酸序列用MegAlign软件进行对比分析。通过Swiss-Model在线软件(http://swissmodel.expasy.org/)对Gcm蛋白的三维结构进行预测。用Sequence Viewer (http://www.ncbi.nlm.nih.gov/projects/sviewer/)和Ensembl Genome Browser (http://www.ensembl.org)对代表物种的Gcm的同源基因在染色体上的位置进行比较。同时,截取代表物种Gcm蛋白的Gcm-motif区域同文昌鱼的Gcm2-like的Gcm-motif结构域氨基酸序列用ClustalX软件进行对比分析。通过MEGA7软件,对不同物种的Gcm蛋白氨基酸序列用Muscle法进行比对,对比完成后进行1 000次bootstrap,分别利用平均连接聚类法(Unweighted Pair Group Method with Arithmetic mean,UPGMA)、邻位相接法(Neighbor Joining,NJ)、最大似然法(Maximum Likelihood,ML)建立关于Gcm蛋白的系统进化树。
1.2.4 RT-PCR利用RT-PCR检测文昌鱼Gcm2-like基因在各组织中的表达情况。本实验中,内参基因为ef1α基因,引物序列为:F:5′-TGCTGATTGTGGCTGCTGGTGGTACTG -3′,R:5′-GGTGTAGGCCAGCAGGGCGTG-3′。PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,35 cycles;72 ℃ 7 min;4 ℃长期保存。PCR产物经琼脂糖凝胶电泳后,在紫外灯下拍照观察。
1.2.5 原位杂交根据扩增得到的青岛文昌鱼Gcm2-like基因序列设计探针,引物序列为:F:5’-AGGGACTGGGACATCAACGA-3’,R:5’-GGTCGTGCGTCCCTTTGG-3’。SP6和T7启动子序列分别加在正向引物和反向引物的5’端。以青岛文昌鱼cDNA为模板,用合成探针时所用的引物进行PCR扩增。PCR产物连接、转化、测序后,以测序正确的质粒为模板,使用Roche公司DIG RNA Labeling kit试剂盒体外转录获得Gcm DIG标记的RNA正义和反义探针。使用Nanodrop仪检测探针的浓度,并通过琼脂糖凝胶电泳检测探针的质量。将文昌鱼组织从多聚甲醛溶液(PFA)中取出后,在石蜡切片机中固定,进行切片,切片厚度为8 μm。展片、捞片、烘片具体操作按照说明书进行。
2 结果 2.1 Gcm基因在文昌鱼及各物种中的鉴定和结构比较通过搜索NCBI数据库,发现Gcm基因有2种类型Gcm1和Gcm2。Gcm1基因仅在从两栖类(比如爪蟾)到哺乳动物(比如人)的物种中存在,而Gcm2 (或Gcm2-like)基因广泛存在于从海葵等低等无脊椎动物到哺乳动物的各物种中(见表 1)。值得注意的是,节肢动物的黑腹果蝇、锯齿果蝇等昆虫存在2个Gcm基因(本文分别称为Gcm2a-like和Gcm2b-like)。它们在染色体上位置相近,而在同是节肢动物的跳蚁、金刻沃氏蚁和家蚕等仅有一个Gcm2-like基因,因此,果蝇的这2个Gcm2-like基因可能是在果蝇的进化过程中由一个Gcm2-like基因的通过复制产生的。
|
|
表 1 在不同物种中鉴定到的Gcm基因 Table 1 Gcm genes identified in different species |
研究还发现,绿头鸭Gcm2 基因有3个不同长度的mRNA剪接体(isoform1-3),其中剪接体isoform1和isoform2编码的蛋白质的氨基酸序列仅在N端存在差异,剪接体isoform3编码的蛋白质的氨基酸序列为isoform1和isoform2剪接体编码蛋白质的一部分。这种一个Gcm基因有多个mRNA剪接体的现象在无脊椎动物中更为常见,比如,星状海葵存在6个不同的mRNA剪接体,编码3个仅在N端有部分差异的蛋白;萼柱珊瑚存在2个不同的mRNA剪接体,编码1个相同的蛋白;棘冠海星存在4个不同的mRNA剪接体,编码1个相同的蛋白;而弗罗里达文昌鱼有6个不同长度的mRNA剪接体(isoform 1-6),这6个转录本仅在5′端存在差异,其它大部分序列都相同。这6个转录本编码6个蛋白,它们的氨基酸序列仅有少数氨基酸不同,其中剪切体isoform 4, isoform5,isoform6编码的蛋白序列完全一样,剪切体isoform1编码的蛋白序列仅比isoform2编码的蛋白多1个氨基酸残基。
通过对人、小鼠、弗罗里达文昌鱼等代表物种Gcm的基因的结构进行比较(见图 1),我们发现脊椎动物Gcm1和Gcm2基因结构在进化上较为保守,均含有2个长度分别为253和113 bp的外显子;但有一个明显不同,即Gcm1基因都有1个长度为129 bp的外显子,而在Gcm2基因中其对应的外显子的长度为126 bp。弗罗里达文昌鱼Gcm2-like基因外显子数量较脊椎动物多1~2个,且大多数外显子略长一些,但整体上保持了与脊椎动物的Gcm基因结构的相似性。
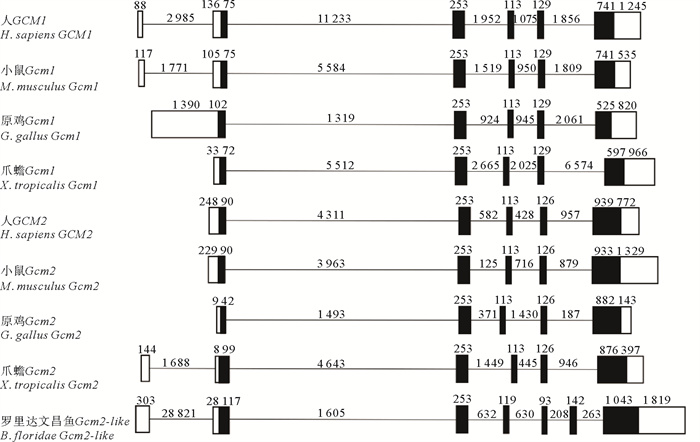
|
( 弗罗里达文昌鱼Gcm2-like基因与人、小鼠、原鸡、爪蟾、斑马鱼Gcm基因的外显子、内含子结构分析。序列来源参见表 1。黑色方框代表编码的外显子,白色方框代表不编码的外显子,直线代表内含子。The exon and intron structure analysis of B. floridae Gcm2-like gene and Gcm genes of human, mouse, chicken, xenopus, and zebrafish. See Table 1 for the sequence sources. The black boxes represent encoded exons, the white boxes represent unencoded exons, and the straight lines represent introns. ) 图 1 弗罗里达文昌鱼Gcm2-like基因与其它代表物种Gcm基因的结构分析 Fig. 1 Structural analysis of B. floridae Gcm2-like gene and Gcm genes of other representative species |
Gcm-motif结构域为Gcm蛋白的典型结构标记,我们从不同门类中选取了几个代表物种首先对该结构域进行了序列相似性分析,结果显示它们都有典型的Gcm-motif结构特点,特别是其半胱氨酸在进化过程中十分保守(见图 2)。
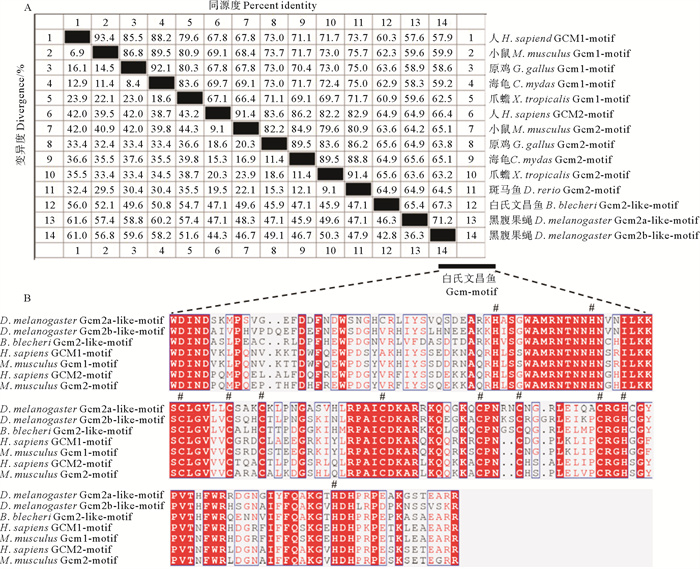
|
( A:白氏文昌鱼Gcm2-like-motif结构域与其它物种Gcm-motif的氨基酸序列用MegAlign软件进行相似性分析。序列来源参见表 1。B: 人、果蝇、白氏文昌鱼Gcm-motif结构域序列用ClustalX进行对比分析,保守的氨基酸用#标注。A: Similarity analysis of amino acid sequences between the Gcm2-like motif domain of B. belcheri and Gcm-motif of other species was conducted by MegAlign software. See Table 1 for the sequence sources. B: The Gcm-motif domain sequences of human, Drosophila melanogaster and B. belcheri were contrastively analyzed by ClustalX, and the conserved amino acids were labeled with #. ) 图 2 白氏文昌鱼Gcm2-like蛋白与其它物种Gcm蛋白的motif结构域氨基酸序列比较 Fig. 2 Comparison of the amino acid sequence of the motif domain of B. belcheri Gcm2-like protein and Gcm protein of other species |
黑腹果蝇、锯齿果蝇等蝇类存在的两种Gcm蛋白的Gcm-motif相似度为71.2%,这两种Gcm蛋白的Gcm-motif与脊椎动物Gcm2-motif结构域部分相似度为63.6%~67.4%,而与脊椎动物Gcm1-motif结构域部分相似度为57.6%~62.5%(见图 2A)。同样的,白氏文昌鱼Gcm蛋白与脊椎动物Gcm2-motif结构域部分相似性要比与脊椎动物Gcm1-motif结构域部分的相似性略高(见图 2B),但相差并不很大。
2.2.2 Gcm的蛋白全长序列比对、结构比较及基因在染色体上的同线性分析将白氏文昌鱼Gcm2-like蛋白与代表性物种人、小鼠、原鸡、果蝇等Gcm蛋白的全长序列进行对比分析,结果显示它们的序列相似度并不高(< 30%)。其中白氏文昌鱼Gcm2-like蛋白与人、小鼠Gcm1蛋白的相似性为26.2%~29.0%,与人、小鼠Gcm2蛋白的相似性为25.1%~27.0%(见图 3A),显示白氏文昌鱼Gcm2-like并不特别类似Gcm1或Gcm2。
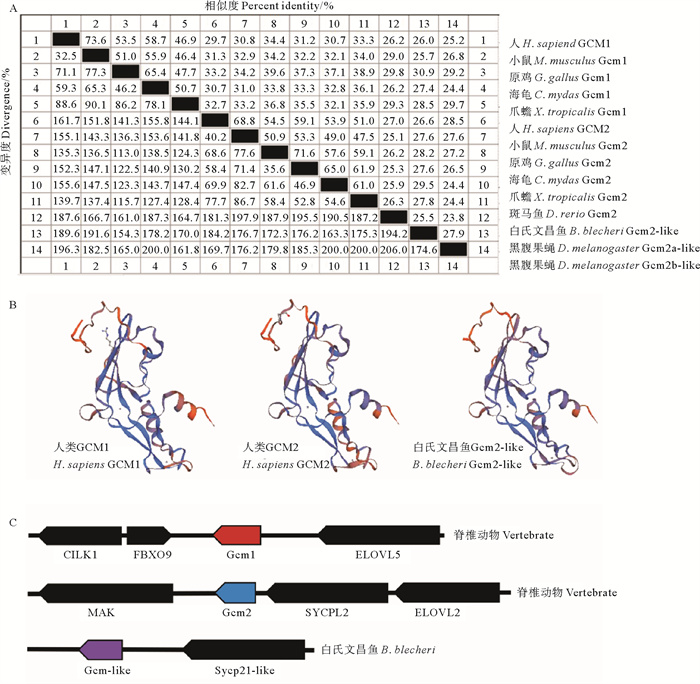
|
( A:白氏文昌鱼Gcm2-like蛋白与其它物种Gcm蛋白的氨基酸序列相似性分析。代表物种Gcm全长蛋白的氨基酸序列用MegAlign软件进行对比分析。序列来源参见表 1;B:白氏文昌鱼Gcm2-like蛋白、人的GCM1及GCM2蛋白预测的三维结构比较。通过Swiss-Model在线软件对Gcm蛋白的三维结构进行预测;C:用Sequence Viewer和Ensembl Genome Browser对表 1列出的所有脊椎动物和白氏文昌鱼的Gcm的同源基因在染色体上的位置进行比较。方框代表基因,箭头代表转录方向。A: Amino acid sequence similarity analysis of Gcm2-like protein of B. belcheri and Gcm protein of other species. The amino acid sequences representing the full-length Gcm protein of the species were analyzed by MegAlign software. See Table 1 for sequence sources; B: Three-dimensional structural comparison of predicted Gcm2-like proteins in B. belcheri, human Gcm1 and Gcm2 proteins. The 3D structure of Gcm protein was predicted by Swiss-Model online software. C: Sequence Viewer and Ensembl Genome Browser were used to compare the locations of homologous genes in Gcm of all vertebrates and amphioxus listed in Table 1 on chromosomes. The boxes represent genes and the arrows represent transcription directions. ) 图 3 白氏文昌鱼Gcm2-like蛋白与其它物种Gcm蛋白的氨基酸序列和三维结构比较,以及基因在染色体上的同线性分析 Fig. 3 Comparison of the amino acid sequence and three-dimensional structure of B. belcheri Gcm2-like protein and Gcm protein of other species, as well as the analysis of synonymy of genes on chromosomes |
对人Gcm1、Gcm2蛋白与白氏文昌鱼的Gcm2-like蛋白的三维结构进行预测,发现它们的三维结构都十分相似,其α螺旋和β折叠的数目均相同(见图 3B),说明虽然Gcm蛋白序列在进化过程中发生了较大的变化,但其仍然保留了较高的三维结构相似性,这可能对维持其功能的保守性具有重要意义。
另外,对脊椎动物染色体上Gcm位点周围基因的分析发现,表 1中列出的所有脊椎动物的Gcm1基因位于Cilk1、Fbxo9、Elovl5基因之间,Gcm2基因位于Mak、Sycp2l、Elovl2基因之间(见图 3C)。这些基因位于染色体的相同位置,且顺序、方向一致,表明在脊椎动物的进化过程中,Gcm基因在染色体上表现出了很好的同线性。类似地,我们发现,白氏文昌鱼Gcm2-like基因在染色体上与sycp2l-like基因位置相临,这与脊椎动物Gcm2基因在染色体上的位置关系相似(见图 3C),暗示文昌鱼Gcm2-like为哺乳动物Gcm2的直系同源基因,而Gcm1基因可能是在进化过程中由Gcm2基因复制而来。
2.3 Gcm基因的系统进化分析为了进一步揭示Gcm基因的进化,本文作者基于Gcm-motif结构域的氨基酸序列,利用UPGMA法构建包含白氏文昌鱼在内的多物种Gcm的系统进化树(见图 4),结果显示:两栖类、鸟类和哺乳动物的Gcm1全部聚为一类,鱼类、两栖类、鸟类和哺乳动物的Gcm2全部聚为一类,无脊椎动物的所有Gcm既不与Gcm1聚为一类,也不与Gcm2聚为一类,而是也全部单独聚为一类(见图 4),而且无脊椎动物的Gcm处于进化的基部。本文作者又分别用NJ法和ML法进行了进化树的构建,结果基本一致(图略)。再者,作者基于Gcm蛋白的全长序列分别利用UPGMA法、NJ法和ML法构建的系统进化树也支持上述结果(图略)。
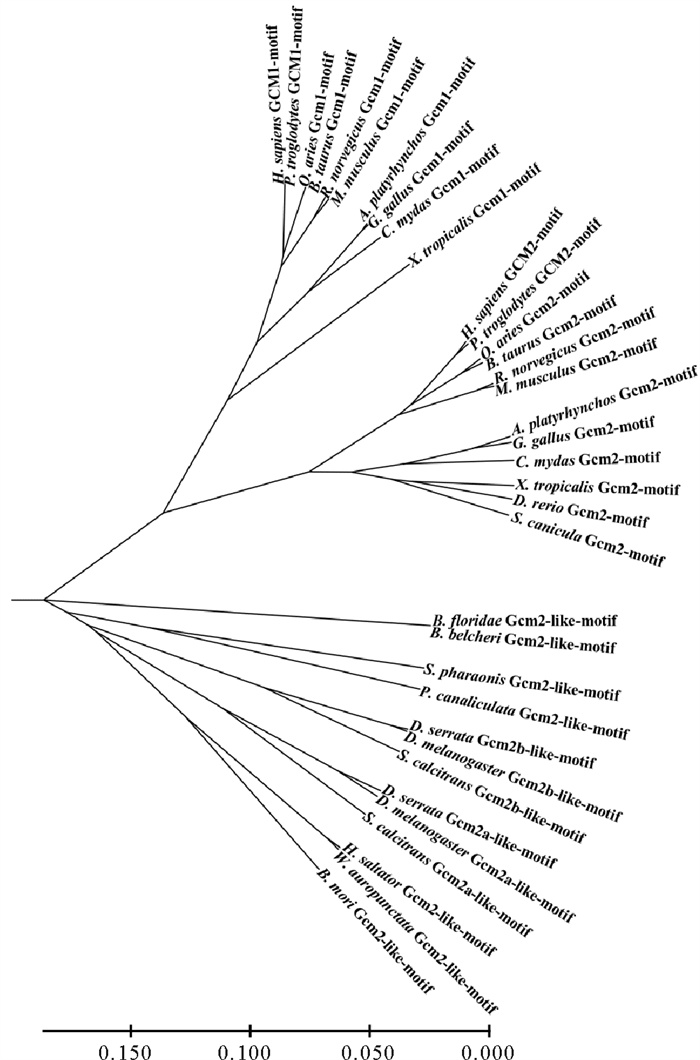
|
( 通过MEGA7软件,对不同物种的Gcm-motif氨基酸序列用Muscle法进行比对,对比完成后进行1 000次bootstrap,利用UPGMA法建立系统进化树。Gcm-motif amino acid sequences of different species were compared with MUSCLE method by MEGA7 software. After comparison, 1 000 Bootstrap was performed, and a phylogenetic tree was established by UPGMA method. ) 图 4 文昌鱼与其他物种Gcm的系统进化分析 Fig. 4 Phylogenetic analysis of Gcm in amphioxus and other species |
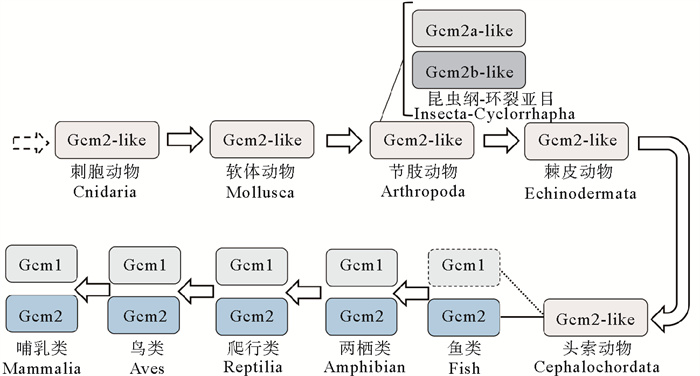
根据以上进化树,作者推测无脊椎动物的Gcm可能代表了脊椎动物Gcm两种类型(Gcm1和Gcm2)分化前的原始形式,无脊椎动物的Gcm没有Gcm1与Gcm2的分化。再结合前面的序列相似性比较及基因在染色体上的同线性分析(文昌鱼的Gcm基因与脊椎动物的Gcm2基因可能为直系同源基因),作者认为Gcm1基因可能是在进化过程中由Gcm2复制而来,脊椎动物的Gcm2可能更多保留了Gcm原始基因的特征。因此,本文将无脊椎动物的Gcm基因全部称为Gcm2-like。最早在刺胞动物中发现Gcm2-like,节肢动物昆虫纲环裂亚目(蝇类)中虽然有两个基因,但也都属于Gcm2-like(本文命名为Gcm2a-like和Gcm2b-like),鱼类中有Gcm2,但未找到Gcm1,两栖类最先产生Gcm1 (见图 5)。
|
( 箭头代表进化方向,方框代表Gcm蛋白类型;昆虫纲的环裂亚目(蝇类)有2个Gcm2-like基因,鱼类Gcm1未被发现。The arrow represents the direction of evolution and the box represents the type of Gcm protein. There are two Gcm2-like genes in Cyclorrhapha (flies) of the class Insecta. Fish Gcm1 has not been found. ) 图 5 Gcm蛋白由刺胞生物进化到哺乳动物简图 Fig. 5 Diagram of the evolution of Gcm protein from cnidaria to mammalia |
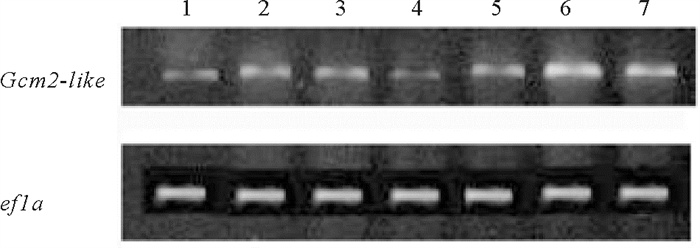
以青岛文昌鱼组织cDNA为模板, PCR扩增Gcm2-like序列,PCR产物测序后经BLAST分析确定属于文昌鱼Gcm2-like。利用RT-PCR检测Gcm2-like在文昌鱼各个组织中的表达情况,结果显示Gcm2-like在文昌鱼鳃和肠中表达量最高,肌肉、精巢及脊索的表达次之,在肝盲囊和卵巢中表达量最少(见图 6)。
|
( 1:肝盲囊;2:肌肉;3:精巢;4:卵巢;:5:脊索: 6:肠;7:鳃。1: Hepatic caecum; 2: Muscles; 3: Testis; 4: Ovaries; 5: Notochord; 6: Intestine; 7: Gills. ) 图 6 RT-PCR检测青岛文昌鱼Gcm2-like基因的组织分布 Fig. 6 Tissue distribution of Gcm2-like gene in B.japonicum was detected by RT-PCR |
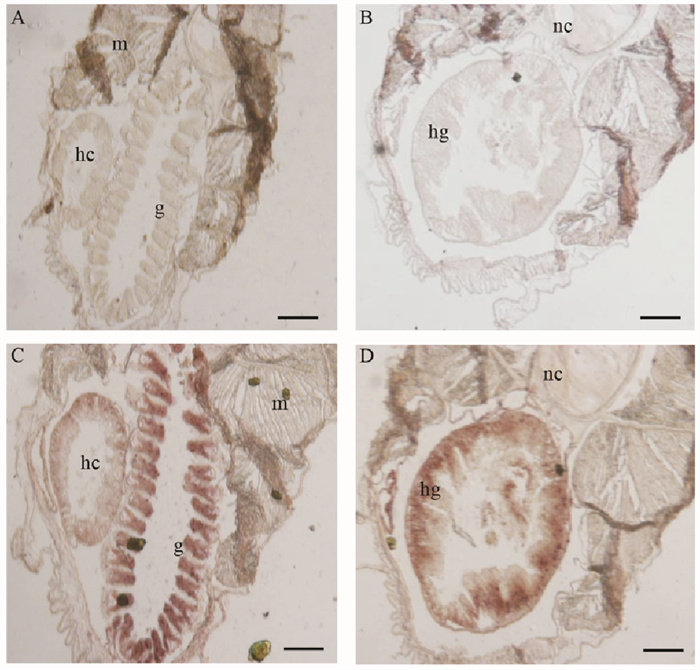
对青岛文昌鱼进行组织切片,原位杂交显示Gcm2-like基因在青岛文昌鱼的鳃、肝盲囊、肠中表达较高,特别是在鳃中有明显的高表达(见图 7)。
|
( hc: 肝盲囊; m: 肌肉; hg: 后肠; nc: 脊索; g: 鳃裂,Bar=100 μm。图A、B为正义探针原位杂交图(阴性对照); C、D为反义探针原位杂交图(实验组)。hc: Hepatic caecum; m: Muscle; hg: Hind-gut; nc: Notochord; g: Gill), Bar=100 μm. A and B are in situ hybridization of the righteous probe (negative control), C and D are in situ hybridization of the antisense probe (experimental group). ) 图 7 青岛文昌鱼Gcm2-like基因的切片原位杂交 Fig. 7 Sectional in situ hybridization of B. japonicum Gcm2-like gene |
最早发现的果蝇Gcm有决定神经元细胞与胶质细胞转化的功能[1-3],而其后在哺乳动物中的研究则显示Gcm同源基因在非神经组织表达[8, 11],这表明脊椎动物的Gcm基因与无脊椎动物的Gcm基因很大可能具有生物学功能上的差异。文昌鱼处于无脊椎动物进化至脊椎动物的过渡类型,因此,对文昌鱼Gcm基因进行研究对于揭示Gcm基因的进化具有重要意义。本文通过生物信息学方法首先对Gcm基因的进化进行了分析,发现其全长序列在各物种中相似性不高,但其Gcm-motif具有很高的保守性,特别是其Gcm蛋白的三维结构十分相似。我们还通过对基因在染色体上的同线性分析,认为文昌鱼Gcm2-like为脊椎动物Gcm2的直系同源基因。系统进化分析显示,Gcm基因在进化中,由无脊椎动物单一的Gcm2-like基因逐步分化为脊椎动物的Gcm1和Gcm2两种基因类型。根据目前的信息,在人、小鼠等哺乳动物及鸡、爪蟾等脊椎动物中,两种类型的Gcm基因都存在,但鱼类中只存在Gcm2,没有发现Gcm1,鱼类的Gcm1基因有可能在进化过程中发生了丢失。
在哺乳动物中,Gcm2基因在咽囊及由此派生出的甲状旁腺中高表达,是甲状旁腺形成所必须的,为甲状旁腺的标记基因[13]。在鱼类中,Gcm2表达在鱼类的咽囊及由咽囊派生出的内鳃蕾中,是内鳃蕾正常形成所必需的[15]。由此,暗示哺乳动物的甲状旁腺和鱼类的内鳃蕾为具有关联的结构,可能有一共同的进化史。本文发现,Gcm2-like基因在文昌鱼的肝盲囊、肠、鳃表达较高,特别是在鳃中表达明显,这与Gcm2基因在鱼类中的表达模式相似。因此,Gcm2可能在整个脊索动物中发挥保守的功能,这也为哺乳动物的甲状旁腺在进化上可能起源于鳃的假设提供了新的证据。
另外,我们的结果还显示Gcm2-like基因在文昌鱼的肌肉、脊索和神经管中也有表达,尽管表达相对较低,暗示其可能还具有其它的功能,比如:鱼类的鳃在钙离子摄取中发挥着重要的作用[18-19]。关于Gcm基因的更多的生物学功能有待于进一步的研究。
| [1] |
Hosoya T, Takizawa K, Nitta K, et al. Glial cells missing: A binary switch between neuronal and glial determination in Drosophila[J]. Cell, 1995, 82(6): 1025-1036. DOI:10.1016/0092-8674(95)90281-3
(  0) 0) |
| [2] |
Jones BW, Fetter RD, Tear G, et al. Glial cells missing: A genetic switch that controls glial versus neuronal fate[J]. Cell, 1995, 82(6): 1013-1023. DOI:10.1016/0092-8674(95)90280-5
(  0) 0) |
| [3] |
Vincent S, Vonesch J L, Giangrande A. Glide directs glial fate commitment and cell fate switch between neuron and glia[J]. Development, 1996, 122(1): 131-139. DOI:10.1242/dev.122.1.131
(  0) 0) |
| [4] |
Akiyama Y, Yasuko T, Hosoya A M, et al. The gcm-motif: A novel DNA-binding motif conserved in Drosophila and mammals[J]. Proc Natl Acad Sci USA, 1996, 93(25): 14912-14916. DOI:10.1073/pnas.93.25.14912
(  0) 0) |
| [5] |
Altshuller Y, Copeland N G, Gilbert D J, et al. Gcm1, a mammalian homolog of Drosophila Glial Cells Missing[J]. Febs Letters, 1996, 393(2-3): 201-204. DOI:10.1016/0014-5793(96)00890-3
(  0) 0) |
| [6] |
Kim J, Jones B W, Zock C, et al. Isolation and characterization of mammalian homologs of the Drosophila gene glial cells missing[J]. Proc Natl Acad Sci USA, 1998, 95(21): 12364-12369. DOI:10.1073/pnas.95.21.12364
(  0) 0) |
| [7] |
Kanemura Y, Hiraga S, Arita N, et al. Isolation and expression analysis of a novel human homologue of the Drosophila glial cells missing (gcm) gene[J]. Febs Letters, 1999, 442(2-3): 151-156. DOI:10.1016/S0014-5793(98)01650-0
(  0) 0) |
| [8] |
Nait-Oumesmar B, Alan B C, Robert A L. Placental expression and chromosomal localization of the human Gcm1 Gene[J]. Journal of Histochemistry & Cytochemistry, 2000, 48(7): 915-922.
(  0) 0) |
| [9] |
Anson-Cartwright L, Dawson K, Holmyard D, et al. The glial cells missing-1 protein is essential for branching morphogenesis in the chorioallantoic placenta[J]. Nature Genetics, 2000, 25(3): 311-314. DOI:10.1038/77076
(  0) 0) |
| [10] |
Stecca B, Nait-Oumesmar B, Kelley KA, et al. Gcm1 expression defines three stages of chorio-allantoic interaction during placental development[J]. Mechanisms of Development, 2002, 115(1-2): 27-34. DOI:10.1016/S0925-4773(02)00095-3
(  0) 0) |
| [11] |
Iwasaki Y. The potential to induce glial differentiation is conserved between Drosophila and mammalian glial cells missing genes[J]. Development, 2003, 130(24): 6027-6035. DOI:10.1242/dev.00822
(  0) 0) |
| [12] |
Gordon J, Bennett A R, Blackburn C C, et al. Gcm2 and Foxn1 mark early parathyroid- and thymus-specific domains in the developing third pharyngeal pouch[J]. Mechanisms of Development, 2001, 103(1-2): 141-143. DOI:10.1016/S0925-4773(01)00333-1
(  0) 0) |
| [13] |
Yamada T, Tatsumi N, Anraku A, et al. Gcm2 regulates the maintenance of parathyroid cells in adult mice[J]. PLoS ONE, 2019, 14(1): e0210662. DOI:10.1371/journal.pone.0210662
(  0) 0) |
| [14] |
Günther T, Chen Z F, Kim J, et al. Genetic ablation of parathyroid glands reveals another source of parathyroid hormone[J]. Nature, 2000, 406(6792): 199-203. DOI:10.1038/35018111
(  0) 0) |
| [15] |
Okabe M, Graham A. The origin of the parathyroid gland[J]. Proc Natl Acad Sci USA, 2004, 101(51): 17716-17719. DOI:10.1073/pnas.0406116101
(  0) 0) |
| [16] |
Hanaoka R, Ohmori Y, Uyemura K, et al. Zebrafish Gcmb is required for pharyngeal cartilage formation[J]. Mechanisms of Development, 2004, 121(10): 1235-1247. DOI:10.1016/j.mod.2004.05.011
(  0) 0) |
| [17] |
Hogan B M, Hunter M P, Oates A C, et al. Zebrafish Gcm2 is required for gill filament budding from pharyngeal ectoderm[J]. Developmental Biology, 2004, 276(2): 508-522. DOI:10.1016/j.ydbio.2004.09.018
(  0) 0) |
| [18] |
Chang W J, Horng J L, Yan J J, et al. The transcription factor, glial cell missing 2, is involved in differentiation and functional regulation of H+-ATPase-rich cells in zebrafish (Danio rerio)[J]. American Journal of Physiology Regulatory Integrative & Comparative Physiology, 2009, 296(4): R1192.
(  0) 0) |
| [19] |
Kumai Y, Kwong R W M, Perry S F. A role for transcription factor glial cell missing 2 in Ca2+ homeostasis in zebrafish, Danio rerio[J]. Pflügers Archiv-European Journal of Physiology, 2015, 467(4): 753-765. DOI:10.1007/s00424-014-1544-9
(  0) 0) |
| [20] |
Shono T, Kurokawa D, Miyake T, et al. Acquisition of glial cells missing 2 Enhancers Contributes to a diversity of ionocytes in zebrafish[J]. PLoS ONE, 2011, 6(8): e23746. DOI:10.1371/journal.pone.0023746
(  0) 0) |
 2021, Vol. 51
2021, Vol. 51


