2. 青岛海洋科学与技术试点国家实验室 海洋药物与生物制品功能实验室,山东 青岛 266237
甲壳素是由β-(1, 4)-N-乙酰氨基葡萄糖组成的线性多糖,在自然界中含量仅次于纤维素,广泛存在于甲壳类动物和昆虫的外骨骼以及植物、真菌和霉菌的细胞壁[1]。这种多糖聚合物结构致密,存在大量分子间和分子内作用力,导致不可溶性限制了其广泛利用[2]。而降解产物甲壳寡糖具有较好的水溶性和生物活性,可用于食品、生物医药、化妆品、农业和环保等[3]。目前甲壳素的降解方法有化学法和酶解法,其中酶解法是一种可循环的、生态友好的、高效制备降解产物的方法,且酶解的专一性可以控制产品的一致性[4]。
甲壳素酶在甲壳素生物降解过程中起着重要作用,在碳水化合物活性酶数据库(Carbohydrate-active enzymes,CAZy)中,根据氨基酸序列同源性将甲壳素酶分为糖苷水解酶18家族(GH18)和糖苷水解酶19家族(GH19)[5],其中GH18家族由一个高度保守的(β-α)8桶结构组成[6]。根据水解机理,甲壳素酶可分为三大类:内切甲壳素酶(EC 3.2.1.14)、外切甲壳素酶(EC 3.2.1.29)和N-乙酰氨基葡萄糖苷酶(EC 3.2.1.30)[7]。内切甲壳素酶的水解产物是甲壳二糖[N, N′-diacetylchitobiose,(GlcNAc)2]为主的甲壳寡糖[8]。相比之下,外切甲壳素酶可以逐步释放产物(GlcNAc)2。N-乙酰氨基葡萄糖苷酶可以进一步裂解内切甲壳素酶和外切甲壳素酶水解生成的甲壳寡糖,生成单体N-乙酰氨基葡萄糖(N-acetyl-D-glucosamine,GlcNAc)[9]。已报道的甲壳素酶多数来自链霉菌,如Streptomyces lividans[10]、S. olivaceoviridis[11]和S. albolongus[12],但来自杆菌状链霉菌(Streptomyces bacillaris)的甲壳素酶尚未有研究报道,且其他来源的甲壳素酶的酶解产物多数不单一,这限制了单一聚合度甲壳寡糖的活性开发与研究。杆状链霉菌是链霉菌家族的重要组成部分,来自杆菌状链霉菌的甲壳素酶可能具有制备单一聚合度甲壳寡糖的能力。同时,研究来自杆状链霉菌的甲壳素酶,可丰富甲壳素酶库,拓展甲壳素酶的性质研究。
本研究从S. bacillaris CGMCC4.1584基因组中克隆出一条编码甲壳素酶的基因,构建大肠杆菌工程菌进行异源表达,通过镍离子亲和层析获得纯酶,探究该甲壳素酶的酶学性质和水解产物。本实验在可控条件下可制备出高纯度甲壳二糖,为其工业制备提供技术支持。
1 材料和方法 1.1 实验材料 1.1.1 材料与试剂甲壳素购自上海源叶生物科技有限公司;快速微型质粒提取试剂盒(TIANprep Mini Plasmid, DP103-02)购自北京天根生物技术有限公司;KOD高保真酶购自日本TOYOBO公司;无缝连接试剂盒(ClonExpress Ultra One Step Cloning Kit, C115)购自诺唯赞生物科技有限公司;Ni-NTA树脂购自北京全式金生物技术有限公司;DNA和蛋白质标准分子量Marker购自索莱宝生物科技有限公司;硅胶板购自德国Merck公司;甲壳寡糖(聚合度2-6)购自青岛博智汇力生物科技有限公司;其他试剂均为分析级试剂,购自国药集团化学试剂有限公司。
1.1.2 菌株与培养基杆菌状链霉菌(S. bacillaris)购自中国普通微生物菌种保藏管理中心(菌株编号:CGMCC4.1584),其培养基成分为:0.4%酵母粉、1%麦芽粉、0.4%葡萄糖、1.5%琼脂(培养基调至pH为7.3)。
大肠杆菌(Escherichia coli) DH5α用于质粒构建(固体培养基)和目的基因质粒扩增(液体培养基)。其培养基成分为:0.5%酵母粉,1%胰蛋白胨,1.5%琼脂(固体培养基)和1% NaCl。
大肠杆菌BL21为目的基因表达菌株,其培养基成分为:0.5%酵母粉、1.0%蛋白胨、0.2% MgSO4、5.0%混合液(1 mmol/L Na2HPO4、1 mmol/L KH2PO4、0.5 mmol/L(NH4)2SO4)、0.5%甘油、0.05%葡萄糖、0.2% α-半乳糖。
1.1.3 仪器与设备BCM-1000型超净工作台购自苏州净化设备有限公司;GI80TW型全自动高压蒸汽灭菌锅购自厦门致微仪器有限公司;Mastercycler nexus型PCR仪、5420型高速小型离心机、5804R型高速冷冻离心机购自德国艾本德股份公司;MULTISKAN FC型酶标仪购自美国赛默飞世尔科技有限公司;SCIENTZ-IID型超声波细胞粉碎机购自宁波新芝生物科技股份有限公司;HH-4A型数显恒温水浴锅购自常州国华仪器制造有限公司;SPX型生化培养箱购自新江南仪器有限公司;DYY-6C型电泳仪购自北京市六一生物科技有限公司;LC-20AT型高效液相色谱仪购自日本岛津公司。
1.2 实验方法 1.2.1 目的基因的生物信息学分析利用SnapGene软件对目的基因进行分析,分析其ORF长度和基因编码理论蛋白量,通过NCBI和CAZy数据库找到与SbChiAJ143相似的基因序列,并用MEGA6.0构建进化树。采用SMART软件进行结构域分析。利用ClustalW对目的蛋白序列与参考序列进行比对,并通过ESPript 3.0完成保守氨基酸分析。利用SWISS model和Pymol软件预测并查看SbChiAJ143的碳水化合物结合模块和催化结构域的三维模型。
1.2.2 目的基因的克隆与重组质粒的构建以S. bacillaris CGMCC4.1584基因组为模板,设计上游引物(5’-GGCCGCAAGCTTCGGCAGGCTGCGGAC-3’)和下游引物(5’-CAAATGGGTCGCGGATCCATGGTGCAACGCTCCG-3’)利用KOD高保真酶克隆目的片段。PCR反应的程序如下:预变性95 ℃ 10 min;30个扩增循环(98 ℃ 10 s,58 ℃ 30 s,68 ℃ 3 min);最后延伸68 ℃ 10 min。PCR产物经琼脂糖凝胶电泳分离并对目的片段进行胶回收,与pET-28a(+)载体进行无缝连接,构建的重组质粒转入感受态细胞大肠杆菌DH5α,涂布在抗性平板进行筛选并进行菌落PCR阳性克隆验证,验证成功后进一步测序鉴定。
1.2.3 重组蛋白的表达与纯化将含有目的基因的重组质粒采用热激法转化到表达菌株大肠杆菌BL21(DE3)中,在20 ℃,220 r/min恒温摇床培养48 h。发酵结束后,4 ℃, 8 000 g离心10 min,收集菌体。与裂解缓冲液(50 mmol/L Tris-HCl,pH=7.4)混合,超声裂解后,再次4 ℃,8 000 g离心20 min,收集的上清即为粗酶液。
粗酶液过0.45 μm滤膜后与Ni-NTA纯化树脂填料充分混合,在Ni-NTA柱中加入平衡缓冲液(50 mmol/L Tris-HCl,500 mmol/L NaCl,10 mmol/L咪唑)以提高特异性结合,然后分别用含20、40、60、100、200和500 mmol/L咪唑、50 mmol/L Tris-HCl和500 mmol/L NaCl梯度的咪唑溶液清洗柱子。用考马斯亮蓝R-250染色的十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)检测目的蛋白的纯度。
1.2.4 蛋白质定量与酶活测定配制不同浓度的牛血清白蛋白(BSA),用Bradford法定量,绘制蛋白定量的标准曲线。纯化出的蛋白用10 kDa超滤管置换出咪唑溶液,根据标准曲线对目的蛋白溶液进行定量。单位酶活性(U)被定义为在特定条件下每分钟生成1 μm还原糖所需要的酶量。SbChiAJ143具体反应条件为10 μL纯酶加到190 μL 1.0%(w/v)胶质甲壳素(pH=6.0的磷酸盐缓冲),在55 ℃反应30 min,然后在沸水浴中灭酶10 min,冷却到室温。采用3, 5-二硝基水杨酸(DNS)法测定反应生成的还原糖含量。根据540 nm处测定不同浓度的N-乙酰氨基葡萄糖与DNS反应吸光度绘制的还原糖定量标准曲线,计算出生成还原糖的量从而得出酶活。
1.2.5 SbChiAJ143酶学性质表征 1.2.5.1 最适pH和pH稳定性为确定酶的最适pH,各取10 μL纯酶加到190 μL不同pH缓冲溶液制备的1.0%(w/v)胶质甲壳素中,在55 ℃下反应30 min,利用DNS法测定纯酶的活性。不同pH缓冲液(50 mmol/L)分别为柠檬酸缓冲液(pH为3.0~6.0)、磷酸盐缓冲液(pH为6.0~8.0)、Tris-HCl缓冲液(pH为7.0~9.0)和甘氨酸-NaOH缓冲液(pH为9.0~10.0)。
pH稳定性的测定方法:在上述缓冲液中加入等量的纯酶,在25 ℃下放置不同时间间隔(0、12、24、36、48、60和72 h)后加入相应的1.0%(w/v)胶质甲壳素底物,55 ℃反应30 min,采用DNS法测定酶的残余活性。
1.2.5.2 最适温度和温度稳定性各取10 μL纯酶加入190 μL 50 mmol/L磷酸盐缓冲液(pH=6.0)配制的1.0%(w/v)胶质甲壳素中,在温度25、30、35、40、45、50、55、60、65、70、75和80 ℃下,反应30 min,DNS法测定酶的残余活性,从而确定最适反应温度。
热稳定性测定是将等量的酶置于温度30、35、40、45、50、55和60 ℃下分别孵育0、30、60、90、120、150和180 min,加到1.0%(w/v)胶质甲壳素(pH=6.0的50 mmol/L磷酸盐缓冲液)中,55 ℃反应30 min,测定酶的残余活性。所有实验重复3次。
1.2.5.3 金属离子对酶活的影响不同金属离子Na+(NaCl)、K+(KCl)、Mg2+(MgCl2)、Ba2+(BaCl2)、Zn2+(ZnSO4)、Ni2+(NiSO4)、Ca2+(CaCl2)、Fe3+(FeCl3)、Mn2+(MnCl2)、Cu2+(CuCl2)、Co2+(CoCl2)、十二烷基硫酸钠(SDS)及Na2EDTA中加纯酶至终浓度为1.4和14 mmol/L。将上述试剂作用的酶加到1.0%(w/v)胶质甲壳素(pH=6.0的50 mmol/L磷酸盐缓冲液)中,55 ℃反应30 min,对照组为不添加上述试剂的反应体系,探究不同浓度、不同金属离子条件下酶活的变化。
1.2.5.4 SbChiAJ143酶促反应动力学参数测定用pH=6.0的50 mmol/L磷酸盐缓冲液配制浓度分别为0.1、0.5、1.0、5、10、15、20、25、30、40、50、60、80和100 mg/mL的胶质甲壳素,取10 μL纯酶与190 μL上述不同浓度的底物混合,55 ℃下反应5 min,终止反应,测定OD540nm。利用米氏方程将所得数据进行非线性回归拟合,计算Km和Vmax的值。
1.2.6 SbChiAJ143酶解产物的高效液相分析SbChiAJ143与1.0%(w/v)胶质甲壳素和甲壳寡糖(DP2-6)在55 ℃反应0 min、2 min、15 min、30 min、60 min和24 h。取20 μL样本注入到高效液相色谱系统(安捷伦1290,德国),分析柱为Sugar Pak I色谱柱(6.5 mm×300 mm,10 μm),流动相为50 mg/L乙二胺四乙酸钙二钠(EDTA-CaNa2),流速为0.5 mL/min,柱温为75 ℃,检测器为示差检测器。
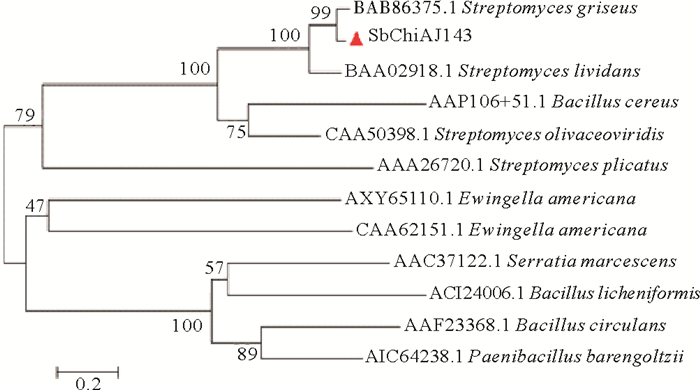
2 结果与分析 2.1 目的基因的生物信息学分析在杆菌状链霉菌(S. bacillaris)基因组中发现一段甲壳素酶编码基因序列,命名为SbChiAJ143(GenBank序列号CP029378.1),全长1 734 bp,编码578个氨基酸,预测相对分子质量为59.2 kDa,等电点为8.66。通过氨基酸序列比对,SbChiAJ143与来自GH18家族灰色链霉菌的甲壳素酶(S. griseus,BAB86375.1)呈现最高相似度(89.12%),与来自同一家族的浅青紫链霉菌的甲壳素酶(S.lividans,BAA02918.1)的相似度次之(73.10%)。根据Neighbor-Joining进化树分析(见图 1),SbChiAJ143属于GH18家族,与蜡样芽孢杆菌(Bacillus cereus)的外切甲壳素酶(AAP10651.1)和橄榄绿链霉菌(S. olivaceoviridis)的外切甲壳素酶(CAA50398.1)有密切的亲缘关系。
|
图 1 SbChiAJ143与GH18其他甲壳素酶氨基酸序列的进化树分析 Fig. 1 Phylogenetic tree analysis of amino acid sequences from SbChiAJ143 and other GH18 chitinases |
基于SMART对蛋白结构域的鉴定和注释,预测SbChiAJ143中存在3个主要结构域(见图 2a):N端碳水化合物结合模块(CBM49,47-168),纤维连接蛋白型结构域(FN3,193-263),C端GH18催化结构域(Glyco18,289-560)。在结合域CBM49中发现了保守的芳香族氨基酸,包括色氨酸(Trp,W)、酪氨酸(Tyr,Y)和苯丙氨酸(Phe,F)(见图 2b),在其两侧分布带负电的残基(Glu-56,Asp-91,Asn-92,Gln-97,Asn-104,Gln-114,Asp-129)形成疏水的沟槽结构,以促进与甲壳素底物结合,这样的结构更有利于该甲壳素酶作用于疏松结构的胶质甲壳素,而不是结构致密的甲壳素粉末(见图 2c)[13-15]。紧挨着结合域的是纤维连接蛋白型结构域FN3,该结构域存在保守的芳香族氨基酸(Trp-213,Tyr-222,Tyr-225,Tyr-250),在细菌甲壳素酶、纤维素酶和淀粉酶中存在同样的结构域,这可能与底物附着有关[16-19]。保守的谷氨酸(Glu,E)和天冬氨酸(Asp,D)在催化结构域中是保守的,它们是参与甲壳素酶酸碱水解机理的关键氨基酸[20]。如图 2d所示,该催化结构域带有隧道状裂隙的(β-α)8桶结构,这是GH-18甲壳素酶典型特征[21-22]。

|
( a:SMART结构域分析;b:多序列比对结果;c:结合域同源建模结构;d:催化结构域同源建模结构。a: Conserved domain analysis of SMART; b: Results of multiple sequence alignment; c: Homology modeling structure of binding domain; d: Homology modeling structure of catalytic domain. ) 图 2 SbChiAJ143多序列比对与结构域分析结果 Fig. 2 Results of multiple sequence alignment and domain analysis of SbChiAJ143 |
重组质粒pET28a-SbChiAJ143在大肠杆菌BL21(DE3)中成功表达。如图 3所示,SDS-PAGE显示纯化的SbChiAJ143为单一条带,其大小约为59.2 kDa,高于一些链霉菌的甲壳素酶,如S.albolongus SaChiA4(47 kDa)[12],Streptomyses sp. CS495(41 kDa)[23]和S. roseolus Chi40(40 kDa)[24]。以胶质甲壳素为底物,纯化酶的比酶活为5.29 U/mg,纯化倍数为11,其粗酶的比酶活为0.48 U/mg。与来自S. olivaceoviridis(酶解产物相对单一)的外切甲壳素酶纯化酶的比酶活(3.50 U/mg)相比,在纯化倍数相当情况下[11],杆菌状链霉菌的甲壳素酶的纯化酶具有较高比酶活。

|
( M:蛋白的标准分子量;泳道1:粗酶液;泳道2:纯酶液。M: Molecular mass marker of proteins; Lane 1: Crude enzyme liquid; Lane 2: Purified enzyme liquid. ) 图 3 蛋白电泳结果 Fig. 3 Results of protein electrophoresis |
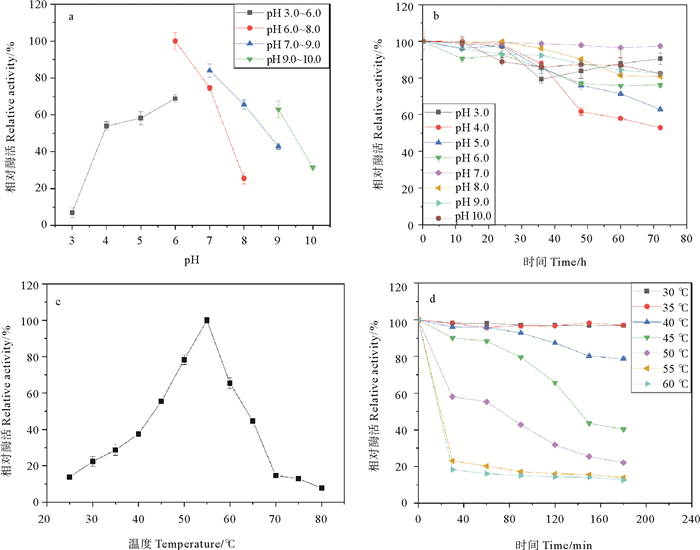
如图 4a所示,SbChiAJ143在pH=6.0的50 mmol/L磷酸盐缓冲液中相对酶活最高,在pH=7.0的Tris-HCl中相对酶活超过80%,与S.chilikensis碱性甲壳素酶最适pH相似[25]。pH为5.0~9.0的相对酶活大于60%,pH=10.0的相对酶活高于pH=3.0,说明该酶在弱酸性和碱性环境中具有较高活性。最适pH为6.0接近中性,更适合工业生产。在pH稳定性方面,在pH为3.0~10.0范围内,72 h后相对酶活仍保持在50%以上(见图 4b),表明在广泛的pH范围内SbChiAJ143是稳定的,与报道的S. albolongus中甲壳素酶一致[12],优于其他细菌和真菌产生的甲壳素酶,如Sanguibacter antarcticus[26],Stenotrophomonas maltophilia[27]和S. anulatus[28]。
|
( a:最适pH;pH 3.0~6.0:柠檬酸盐缓冲液;pH 6.0~8.0:磷酸盐缓冲液;pH 7.0~9.0:Tris-HCl缓冲液;pH 9.0~10.0:甘氨酸-NaOH缓冲液;b:pH稳定性;c:最适温度;d:温度稳定性。a: The optimum pH; pH 3.0~6.0: Citrate buffer; pH 6.0~8.0: Phosphate buffer; pH 7.0~9.0: Tris-HCl buffer; pH 9.0~10.0: Glycline-NaOH buffer; b: The pH stability; c: The optimum temperature; d: The temperature stability. ) 图 4 pH和温度对SbChiAJ143纯酶活性和稳定性的影响 Fig. 4 Effects of pH and temperature on activity and stability of purified SbChiAJ143 |
SbChiAJ143在55 ℃时相对酶活最高(见图 4c),与来自S. albolongus菌株的甲壳素酶SaChiA4[12]和Paenibacillus barengoltzii菌株的甲壳素酶PbChi70相当[29],具有较高的最适反应温度,可防止反应过程中其他微生物的污染,保障反应过程的安全性。当温度高于55 ℃时,甲壳素酶SbChiAJ143的活性迅速显著下降,达到65 ℃时,其活性不到最高活性的一半。在45~60 ℃时,SbChiAJ143的残余酶活相对较高。在50 mmol/L磷酸盐缓冲液(pH=6.0)中将SbChiAJ143于30~45 ℃温度范围孵育180 min,残留酶活仍保留70%以上(见图 4d)。
2.3.3 金属离子对酶活的影响如表 1所示,Ca2+和Ba2+对SbChiAJ143催化活性有轻微的促进作用,且促进效果随着浓度增大而略增强。在高浓度(5 mmol/L)时,Mg2+呈现轻微的激活作用。大部分金属离子在低浓度和高浓度下都对该酶有一定的抑制作用,如Zn2+、Fe3+、Mn2+、Cu2+、Co2+、Ni2+。其中高浓度的Zn2+、Fe3+、Cu2+、Co2+抑制作用更明显。添加低浓度(1 mmol/L)和高浓度(5 mmol/L)表面活性剂SDS,酶活下降至未添加时酶活的57.6%和16.9%,说明表面活性剂可以明显抑制该酶的活性。1和5 mmol/L的Na2EDTA对该酶有轻微抑制作用,且抑制效果相当,说明SbChiAJ143不是金属依赖型酶。
|
|
表 1 金属离子对酶活的影响 Table 1 Effect of metal ions on enzymatic activity |
利用米氏方程非线性拟合得到该酶的Vmax值为17.14 μmol/(min·mg),Km值为3.54 mg/mL。与来源于Paenibacillus pasadenensis CS0611[30]和Streptomyces sp.[31]的甲壳素酶的Km值相比(Km值分别为4.41和6.74 mg/mL),该酶的Km值较小,对底物胶质甲壳素的亲和力较好,但比来源于Streptomyces sp. CS495[23]的甲壳素酶的Km值略大(Km值为1.34 mg/mL)。
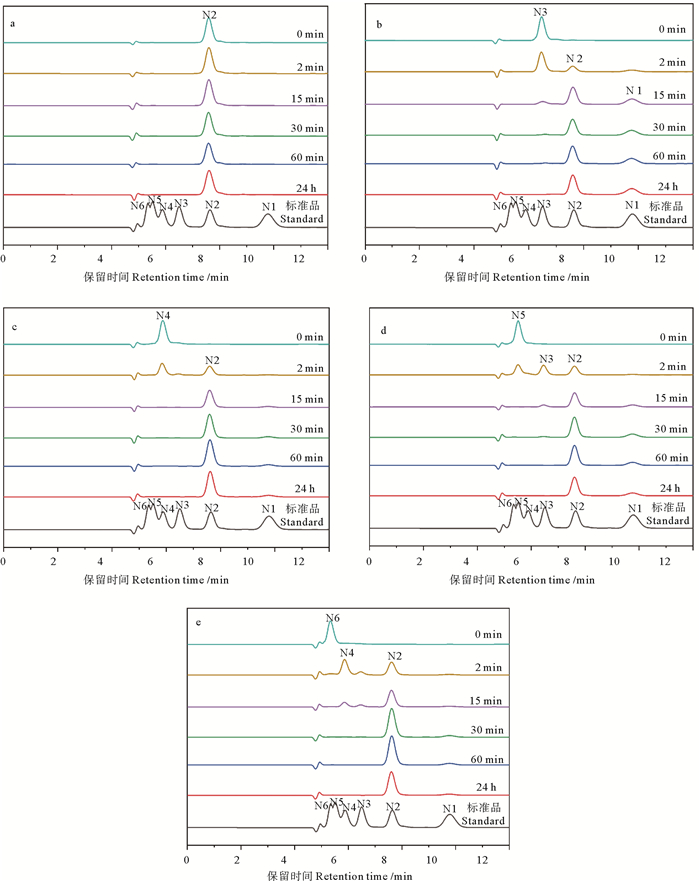
2.4 SbChiAJ143水解产物的高效液相分析采用高效液相色谱法测定了甲壳寡糖(DP 2-6)的水解过程。(GlcNAc)2未被降解,(GlcNAc)3被水解成(GlcNAc)2和GlcNAc;反应2 min后,(GlcNAc)4转化为(GlcNAc)2,伴以少量(GlcNAc)3和GlcNAc;(GlcNAc)5降解为(GlcNAc)3和(GlcNAc)2,(GlcNAc)6生成(GlcNAc)4、(GlcNAc)2和微量(GlcNAc)3,最终降解为(GlcNAc)2和极少量的GlcNAc(见图 5)。上述结果表明,SbChiAJ143甲壳素酶能识别的最小单位为(GlcNAc)3,这与目前有关甲壳素酶产生(GlcNAc)2的研究结果一致。
|
( a:(GlcNAc)2水解产物的HPLC分析;b:(GlcNAc)3水解产物的HPLC分析;c:(GlcNAc)4水解产物的HPLC分析;d:(GlcNAc)5水解产物的HPLC分析;e:(GlcNAc)6水解产物的HPLC分析。甲壳寡糖标准品:N1: N-乙酰氨基葡萄糖;N2: 甲壳二糖;N3: 甲壳三糖;N4: 甲壳四糖;N5: 甲壳五糖;N6: 甲壳六糖。a: HPLC analysis of the hydrolysis products of (GlcNAc)2; b: HPLC analysis of the hydrolysis products of (GlcNAc)3; c: HPLC analysis of the hydrolysis products of (GlcNAc)4; d: HPLC analysis of the hydrolysis products of (GlcNAc)5; e: HPLC analysis of the hydrolysis products of (GlcNAc)6. Standards: N1: GlcNAc; N2: (GlcNAc)2; N3: (GlcNAc)3; N4: (GlcNAc)4; N5: (GlcNAc)5; N6: (GlcNAc)6. ) 图 5 SbChiAJ143酶解甲壳寡糖产物的高效液相色谱法分析 Fig. 5 HPLC analysis of the hydrolysis products from chitin oligosaccharide by SbChiAJ143 |
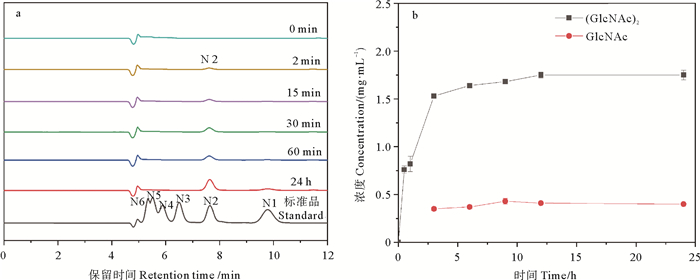
SbChiAJ143酶解过量的胶质甲壳素结果如图 6所示,水解产物为(GlcNAc)2,伴随着少量的GlcNAc。产物定量结果表明在酶解2 h内产物为单一的(GlcNAc)2,从3 h开始出现少量GlcNAc,且(GlcNAc)2随着反应时间的延长而增加,而GlcNAc浓度基本不变。在酶解6 h内,(GlcNAc)2的含量有明显的上升趋势,然后上升不再显著。反应24 h后,胶质甲壳素产(GlcNAc)2和GlcNAc含量分别达到1.75和0.40 mg/mL。在整个过程中产生的GlcNAc的量非常小,几乎是恒定的。可能此酶优先水解胶质甲壳素为偶数甲壳寡糖,然后迅速切割偶数甲壳寡糖为(GlcNAc)2。这对于甲壳素酶的酶切机理深入研究具有一定的参考价值。(GlcNAc)2是大多数GH18家族甲壳素酶的主要产物,并存在一定量的GlcNAc干扰纯度[32]。但SbChiAJ143水解胶质甲壳素时,GlcNAc的产量并没有随水解时间的延长而增加,这有利于单一(GlcNAc)2的工业化生产,也为快速生产高纯度的(GlcNAc)2提供了重要参考。
|
( a:胶质甲壳素水解产物的HPLC分析。甲壳寡糖标准品:N1:N-乙酰氨基葡萄糖;N2:甲壳二糖;N3:甲壳三糖;N4:甲壳四糖;N5:甲壳五糖;N6:甲壳六糖。b:胶质甲壳素水解产物的定量分析。a: HPLC analysis of the hydrolysis products of colloidal chitin. Standards: N1: GlcNAc; N2: (GlcNAc)2; N3: (GlcNAc)3; N4: (GlcNAc)4; N5: (GlcNAc)5; N6: (GlcNAc)6. b: Quantitative analysis of the hydrolysis products of colloidal chitin. ) 图 6 胶质甲壳素水解产物的分析 Fig. 6 Analysis of the hydrolysis products of colloidal chitin |
本研究成功克隆和表达了新的甲壳素酶SbChiAJ143,进化树分析和多序列比对表明该酶属于GH18家族。在pH=6.0的50 mmol/L磷酸盐缓冲液中55 ℃时该酶的活性最优,具有广泛的pH稳定性。大部分金属离子和SDS都可抑制该酶的活性,Ca2+和Ba2+对酶活有轻微的促进作用,该酶属于非金属依赖型酶。该酶的Km值为3.54 mg/mL,Vmax值为17.14 μmol/(min·mg),对底物胶质甲壳素的亲和力较好。该酶具有生产单一(GlcNAc)2的能力,如果能够通过基因定向进化或基因突变提高该酶的活性, 该酶在工业生产中将更具产业价值。
| [1] |
Tharanathan R N, Kittur F S. Chitin-the undisputed biomolecule of great potential[J]. Critical Reviews in Food Science and Nutrition, 2003, 43(1): 61-87. DOI:10.1080/10408690390826455 (  0) 0) |
| [2] |
Yeul V S, Rayalu S S. Unprecedented chitin and chitosan: A chemical overview[J]. Journal of Polymers and the Environment, 2013, 21(2): 606-614. DOI:10.1007/s10924-012-0458-x (  0) 0) |
| [3] |
Synowiecki J, Al-Khateeb N A. Production, properties, and some new applications of chitin and its derivatives[J]. Critical Reviews in Food Science and Nutrition, 2003, 43(2): 145-171. DOI:10.1080/10408690390826473 (  0) 0) |
| [4] |
Zhang A, Gao C, Wang J, et al. An efficient enzymatic production of N-acetyl-D-glucosamine from crude chitin powders[J]. Green Chemistry, 2016, 18(7): 2147-2154. DOI:10.1039/C5GC02242H (  0) 0) |
| [5] |
Lombard V, Ramulu H G, Drula E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2013, 42: D490-D495. (  0) 0) |
| [6] |
Funkhouser J D, Aronson N N. Chitinase family GH18: Evolutionary insights from the genomic history of a diverse protein family[J]. BMC Evolutionary Biology, 2007, 7: 96. DOI:10.1186/1471-2148-7-96 (  0) 0) |
| [7] |
Sahai A S, Manocha M S. Chitinases of fungi and plants: Their involvement in morphogenesis and host-parasite interaction[J]. FEMS Microbiology Reviews, 1993, 11(4): 317-338. DOI:10.1111/j.1574-6976.1993.tb00004.x (  0) 0) |
| [8] |
Lan X Q, Zhang X, Hu J H, et al. Cloning, expression, and characterization of a chitinase from the chitinolytic bacterium aeromonas Hydrophila strain SUWA-9[J]. Bioscience, Biotechnology, and Biochemistry, 2006, 70(10): 2437-2442. DOI:10.1271/bbb.60169 (  0) 0) |
| [9] |
Cohen-Kupiec R, Chet I. The molecular biology of chitin digestion[J]. Current Opinion in Biotechnology, 1998, 9(3): 270-277. DOI:10.1016/S0958-1669(98)80058-X (  0) 0) |
| [10] |
Miyashita K, Fujii T. Nucleotide sequence and analysis of a gene (chiA) for a chitinase from Streptomyces lividans 66[J]. Bioscie-nce, Biotechnology, and Biochemistry, 1993, 57(10): 1691-1698. DOI:10.1271/bbb.57.1691 (  0) 0) |
| [11] |
Blaak H, Schnellmann J, Walter S, et al. Characteristics of an exochitinase from Streptomyces olivaceoviridis, its corresponding gene, putative protein domains and relationship to other chitinases[J]. European Journal of Biochemistry, 1993, 214(3): 659-669. DOI:10.1111/j.1432-1033.1993.tb17966.x (  0) 0) |
| [12] |
Gao L, Sun J, Secundo F, et al. Cloning, characterization and substrate degradation mode of a novel chitinase from Streptomyces albolongus ATCC 27414[J]. Food Chemistry, 2018, 261: 329-336. DOI:10.1016/j.foodchem.2018.04.068 (  0) 0) |
| [13] |
Alahuhta M, Xu Q, Bomble Y J, et al. The unique binding mode of cellulosomal CBM4 from Clostridium thermocellum cellobiohydrolase A[J]. Journal of Molecular Biology, 2010, 402(2): 374-387. DOI:10.1016/j.jmb.2010.07.028 (  0) 0) |
| [14] |
Johnson P E, Joshi M D, Tomme P, et al. Structure of the N-terminal cellulose-binding domain of Cellulomonas fimi CenC determined by nuclear magnetic resonance spectroscopy[J]. Biochemistry, 1996, 35(45): 14381-14394. DOI:10.1021/bi961612s (  0) 0) |
| [15] |
Sitrit Y, Vorgias C E, Chet I, et al. Cloning and primary structure of the chiA gene from Aeromonas caviae[J]. Journal of Bacteriology, 1995, 177(14): 4187-4189. DOI:10.1128/jb.177.14.4187-4189.1995 (  0) 0) |
| [16] |
Barboza-Corona J E, Nieto-Mazzocco E, Velázquez-Robledo R, et al. Cloning, sequencing, and expression of the chitinase gene chiA74 from Bacillus thuringiensis[J]. Applied and Environmental Microbiology, 2003, 69(2): 1023-1029. DOI:10.1128/AEM.69.2.1023-1029.2003 (  0) 0) |
| [17] |
Thamthiankul S, Suan-Ngay S, Tantimavanich S, et al. Chitinase from Bacillus thuringiensis subsp. Pakistani[J]. Applied Microbiology and Biotechnology, 2001, 56(3-4): 395-401. DOI:10.1007/s002530100630 (  0) 0) |
| [18] |
Meinke A, Braun C, Gilkes N R, et al. Unusual sequence organization in CenB, an inverting endoglucanase from Cellulomonas fimi[J]. Journal of Bacteriology, 1991, 173(1): 308-314. DOI:10.1128/jb.173.1.308-314.1991 (  0) 0) |
| [19] |
Candussio A, Schmid G, Böck A. Biochemical and genetic analysis of a maltopentaose-producing amylase from an alkaliphilic Gram-positive bacterium[J]. European Journal of Biochemistry, 1990, 191(1): 177-185. DOI:10.1111/j.1432-1033.1990.tb19108.x (  0) 0) |
| [20] |
Watanabe T, Kobori K, Miyashita K, et al. Identification of glutamic acid 204 and aspartic acid 200 in chitinase A1 of Bacillus circulans WL-12 as essential residues for chitinase activity[J]. The Journal of Biological Chemistry, 1993, 268(25): 18567-18572. DOI:10.1016/S0021-9258(17)46665-8 (  0) 0) |
| [21] |
Nakamura T, Mine S, Hagihara Y, et al. Structure of the catalytic domain of the hyperthermophilic chitinase from Pyrococcus furiosus[J]. Acta Crystallographica Section F Structural Biology and Crystallization Communications, 2007, 63(1): 7-11. DOI:10.1107/S1744309106051773 (  0) 0) |
| [22] |
Davies G, Henrissat B. Structures and mechanisms of glycosyl hydrolases[J]. Structure, 1995, 3(9): 853-859. DOI:10.1016/S0969-2126(01)00220-9 (  0) 0) |
| [23] |
Pradeep G C, Choi Y H, Choi Y S, et al. An extremely alkaline novel chitinase from Streptomyces sp. CS495[J]. Process Biochemistry, 2014, 49(2): 223-229. DOI:10.1016/j.procbio.2013.12.002 (  0) 0) |
| [24] |
Jiang X Y, Chen D C, Hong S L, et al. Identification, characterization and functional analysis of a GH-18 chitinase from Streptomyces roseolus[J]. Carbohydrate Polymers, 2012, 87(4): 2409-2415. DOI:10.1016/j.carbpol.2011.11.008 (  0) 0) |
| [25] |
Ray L, Panda A N, Mishra S R, et al. Purification and characterization of an extracellular thermo-alkali stable, metal tolerant chitinase from Streptomyces chilikensis RC1830 isolated from a brackish water lake sediment[J]. Biotechnology Reports, 2019, 21: 311. (  0) 0) |
| [26] |
Lee S G, Koh H Y, Han S J, et al. Expression of recombinant endochitinase from the antarctic bacterium, sanguibacter antarcticus KOPRI 21702 in Pichia pastoris by codon optimization[J]. Protein Expression and Purification, 2010, 71(1): 108-114. DOI:10.1016/j.pep.2010.01.017 (  0) 0) |
| [27] |
Suma K, Podile A R. Chitinase a from Stenotrophomonas maltophilia shows transglycosylation and antifungal activities[J]. Bioresource Technology, 2013, 133: 213-220. DOI:10.1016/j.biortech.2013.01.103 (  0) 0) |
| [28] |
Mander P, Cho S S, Choi Y H, et al. Purification and characteri-zation of chitinase showing antifungal and biodegradation properties obtained from Streptomyces anulatus CS242[J]. Archives of Pharmacal Research, 2016, 39(7): 878-886. DOI:10.1007/s12272-016-0747-3 (  0) 0) |
| [29] |
Yang S Q, Fu X, Yan Q J, et al. Cloning, expression, purification and application of a novel chitinase from a thermophilic marine bacterium Paenibacillus barengoltzii[J]. Food Chemistry, 2016, 192: 1041-1048. DOI:10.1016/j.foodchem.2015.07.092 (  0) 0) |
| [30] |
Guo X X, Xu P, Zong M H, et al. Purification and characterization of alkaline chitinase from Paenibacillus pasadenensis CS0611[J]. Chinese Journal of Catalysis, 2017, 38(4): 665-672. DOI:10.1016/S1872-2067(17)62787-6 (  0) 0) |
| [31] |
Karthik N, Binod P, Pandey A. Purification and characterisation of an acidic and antifungal chitinase produced by a Streptomyces sp.[J]. Bioresource Technology, 2015, 188: 195-201. DOI:10.1016/j.biortech.2015.03.006 (  0) 0) |
| [32] |
Ilankovan P, Hein S, Ng C H, et al. Production of N-acetyl chitobiose from various chitin substrates using commercial enzymes[J]. Carbohydrate Polymers, 2006, 63(2): 245-250. DOI:10.1016/j.carbpol.2005.08.060 (  0) 0) |
2. Laboratory for Marine Drugs and Bioproducts, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China
 2022, Vol. 52
2022, Vol. 52


