2. 中国科学院青岛生物能源与过程研究所,中国科学院生物燃料重点实验室,山东 青岛 266101
蓝细菌是地球上一类能够进行放氧光合作用的原核微生物,能够利用光能将CO2和水转化为各类有机物,并释放氧气,在地球物质循环与能量流动中起着重要的作用,它们广泛分布于淡水、海洋、陆地甚至各类极端环境中(如贫瘠的土壤、盐湖、火山和热泉等)[1]。相较于真核微藻和高等植物,蓝细菌具有结构简单、易于培养、生长迅速且遗传操作相对简单等优点,长期以来被作为研究光合作用机制以及光合自养生物环境适应性的模式种类而被广泛研究[2]。另一方面,相较于大肠杆菌、枯草芽孢杆菌和酿酒酵母等模式异养微生物,蓝细菌又因其高效的光合作用能力,被认为是极具潜力的光合微生物细胞工厂之一[3-4]。
很多研究已表明,蓝细菌在盐胁迫的条件下在细胞内迅速从头合成一类小分子相容性物质(如海藻糖、蔗糖、甘油葡糖苷[glucosylglycerol, GG]、甘油葡萄糖酸酯[glucosylglycerate, GGA]、甘氨酸甜菜碱[glycine betaine, GB]等)来抵抗逆境[5]。基于这一生理特性,国内外多个研究小组通过基因工程改造,构建了一系列高效合成相容性物质的蓝细菌基因工程菌株[6-10]。以被广泛用作食品调味剂和微生物发酵原料的蔗糖为例,通过过量表达蔗糖合成关键基因sps(蔗糖磷酸合酶基因),Synechocystis sp. PCC 6803(集胞藻PCC 6803)盐胁迫下蔗糖合成能力提高了将近3倍[7]。在Synechococcus elongatus PCC 7942(细长聚球藻PCC 7942,S. elongatus PCC 7942)中过表达sps,盐胁迫条件下藻细胞内蔗糖积累量也提高了将近1倍[11];进一步向S. elongatus PCC 7942中引入一个来自大肠杆菌的蔗糖转运蛋白CscB,成功实现了蔗糖从胞内向胞外的高效分泌,细胞蔗糖合成能力提高近10倍;该过程中通过光合作用固定的CO2,其中约80%都被导向了蔗糖合成[12]。目前,基因工程蓝细菌达到的最高蔗糖产率为36.1 mg·L-1·h-1,其相应的理论产率已超过部分地区甘蔗蔗糖生产水平,展现出利用基因工程蓝细菌规模化生产蔗糖的潜力[12]。而在蓝细菌GG细胞工厂的构建过程中,通过敲除Synechocystis sp. PCC 6803中ggtCD基因,造成细胞GG吸收转运蛋白无法组装,成功实现了GG从胞内向细胞外的分泌,使得工程菌株GG的产量较野生型菌株提高了约1.5倍;进一步敲除一个相关转录因子基因ggpR,工程菌株△ggtCD△ggpR中GG的产量进一步提高16% [13]。
然而,与其它利用基因工程蓝细菌生产各种燃料分子或化学品所遇到的问题一样,通过对合成途径关键基因的过表达、竞争途径的敲除、基因转录表达效率优化等传统研究手段,已很难在数量级上进一步提高蓝细菌中目标相容性物质的产量。这迫切需要我们全面认识蓝细菌响应盐胁迫积累相容性物质的生理过程,尤其是深入了解其代谢调控的内在机制,进而指导新的基因工程改造策略、培养条件优化策略,以及产品获取、加工策略,实现蓝细菌相容性物质合成能力的继续提升以及相关生产工艺的建立与优化。在本文中,我们对蓝细菌应对盐胁迫合成相容性物质调控机制的研究现状进行了总结,从基因表达(包括转录、翻译)和酶学活性两个层面对该领域研究进展进行了详细阐述,并对其未来发展方向进行了展望。这对我们进一步认识微生物响应、适应盐胁迫的分子机理和指导高效蓝细菌细胞工厂构建等都具有重要参考价值。
1 蓝细菌盐胁迫下合成相容性物质的种类盐胁迫是环境中一种常见的非生物胁迫,其主要通过增加外界环境的渗透势,打破细胞内外的渗透平衡,从而引起对细胞膜、细胞器结构的损伤。此外,胞外离子向胞内的大量涌入,抑制一些代谢关键酶功能的发挥,从而严重影响细胞正常的生命活动过程[5]。为了应对盐胁迫,蓝细菌主要从两方面来调整细胞活动:一方面,细胞通过膜上的离子转运蛋白主动泵出无机盐离子(如Na+、Cl-),以降低胞内离子浓度,进而维持细胞内的离子稳态;另一方面,蓝细菌在细胞内迅速合成并积累相容性物质,以实现细胞内外的渗透压平衡[14]。
相容性物质是一类易溶于水的低分子量有机化合物,通常不带电荷,能够在细胞内积累到较高浓度而不干扰细胞的新陈代谢过程[15]。细胞内高浓度的相容性物质一方面可以平衡细胞内外的渗透势,防止细胞脱水,维持细胞的体积和形态;另一方面,这些相容物还可以稳定细胞内的大分子结构(如蛋白质、核酸等),减小高浓度的盐离子对其结构的破坏作用[16-18]。目前,微生物中已发现的相容性物质按照结构特点可以分为糖类(如蔗糖)、糖苷类(如GG)、氨基酸(如脯氨酸)及其衍生物(如四氢嘧啶)、多元醇(如山梨糖醇)及其衍生物(如邻甲基肌醇)、甲胺类(如N-三甲胺氧化物)等[19, 20]。
通过对多株蓝细菌的研究发现,蓝细菌中合成的相容性物质种类与其盐耐受程度有一定相关性(见表 1)[14, 21]。一般来说,低耐盐度蓝细菌(最高耐受0.6 mol/L NaCl,相当于海水盐度)主要合成海藻糖和/或蔗糖作为相容性物质,如S. elongatus PCC 7942在盐胁迫下专一性合成蔗糖,而Anabaena sp. PCC 7120(鱼腥藻PCC 7120,A. sp. PCC 7120)则同时合成海藻糖和蔗糖;中等耐盐蓝细菌(最高耐受1.8 mol/L NaCl,相当于3倍海水盐度)往往可以合成多种相容性物质,多以GG作为主要相容性物质,以海藻糖、蔗糖、GGA等作为次要相容性物质,如Synechocystis sp. PCC 6803在盐胁迫下同时合成蔗糖与GG来抵抗逆境;高盐耐受性蓝细菌(最高能耐受3 mol/L NaCl)则主要合成甘氨酸甜菜碱(GB)作为主要相容性物质,如Spirulina subsalsa(盐泽螺旋藻)。除了上述相容性物质,最新研究表明,海洋蓝细菌Trichodesmium erythraeum IMS101(汉氏束毛藻IMS101)合成一种新的相容性物质——高丝氨酸甜菜碱来应对盐胁迫[22]。
|
|
表 1 部分蓝细菌中盐胁迫诱导合成相容性物质种类情况 Table 1 Compatible solutes synthesized in some cyanobacteria |
在蓝细菌合成的相容性物质中,GG、蔗糖和海藻糖因其在皮肤护理、功能食品、微生物发酵原料等方面的应用潜力,已成为当前基因工程蓝细菌生物合成相容性物质的主要聚焦点[14, 38]。对构建高效蓝细菌细胞工厂的迫切需求,要求我们对盐胁迫诱导蓝细菌相容性物质积累的分子调控机理有更为深入的认识。我们在此对相关研究进行了全面总结,重点阐述蓝细菌积累GG、蔗糖和海藻糖的代谢调控机制,尝试探索其中的共性规律,为构建新一代高效蓝细菌细胞工厂提供新指导。
2.1 蓝细菌中GG代谢途径及其调控在盐胁迫的条件下,蓝细菌细胞中相容性物质(如GG)的积累实际上是合成和降解协同作用的结果,通过这种方式,细胞动态地调节胞内相容性物质的水平,以适应环境盐度的变化。对GG而言,其在蓝细菌中的代谢过程已被清楚鉴定(见图 1)。GG的合成包含两步反应:首先,ADP-葡萄糖和甘油-3-磷酸在GG磷酸合酶GgpS(EC 2.4.1.213)的作用下合成GG-6-磷酸,并释放一个ADP分子;随后,GG磷酸磷酸酶GgpP(EC 3.1.3.69)催化GG-6-磷酸水解产生GG[39-40]。GG的降解则是由gghA基因编码的GG水解酶GghA(EC 3.2.1.21)催化完成,该酶将1分子的GG水解为1分子的甘油和1分子的葡萄糖(见图 1)[41-42]。
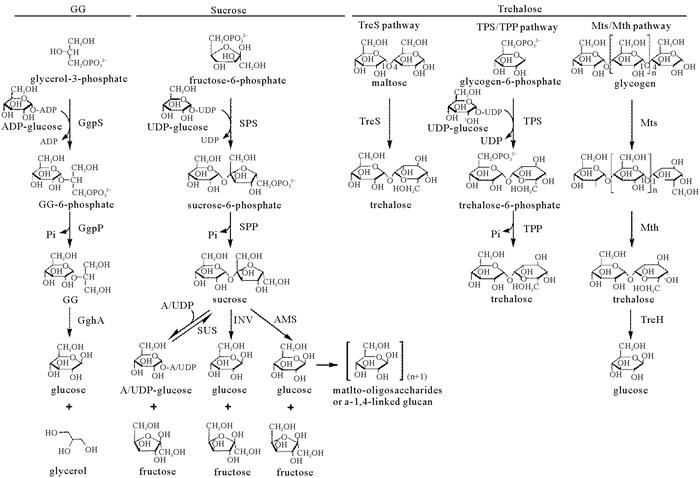
|
(甘油葡糖苷:glucosylglycerol,简称GG;GgpS:GG磷酸合酶;GgpP:GG磷酸磷酸酶;GghA:GG水解酶;SPS:蔗糖磷酸合酶;SPP:蔗糖磷酸磷酸酶;SUS:蔗糖合酶;INV:蔗糖酶;AMS:淀粉蔗糖酶;TreS:海藻糖合酶;TPS:海藻糖磷酸合酶;TPP:海藻糖磷酸磷酸酶;Mts:麦芽寡糖基海藻糖合酶;Mth:麦芽寡糖基海藻糖水解酶;TreH:海藻糖酶。glucosylglycerol is abbreviated as GG; GgpS: GG phosphate synthase; GgpP: GG phosphate phosphatase; GghA: GG hydrolase; SPS: sucrose phosphate synthase; SPP: sucrose phosphate phosphatase; SUS: sucrose synthase; INV: invertase; AMS: amylosucrase; TreS: trehalose synthase; TPS: trehalose phosphate synthase; TPP: trehalose phosphate phosphatase; Mts: maltooligosyltrehalose synthase; Mth: maltooligosyltrehalose trehalohydrolase; TreH: trehalase. ) 图 1 蓝细菌中甘油葡糖苷、蔗糖和海藻糖的代谢途径 Fig. 1 Metabolic pathways of glucosylglycerol, sucrose, and trehalose in cyanobacteria |
与已被深入研究的GG代谢途径相比,盐诱导GG积累调控机制的研究仍然有限,相关研究主要集中在Synechocystis sp. PCC 6803中。淡水单细胞蓝细菌Synechocystis sp. PCC 6803在面对盐胁迫时,细胞内会迅速合成两种相容性物质——GG(主要)和蔗糖[7]。研究表明,Synechocystis sp. PCC 6803中盐诱导GG合成的调控发生在多个水平上——关键基因的转录、翻译和关键酶的生物化学活性(见图 2)。在标准培养条件下(无盐胁迫),GG合成限速酶GgpS的基因转录和翻译维持基础水平,细胞内几乎没有GG合成;而在NaCl胁迫条件下,ggpS基因的转录迅速上调,且呈现明显的盐浓度正相关性,与之对应,GgpS的表达量在胁迫后明显提高,GG开始迅速积累[43, 44]。进一步研究发现,RNA聚合酶选择性σ因子SigF突变株中,盐胁迫条件下ggpS基因的转录水平较野生型明显降低,同时突变株应对盐胁迫的能力也明显受损,表明SigF因子参与了ggpS基因的盐依赖性转录调节[43]。此外,敲除转录因子LexA和GgpR,ggpS基因的转录均明显上调,表明LexA和GgpR很可能作为转录抑制因子参与了ggpS的表达调控[13, 45-46]。值得注意的是,转录因子LexA的调控作用可能是全局性的,很多研究表明LexA参与了多种细胞生理过程的调控(如在Synechocystis sp. PCC 6803中参与了涉及GG合成和吸收的多个相关基因的表达调控;在大肠杆菌中作为SOS应急反应的重要调节因子之一,调控了约50个相关基因的表达)[45, 47];而对于GgpR,其编码基因位于ggpS上游,可能介导了对GG合成的特异性调控[46]。但多个调控因子之间如何协同配合完成基因表达调控还有待深入研究。除了转录翻译水平的调控,Hagemann等的研究还表明Synechocystis sp. PCC 6803中GG的合成调控也可能发生在基因的翻译后水平。他们发现利用氯霉素阻断细胞中的蛋白合成后,对细胞施加684 mmol·L-1 NaCl胁迫,仍然导致细胞内GG水平的迅速上升[44]。2011年,Novak等的研究进一步发现这种翻译后水平的调控主要通过胞内离子浓度对GgpS酶活力的改变来实现,并且核酸在GgpS酶活发挥中起着抑制作用[48]。在正常培养条件下,胞内基础性表达的GgpS蛋白可能通过静电作用与核酸非特异性结合,致使GgpS处于酶活抑制状态,该状态下细胞几乎不合成GG;而当细胞遇到盐胁迫环境后,细胞内升高的离子浓度破坏了GgpS与核酸的静电结合,释放出活性状态的GgpS蛋白,进而催化GG的迅速合成[48]。
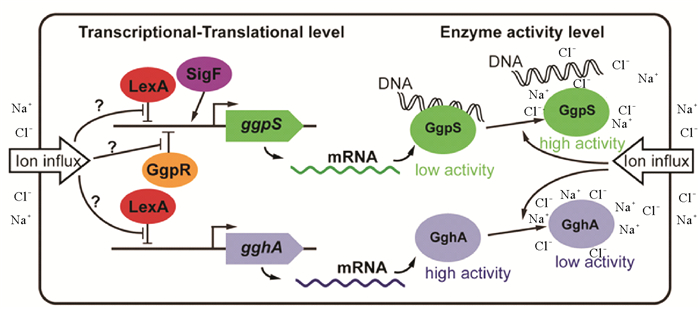
|
图 2 NaCl诱导Synechocystis sp. PCC 6803积累甘油葡糖苷的调控机制示意图 Fig. 2 Schematic diagram of the regulatory mechanism of NaCl-induced glucosylglycerol accumulation in Synechocystis sp. PCC 6803 |
在GG的降解方面,研究表明Synechocystis sp. PCC 6803中GG水解酶基因gghA的表达和GghA蛋白的酶学活性同样受到盐胁迫的影响(见图 2)。在标准培养条件下(无盐胁迫),细胞中gghA的转录和翻译维持在低水平,而在550 mmol·L-1 NaCl胁迫条件下,gghA基因的转录迅速上调[49]。进一步研究发现转录因子LexA的敲除后,gghA基因的转录明显上调,表明LexA可能作为转录抑制因子也参与了对gghA的表达调控[45]。gghA基因转录上调是否意味着细胞中GG降解也随之加快呢?Kirsch等的研究发现情况并不是这样。他们对GghA酶学活性的分析表明,GghA酶活力与GgpS一样也受到了胞内离子浓度的调控,然而其调控方式与GgpS正好相反,即胞内离子浓度的升高,明显抑制了GghA的活性,而离子浓度的下降,则激活GghA的活力[42]。这样,由高离子浓度引起的对GghA活性的抑制作用,一方面很大程度上抵消了gghA基因上调表达对胞内GG积累的影响;另一方面,它与高离子浓度介导的GgpS活性激活一起,协同调控,共同实现了胞内GG含量迅速积累。这样,蓝细菌通过胞内离子浓度对GG合成/降解关键酶的协同调控,实现了对环境盐度变化的动态适应。
与对基因的转录、翻译调控相比,这种通过离子浓度介导的GG代谢关键酶学活性的协调调控,保证了蓝细菌能以一种更为快速的方式实现细胞生理、生化响应,因此也很可能是微生物界普遍采用的一种盐胁迫环境适应调控机制。
2.1.2 Synechococcus sp. PCC 7002中GG代谢的调控除了Synechocystis sp. PCC 6803,GG的代谢调控也在模式海洋蓝细菌Synechococcus sp. PCC 7002(聚球藻PCC 7002,S. sp. PCC 7002)中被研究,但相关研究仅限于GG的合成过程,并未涉及GG降解过程。S. sp. PCC 7002是一种中等耐盐蓝细菌,其在面对盐胁迫时,会在胞内能合成多种相容性物质——GG(主要)、蔗糖以及GGA[32]。研究表明,S. sp. PCC 7002中GG合成的调控与Synechocystis sp. PCC 6803明显不同,其GG合成仅在基因表达水平受到调控,没有发现类似Synechocystis sp. PCC 6803中酶学水平上的调控[44]。当S. sp. PCC 7002细胞受到684 mmol·L-1 NaCl胁迫后,ggpS基因的转录水平迅速提高,在40 min左右达到最高水平;与转录上调相对应的是,盐胁迫后的24 h内,胞内GgpS蛋白含量也显著上升,胞内GG的单位含量也在5 h左右达到峰值,并在随后近20 h内维持稳定[44]。而在培养基中加入氯霉素阻断蛋白合成,盐胁迫下的S. sp. PCC 7002细胞提取液中GgpS活性不受反应体系中NaCl的影响,表明该蓝细菌中GgpS蛋白调控方式与Synechocystis sp. PCC 6803中不同,其酶活并不受到离子激活[44]。
由此可见,不同蓝细菌种类中,盐胁迫诱导GG合成的调控机制并不完全相同。有的种类主要依靠对关键基因的转录、翻译的调控来加速GG合成,有的则主要依赖酶学活性调控来实现GG的快速积累。
2.2 蓝细菌中蔗糖代谢途径及其调控如前所述,很多蓝细菌在盐胁迫下合成蔗糖来作为相容性物质。与GG合成相似,蓝细菌中蔗糖的合成也是通过两步酶学反应来实现(见图 1):首先,UDP-葡萄糖和果糖-6-磷酸在蔗糖磷酸合酶SPS(EC 2.4.1.14)的作用下,合成蔗糖-6-磷酸,同时释放一分子UDP,该步反应是蔗糖合成的限速步骤;随后,蔗糖-6-磷酸在蔗糖磷酸磷酸酶SPP(EC 3.1.3.24)的作用下水解生成蔗糖[24, 50]。相比蔗糖合成较为单一的途径,目前为止,蓝细菌中已鉴定出三条蔗糖降解途径(见图 1):(1)由蔗糖酶INV(EC 3.2.1.26)催化的蔗糖降解,该过程不可逆地催化蔗糖水解成游离的葡萄糖和果糖,是目前蓝细菌中最为广泛采用的一种蔗糖降解策略[51];(2)由蔗糖合酶SUS(EC 2.4.1.13)催化的蔗糖降解,SUS主要存在于能形成异形胞的蓝细菌种类中(如A. sp. PCC 7120),在藻细胞的固氮过程中起着重要的作用[52-54]。SUS可催化从蔗糖、(A/U)DP到(A/U)DP-葡萄糖、果糖的可逆转化,但在蓝细菌细胞内,该酶主要催化蔗糖的裂解反应而非合成反应[53];(3)由淀粉蔗糖酶AMS(EC 2.4.1.4)催化的蔗糖降解,该途径由Perez-Cenci等2014年在S. sp. PCC 7002中发现。AMS可催化蔗糖分解为游离果糖和葡萄糖,葡萄糖进一步通过糖苷键连到寡糖或者糖原上[55]。
目前,关于蓝细菌中盐诱导蔗糖积累的调控机制研究主要集中在一些模式种类中,如Synechocystis sp. PCC 6803、A. sp. PCC 7120和S. elongatus PCC 7942。与GG积累的调控相似,盐胁迫诱导蔗糖积累的调控同样发生在多个水平上——关键基因的转录、翻译和关键酶的生物化学活性,但是不同蓝细菌种类采用的调控方式又不完全相同。
2.2.1 Anabaena sp. PCC 7120中蔗糖代谢的调控有关蓝细菌中蔗糖代谢调控的研究最早在淡水固氮种类A. sp. PCC 7120中展开[56]。鱼腥藻A. sp. PCC 7120属于丝状固氮蓝细菌,耐盐能力较低,其在面对盐胁迫时细胞内能快速合成蔗糖(主要)和海藻糖两种相容性物质。Salerno等的研究发现,在标准培养条件下(无盐胁迫),A. sp. PCC 7120中蔗糖合成限速酶基因spsA的转录和翻译维持低的基础水平,细胞内几乎没有蔗糖合成;而在80 mmol·L-1 NaCl胁迫条件下,spsA基因的转录、翻译迅速上调,胞内蔗糖浓度也迅速增加[25]。这一结果表明A. sp. PCC 7120中蔗糖合成在基因表达水平上受到了盐胁迫的调控。Ehira等的研究进一步发现,Hik-Rre双元信号转导系统可能参与了A. sp. PCC 7120中蔗糖合成的调控[57]。Hik-Rre即组氨酸激酶-应答调控蛋白,该双元信号转导系统是包括蓝细菌在内的原核微生物感知、传导并响应各种环境变化的重要途径,Hik负责感知外界信号,Rre接收Hik传递的信号后,通常借助转录因子调控下游过程[58]。A. sp. PCC 7120中的orrA基因(alr3768)编码一个NarL型的Rre蛋白OrrA,研究发现,orrA敲除株中,spsA的转录不再受盐胁迫调控;而orrA过表达菌株中,spsA基因的转录在无盐胁迫条件下明显上调[57]。此外,RNA聚合酶σ因子SigB2敲除后,A. sp. PCC 7120细胞在盐胁迫条件下,其spsA基因转录上调水平在胁迫初期较野生型明显降低,表明SigB2也参与到了蓝细菌对盐胁迫的早期转录调控[57]。除了在基因表达水平上的调控,Hagemann等还对A. sp. PCC 7120中SPS酶学活性也进行了研究,他们发现细胞破碎液中SPS酶活并没有受到反应体系中盐浓度的影响,这表明盐离子浓度的变化并不能直接调控A. sp. PCC 7120中蔗糖合成限速酶SPS的酶学活性[24]。综上A. sp. PCC 7120中盐胁迫诱导的蔗糖合成的调控主要发生在关键基因的表达水平。
2.2.2 Synechocystis sp. PCC 6803中蔗糖代谢的调控作为蓝细菌研究中的模式种类,Synechocystis sp. PCC 6803在盐胁迫下蔗糖(次要相容性物质)代谢调控机制,也获得了研究人员的关注。研究发现,与A. sp. PCC 7120相似,Synechocystis sp. PCC 6803中蔗糖合成的调控主要发生在sps基因的表达水平。在684 mmol·L-1 NaCl胁迫下,sps基因的转录水平迅速上调[59]。Song等进一步发现,敲除双元信号转导蛋白Rre39的编码基因slr1588后,sps基因的转录水平上调,表明Rre39参与了对sps的表达调控[60]。此外,对Synechocystis sp. PCC 6803中SPS酶活的研究显示,纯化的SPS蛋白在含有不同NaCl浓度的反应体系中,其活性并无明显差别,不受离子浓度的调控[24]。
研究表明,Synechocystis sp. PCC 6803中的蔗糖降解过程同样在基因表达水平受到了调控。Billis等的转录组学分析发现,550 mmol·L-1 NaCl胁迫Synechocystis sp. PCC 6803 1 h后,细胞内inv基因(蔗糖酶基因)的转录水平明显下降,表明盐胁迫抑制了inv基因的表达[49]。Kirsch等进一步详细分析了蔗糖酶INV的酶学活性。结果发现,纯化的INV蛋白的活性受到盐浓度的影响。与不添加NaCl的条件相比,当反应体系中NaCl浓度升高到75 mmol·L-1时,INV活性降低到约初始水平的50%,而当NaCl浓度继续升高到145 mmol·L-1时,INV活性仅残留15%[61]。由此可见,Synechocystis sp. PCC 6803中蔗糖降解基因inv的表达和关键酶INV的活性同时受到了盐胁迫的调控。
综上所述,蔗糖作为Synechocystis sp. PCC 6803中次要的相容性物质,其盐诱导的积累主要是通过迅速sps基因的转录上调、inv基因的转录下调,以及对INV酶活性的抑制来实现的。
2.2.3 Synechococcus elongatus PCC 7942中蔗糖代谢的调控S. elongatus PCC 7942是一种被广为研究的低耐盐性淡水蓝细菌,在盐胁迫下其合成蔗糖作为唯一相容性物质抵御逆境。近期,其蔗糖代谢调控机制(见图 3)也被系统研究[62]。在S. elongatus PCC 7942中,蔗糖合成的关键酶SPS和SPP是以融合蛋白(记作7942-SPS)的形式存在的,由Synpcc7942_0808基因编码(记作Syn0808)。研究发现,细胞经300 mmol·L-1 NaCl胁迫0.5 h后,Syn0808基因的转录水平明显上调;此外蛋白水平分析也表明,相同胁迫条件下7942-SPS蛋白含量也逐渐升高,在胁迫8 h达到初始水平的2倍[62]。尽管Syn0808基因的表达在盐胁迫条件下受到了一定的调控作用,但这种7942-SPS水平有限的升高,似乎并不足以解释蔗糖的快速积累过程(2 h内可到达稳定水平的80%),暗示其蔗糖合成可能还有其它调控方式。Hagemann等发现,在S. elongatus PCC 7942近似种类Synechococcus elongatus PCC 6301(细长聚球藻PCC 6301,S. elongatus PCC 6301)中,细胞破碎液中SPS的活性明显受到NaCl的激活,暗示7942-SPS活性也可能受到离子浓度的直接调控[24]。Liang等进一步的酶学分析表明,异源表达纯化的7942-SPS蛋白其活性能被高浓度的盐离子显著激活,在200 mmol·L-1 NaCl浓度条件下,7942-SPS的单位酶活性较无盐条件提高了近40倍;进一步研究表明这种激活调控发生在SPS结构域而非SPP结构域上[62]。但高盐离子激活SPS结构域的深入机制并没能得到阐明,是类似Synechocystis sp. PCC 6803中GgpS的离子激活机制,还是依赖其它方式,仍有待后续研究。相比于基因表达水平的调控,这种离子浓度介导的7942-SPS酶活调控保证了S. elongates PCC 7942能以一种更为快速的方式实现细胞的生化响应,从而迅速应对环境中的盐度变化。有趣的是,该蓝细菌中的蔗糖降解酶INV像Synechocystis sp. PCC 6803中一样,其活性也受到离子浓度变化的影响,且以与7942-SPS完全相反的方式受到调控,即高离子浓度抑制其活性,低离子浓度促进其活性。这样,S. elongatus PCC 7942细胞内动态变化的离子浓度以完全相反的方式调控着蔗糖合成和降解的关键酶,从而实现对环境盐度变化的动态响应。
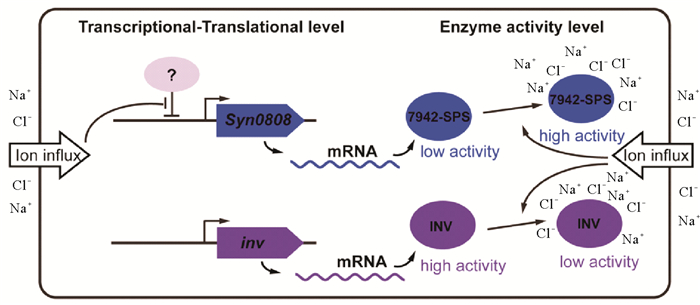
|
图 3 NaCl诱导Synechococcus elongatus PCC 7942积累蔗糖的调控机制示意图 Fig. 3 Schematic diagram of the regulatory mechanism of NaCl-induced sucrose accumulation in Synechococcus elongatus PCC 7942 |
综上所述,对于仅以蔗糖为唯一相容性物质的S. elongates PCC 7942,其盐诱导的蔗糖积累主要以这种“离子浓度介导的酶活协同调控”方式进行调控,其合成活性与降解活性的调控以一正一反的方式相互配合,维持细胞内蔗糖含量的动态平衡。
2.3 蓝细菌中海藻糖代谢途径及其调控很多蓝细菌种类在盐胁迫条件下也能合成海藻糖。与蔗糖和GG的合成途径相比,蓝细菌中海藻糖的合成途径相对更加多样化。目前已在不同蓝细菌中鉴定出三条海藻糖合成途径(见图 1):(1)TreS途径,已在Spirulina platensis NIES-39(钝顶螺旋藻NIES-39,S. platensis NIES-39)、Cyanothece sp. PCC 7424(蓝杆藻PCC 7424)、Microcoleus chthonoplastes PCC 7420(原型微鞘藻PCC 7420)等种类中发现[5]。海藻糖合酶TreS(EC 5.4.99.16)能够通过分子内转糖基作用,特异性的将麦芽糖的α-1, 4糖苷键转化为α-1, 1糖苷键生成海藻糖;(2)TPS/TPP途径,该途径与蓝细菌GG(GgpS/GgpP途径)和蔗糖(SPS/SPP途径)合成过程类似,由一个合酶(synthase)和一个磷酸酶(phosphatase)催化完成。首先,在海藻糖磷酸合酶TPS(EC 2.4.1.15)催化下,UDP-葡萄糖和葡萄糖-6-磷酸转化为海藻糖-6-磷酸并释放一分子UDP,随后海藻糖-6-磷酸进一步被海藻糖磷酸磷酸酶TPP(EC 3.1.3.12)水解,生成海藻糖。该途径目前仅发现于海洋固氮蓝细菌Crocosphaera watsonii WH8501(瓦氏鳄球藻WH8510,C. watsonii WH8501)中。对C. watsonii WH8501基因组的分析表明,该蓝细菌中TPS和TPP由同一个基因编码(otsAB基因),产生一个融合型的TPS-TPP蛋白[29];(3)Mts/Mth途径(或TreY/TreZ途径),该途径主要存在于A. sp. PCC 7120和近似种类中(如Nostoc flagelliforme[发状念珠藻,N. flagelliforme]、S. platensis NIES-39)[26, 27]。这一途径由两种酶催化——麦芽寡糖基海藻糖合酶Mts(也称TreY,EC 5.4.99.15)和麦芽寡糖基海藻糖水解酶Mth(也称TreZ,EC 3.2.1.141)。细胞利用葡萄糖聚合物(如糖原和麦芽低聚糖)作为海藻糖合成的前体,由Mts首先将分子骨架中的α-1, 4糖苷键转换成α-1, 1糖苷键,随后Mth水解上述高分子量前体,释放出海藻糖分子[26]。相对于研究较多的海藻糖合成途径,目前在蓝细菌中仅发现一种海藻糖降解途径,即由海藻糖酶TreH催化水解海藻糖,生成两个葡萄糖分子。该途径中treH基因通常与负责海藻糖合成的mts和mth基因位于同一个操纵子中[26]。
与GG和蔗糖代谢调控研究相比,人们对盐胁迫如何诱导蓝细菌积累海藻糖的认识还相对有限,目前只在少数种类中有过零星的研究。如对N. flagelliforme、S. platensis NIES-39和C. watsonii WH8501的研究发现,尽管这三种蓝细菌采用不同的途径催化海藻糖的合成,其海藻糖合成关键基因(即Nostoc的mts和mth、Spirulina的mth和C. watsonii的ostAB)的转录均受到了盐胁迫的诱导上调[27, 29, 34]。而负责海藻糖降解的海藻糖酶基因treH因与mts、mth位于同一操纵子中,其表达水平也会和mts、mth一样受到盐胁迫的诱导上调(见图 4)[26, 28]。2009年,Yoshida和Sakamoto分析了丝状固氮蓝细菌Nostoc punctiforme IAM M-15(点状念珠藻IAM M-15,N. punctiforme IAM M-15)中Mts、Mth和TreH酶学活性[28],结果发现细胞破碎液中Mts、Mth酶活并没有受到反应体系中盐浓度的影响,表明该蓝细菌中海藻糖的合成不受盐离子浓度的直接调控。然而有趣的是,海藻糖酶TreH的酶活性却受到盐浓度的明显影响。在50 mmol·L-1 NaCl条件下,不论是利用细胞破碎液或是异源表达纯化的蛋白,其TreH活性都会随着反应体系中盐浓度的升高而迅速下降[28]。因此,在N. punctiforme IAM M-15中,盐胁迫条件下海藻糖在胞内的积累主要是通过对mts、mth基因表达的诱导以及对TreH酶活的抑制而实现的(见图 4)。
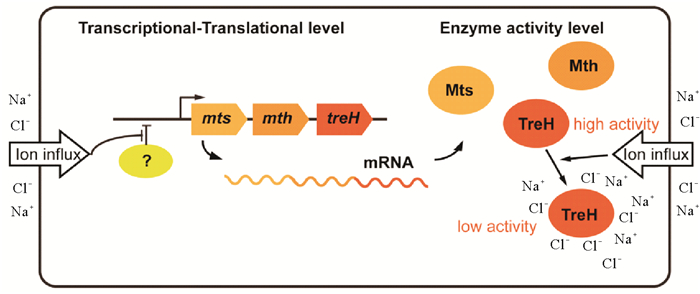
|
图 4 NaCl诱导Nostoc punctiforme IAM M-15积累海藻糖的调控机制示意图 Fig. 4 Schematic diagram of the regulatory mechanism of NaCl-induced trehalose accumulation in Nostoc punctiforme IAM M-15 |
在很多蓝细菌中,海藻糖往往作为一种应对盐胁迫的次要相容性物质被合成,其合成水平与主要相容性物质的合成水平密切相关[26-27, 34],这一生理特征也暗示海藻糖合成调控方式可能更为复杂,因此未来有必要针对一种具体的蓝细菌种类做更为系统的研究。
3 总结与展望本文总结了蓝细菌相容性物质代谢调控方面的研究进展,重点阐述了三种典型的蓝细菌相容性物质GG、蔗糖和海藻糖盐诱导积累的调控机制。蓝细菌相容性物质的积累是合成和降解协同作用的结果,其调控可以发生在两个水平——关键基因的表达(包括转录、翻译)和关键酶的活性上。然而对于不同蓝细菌,这两个水平在相容性物质代谢调控中的重要性存在诸多差异。
有些蓝细菌中,细胞遭遇盐胁迫后通过促进关键合成酶基因的表达,同时抑制降解关键酶活性的方式来实现相容性物质的积累,如Synechocystis sp. PCC 6803中蔗糖的积累、N. punctiforme中海藻糖的积累。尽管对关键合成酶基因表达的促进有利于相容物的合成,但该过程需要经历转录、翻译等一系列过程,并不能立即实现细胞的生化响应,所以对盐胁迫前期的快速响应主要是依赖对降解酶活性的抑制来实现,因此相容性物质的积累量相对有限,很可能是这类小分子没有被作为主要相容物而被积累的原因之一。
而对于蓝细菌中被作为主要抗逆物而积累的相容性物质(如Synechocystis sp. PCC 6803的GG、S. elongatus PCC 7942中的蔗糖),其代谢调控则主要通过对合成关键酶与降解关键酶的协同调控来实现。环境中升高的盐浓度,不仅有效的激活了合成关键酶,同时还显著的抑制了降解代谢关键酶。通过这种方式,蓝细菌细胞可以在盐胁迫后的短时间内,迅速完成相容物质的积累,并达到较高的水平。由于这种“离子浓度介导的酶活协同调控”方式能快速的让细胞做出生化响应,因此很可能是微生物界广泛采用的一种盐适应调控策略。
不同蓝细菌相容性物质积累的调控机制不同,这提示着我们在相关代谢工程研究中要根据具体的目标选择合适的技术策略。在以往的代谢工程研究中,研究者采用的策略多包括启动子优化、替换或者增加基因拷贝数的方式,大量表达关键合成酶,同时敲除关键降解酶基因。但这些策略可能存在两方面的问题:(1)细胞内能承载的蛋白量有限。这对于仅依赖关键基因表达方式合成相容性物质的蓝细菌,提高工程菌株中目标相容性物质产量的前景是有限的。若能引入依赖酶学水平调控的关键酶替换原始底盘细胞内的关键酶,可能会明显提高工程菌株的生产效率;若进一步通过酶工程改造,获得“始终激活”的突变酶,可能会实现“非盐胁迫”合成相容性物质。(2)蓝藻细胞相容性物质的积累是在高盐条件下,细胞内合成和降解代谢动态平衡的结果。因此,单纯敲除竞争途径,胞内相容性物质积累的浓度足以与细胞外的渗透压平衡时,其浓度就难进一步提高。由此可见,进一步实现相容物的胞外分泌也是十分必要的,如在S. elongatus PCC 7942工程菌株中进一步中引入蔗糖转运蛋白CscB,实现蔗糖分泌,明显提高了工程菌株的蔗糖产量。综合上述分析,我们应该针对不同蓝细菌底盘细胞合成相容性物质特异性调控策略进行相应地基因工程改造,通过系统性、差异性地调控转录、翻译、酶活、分泌等过程,来进一步挖掘蓝细菌相容性物质生物合成的应用潜力。
未来,随着对蓝细菌盐诱导相容性物质调控机制的深入解析,光合固碳调控网络等方面认识的不断加深,以及合成生物学和代谢工程手段的丰富和完善,光驱固碳高效定向生物合成相容性物质的研究将会取得新的突破。
| [1] |
Vincent W F. Cyanobacteria[M]. Vincent WF eds. Encyclopedia of Inland Waters, Holland: Elsevier, 2009: 226-232.
(  0) 0) |
| [2] |
Singh H. Desiccation and radiation stress tolerance in cyanobacteria[J]. Journal of Basic Microbiology, 2018, 58(10): 813-826. DOI:10.1002/jobm.201800216
(  0) 0) |
| [3] |
Ducat D C, Way J C, Silver P A. Engineering cyanobacteria to generate high-value products[J]. Trends in Biotechnology, 2011, 29(2): 95-103. DOI:10.1016/j.tibtech.2010.12.003
(  0) 0) |
| [4] |
Hagemann M, Hess W R. Systems and synthetic biology for the biotechnological application of cyanobacteria[J]. Curr Opin Biotechnol, 2018, 49: 94-99. DOI:10.1016/j.copbio.2017.07.008
(  0) 0) |
| [5] |
Klähn S, Hagemann M. Compatible solute biosynthesis in cyanobacteria[J]. Environmental Microbiology, 2011, 13(3): 551-562. DOI:10.1111/j.1462-2920.2010.02366.x
(  0) 0) |
| [6] |
Tan X, Du W, Lu X. Photosynthetic and extracellular production of glucosylglycerol by genetically engineered and gel-encapsulated cyanobacteria[J]. Appl Microbiol Biotechnol, 2015, 99(5): 2147-2154. DOI:10.1007/s00253-014-6273-7
(  0) 0) |
| [7] |
Du W, Liang F, Duan Y, et al. Exploring the photosynthetic production capacity of sucrose by cyanobacteria[J]. Metabolic Engineering, 2013, 19: 17-25. DOI:10.1016/j.ymben.2013.05.001
(  0) 0) |
| [8] |
Song K, Tan X, Liang Y, et al. The potential of Synechococcus elongatus UTEX 2973 for sugar feedstock production[J]. Appl Microbiol Biotechnol, 2016, 100(18): 7865-7875. DOI:10.1007/s00253-016-7510-z
(  0) 0) |
| [9] |
Qiao C, Duan Y, Zhang M, et al. Effects of reduced and enhanced glycogen pools on salt-induced sucrose production in a sucrose-secreting strain of Synechococcus elongatus PCC 7942[J]. Applied and Environmental Microbiology, 2018, 84(2): e02023-17.
(  0) 0) |
| [10] |
Lin P-C, Zhang F, Pakrasi H B. Enhanced production of sucrose in the fast-growing cyanobacterium Synechococcus elongatus UTEX 2973[J]. Scientific Reports, 2020, 10(1): 1-8. DOI:10.1038/s41598-019-56847-4
(  0) 0) |
| [11] |
Duan Y, Luo Q, Liang F, et al. Sucrose secreted by the engineered cyanobacterium and its fermentability[J]. Journal of Ocean University of China, 2016, 15(5): 890-896. DOI:10.1007/s11802-016-3007-8
(  0) 0) |
| [12] |
Ducat D C, Avelar-Rivas J A, Way J C, et al. Rerouting carbon flux to enhance photosynthetic productivity[J]. Applied and Environmental Microbiology, 2012, 78(8): 2660-2668. DOI:10.1128/AEM.07901-11
(  0) 0) |
| [13] |
Tan X, Du W, Lu X. Photosynthetic and extracellular production of glucosylglycerol by genetically engineered and gel-encapsulated cyanobacteria[J]. Applied Microbiology and Biotechnology, 2014, 99(5): 2147-2154.
(  0) 0) |
| [14] |
Pade N, Hagemann M. Salt acclimation of cyanobacteria and their application in biotechnology[J]. Life, 2014, 5(1): 25-49. DOI:10.3390/life5010025
(  0) 0) |
| [15] |
Brown A D, Simpson J R. Water relations of sugar-tolerant Yeasts: the role of intracellular polyols[J]. Journal of General Microbiology, 1972, 72(3): 589-591. DOI:10.1099/00221287-72-3-589
(  0) 0) |
| [16] |
Welsh D T. Ecological significance of compatible solute accumulation by micro-organisms from single cells to global climate[J]. Fems Microbiology Reviews, 2000, 24(3): 263-290. DOI:10.1111/j.1574-6976.2000.tb00542.x
(  0) 0) |
| [17] |
Arakawa T, Timasheff S N. The stabilization of proteins by osmolytes[J]. Biophysical Journal, 1985, 47(3): 411-414.
(  0) 0) |
| [18] |
Kurz M. Compatible solute influence on nucleic acids: Many questions but few answers[J]. Saline Systems, 2008, 4(6): 1-13.
(  0) 0) |
| [19] |
Kirsch F, Klähn S, Hagemann M. Salt-regulated accumulation of the compatible solutes sucrose and glucosylglycerol in cyanobacteria and its biotechnological potential[J]. Frontiers in Microbiology, 2019, 10(2139): 1-17.
(  0) 0) |
| [20] |
Yancey P H. Organic osmolytes as compatible, metabolic and counteracting cytoprotectants in high osmolarity and other stresses[J]. The Journal of Experimental Biology, 2005, 208(15): 2819-2830. DOI:10.1242/jeb.01730
(  0) 0) |
| [21] |
Hagemann M. Molecular biology of cyanobacterial salt acclimation[J]. Fems Microbiology Reviews, 2011, 35(1): 87-123. DOI:10.1111/j.1574-6976.2010.00234.x
(  0) 0) |
| [22] |
Pade N, Michalik D, Ruth W, et al. Trimethylated homoserine functions as the major compatible solute in the globally significant oceanic cyanobacterium Trichodesmium[J]. Proceedings of the National Academy of Sciences, 2016, 113(46): 13191-13196. DOI:10.1073/pnas.1611666113
(  0) 0) |
| [23] |
Fulda S, Huckauf J, Schoor A, et al. Analysis of stress responses in the cyanobacterial strains Synechococcus sp. PCC 7942, Synechocystis sp. PCC 6803, and Synechococcus sp. PCC 7418: osmolyte accumulation and stress protein synthesis[J]. Journal of Plant Physiology, 1999, 154(2): 240-249. DOI:10.1016/S0176-1617(99)80215-6
(  0) 0) |
| [24] |
Hagemann M, Marin K. Salt-induced sucrose accumulation is mediated by sucrose-phosphate-synthase in cyanobacteria[J]. Journal of Plant Physiology, 1999, 155(3): 424-430. DOI:10.1016/S0176-1617(99)80126-6
(  0) 0) |
| [25] |
Salerno G L, Porchia A C, Vargas W A, et al. Fructose-containing oligosaccharides: novel compatible solutes in Anabaena cells exposed to salt stress[J]. Plant Science, 2004, 167(5): 1003-1008. DOI:10.1016/j.plantsci.2004.05.029
(  0) 0) |
| [26] |
Higo A. The role of a gene cluster for trehalose metabolism in dehydration tolerance of the filamentous cyanobacterium Anabaena sp. PCC 7120[J]. Microbiology, 2006, 152(4): 979-987. DOI:10.1099/mic.0.28583-0
(  0) 0) |
| [27] |
Wu S. Molecular cloning of maltooligosyltrehalose trehalohydrolase gene from Nostoc flagelliforme and trehalose-related response to stresses[J]. Journal of Microbiology and Biotechnology, 2011, 21(8): 830-837. DOI:10.4014/jmb.1101.10068
(  0) 0) |
| [28] |
Yoshida T, Sakamoto T. Water-stress induced trehalose accumulation and control of trehalase in the cyanobacterium Nostoc punctiforme IAM M-15[J]. Journal of General and Applied Microbiology, 2009, 55(2): 135-145.
(  0) 0) |
| [29] |
Pade N, Compaoré J, Klähn S, et al. The marine cyanobacterium Crocosphaera watsonii WH8501 synthesizes the compatible solute trehalose by a laterally acquired OtsAB fusion protein[J]. Environmental Microbiology, 2012, 14(5): 1261-1271. DOI:10.1111/j.1462-2920.2012.02709.x
(  0) 0) |
| [30] |
Hershkovitz N, Oren A, Cohen Y. Accumulation of trehalose and sucrose in cyanobacteria exposed to matric water stress[J]. Appl Environ Microbiol, 1991, 57(3): 645-648.
(  0) 0) |
| [31] |
Page-Sharp M, Behm C A, Smith G D. Involvement of the compatible solutes trehalose and sucrose in the response to salt stress of a cyanobacterial Scytonema species isolated from desert soils[J]. Biochimica et Biophysica Acta, 1999, 1472(3): 519-528. DOI:10.1016/S0304-4165(99)00155-5
(  0) 0) |
| [32] |
Klähn S, Steglich C, Hess W R, et al. Glucosylglycerate: a secondary compatible solute common to marine cyanobacteria from nitrogen-poor environments[J]. Environmental Microbiology, 2010, 12(1): 83-94. DOI:10.1111/j.1462-2920.2009.02045.x
(  0) 0) |
| [33] |
Reed R H, Warr S R C, Richardson D L, et al. Multiphasic osmotic adjustment in a euryhaline cyanobacterium[J]. Fems Microbiology Letters, 1985, 28(3): 225-229. DOI:10.1111/j.1574-6968.1985.tb00796.x
(  0) 0) |
| [34] |
Ohmori K, Ehira S, Kimura S, et al. Changes in the amount of cellular trehalose, the activity of maltooligosyl trehalose hydrolase, and the expression of its gene in response to salt stress in the cyanobacterium Spirulina platensis[J]. Microbes and Environments, 2009, 24(1): 52-56. DOI:10.1264/jsme2.ME08537
(  0) 0) |
| [35] |
Mohammad F A A, Reed R H, Stewart W D P. The halophilic cyanobacterium Synechocystis DUN52 and its osmotic responses[J]. Fems Microbiology Letters, 1983, 16(2-3): 287-290. DOI:10.1111/j.1574-6968.1983.tb00304.x
(  0) 0) |
| [36] |
Goh F, Barrow K D, Burns B P, et al. Identification and regulation of novel compatible solutes from hypersaline stromatolite-associated cyanobacteria[J]. Archives of Microbiology, 2010, 192(12): 1031-1038. DOI:10.1007/s00203-010-0634-0
(  0) 0) |
| [37] |
Gabbay-Azaria R, Tel-Or E, Schonfeld M. Glycinebetaine as an osmoregulant and compatible solute in the marine cyanobacterium Spirulina subsalsa[J]. Archives of Biochemistry and Biophysics, 1988, 264(1): 333-339. DOI:10.1016/0003-9861(88)90601-7
(  0) 0) |
| [38] |
Tan X, Luo Q, Lu X. Biosynthesis, biotechnological production, and applications of glucosylglycerols[J]. Applied Microbiology and Biotechnology, 2016, 100(14): 6131-6139. DOI:10.1007/s00253-016-7608-3
(  0) 0) |
| [39] |
Marin K, Zuther E, Kerstan T, et al. The ggpS gene from Synechocystis sp. strain PCC 6803 encoding glucosyl-glycerol-phosphate synthase is involved in osmolyte synthesis[J]. Journal of Bacteriology, 1998, 180(18): 4843-4849. DOI:10.1128/JB.180.18.4843-4849.1998
(  0) 0) |
| [40] |
Hagemann M, Schoor A, Jeanjean R, et al. The stpA gene form Synechocystis sp. strain PCC 6803 encodes the glucosylglycerol-phosphate phosphatase involved in cyanobacterial osmotic response to salt shock[J]. Journal of Bacteriology, 1997, 179(5): 1727-1733. DOI:10.1128/JB.179.5.1727-1733.1997
(  0) 0) |
| [41] |
Savakis P, Tan X, Qiao C, et al. Slr1670 from Synechocystis sp. PCC 6803 is required for the re-assimilation of the osmolyte glucosylglycerol[J]. Front Microbiol, 2016, 7(1350): 1-10.
(  0) 0) |
| [42] |
Kirsch F, Pade N, Klähn S, et al. The glucosylglycerol-degrading enzyme GghA is involved in acclimation to fluctuating salinities by the cyanobacterium Synechocystis sp. strain PCC 6803[J]. Microbiology, 2017, 163(9): 1319-1328. DOI:10.1099/mic.0.000518
(  0) 0) |
| [43] |
Marin K, Huckauf J, Fulda S, et al. Salt-dependent expression of glucosylglycerol-phosphate synthase, involved in osmolyte synthesis in the syanobacterium Synechocystis sp. Strain PCC 6803[J]. Journal of Bacteriology, 2002, 184(11): 2870-2877. DOI:10.1128/JB.184.11.2870-2877.2002
(  0) 0) |
| [44] |
Engelbrecht F, Marin K, Hagemann M. Expression of the ggpS gene, involved in osmolyte synthesis in the marine cyanobacterium Synechococcus sp. Strain PCC 7002, revealed regulatory differences between this strain and the freshwater strain Synechocystis sp. strain PCC 6803[J]. Applied and Environmental Microbiology, 1999, 65(11): 4822-4829. DOI:10.1128/AEM.65.11.4822-4829.1999
(  0) 0) |
| [45] |
Kizawa A, Kawahara A, Takimura Y, et al. RNA-seq profiling reveals novel target genes of LexA in the cyanobacterium Synechocystis sp. PCC 6803[J]. Frontiers in Microbiology, 2016, 7(193): 1-14.
(  0) 0) |
| [46] |
Klähn S, Höhne A, Simmon E, et al. The gene ssl3076 encodes a protein mediating the salt-induced expression of ggpS for the biosynthesis of the compatible solute glucosylglycerol in Synechocystis sp. strain PCC 6803[J]. Journal of Bacteriology, 2010, 192(17): 4403-4412. DOI:10.1128/JB.00481-10
(  0) 0) |
| [47] |
Butala M, Zgur-Bertok D, Busby S J. The bacterial LexA transcriptional repressor[J]. Cellular and Molecular Life Sciences: CMLS, 2009, 66(1): 82-93. DOI:10.1007/s00018-008-8378-6
(  0) 0) |
| [48] |
Novak J F, Stirnberg M, Roenneke B, et al. A novel mechanism of osmosensing, a salt-dependent protein-nucleic acid interaction in the cyanobacterium Synechocystis species PCC 6803[J]. Journal of Biological Chemistry, 2011, 286(5): 3235-3241. DOI:10.1074/jbc.M110.157032
(  0) 0) |
| [49] |
Billis K, Billini M, Tripp H J, et al. Comparative transcriptomics between Synechococcus PCC 7942 and Synechocystis PCC 6803 provide insights into mechanisms of stress acclimation[J]. PLoS ONE, 2014, 9(10): e109738. DOI:10.1371/journal.pone.0109738
(  0) 0) |
| [50] |
Lunn J E. Evolution of sucrose synthesis[J]. Plant Physiology, 2002, 128(4): 1490-1500. DOI:10.1104/pp.010898
(  0) 0) |
| [51] |
Vargas W A, Salerno G L. The cinderella story of sucrose hydrolysis: alkaline/neutral invertases, from cyanobacteria to unforeseen roles in plant cytosol and organelles[J]. Plant Science, 2010, 178(1): 1-8.
(  0) 0) |
| [52] |
Curatti L, Flores E, Salerno G. Sucrose is involved in the diazotrophic metabolism of the heterocyst-forming cyanobacterium Anabaena sp.[J]. FEBS Letters, 2002, 513(2-3): 175-178. DOI:10.1016/S0014-5793(02)02283-4
(  0) 0) |
| [53] |
Kolman M, Nishi C, Perez-Cenci M, et al. Sucrose in cyanobacteria: from a salt-response molecule to play a key role in nitrogen fixation[J]. Life, 2015, 5(1): 102-126. DOI:10.3390/life5010102
(  0) 0) |
| [54] |
Koch K. Sucrose metabolism: regulatory mechanisms and pivotal roles in sugar sensing and plant development[J]. Current Opinion in Plant Biology, 2004, 7(3): 235-246. DOI:10.1016/j.pbi.2004.03.014
(  0) 0) |
| [55] |
Perez-Cenci M, Salerno G L. Functional characterization of Synechococcus amylosucrase and fructokinase encoding genes discovers two novel actors on the stage of cyanobacterial sucrose metabolism[J]. Plant Science, 2014, 224: 95-102. DOI:10.1016/j.plantsci.2014.04.003
(  0) 0) |
| [56] |
Schwartz S H, Black T A, Jäger K, et al. Regulation of an osmoticum-responsive gene in Anabaena sp. strain PCC 7120[J]. Journal of Bacteriology, 1998, 180(23): 6332-6337. DOI:10.1128/JB.180.23.6332-6337.1998
(  0) 0) |
| [57] |
Ehira S, Kimura S, Miyazaki S, et al. Sucrose synthesis in the nitrogen-fixing cyanobacterium Anabaena sp. strain PCC 7120 is controlled by the two-component response regulator OrrA[J]. Appl Environ Microbiol, 2014, 80(18): 5672-5679. DOI:10.1128/AEM.01501-14
(  0) 0) |
| [58] |
Los D A, Zorina A, Sinetova M, et al. Stress sensors and signal transducers in cyanobacteria[J]. Sensors, 2010, 10(3): 2386-2415. DOI:10.3390/s100302386
(  0) 0) |
| [59] |
Desplats P, Folco E, Salerno G L. Sucrose may play an additional role to that of an osmolyte in Synechocystis sp. PCC 6803 salt-shocked cells[J]. Plant Physiology and Biochemistry, 2005, 43(2): 133-138. DOI:10.1016/j.plaphy.2005.01.008
(  0) 0) |
| [60] |
Song K, Hagemann M, Tan X, et al. The response regulator Slr1588 regulates spsA but is not crucial for salt acclimation of Synechocystis sp. PCC 6803[J]. Frontiers in Microbiology, 2017, 8(1176): 1-10.
(  0) 0) |
| [61] |
Kirsch F, Luo Q, Lu X, et al. Inactivation of invertase enhances sucrose production in the cyanobacterium Synechocystis sp. PCC 6803[J]. Microbiology, 2018, 164(10): 1220-1228. DOI:10.1099/mic.0.000708
(  0) 0) |
| [62] |
Liang Y, Zhang M, Wang M, et al. Freshwater cyanobacterium Synechococcus elongatus PCC 7942 adapts to salt stress environment via ion-induced enzymatic balance of compatible solute[J]. Appl Environ Microbiol. DOI:10.1128/AEM.02904-19
(  0) 0) |
2. The Key Laboratory of Biofuels, Qingdao Institute of Bioenergy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao 266101, China
 2021, Vol. 51
2021, Vol. 51


