2. 青岛海洋科学与技术试点国家实验室 海洋渔业科学与食物产出过程功能实验室,山东 青岛 266237
RNA干扰(RNA interference,RNAi)是由双链RNA诱导,抑制内源基因表达的一种调控机制,在mRNA降解、翻译抑制、转座子沉默和维持基因组完整性等方面有重要作用[1-3]。RNAi的主要参与成分是RNA诱导的沉默复合体(RNA-induced silencing complex,RISC),Argonaute蛋白作为RISC的重要组成部分,通过与RISC的装配参与到RNAi过程中[4]。许多研究表明,RNAi作用方式的多样性与Argonaute家族基因的分化有关[5]。Argonaute家族包含两个亚家族:AGO亚家族和PIWI亚家族。AGO蛋白多在生物体各组织中广泛表达,主要与siRNA/miRNA结合装配进入RISC,从而介导靶mRNA的沉默;PIWI蛋白多在生殖细胞中表达,与piRNA结合形成piRNA复合物(piRNA complex,piRC),在生殖系转座子和基因沉默中发挥作用[6-7]。近年的研究表明,RNAi过程中与抗病毒(siRNA)和转座子沉默(piRNA)相关的基因常表现出快速进化的特征,例如siRNA和piRNA途径相关基因的快速进化可能与病毒或转座子同宿主的协同进化有关[5, 8]。目前相关研究多报道于无脊椎动物中,特别是基于果蝇的研究最为详细[8-9]。而在脊椎动物中,piRNA途径可以通过对转座子和基因的调控在维持生殖细胞基因组的稳定性和完整性、调控生殖细胞增殖、减数分裂和精子发生等过程中发挥重要作用[10-13]。然而,目前对RNAi不同途径在脊椎动物特别是硬骨鱼谱系中的进化模式和功能的报道还相对较少。
硬骨鱼类作为低等脊椎动物,是脊椎动物门中最大的类群,其繁殖发育策略丰富多样,有单性生殖的、也有雌雄异体和雌雄同体的。雌雄异体鱼的性腺发育包括:
(1) 未分化型:原始性腺先发育成类似卵巢的结构,随后一半个体的性腺发育成精巢,另一半个体的性腺发育成卵巢,如斑马鱼[14]。
(2) 分化型:原始性腺直接发育成精巢或卵巢,如鲤鱼[15]。
雌雄同体的鱼类又分为:
(1) 同步雌雄同体:个体性成熟后,性腺中同时存在精巢和卵巢,但精子和卵子在不同时期成熟以避免自体受精,如乌鳢[16]。
(2) 次序雌雄同体,又分为雌性先熟和雄性先熟型,前者在性成熟前均为雌性,性成熟产卵后,卵巢转变为精巢,表现为雄性,如黄鳝[17];后者则相反,性成熟前为雄性,性成熟后转变为雌性,如黑鲷[18]。此外,环境因素(温度、盐度等)对鱼类的性别决定和性腺分化也有巨大影响[19-23]。
鱼类生活的水生环境复杂多样,天敌种类繁多,多数鱼类的繁殖方式为体外受精,精卵暴露在复杂多变的水域环境中,长期进化形成了怀卵量大等特点以保证受精率,这都为其繁殖发育、配子发生、受精、胚胎发育以及遗传改良育种等研究提供了优良的材料。另外相比较哺乳类,鱼类基因组中转座子种类更多且更加活跃,更易对基因组造成损伤[24]。越来越多的研究表明,作为RNAi中转录和转录后基因表达的重要调控因子,多种非编码RNA(如miRNA和piRNA等)都在动物生殖细胞中表达,对原始生殖细胞的干性维持、自我更新和分化、生殖细胞减数分裂和精卵发生过程具有重要的调控作用[4, 25]。其中piRNA参与生殖系中转座子沉默并维持基因组的稳定,对鱼类的繁殖发育至关重要。已有研究表明,在雌雄异体的斑马鱼中,两种Piwi蛋白之一的Ziwi蛋白是维持生殖系所必需的,Ziwi蛋白的丢失会导致减数分裂前的生殖细胞凋亡[26];在雌雄同体的黄鳝中,piRNA途径基因在三种类型的性腺(卵巢、卵精巢、精巢)中均呈现高表达,表明piRNA途径基因在性逆转过程中发挥关键作用[27]。花鲈(Lateolabrax maculatus)隶属鲈形目、鮨科、花鲈属,为广盐、广温性鱼类,是中国传统的经济养殖鱼类,近年来其养殖产量连续位居中国海水养殖鱼类前列。花鲈经淡水化处理后也可在淡水中进行养殖,但成鱼必须在海水中才能完成性成熟及繁殖过程[28]。因此,本研究鉴定了花鲈Argonaute家族基因,对哺乳动物和硬骨鱼类siRNA/miRNA与piRNA途径基因进行了系统的分子进化分析,利用鱼类转录组数据比较了siRNA/miRNA和piRNA基因的表达特征,为硬骨鱼类,特别是花鲈的生殖发育研究提供重要理论依据。
1 材料与方法 1.1 Argonaute家族基因的鉴定及结构分析为鉴定花鲈基因组中的Argonaute家族基因,本文首先检索了NCBI(http://www.ncbi.nlm.nih.gov)和Ensembl(http://www.ensembl.org)数据库,收集到14种硬骨鱼(牙鲆(Paralichthys olivaceus)、半滑舌鳎(Cynoglossus semilaevis)、大菱鲆(Scophthalmus maximus)、斑马鱼(Danio rerio)、墨西哥脂鲤(Astyanax mexicanus)、大西洋鳕(Gadus morhua)、三棘鱼(Gasterosteus aculeatus)、红鳍东方鲀(Takifugu rubripes)、绿斑鲀(Tetraodon nigroviridis)、罗非鱼(Oreochromis niloticus)、青鳉(Oryzias latipes)、花鳉(Poecilia formosa)、斑剑尾鱼(Xiphophorus maculatus)、亚洲鲈(Lates calcarifer)) 和12种哺乳类(人(Homo sapiens)、小鼠(Mus musculus)、负鼠(Monodelphis domestica)、大象(Loxodonta africana)、马(Equus caballus)、狗(Canis familiaris)、猪(Sus scrofa)、牛(Bos taurus)、猕猴(Macaca mulatta)、兔(Oryctolagus cuniculus)、羊(Ovis aries)、大鼠(Rattus norvegicus)),共26个物种Argonaute家族基因的核酸序列,通过本地BLAST (e-value=1e-5)比对花鲈基因组(GCA_004023545.1)获得花鲈Argonaute基因家族成员序列。随后,根据比对得到的花鲈Argonaute家族成员的蛋白序列,利用SMART在线工具(http://smart.embl-heidelberg.de)预测花鲈Argonaute蛋白序列的结构域。利用Gene Structure Display Server2.0网站(http://gsds.cbi.pku.edu.cn/)分析花鲈Argonaute家族基因的基因结构。
1.2 Argonaute家族基因系统发生分析根据从Ensembl和NCBI数据库获得的26个选定物种(12种哺乳动物和14种鱼类)和本地BLAST鉴定得到的花鲈(共27个物种)的Argonaute基因(Piwil1、Piwil2、Piwil3、Piwil4、Ago1、Ago2、Ago3和Ago4)的编码序列,本文利用Muscle软件对这些核酸序列进行多序列比对。随后使用MEGA 7.0软件构建最大似然法(Maximum Likelihood, ML)系统发生树,基于最优模型(GTR+G+I模型),自举值(Bootstrap)为1 000次。
1.3 Argonaute家族基因共线性分析共线性分析是基于对花鲈Argonaute家族基因和其它物种对应基因间的临近基因进行比较建立起来的。本文利用花鲈Argonaute成员序列号从花鲈基因数据库中检索得到各成员上下游基因的核酸序列,利用NCBI中的BLAST功能确认花鲈Argonaute家族各成员上下游基因的名称、方向及其在染色体上的定位,利用Genomicus数据库(https://www.genomicus.biologie.ens.fr/genomicus)查找其它物种Argonaute基因上下游基因的线性排列。
1.4 分子进化分析本文通过分析15种硬骨鱼(包括花鲈)和12种哺乳动物的编码序列,来检测它们在siRNA/miRNA和piRNA途径中的不同选择压力。每个基因的编码序列通过多序列比对,使用MEGA 7.0生成ML树,确定各基因的系统发生关系。分子进化分析使用PAML v4.9的Codeml程序,通过ML方法分别进行点模型(Site model,SM)、枝模型(Branch model,BM)和枝-点模型(Branch-site model,BSM)检验[29-30]。
点模型采用ML估计非同义替换与同义替换的比率(dN/dS,ω值)和似然率检验(Likelihood ratio tests,LRTs)。本研究共使用六个点模型测试各密码子中的正选择位点:M0(单一比率)假设所有位点具有相同的ω值;M3(离散)假设有3类ω值,所有位点ω值呈简单的离散分布趋势;M1a(近中性)假设仅有保守位点(0<ω<1)和中性位点(ω=1)而没有正选择位点(ω>1)存在;M2a(正选择)在M1a基础上增加了第三类ω值,即假设除了保守位点和中性位点外,还存在正选择位点(ω>1);M7(beta)假设所有位点的ω值属于矩阵(0,1)并呈beta分布;M8(beta&ω)在M7基础上增加另一类ω值(ω>1)。通过卡方(χ2)检验计算配对模型(M0对M3;M1a对M2a;M7对M8)的ML值之间的两倍差(2ΔlnL),自由度等于配对模型之间参数数量的差,判断两个模型的ω是否差异显著(M3对M0),并确定是否存在正选择位点(M2a对M1a和M8对M7)。贝叶斯经验(The Bayes empirical Bayes,BEB)[29]用于评估正选择下点模型的贝叶斯后验概率。使用点模型计算的ω值(dN/dS)用于比较各基因在哺乳动物和硬骨鱼谱系中的进化速率[29-30]。
为进一步检验各基因在不同谱系分枝(哺乳动物和硬骨鱼)中的选择压力,本文进行了枝模型检验[30]。单比率模型(One-ratio model)是零假设,即假设整个树具有一个ω值。然后将双比率模型(Two-ratio model,背景枝为ω0,前景枝为ω1)与单比率模型进行配对比较,通过LRTs来确定不同分枝是否有不同的ω值。将硬骨鱼谱系分枝设置为前景枝,哺乳类分枝设置为背景枝,比较硬骨鱼和哺乳类的进化速率。此外,将花鲈分枝设置为前景枝,其它硬骨鱼分枝设置为背景枝,来比较花鲈和其它硬骨鱼的进化速率。通过χ2分析2ΔlnL,确定LRT的显著性。
为进一步检验各基因在特定谱系中是否存在正选择位点,本文进行了枝-点模型检验[30]。将硬骨鱼分枝和花鲈分枝分别设置为前景枝,并将其它分枝设置为背景枝。在model A中,为前景枝设置了3个ω值(0<ω0<1,ω1=1,ω2>1),为背景枝设置了2个ω值(0<ω0<1和ω1=1)。null model除ω2=1外,其它参数与model A的参数相似。使用标准自由度,通过计算model A与null model之间的2ΔlnL来进行χ2分析,确定LRT的显著性,并根据BEB后验概率分析正选择位点。最后将这些正选择位点与Pfam数据库中的蛋白序列和功能域进行比对。
1.5 Argonaute家族基因表达分析为比较siRNA/miRNA与piRNA途径基因在各物种中的表达模式,本文从NCBI分别下载花鲈和亚洲鲈转录组数据(花鲈:腮-SRR7528883、胃-SRR7528884、肝-SRR7528886、脑-SRR7528887、脾-SRR752888、精巢-SRR7528885、卵巢-SRR2937376;亚洲鲈:卵巢-SRR1791597.1、精巢-SRR1791598.1、脑-SRR1791593.1)。牙鲆和半滑舌鳎[31]的基因表达从课题组转录组数据获得。首先用fastqc检测质量,使用trimmomatic去除接头和低质量序列,使用hisat将优化后序列比对到各物种参考基因组,使用StringTie进行拼接组装和表达量分析。斑马鱼[32]、肺鱼[33]、腔棘鱼[33]、黄鳝[27]、罗非鱼[34]各基因表达情况均从文献中引用。得到花鲈各基因的FPKM值后,计算log2(FPKM+1)值,通过MeV 4.90软件对花鲈各基因的表达情况绘制热图。
2 结果与分析 2.1 Argonaute家族基因的鉴定及拷贝数分析通过本地BLAST比对花鲈基因组序列(GCA_004023545.1),本文共鉴定得到7个花鲈Argonaute家族成员,分为2个亚家族:AGO亚家族和PIWI亚家族。花鲈PIWI亚家族包含Piwil1和Piwil2,AGO亚家族包含Ago1、Ago2、Ago3a、Ago3b和Ago4共5个成员,其中Ago3具有两个拷贝,在系统发生分析后分别命名为Ago3a和Ago3b。本文对可获得的脊椎动物基因组进行了同源搜索,比较硬骨鱼类与哺乳类Argonaute家族基因的拷贝数。如表 1中,哺乳动物多有7~8个Argonaute家族成员,其中包含4个AGO亚家族成员(Ago1、Ago2、Ago3和Ago4)和4个PIWI亚家族成员(Piwil1、Piwil2、Piwil3和Piwil4),仅在小鼠、大鼠和大象中未发现Piwil3。硬骨鱼多包含5~7个Argonaute家族基因成员,其中所有硬骨鱼均含有Piwil1和Piwil2,但与哺乳类不同的是,硬骨鱼中不存在Piwil3和Piwil4。斑马鱼、大西洋鳕、牙鲆、大菱鲆、三棘鱼、花鳉、罗非鱼与花鲈中均发现两个Ago3,分别为Ago3a和Ago3b;半滑舌鳎、红鳍东方鲀、青鳉、斑剑尾鱼、墨西哥脂鲤、亚洲鲈中只有1个Ago3;绿斑鲀斑点雀鳝Ago3基因缺失,大西洋鳕缺失Ago1基因。拷贝数结果显示,各物种Argonaute家族成员数量上有所差别,但组成情况基本相似,是高度保守的基因家族。在高等脊椎动物中,一般都含有4个Ago基因(Ago1、Ago2、Ago3和Ago4),但在硬骨鱼中却有5个Ago成员,这可能是由于大约350万年前,在脊椎动物中发生了两次全基因组复制事件后,在硬骨鱼中又发生了一次其特有的第三次全基因组复制事件[35],产生了一个硬骨鱼特有的全基因组复制起源的Ago3基因。
|
|
表 1 脊椎动物Argonaute家族基因的拷贝数 Table 1 Copy number of Argonaute family genes among different vertebrates |
花鲈Argonaute家族基因结构见图 1A。其中Ago1含有18个外显子和17个内含子;Ago2含有18个外显子和17个内含子;Ago3a含21个外显子和20个内含子;Ago3b含19个外显子和18个内含子;Ago4含16个外显子和15个内含子;Piwil1含18个外显子和17内含子;Piwil2含有19个外显子和18个内含子。我们使用SMART在线工具预测了花鲈Argonaute家族蛋白质序列的结构域,如图 1B所示,花鲈Argonaute家族蛋白成员都有一个完整的PIWI结构域,除Ago4外都有一个位于PIWI结构域上游的PAZ结构域。此外,AGO亚家族成员(Ago1、Ago2、Ago3a、Ago3b和Ago4)在PAZ结构域上游都有一个DUF1785结构域,且两者距离很近。总体而言,花鲈Argonaute家族蛋白质的各结构域均较保守,PIWI结构域较PAZ结构域的保守性更强,PAZ结构和DUF1785结构相距很近,可能与其功能有关。其它脊椎动物Argonaute家族蛋白中也都含有PAZ结构域和PIWI结构域,二者分别可以特异性结合small RNA的3’端和5’端[36-37]。重要结构域的保守性与物种的进化有一定联系,由于功能位点的特异性,这些结构域在进化过程中往往出现较少的变异位点。在真核生物中,PAZ结构域是位于N端包含130个氨基酸的区域,可以形成一个口袋状的结构结合small RNA的3’端;PIWI结构域是位于C端包含300个氨基酸的区域,类似于核糖核酸酶H的结构域,具有核酸酶活性,可以结合small RNA的5’端并介导靶RNA的降解[38-39]。
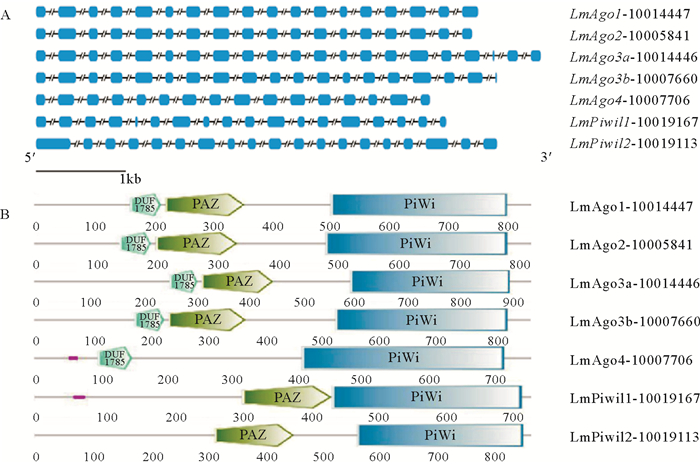
|
(图A中方框代表外显子,斜线隔断的直线代表内含子(内含子被设置为等长)。基因名后的编号为花鲈数据库里的基因号。In panel A, exons are shown by the rectangle box, the straight line separated by diagonal lines represents introns (the introns are set to be equal in length). The numbers behind the gene name represents the gene ID in the L. amaculatus genome database.) 图 1 花鲈Argonaute家族基因结构(A)与蛋白结构域(B)分析 Fig. 1 Gene structure (A) and functional domain (B) of Argonaute family in L. amaculatus |
本文使用MEGA7.0构建了哺乳动物和硬骨鱼类Argonaute家族基因的系统进化树。如图 2所示,脊椎动物Argonaute家族成员可分为2大枝,支持Argonaute家族基因的2个亚家族(PIWI亚家族和AGO亚家族)。各Argonaute基因分枝中,哺乳类与硬骨鱼类各聚为一枝,花鲈与硬骨鱼类聚成一枝,均符合物种进化地位。在PIWI亚家族中,哺乳动物Piwil1先与Piwil3和Piwil4聚为一枝,随后再与硬骨鱼类的Piwil1聚类;而所有脊椎动物的Piwil2则聚为另一支。Ago亚家族主要包含4个分支:Ago1、Ago2、Ago3(Ago3a和Ago3b)和Ago4。Ago3基因可分为两枝:哺乳类Ago3和硬骨鱼类Ago3,其中硬骨鱼类Ago3基因又分别聚类为Ago3a与Ago3b两枝,进一步证明这两个基因起源于硬骨鱼特有的第三次全基因组复制事件。
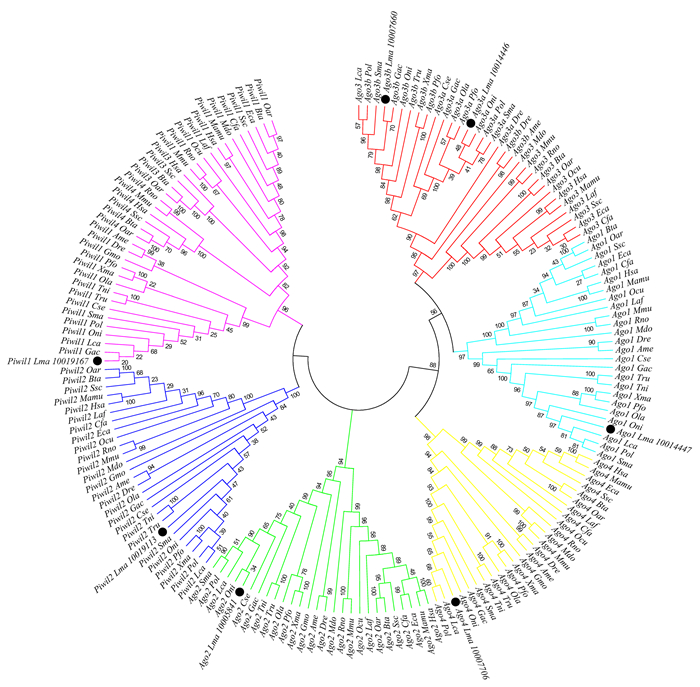
|
(花鲈Argonaute基因用黑点标记。Hsa:人,Mdo:负鼠,Mmu:小鼠,Laf:大象,Eca:马,Cfa:狗,Ssc:猪,Bta:牛,Mamu:猕猴, Ocu:兔, Oar:羊, Rno:大鼠,Dre:斑马鱼,Ame:墨西哥脂鲤,Gmo:大西洋鳕,Gac:三棘鱼, Tru:红鳍东方鲀,Tni:绿斑鲀,Oni:罗非鱼,Ola:青鳉,Xma:斑剑尾鱼,Pfo:花鳉,Pol:牙鲆,Cse:半滑舌鳎,Sma:大菱鲆,Lca:亚洲鲈,Lma:花鲈。Argonaute genes in L. maculatus are marked with black dots. Hsa: Homo sapiens, Mdo: Monodelphis domestica, Mmu: Mus musculus, Laf: Loxodonta africana, Eca: Equus caballus, Cfa: Canis familiaris, Ssc: Sus scrofa, Bta: Bos taurus, Mamu: Macaca mulatta, Ocu: Oryctolagus cuniculus, Oar: Ovis aries, Rno: Rattus norvegicus, Dre: Danio rerio, Ame: Astyanax mexicanus, Gmo: Gadus morhua, Gac: Gasterosteus aculeatus, Tru: Takifugu rubripes, Tni: Tetraodon nigroviridis, Oni: Oreochromis niloticus, Ola: Oryzias latipes, Xma: Xiphophorus maculatus, Pfo: Poecilia formosa, Pol: Paralichthys olivaceus, Cse: Cynoglossus semilaevis, Sma: Scophthalmus maximus, Lca: Lates calcarifer, Lma: Lateolabrax maculatus.) 图 2 脊椎动物Argonaute家族基因系统发生分析 Fig. 2 Phylogenetic relationships of Argonaute genes among vertebrates |
为比较Argonaute家族基因在哺乳类和硬骨鱼类中的起源和进化过程,本文分析了物种间Argonaute家族基因的共线性关系。本文选用人、鼠、狗和马四种哺乳动物以及斑马鱼、花鲈、墨西哥脂鲤和斑点叉尾鮰四种硬骨鱼(见图 3A)。哺乳动物中AGO亚家族的上下游基因在染色体上的排列顺序比较保守,其中Ago4、Ago1和Ago3多串联排列于同一条染色体,呈连锁分布,Ago2单独位于另一条染色体。以人为例,Ago4、Ago1和Ago3自上而下依次排列,其上游基因分别是CLAPN、C1orf216、PSMB2、PSMB2、TFAP2E,下游基因分别是TEKT2、ADPRHL2、COL8A2、TRAPPC3、AMAP7D1。在硬骨鱼中,AGO亚家族基因的共线性与哺乳类存在较大差异(见图 3A),硬骨鱼中Ago1和Ago3a呈连锁分布于同一条染色体,Ago2和Ago4多位于另一条染色体且两者间距离较远。Ago3b的分布有两种类型:斑马鱼和花鲈中,Ago3b、Ago2和Ago4位于同一条染色体上,但三个基因在斑马鱼和花鲈中的分布顺序不同,在斑马鱼中为Ago3b-Ago2-Ago4;而在花鲈中为Ago4-Ago3b-Ago2;另一种情况在墨西哥脂鲤和斑点叉尾鮰中,Ago3b既不与Ago2和Ago4共染色体,也不与Ago1和Ago3a共染色体,而是单独位于另一条染色体上。
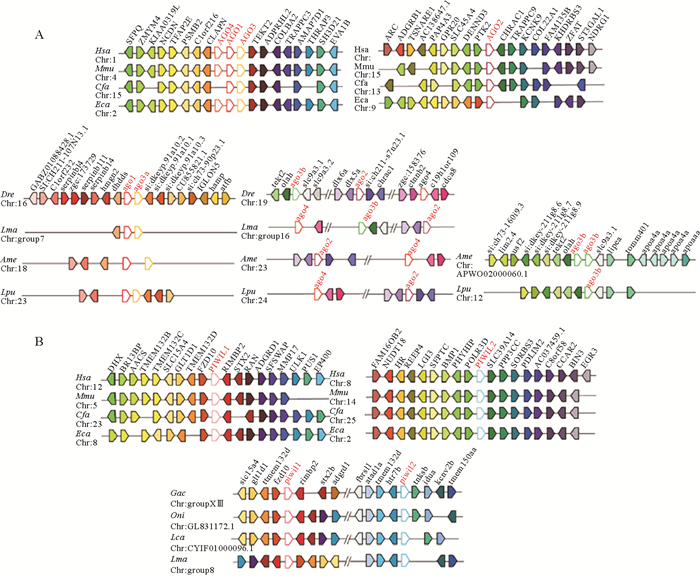
|
(A.AGO亚家族共线性分析;B.PIWI亚家族共线性分析。不同颜色的框代表不同的基因,Argonaute基因为红色字体。A. Synteny analysis of AGO subfamily; B. Synteny analysis of PIWI subfamily. Boxes with different colors indicate different genes, and Argonaute gene is in red font.) 图 3 脊椎动物Argonaute家族基因共线性分析 Fig. 3 Synteny analysis of Argonaute genes among vertebrates |
PIWI亚家族的共线性分析,本文选用人、鼠、狗和马四种哺乳动物以及三棘鱼、罗非鱼、亚洲鲈和花鲈四种硬骨鱼(见图 3B)。哺乳动物中Piwil1和Piwil2分布在不同的染色体上,以人为例,Piwil1的上游基因有FZD10、TMEM132D、GLT1D1、SLC15A4,下游基因有RIMBP2、STX2、RAN、ADGRD1;Piwil2的上游基因有POLR3D、PHYHIP、BMP1、SFPTC,下游基因有SLC39A14、PPP3CC、SORBS3、PDLIM2。其它哺乳动物中Piwil1和Piwil2的上下游排布方式与此类似,只存在个别基因缺失的现象。硬骨鱼中Piwil1和Piwil2位于同一条染色体上,硬骨鱼的Piwil1与Piwil2基因的上下游基因都较为保守,我们以三棘鱼为例,Piwil1的上游基因依次为fzd10、tmem132d、glt1d1、slc15a4,下游基因有rimbp2、stx2b、adgrd1,类似于哺乳动物Piwil1上下游基因排布,而Piwil2上下游基因排布与哺乳类Piwil2上下游基因排布差异较大,没有共线性。
2.5 Argonaute家族基因分子进化分析为比较哺乳动物和硬骨鱼类piRNA途径基因(Piwil1,Piwil2,Vasa,Mov10l1和Henmt1)和miRNA/siRNA途径基因(Ago1、Ago2、Ago3a、Ago3b和Ago4)的分子进化模式,本文使用PAML v4.9中的codeML程序进行了一系列模型检验,包括点模型、枝模型和枝-点模型检验。
2.5.1 点模型检验哺乳动物和硬骨鱼类Argonaute家族成员的dN/dS值由点模型可知(见图 4),piRNA途径基因的dN/dS明显高于miRNA/siRNA途径基因,说明piRNA途径基因的进化速率比miRNA/siRNA途径基因更快。比较哺乳动物和硬骨鱼谱系间的dN/dS值,除Henmt1外,piRNA途径基因在硬骨鱼中的进化速率均高于哺乳动物,而miRNA/siRNA途径基因中只有硬骨鱼的Ago2和Ago3b的dN/dS值高于哺乳动物(见图 4)。
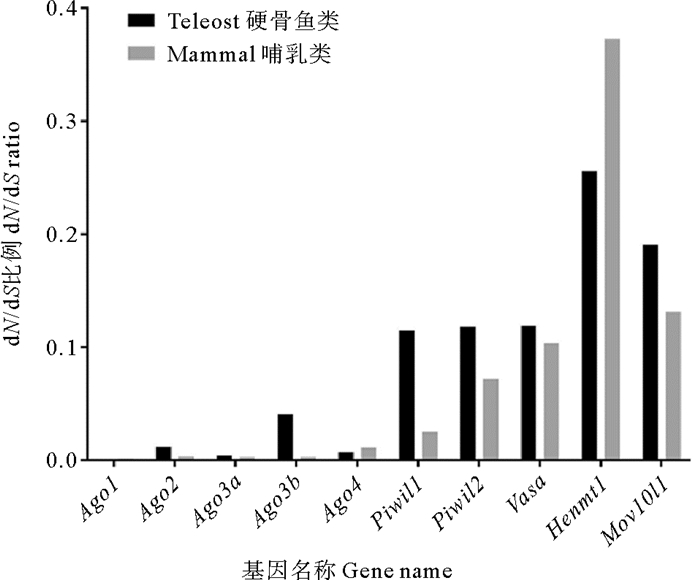
|
(Ago1, Ago2, Ago3a, Ago3b和Ago4为miRNA/siRNA途径相关基因,Piwil1, Piwil2, Vasa, Mov10l和Henmt1为piRNA途径相关基因。Ago1, Ago2, Ago3a, Ago3b, and Ago4 are involved in miRNA/siRNA pathway, and Piwil1, Piwil2, Vasa, Mov10l1, and Henmt1 are involved in piRNA pathway. ) 图 4 硬骨鱼和哺乳动物miRNA/siRNA和piRNA途径基因的dN/dS比较 Fig. 4 dN/dS ratio of miRNA/siRNA and piRNA pathway genes in teleost and mammal lineages |
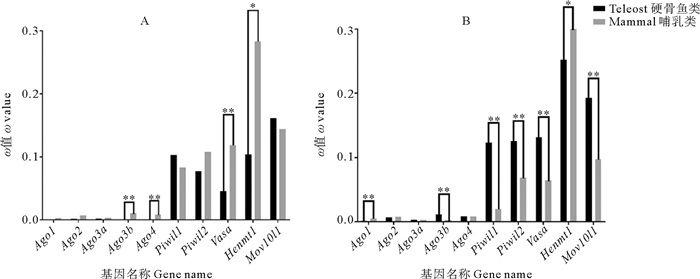
枝模型检验可比较基因在不同谱系分枝上所受选择压力的差异。本文分别对硬骨鱼祖先枝和所有硬骨鱼谱系进行前景枝标记。标记硬骨鱼祖先枝为前景枝的结果(见图 5A)与点模型结果相似,piRNA途径基因的ω值明显高于miRNA/siRNA途径基因。但除Piwil1和Mov10l1外,所有哺乳动物siRNA/miRNA途径基因和piRNA途径基因的ω0均高于硬骨鱼的ω1,其中Ago3b、Ago4、Vasa和Henmt1在哺乳动物中的进化速率显著高于硬骨鱼(P<0.05)。标记所有硬骨鱼谱系为前景枝的结果(见图 5B)显示,piRNA途径基因的ω值仍明显高于miRNA/siRNA途径基因。除Ago2、Ago3a和Ago4外,哺乳类与硬骨鱼的ω值均有显著差异。其中除Henmt1外,硬骨鱼piRNA途径基因的ω1均极显著高于哺乳动物的ω0(P<0.01)。枝模型检验的结果与点模型相似,进一步证明除Henmt1外,piRNA途径基因在硬骨鱼中比在哺乳动物中进化得更快。
|
(A.标记硬骨鱼祖先枝为前景枝ω1;B.标记全部硬骨鱼谱系为前景枝ω1。黑色柱表示硬骨鱼中各基因的ω1值,灰色柱表示哺乳动物中各基因的ω0值。*表示似然比检验(LRT)P<0.05; **表示P<0.01。A. Lable the teleost ancestral lineage as the foreground branch ω1; B. Lable all teleost lineages as foreground branch ω1. Black column indicates ω1 value of each gene in teleost lineages, and gray column indicates ω0 value of the mammalian lineages.* indicates P<0.05 and ** indicates P<0.01via LRT.) 图 5 硬骨鱼和哺乳动物miRNA/siRNA和piRNA途径基因枝模型检验 Fig. 5 Branch model tests for miRNA/siRNA and piRNA pathway genes in teleost and mammal lineages |
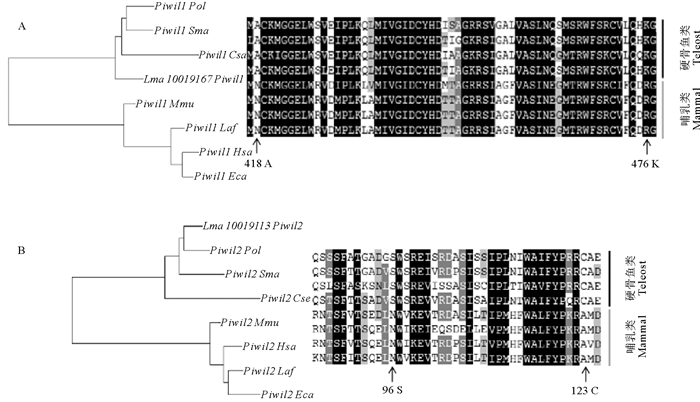
本文进一步通过枝-点模型检验筛选piRNA/miRNA途径基因中可能存在的正选择位点。本文分别标记硬骨鱼祖先枝和所有硬骨鱼谱系为前景枝进行分析。在标记硬骨鱼祖先枝时(见表 2),除Henmt1外,piRNA途径基因均检测到正选择位点(P<0.01),其中Mov10l1检测到13个正选择位点,Piwil1有8个正选择位点,Piwil2有2个,Vasa只有1个。Ago亚家族中并没有检测到正选择位点。图 6中展示了Piwil1和Piwil2中的部分正选择位点,其中Piwil1有3个正选择位点位于PAZ结构域中,可能与piRNA诱导靶mRNA切割的RNAi过程相关。
|
|
表 2 枝-点模型检验miRNA/siRNA和piRNA途径基因正选择位点(标记硬骨鱼祖先枝为前景枝) Table 2 Branch-site model tests for detection of positively selected sites of miRNA/siRNA and piRNA pathway genes (Lable teleost ancestral lineage as the foreground branch) |
|
(左边为4种硬骨鱼和4种哺乳类基于核酸序列的Piwil1和Piwil2的系统进化关系,右边为这8个物种Piwil1和Piwil2部分氨基酸序列比对,箭头所指是通过枝-点模型检测到的正选择位点。The left panel is the phylogenetic analysis Piwil1 and Piwil2 based on nuclear acid sequences of 4 teleost and 4 mammalian species, and the right panel is the partial aligned amino acid sequence of Piwil1 and Piwil2 of the 8 species. The arrow indicates the positively selected sites detected via branch-site model test.) 图 6 Piwil1(A)和Piwil2(B)的枝-点模型检验的正选择位点(标记硬骨鱼祖先枝为前景枝) Fig. 6 The positively selected sites of Piwil1(A) and Piwil2(B) from branch-site model test(Lable teleost ancestral lineage as foreground branch) |
标记所有硬骨鱼谱系为前景枝的枝-点模型检验结果(见表 3)表明,正选择位点仅存在于Piwil1、Piwil2和Mov10l1中,且在Mov10l1中发现了多达75个的正选择位点,在Piwil1中发现43个正选择位点,Piwil2中仅发现9个正选择位点,但这些正选择位点的结果均不显著(P>0.05)。
|
|
表 3 枝-点模型检验miRNA/siRNA和piRNA途径基因正选择位点(标记所有硬骨鱼谱系为前景枝) Table 3 Branch-site model tests for detection of positively selected sites of miRNA/siRNA and piRNA pathway genes (Lable all teleost lineages as foreground branch) |
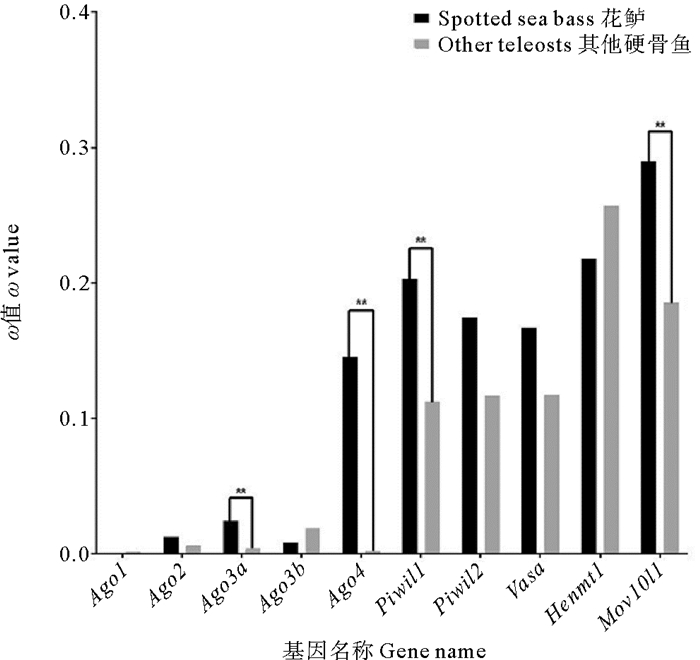
最后本文以花鲈为前景枝,其它硬骨鱼为背景枝,对花鲈和其它硬骨鱼类谱系的Argonaute家族基因进行枝模型检验(见图 7)。piRNA途径基因的ω值仍明显高于miRNA/siRNA途径基因。其中,花鲈和其它硬骨鱼之间的ω值在Ago3a、Ago4、Piwil1和Mov10l1中有极显著差异(P<0.01),说明相比于其它硬骨鱼类,花鲈的Ago3a、Ago4、Piwil1和Mov10l1可能受到更大的选择压力。
|
(黑色柱表示花鲈各基因的ω值,灰色柱表示其它硬骨鱼各基因的ω值。*表示似然比检验(LRT)P<0.05; **表示P<0.01。Black column indicates ω value of each gene in L. maculatus, and gray column indicates ω value of other teleost lineages. *indicates P<0.05 and **indicates P<0.01 via LRT.) 图 7 硬骨鱼谱系中miRNA/siRNA和piRNA途径基因的枝模型检验 Fig. 7 Branch model tests for miRNA/siRNA and piRNA genes in teleost lineages |
在花鲈与其它硬骨鱼类的枝-点模型检验中,正选择位点存在于Ago4、Piwil1、Piwil2、Vasa和Mov10l1基因中(见表 4)。其中,在Ago4中得到了19个正选择位点;Mov10l1中有5个正选择位点;在Piwil1中仅发现3个正选择位点;在Piwil2和Vasa中均只发现2个正选择位点,其中Piwil2有一个正选择位点位于PAZ结构域中。
|
|
表 4 枝-点模型检验花鲈miRNA/siRNA和piRNA途径基因正选择位点 Table 4 Branch-site model tests for detection of positively selected sites of miRNA/siRNA and piRNA pathway genes in L. maculatus |
为比较花鲈miRNA/siRNA和piRNA途径基因的表达模式,本文首先对花鲈7个成体组织(脑、肠、肝脏、脾脏、鳃、卵巢和精巢)的转录组数据进行表达分析。如图 8所示,5个Ago基因(Ago1、Ago2、Ago3a、Ago3b和Ago4)在不同组织中呈现泛表达。其中Ago2主要在脑和脾中表达。Ago3a在各组织中低表达,而Ago3b主要表达于脾和鳃,Ago4主要表达于脑、脾、鳃和精巢。此外,5个piRNA基因(Piwil1、Piwil2、Vasa、Mov10l1和Henmt1)均在花鲈性腺中特异高表达(见图 8),表明它们可能在花鲈的生殖发育中有重要作用。

|
(标尺中“0~9”表示log2(FPKM+1)值。The value of log2(FPKM+1) is represented by "0~9" on the scale.) 图 8 花鲈各组织中miRNA/siRNA与piRNA途径基因的表达模式 Fig. 8 Expression profile of miRNA/siRNA and piRNA pathway genes in L. maculatus tissues |
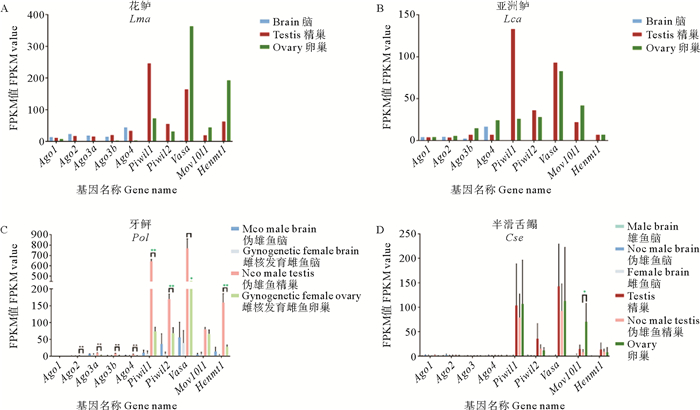
此外,本文通过转录组分析比较了不同生殖类型鱼类(花鲈、亚洲鲈、牙鲆和半滑舌鳎)miRNA/siRNA和piRNA基因在性腺和脑中的表达情况(见图 9)。在这四种硬骨鱼中,miRNA/siRNA途径基因的表达均低于piRNA途径基因,piRNA基因在性腺中的表达普遍高于在脑中的表达,且在性腺中Piwil1的表达多高于Piwil2。就piRNA途径基因而言,在适应广温广盐水域的花鲈中,Piwil1和Piwil2在精巢中的表达高于卵巢,而Vasa、Henmt1和Mov10l1的表达情况则相反,在卵巢中的表达高于精巢;在雌雄同体,先雄后雌的亚洲鲈中,Piwil1、Piwil2和Vasa在精巢中的表达高于卵巢,而Mov10l1在卵巢中的表达高于精巢,Henmt1在精卵巢中的表达没有明显差异;在存在XX性逆转伪雄鱼的牙鲆中,piRNA途径基因在伪雄鱼精巢中的表达均高于卵巢,表现出精巢高表达。除Mov10l1外,其它基因在精卵巢中的表达均有极显著差异;在存在ZW性逆转伪雄鱼的半滑舌鳎中,Piwil1在精巢和卵巢中的表达没有明显差异,Piwil2、Vasa和Henmt1在精巢中的表达高于卵巢,而Mov10l1在卵巢中表现出高表达。性逆转伪雄鱼精巢中的表达多低于正常精卵巢中的表达。除Mov10l1在正常发育雌鱼的卵巢和性逆转伪雄鱼精巢中的表达存在显著差异外,其余基因在雌雄性腺中的差异并不显著。
|
(miRNA/siRNA和piRNA途径基因在花鲈(A);亚洲鲈(B);雌核发育牙鲆(C);半滑舌鳎雌、雄和伪雄鱼(D)脑和性腺中的表达。对牙鲆和半滑舌鳎各基因在性腺中的表达量进行显著性分析,**表示P<0.01,*表示P<0.05。Brain:脑;Testis:精巢;Ovary:卵巢;Male brain:雄鱼脑;Female brain:雌鱼脑;Neo male brain:伪雄鱼脑;Gynogenetic female brain:雌核发育雌鱼脑;Neo male testis:伪雄鱼精巢;Gynogenetic female ovary:雌核发育雌鱼卵巢。The expression of miRNA/siRNA and piRNA pathway genes in brain and gonads of L.maculatus (A); L. calcarifer (B); gynogenetic P. olivaceus (C); C. semilaevis (D). The expression of each gene in the gonad of Pol and Cse was analyzed significantly. ** indicates P<0.01, * indicates P<0.05.) 图 9 四种硬骨鱼脑和性腺中miRNA/siRNA与piRNA途径基因的表达分析 Fig. 9 Expression of miRNA/siRNA and piRNA pathway genes in brain and gonads of four teleost fishes |
Argonaute基因家族几乎存在于所有真核生物中,但不同物种中其成员数有所差别。本研究共鉴定得到7个花鲈Argonaute基因家族成员(Ago1、Ago2、Ago3a、Ago3b、Ago4、Piwil1和Piwil2),而Piwil3和Piwil4仅在哺乳动物中存在,在硬骨鱼中却未发现,这种差异是在哺乳动物谱系中独立获得还是在鱼类谱系中丢失需进一步通过低等脊椎动物的比较分析进行验证。据报道,Piwil3位于基因组中一个不稳定的区域,该区域在哺乳动物中经历了多次复杂的重排,这可能是导致小鼠、大鼠和大象缺失Piwil3基因的原因[40]。此外,硬骨鱼中还出现了一个由全基因组复制产生的Ago3复制。有研究发现斑马鱼位于16号和19号染色体上的两个Ago3基因与人类位于1号染色体上的Ago3基因在染色体上的位置具有保守性[32]。斑马鱼的16号和19号染色体均由硬骨鱼特有的全基因组复制过程中硬骨鱼原染色体b复制产生,因此硬骨鱼中的Ago3a和Ago3b都是哺乳动物Ago3基因的同源基因,且是由全基因组复制产生的[41]。基因复制产生的同源基因在进化过程中可能经历不同的命运,包括去功能化(Nonfunctionalization),亚功能化(Subfunctionalization)和新功能化(Neofunctionalization)[42]。点模型结果显示,硬骨鱼Ago3b的dN/dS高于哺乳动物Ago3,而Ago3a的dN/dS与哺乳动物Ago3无明显差异。此外,在标记硬骨鱼祖先枝的枝模型中,硬骨鱼Ago3b的ω值显著低于哺乳动物Ago3;在标记全部硬骨鱼谱系的枝模型中,硬骨鱼Ago3b的ω值显著高于哺乳动物Ago3,而硬骨鱼Ago3a与哺乳动物Ago3的ω值差异并不显著。标记全部硬骨鱼谱系的枝模型结果与点模型结果更为相似,这可能是由于点模型是基于硬骨鱼分枝的每个位点分析,这与标记所有硬骨鱼分枝的枝模型更相似。Ago3b在硬骨鱼中的进化速率显著高于哺乳类,这种快速进化可能导致基因的新功能化。例如在斑马鱼、黄鳝和罗非鱼中均有研究显示,Ago3b在卵巢的表达量明显高于精巢,而Ago3a在精卵巢中的表达并无显著差异[27, 32, 34]。另有研究显示,腔棘鱼Ago3在卵巢中的表达量比精巢高[33]。本研究在花鲈基因组中也鉴定到了两个Ago3基因,表达分析显示花鲈Ago3a和Ago3b均在性腺中表达,其精巢表达均高于卵巢。分子进化结果显示花鲈Ago3a的进化速率更高,并且与其他硬骨鱼的Ago3a具有极显著差异。因此花鲈Ago3a的快速进化及表达模式可能反映其在生殖发育中的重要作用。
3.2 piRNA途径基因的快速进化RNAi过程中与抗病毒(siRNA)和转座子沉默(piRNA)相关的基因常表现出快速进化的特征,例如siRNA和piRNA途径相关基因的快速进化可能与病毒或转座子与宿主RNAi途径的协同进化有关[5, 8]。与哺乳动物基因组相比,硬骨鱼的基因组更简洁,但其转座子类型却更多样,多数硬骨鱼中含有丰富的DNA转座子,所有在脊椎动物中发现的转座子超家族(Gypsy,BEL/Pao,ERV和DIRS等)大多数都存在于硬骨鱼中[43]。这些转座子的活性可能会影响个体对环境的长期适应和生殖细胞基因组的稳定性[44]。由于硬骨鱼类多为体外受精,必须形成更多配子以保证受精率和后代成活率,因此硬骨鱼配子发生的过程更易受到转座子等的影响,piRNA途径基因作为调控转座子沉默的关键基因也将面临更大的选择压力。本文在点模型与枝模型检验中均得到piRNA途径基因在硬骨鱼中进化速率更快的结论,进一步证明在硬骨鱼中piRNA基因面临更大选择压力。牙鲆和黄鳝中也有研究显示piRNA途径基因的快速进化[27, 45]。在枝-点模型中,piRNA途径基因有更多的正选择位点,且部分正选择位点位于PAZ和PIWI结构域中,这些位点可以提供不同的piRNA结合位点,形成Piwi-piRNA复合体,并调控多种类型的转座子来维持基因组稳定。对花鲈Argonaute基因的进化分析发现,花鲈Piwil1基因比其它硬骨鱼进化更快,预示花鲈生殖系中也可能存在针对转座子多样性的适应性进化。
3.3 硬骨鱼类piRNA途径的功能探讨PIWI蛋白通常特异性地在动物生殖系细胞中表达,可与piRNA结合形成PIWI/piRNA复合物,抑制生殖系中移动遗传元件、抵御转座元件对基因组的侵袭和破坏,同时还参与调控蛋白编码基因的表达,维持生殖细胞的发育分化及配子形成[6-7, 46-48]。在模式生物线虫、果蝇和小鼠中,Piwi基因突变会引起生殖细胞发育缺陷,如生殖系建立失败,生殖干细胞减数分裂停滞,精子发生受阻,最后造成不育[8, 11, 46]。除了模式生物以外,Piwi基因的表达模式在硬骨鱼中也有报道。斑马鱼中Piwi基因特异性在精巢和卵巢中表达,Ziwi的突变会导致转座子转录水平增加以及阻遏生殖细胞的分化,最终导致生殖细胞的凋亡[26]。中华鲟(Acipenser sinensis)Piwil1在生殖细胞中特异表达[49];青鳉(Oryzias latipes)Piwil1和Piwil2主要在性腺中表达[50];牙鲆Piwil1主要在性腺中表达且在精巢中的表达量高于卵巢[45]。本研究中,花鲈和亚洲鲈均为广盐性鱼类,在淡水中可以生长但不能完成性腺发育和配子成熟[28, 51]。花鲈和亚洲鲈Piwil1和Piwil2在精巢中的表达高于卵巢,且Piwil1的表达量明显高于Piwil2,这表明Piwil1和Piwil2基因可能参与不同的piRNA加工过程,并在精子发生过程中起着更重要的调控作用。牙鲆和半滑舌鳎都存在性别分化关键期受环境影响而发生的遗传雌性向雄性转变的性逆转现象,牙鲆Piwil1和Piwil2在精巢中的表达量高于卵巢,半滑舌鳎Piwil2在精巢中的表达量高于卵巢,两者Piwil1在性腺的表达量明显高于Piwil2,说明Piwil1可能在伪雄鱼与正常雄鱼的生精过程中发挥不同的调控作用。然而Piwi基因在硬骨鱼配子发生中的调控作用还需要进一步的功能实验来进行验证。
| [1] |
Zamore P D, Tuschl T, Sharp P A, et al. RNAi: double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals[J]. Cell, 2000, 101(1): 25-33. DOI:10.1016/S0092-8674(00)80620-0
(  0) 0) |
| [2] |
Aravin A A, Sachidanandam R, Girard A, et al. Developmentally regulated piRNA clusters implicate MILI in transposon control[J]. Science, 2007, 316(5825): 744-747. DOI:10.1126/science.1142612
(  0) 0) |
| [3] |
Sato K, Siomi H. PIWI proteins and their slicer activity in piRNA biogenesis and transposon silencing[J]. The Enzymes, 2012, 32: 137-162.
(  0) 0) |
| [4] |
Peters L, Meister G. Argonaute proteins: Mediators of RNA silencing[J]. Molecular Cell, 2007, 26(5): 611-623. DOI:10.1016/j.molcel.2007.05.001
(  0) 0) |
| [5] |
Wynant N, Santos D, Vanden Broeck J. The evolution of animal Argonautes: Evidence for the absence of antiviral AGO Argonautes in vertebrates[J]. Scientific Reports, 2017, 7(1): 477. DOI:10.1038/s41598-017-00490-4
(  0) 0) |
| [6] |
Hutvagner G, Simard M J. Argonaute proteins: Key players in RNA silencing[J]. Molecular and Cellular Biology, 2008, 9(1): 22-32.
(  0) 0) |
| [7] |
Gainetdinov I, Colpan C, Arif A, et al. A single mechanism of biogenesis, initiated and directed by PIWI proteins, explains piRNA production in most animals[J]. Molecular Cell, 2018, 71(5): 775-790. DOI:10.1016/j.molcel.2018.08.007
(  0) 0) |
| [8] |
Parhad S S, Theurkauf W E. Rapid evolution and conserved function of the piRNA pathway[J]. Royal Society Open Biology, 2019, 9(1): 180-181.
(  0) 0) |
| [9] |
William H P, Jarrod D H, Darren J O. RNA-interference pathways display high rates of adaptive protein evolution in multiple invertebrates[J]. Genetics, 2018, 208(4): 1585-1599. DOI:10.1534/genetics.117.300567
(  0) 0) |
| [10] |
Ozata M D, Ildar G, Ansgar Z, et al. PIWI-interacting RNAs: Small RNAs with big functions[J]. Nature Reviews Genetics, 2019, 20(2): 89-108. DOI:10.1038/s41576-018-0073-3
(  0) 0) |
| [11] |
Malone C D, Hannon G J. Small RNAs as Guardians of the Genome[J]. Cell, 2009, 136(4): 656-668. DOI:10.1016/j.cell.2009.01.045
(  0) 0) |
| [12] |
Luo L F, Hou C C, Yang W X. Small non-coding RNAs and their associated proteins in spermatogenesis[J]. Gene, 2016, 578(2): 141-157. DOI:10.1016/j.gene.2015.12.020
(  0) 0) |
| [13] |
Christina E, Duncan T O, Claudia K. The emergence of piRNAs against transposon invasion to preserve mammalian genome integrity[J]. Nature Communications, 2017, 8(1): 1411. DOI:10.1038/s41467-017-01049-7
(  0) 0) |
| [14] |
Orban L, Sreenivasan R, Olsson P E. Long and winding roads: Testis differentiation in zebrafish[J]. Molecular and Cellular Endocrinology, 2009, 312(1): 35-41.
(  0) 0) |
| [15] |
Komen J, De B P, Richter C. Male sex reversal in gynogenetic XX females of common carp (Cyprinus carpio L.) by a recessive mutation in a sex-determining gene[J]. Journal of Heredity, 1992, 83(6): 431-434. DOI:10.1093/oxfordjournals.jhered.a111246
(  0) 0) |
| [16] |
Hong W S, Chen S X, Zheng W Y, et al. Hermaphroditism in Cultured Chinese Black Sleeper (Bostrichthys sinensis L.)[J]. Journal of the World Aquaculture Society, 2006, 37(4): 363-369. DOI:10.1111/j.1749-7345.2006.00049.x
(  0) 0) |
| [17] |
刘建康, 顾国彦. 鳝鱼性别逆转时生殖腺组织改变[J]. 科学, 1950(3): 91. Liu J K, Gu G Y. Morphological changes in the gonad of monopterus during sex reversal[J]. Science, 1950(3): 91. (  0) 0) |
| [18] |
Wu G C, Tomy S, Lee M F, et al. Sex differentiation and sex change in the protandrous black porgy, Acanthopagrus schlegeli[J]. General and Comparative Endocrinology, 2010, 167(3): 417-421. DOI:10.1016/j.ygcen.2009.11.003
(  0) 0) |
| [19] |
Ospina-álvarez N, Piferrer F. Temperature-dependent sex determination in fish revisited: Prevalence, a single sex ratio response pattern, and possible effects of climate change[J]. PLoS One, 2008, 3(7): e2837. DOI:10.1371/journal.pone.0002837
(  0) 0) |
| [20] |
Baroiller F J, D′Cotta H. Environment and sex determination in farmed fish[J]. Comparative Biochemistry and Physiology-Part C: Toxicology & Pharmacology, 2001, 130(4): 399-409.
(  0) 0) |
| [21] |
Wan R. Reproductive behavior and early development of Lateolabrax japonicus (Cuvier & Valenciennes) in the Bohai Sea[J]. Marine Fisheries Research, 1988, 9: 203-211.
(  0) 0) |
| [22] |
Luckenbach J, Borski R, Daniels H, et al. Sex determination in flatfishes: Mechanisms and environmental influences[J]. Seminars in Cell & Developmental Biology, 2009, 20(3): 256-263.
(  0) 0) |
| [23] |
Rubin D. Effect of pH on sex ratio in cichlids and a poecilliid (Teleostei)[J]. Copeia, 1985(1): 233-235.
(  0) 0) |
| [24] |
Volff J. Genome evolution and biodiversity in teleost fish[J]. Heredity, 2005, 94(3): 280-294. DOI:10.1038/sj.hdy.6800635
(  0) 0) |
| [25] |
Izumi N, Shoji K, Sakaguchi Y, et al. Identification and functional analysis of the pre-piRNA 3′ trimmer in silkworms[J]. Cell, 2016, 164(5): 962-973. DOI:10.1016/j.cell.2016.01.008
(  0) 0) |
| [26] |
Houwing S, Berezikov E, Ketting R F. Zili is required for germ cell differentiation and meiosis in zebrafish[J]. The EMBO Journal, 2008, 27(20): 2702-2711. DOI:10.1038/emboj.2008.204
(  0) 0) |
| [27] |
Yi M, Chen F, Luo M, et al. Rapid evolution of piRNA pathway in the teleost fish: Implication for an adaptation to transposon diversity[J]. Genome Biology and Evolution, 2014, 6(6): 1393-407. DOI:10.1093/gbe/evu105
(  0) 0) |
| [28] |
Wang Z P, Wang D, Wang C L, et al. Transcriptome characterization of HPG axis from Chinese sea perch Lateolabrax maculatus[J]. Journal of Fish Biology, 2017, 91(5): 1407-1418. DOI:10.1111/jfb.13459
(  0) 0) |
| [29] |
Yang Z, Wong W, Rasmus N. Bayes empirical bayes inference of amino acid sites under positive selection[J]. Molecular Biology and Evolution, 2005, 22(4): 1107-1118. DOI:10.1093/molbev/msi097
(  0) 0) |
| [30] |
Yang Z. PAML 4: phylogenetic analysis by maximum likelihood[J]. Molecular Biology and Evolution, 2007, 24(8): 1586-1591. DOI:10.1093/molbev/msm088
(  0) 0) |
| [31] |
Liu J, Liu X, Jin C, et al. Transcriptome profiling insights the feature of sex reversal induced by high temperature in tongue sole Cynoglossus semilaevis[J]. Frontiers in Genetics, 2019, 10: 522. DOI:10.3389/fgene.2019.00522
(  0) 0) |
| [32] |
Mcfarlane L, Svingen T, Braasch I, et al. Expansion of the Ago gene family in the teleost clade[J]. Development Genes and Evolution, 2011, 221(2): 95-104. DOI:10.1007/s00427-011-0363-7
(  0) 0) |
| [33] |
Assunta B M, Adriana C, Mariko F, et al. The small non-coding RNA processing machinery of two living fossil species, lungfish and coelacanth, gives new insights into the evolution of the Argonaute protein family[J]. Genome Biology and Evolution, 2017, 9(3): 438-453. DOI:10.1093/gbe/evx017
(  0) 0) |
| [34] |
Tao W, Sun L, Chen J, et al. Genomic identification, rapid evolution, and expression of Argonaute genes in the tilapia, Oreochromis niloticus[J]. Development Genes and Evolution, 2016, 226(5): 339-348. DOI:10.1007/s00427-016-0554-3
(  0) 0) |
| [35] |
周莉, 汪洋, 桂建芳. 鱼类特异的基因组复制[J]. 动物学研究, 2006(5): 525-532. Zhou L, Wang Y, Gui J F. Fish-specific genome duplication[J]. Zoological Research, 2006(5): 525-532. (  0) 0) |
| [36] |
Hall T M. Structure and function of argonaute proteins[J]. Structure, 2005, 13(10): 1403-1408. DOI:10.1016/j.str.2005.08.005
(  0) 0) |
| [37] |
李超, 杜志游, 陈集双. 解读AGO蛋白结构及其功能[J]. 中国生物化学与分子生物学报, 2009, 25(11): 969-976. Li C, Du Z Y, Chen J S. Structural and functional elucidation of AGO proteins[J]. Chinese Journal of Biochemistry and Molecular Biology, 2009, 25(11): 969-976. (  0) 0) |
| [38] |
Andreas L, Bernd S, Elisa I, et al. Structure and nucleic-acid binding of the Drosophila Argonaute 2 PAZ domain[J]. Nature, 2003, 426(6965): 465-469. DOI:10.1038/nature02123
(  0) 0) |
| [39] |
Cerutti L, Mian N, Bateman A. Domains in gene silencing and cell differentiation proteins: The novel PAZ domain and redefinition of the Piwi domain[J]. Trends in Biochemical Sciences, 2000, 25(10): 481-482. DOI:10.1016/S0968-0004(00)01641-8
(  0) 0) |
| [40] |
Lim S L, Tsend-Ayush E, Kortschak R D, et al. Conservation and expression of PIWI-interacting RNA pathway genes in male and female adult gonad of amniotes[J]. Biology of Reproduction, 2013, 89(6): 136.
(  0) 0) |
| [41] |
Kassahn K S, Dang V T, Wilkins S J, et al. Evolution of gene function and regulatory control after whole-genome duplication: Comparative analyses in vertebrates[J]. Genome Research, 2009, 19(8): 1404-1418. DOI:10.1101/gr.086827.108
(  0) 0) |
| [42] |
Venkatachalam A B, Parmar M B, Wright J M. Evolution of the duplicated intracellular lipid-binding protein genes of teleost fishes[J]. Molecular Genetics and Genomics, 2017, 292(4): 699-727. DOI:10.1007/s00438-017-1313-5
(  0) 0) |
| [43] |
Domitille C, Magali N, Floriane P, et al. Comparative analysis of transposable elements highlights mobilome diversity and evolution in vertebrates[J]. Genome Biology and Evolution, 2015, 7(2): 567-580. DOI:10.1093/gbe/evv005
(  0) 0) |
| [44] |
Malone C D, Brennecke J, Dus M, et al. Specialized piRNA pathways act in germline and somatic tissues of the drosophila ovary[J]. Cell, 2009, 137(3): 522-535. DOI:10.1016/j.cell.2009.03.040
(  0) 0) |
| [45] |
Song H, Xing C, Lu W, et al. Rapid evolution of piRNA pathway and its transposon targets in Japanese flounder (Paralichthys olivaceus)[J]. Comparative Biochemistry and Physiology-Part D: Genomics and Proteomics, 2019, 31: 100609. DOI:10.1016/j.cbd.2019.100609
(  0) 0) |
| [46] |
Houwing S, Kamminga L M, Berezikov E, et al. A role for piwi and pirnas in germ cell maintenance and transposon silencing in zebrafish[J]. Cell, 2007, 129(1): 69-82. DOI:10.1016/j.cell.2007.03.026
(  0) 0) |
| [47] |
Ni F, Yu H, Liu Y, et al. Roles of piwil1 gene in gonad development and gametogenesis in Japanese flounder, Paralichthys olivaceus[J]. Gene, 2019, 701: 104-112. DOI:10.1016/j.gene.2019.03.045
(  0) 0) |
| [48] |
Vourekas A, Zheng Q, Alexiou P, et al. Mili and Miwi target RNA repertoire reveals piRNA biogenesis and function of Miwi in spermiogenesis[J]. Nature Structural & Molecular Biology, 2012, 19(8): 773-781.
(  0) 0) |
| [49] |
李创举, 杨晓鸽, 岳华梅, 等. 中华鲟piwil1基因的克隆表达特征研究[J]. 淡水渔业, 2016, 46(6): 20-25. Li C J, Yang X G, Yue H M, et al. Molecular cloning and expression analysis of piwil1 gene in Acipenser sinensis[J]. Freshwater Fisheries, 2016, 46(6): 20-25. (  0) 0) |
| [50] |
Zhao H, Duan J, Cheng N, et al. Specific expression of Olpiwi1and Olpiwi2in medaka (Oryzias latipes) germ cells[J]. Biochemical and Biophysical Research Communications, 2012, 418(4): 592-597. DOI:10.1016/j.bbrc.2011.12.062
(  0) 0) |
| [51] |
Athauda S, Anderson T, Nys R D. Effect of rearing water temperature on protandrous sex inversion in cultured Asian seabass (Lates calcarifer)[J]. General and Comparative Endocrinology, 2011, 175(3): 416-423.
(  0) 0) |
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
 2021, Vol. 51
2021, Vol. 51


