Fem-1(Ferminazation-1)基因最早是在秀丽隐杆线虫(Caenorhabditis elegans)的研究中被发现,并且在其性别决定通路中发挥了十分关键的作用。该基因不但对雄性体组织具有致雄化作用,还可以对雄性和雌雄同体个体中的雄性生殖细胞产生调控作用[1-4]。在秀丽隐杆线虫的性别决定通路中,Fem基因既可以负调控下游的Tra-1基因,又受到了上游Tra-2和Tra-3的负调控[5-7]。除了线虫以外,Fem-1基因家族也在人类、小鼠和斑马鱼中被发现[8-11]。截止目前的研究表明,Fem-1基因家族包含Fem-1a,Fem-1b和Fem-1c三个基因,尽管这3个基因的具体功能还不明确,但是它们编码的氨基酸序列存在一定的相似性而且在脊椎动物和线虫之间存在一定的保守性。由此推测脊椎动物中Fem-1基因的功能可能与线虫相似,在调节细胞凋亡方面发挥重要作用[12]。迄今,双壳贝类中Fem-1基因的研究目前仅在长牡蛎基因组和转录组的研究中有所报道[13]。
长牡蛎(Crassostrea gigas)又称太平洋牡蛎,隶属软体动物门(Mollusca)瓣鳃纲(Lamellivranchia)翼形亚纲(Pterimorphia)珍珠贝目(Pterioida)牡蛎科(Ostridae)巨蛎属(Crassostrea)。作为我国优良的海水养殖贝类,长牡蛎具有抗逆性强、体型大、生长速度快、营养丰富、肉质鲜美等特点,具有重要的市场经济价值,现已成为我国北方牡蛎养殖的主要品种。然而,长牡蛎在繁殖过程中存在很多特殊的繁殖生理现象,比如性逆转和雌雄同体等现象。截止目前,这些现象的机理还不清楚,长牡蛎性别决定和分化机制还不明确[13-15]。本文通过RACE技术和荧光定量技术对长牡蛎Fem-1b和Fem-1c两个基因进行克隆并且利用采集的周年样品和不同胚胎发育时期的样品进行表达分析。通过本研究,旨在为长牡蛎性腺发育的分子机制研究提供基础数据。
1 材料与方法 1.1 实验动物2015年5月—2016年4月每月在齐东路水产市场购买来自乳山海域的长牡蛎20~25个。对样品进行解剖得到一部分性腺组织保存于波恩氏液中固定24 h,经过数次酒精洗涤后最终保存于70%酒精溶液中等待组织切片的制作,用于观察确定样品所处的性腺发育时期。解剖取得的目标组织保存于RNA保存液中,-30 ℃待用。目标组织包括所有样品的性腺组织,以及5—8月样品的外套膜、唇瓣、消化腺、闭壳肌和鳃组织。2016年8月采集长牡蛎胚胎各发育时期样品,置于RNA保存液中-30 ℃保存待用。
1.2 长牡蛎Fem-1b和Fem-1c基因克隆采用Trizol法对长牡蛎各组织总RNA进行提取。提取结束后利用Nanodrop检测所提的RNA的浓度,接着利用1.5%琼脂糖凝胶电检测所提RNA完整性。利用试剂盒SMARTTM RACE cDNA完成RACE cDNA第一链的合成。在NCBI数据库中下载人(Homo sapiens)、小鼠(Mus musculus)、池蝶蚌(Hyriopsis schlegelii)和加州海兔(Aplisia californica)等物种的Fem-1b和Fem-1c蛋白序列,利用这些序列与长牡蛎基因组数据库进行比对获得预测长牡蛎Fem-1b和Fem-1c基因片段。根据所得片段设计引物扩增核心序列并验证。根据验证所得的核心序列设计RACE引物,采用巢式PCR的方法扩增得到2个基因的3′和5′端序列。实验所用引物见表 1。利用1.5%琼脂糖凝胶电检测得到PCR产物长度符合预期后采用DNA凝胶回收纯化试剂盒的操作对目的片段进行纯化回收。将目的片段连接至pEASY-T1载体,后转化至大肠杆菌感受态细胞中。通过蓝白斑筛选的方式挑取阳性克隆,经过菌液PCR检测。根据检测结果挑选合适的菌液送至北京华大基因测序。根据测序结果对目的基因片段进行拼接得到目的基因cDNA全长序列。
|
|
表 1 Fem-1b和Fem-1c引物信息 Table 1 The information of the Fem-1b and Fem-1c genes primers in Crassostrea gigas |
利用ORF finder软件对目的基因开放阅读框进行预测。氨基酸序列的同源性比对由NCBI网站上的BLAST程序完成。通过NJ法建立的进化树通过MEGA5.0软件完成。除此之外,等电点以及相对分子质量的预测则是通过在线软件工具(http://web.expasy.org/compute_pi/)进行预测。
1.4 RT-PCR分析根据周年样品性腺石蜡切片的结果将长牡蛎性腺样品分为增殖期、生长期、成熟期、排放期和休止期(休止期无法分辨雌雄)5个时期。选择每月占多数时期的样品雌雄各3个组成周年样品。他们对应的生长发育时期依次是10月至次年2月为休止期,3月为增殖期,4、5月为生长期,6、7月为成熟期,8、9月为排放期。组织分布的分析选择6、7月中处于成熟期个体的雌雄性腺、外套膜、闭壳肌、唇瓣、消化腺和鳃。不同胚胎时期的样品包括受精卵、2细胞期、4细胞期、囊胚期、壳顶幼虫,D形幼虫和眼点幼虫。通过Trizol法对所有目的组织样品进行总RNA的提取,然后利用PrimeScriptTMRT reagent Kit进行cDNA第一链的合成。根据克隆所得的目的基因片段设计荧光定量特异性引物。成熟期各组织表达分析方面,利用EF2基因的表达量作为内参而对于胚胎幼虫表达分析采用18S基因的表达量作为内参[16]。实验所用引物见表 1。实验使用Roche480荧光定量PCR仪对目标实验样品进行分析,每一组设置3个样品。PCR实验的反应条件如下:94 ℃条件下5 s,60 ℃ 30 s总共进行45个循环。采用2-ΔΔCt法对基因的相对表达量进行分析。所得数据以3样本平均值±标准误(X±SE)来表示, 使用SPSS 16.0进行单因素方差分析(One-Way ANOVA), 使用Turkey B检验法对其进行两两差异比较, 以P<0.05作为显著性差异水平。
2 实验结果 2.2 长牡蛎Fem-1b和Fem-1c基因序列特征及系统进化分析通过测序结果进行拼接得到长牡蛎Fem-1b和Fem-1c cDNA全长序列(见图 1)。Cg-Fem-1b cDNA序列全长为2 580 bp,开放阅读框1 911 bp,共编码了636个氨基酸。通过在线软件Expasy compute Pi/Mw推测Cg-Fem1b的分子量为71.32 kDa,等电点为5.46。Cg-Fem-1c基因全长为2 147 bp,其中开放阅读框1 869 bp,编码622个氨基酸,等电点为4.93,相对分子质量为17.71 kDa。

|
(小写字母表示非编码区序列,大写字母表示编码区序列。The lowercase indicates the untranslated region of Cg-Fem1b and Cg-Fem-1c, while the uppercase indicates the open reading frame.) 图 1 Fem-1b和Fem-1c基因核苷酸和预测的氨基酸序列信息 Fig. 1 Nucleotide and deduced amino acid sequence of Fem-1b and Fem1c gene from Crassostrea gigas |
Fem-1b和Fem-1c编码蛋白结构的预测显示,Cg-Fem-1b的氨基酸序列具有6个典型的锚定蛋白重复序列(Anyrin repeat)模体结构而Cg-Fem-1c序列含有7个ANK模体结构(见图 2)。
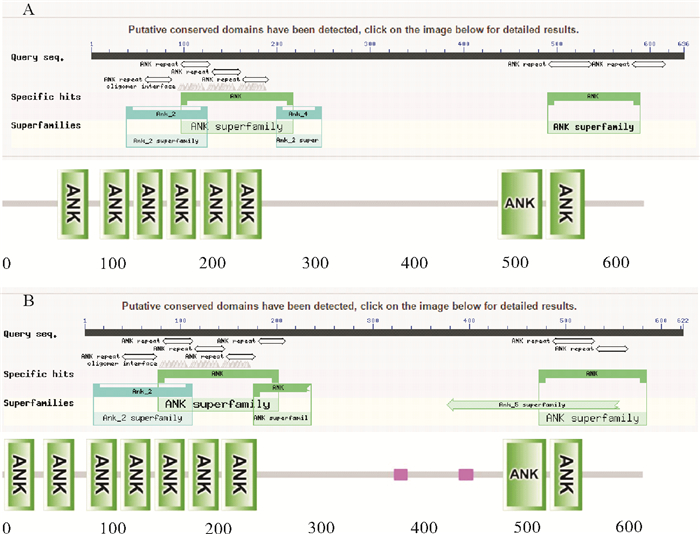
|
(A. Cg-Fem-1b码蛋白结构的预测;B. Cg-Fem-1c编码蛋白结构的预测。A. Prediction of protein structure of Cg-Fem-1b. B. Prediction of protein structure of Cg-Fem-1c.) 图 2 Cg-Fem-1b和Cg-Fem-1c编码蛋白结构的预测 Fig. 2 Prediction of protein structure of Cg-Fem-1b and Cg-Fem-1c |
Cg-Fem-1b和Cg-Fem-1c两序列比对结果显示,两序列之间同源性较低,只有37%。然而在与其他物种的氨基酸的序列比对中却发现它们与相应基因的具有较高的同源性。Cg-Fem-1b与池蝶蚌的相似度可以达到59%。此外,相似度较高的物种大多是双壳贝类,比如Cg-Fem-1b与加州海兔和光滑双脐螺(Biomphalaria glabrata)的相似度分别为50%和51%。Cg-Fem-1c与池蝶蚌的相似度高达79%。除此之外,与章鱼(Octopus bimaculoides)和光滑双脐螺也具有较高的相似度,分别是72%和64%。通过NCBI的数据库下载获得大量模式动物及近缘种的Fem-1基因的氨基酸序列(见表 2),使用软件MEGA5.0通过NJ法建立系统进化树(见图 3)。经过对长牡蛎基因组的筛选,我们得到包括本文2个目的基因在内的一共8个被标注为长牡蛎Fem-1基因的序列。通过8个序列与模式动物中Fem-1a、Fem-1b、Fem-1c以及近缘基因Fem-3基因的进化树构建,我们发现Cg-Fem-1b与其他模式动物的Fem-1b基因聚为一支而Cg-Fem-1c与其他双壳贝类的Fem-1c聚为一支。除此之外,其他被标注为长牡蛎Fem-1基因的序列则没有与模式动物的Fem-1基因聚类。具体的聚类分支结果与相似性比对结果基本一致,Cg-Fem-1b和Cg-Fem-1c都优先与池蝶蚌的相应基因聚为一支,与脊椎动物进化关系较远。
|
|
表 2 Fem-1b和Fem-1c系统进化树中氨基酸序列的来源信息 Table 2 The Fem-1b and Fem-1c protein information used for phylogenic analysis |
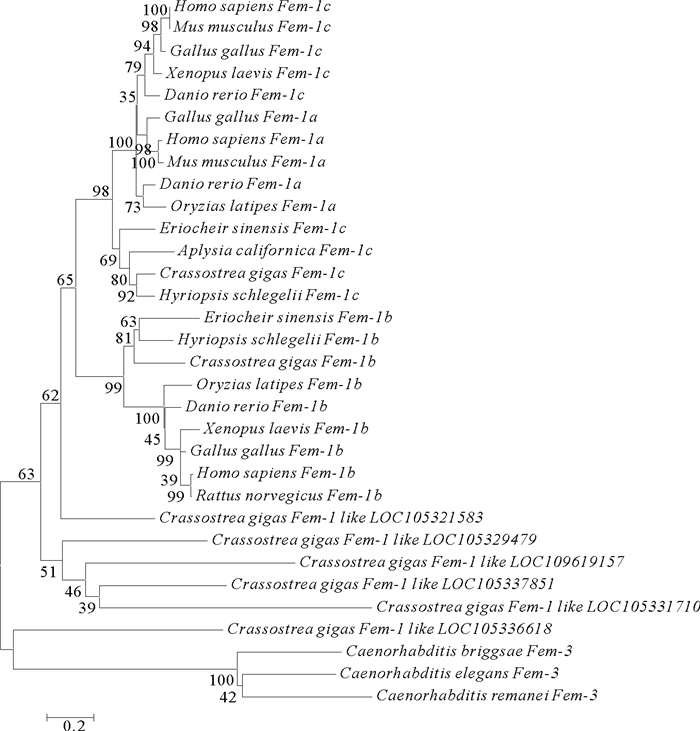
|
(GenBank注册号见表 2 Genbank Accession numbers are indicated in Table 2) 图 3 长牡蛎与其他代表物种Fem-1b和Fem-1c氨基酸序列的系统进化分析 Fig. 3 A phylogenetic tree based on the Fem-1b and Fem-1c family members of Crassostrea gigas and other representative species |
通过荧光定量PCR实验分别分析了Cg-Fem-1b和Cg-Fem-1c在处于成熟期的长牡蛎个体的各组织(闭壳肌,鳃,消化腺,外套膜,唇瓣以及雌性性腺)的表达模式。整体上看,在各组织的内参基因CT值均小于25且各组织目的基因的CT值较小,因此我们可以发现Cg-Fem-1b和Cg-Fem-1c的表达具有广泛表达的特点,且精巢的表达量显著高于其他组织(P<0.05)。除此之外,Cg-Fem-1b在唇瓣,闭壳肌中也有相对于除精巢外其他组织显著高的表达量(P<0.05),而Cg-Fem-1c在除精巢外的其他组织之间不存在显著性差异(P>0.05)(见图 4)。
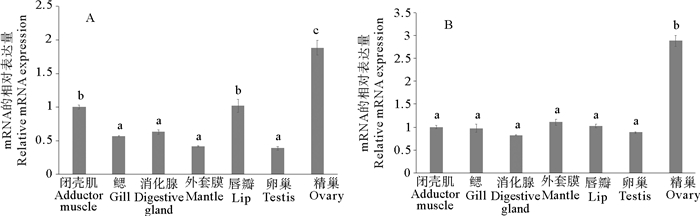
|
(A. Cg-Fem-1b在不同组织中的表达情况; B. Cg-Fem-1c在不同组织中的表达情况。不同的字母代表差异性显著(P < 0.05,平均值±标准差,n=3)。A. Relative expression of Cg-Fem-1 b. B. Relative expression of Cg-Fem-1c. Different letters indicate significant differences statistically(P < 0.05,Means±SE, n=3).) 图 4 Cg-Fem-1b和Cg-Fem-1c在不同组织中的表达情况 Fig. 4 Relative expression of Cg-Fem-1b and Cg-Fem-1c in different tissues |
Cg-Fem-1b和Cg-Fem-1c表达具有明显的性别二态性,即精巢中的表达量显著高于卵巢(见图 4)。除此之外,休止期(2015年10月—2016年2月)的表达量显著低于其他性腺发育时期(P < 0.05)。精巢中Cg-Fem-1c在3月(增殖期)和4月(生长期)表达量显著高于其他组(P < 0.05)。相比之下,Cg-Fem-1b在6月(成熟期)才出现显著地上调,并在之后的7月(成熟期)保持在了显著高于其他组的水平(P < 0.05)。相比之下,卵巢中两基因的表达量,除了7月表达量显著高于其他组外,其他月份卵巢中的表达量之间没有明显变化(P>0.05)(见图 5)。

|
(A. Cg-Fem-1b在周年性腺样品中的表达情况。B.Cg-Fem-1c在周年性腺样品中的表达情况。不同的字母代表差异性显著(P < 0.05,平均值±标准差,n=3)。A. Relative expression of Cg-Fem-1b uring different months.B. Relative expression of Cg-Fem-1c during different months. Different letters indicate significant differences statistically(P < 0.05,Means±SE, n=3).) 图 5 Cg-Fem-1b和Cg-Fem-1c在周年性腺样品中的表达情况 Fig. 5 Relative expression of Cg-Fem-1b and Cg-Fem-1c during different months |
针对不同胚胎幼虫发育阶段的荧光定量实验结果显示,Cg-Fem-1b和Cg-Fem-1c的整体表达模式类似,最高表达量都出现在囊胚期并且显著高于其他时期(P < 0.05)。Cg-Fem-1b在囊胚期的表达量是受精卵表达量的5.56倍,而Cg-Fem-1c在囊胚期的表达量是受精卵时期表达量的18.38倍(见图 6)。除此之外,Cg-Fem-1b在2细胞期、4细胞期以及幼虫期显著高于除囊胚期外的其他时期(P < 0.05)。Cg-Fem-1c和Cg-Fem-1c在胚胎发育早期,即2细胞期和4细胞期以及幼虫期也有显著高于除囊胚期外其他时期的高表达模式(P < 0.05)。Cg-Fem-1c和Cg-Fem-1c在受精卵、担轮幼虫、D形幼虫、壳顶幼虫以及眼点幼虫表达量显著低于其他时期(P < 0.05)且彼此之间不存在显著性差异(P>0.05)。
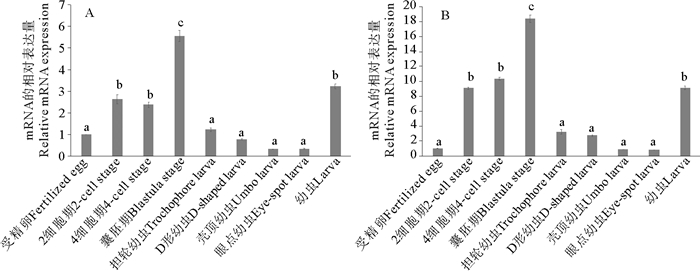
|
(A. Cg-Fem-1b在不同胚胎及幼虫发育时期中的表达情况; B. Cg-Fem-1c在不同胚胎及幼虫发育时期中的表达情况。不同的字母代表差异性显著(P < 0.05,平均值±标准差,n=3)。A. Relative expression of Cg-Fem-1b during different embryonic and larval developmental stages; B. Relative expression of Cg-Fem-1c during different embryonic and larval developmental stages. Different letters indicate significant differences statistically(P < 0.05,Means±SE, n=3).) 图 6 Cg-Fem-1b和Cg-Fem-1c在不同胚胎及幼虫发育时期中的表达情况 Fig. 6 Relative expression of Cg-Fem-1b and Cg-Fem-1c during different embryonic and larval developmental stages |
总体来看, Cg-Fem-1b和Cg-Fem-1c在组织分布情况中具有类似的表达模式。Cg-Fem-1b和Cg-Fem-1c在所有检测组织(闭壳肌,鳃,消化腺,外套膜,唇瓣以及雌性性腺)均有表达。在这种广泛表达的特征在许多动物的研究中都有所体现,比如池蝶蚌、线虫、东亚飞蝗和毛蟹等物种[17-20]。这种不同存在于不同物种之间相似的表达特征说明了Fem-1基因在基因表达上具有一定的保守性。由此我们推测长牡蛎中Fem-1b和Fem-1c两个基因可能参与了基础并且广泛的生物学过程,比如在秀丽隐杆线虫中Fem-1基因参与细胞凋亡的调控[12]。尽管Fem-1基因在不同物种中都有广泛表达的模式,但是其具体的表达模式却各有特点。在人类和小鼠的研究中发现,Fem-1a在成体骨骼肌和心脏中具有高表达的特征,而Fem-1b和Fem-1c则在睾丸中表达丰富,研究者推测Fem-1b参与了雄性性别决定的过程,而Fem-1c在精子发生过程中发挥了重要的作用[9-10]。在本实验中Cg-Fem-1b除了在精巢中高表达之外,在闭壳肌以及唇瓣中也有显著高于其他组织的表达特点。东亚飞蝗的研究表明,Fem-1基因在精巢中的表达量显著高于其他组织[21-23]。在毛蟹的研究中发现Fem-1a、Fem-1b和Fem-1c三个基因的最高表达量分别出现在肝胰腺、精巢和肌肉中[20]。这种表达模式被认为与毛蟹的性别决定和分化过程具有重要的关联,因为有研究表明在毛蟹中肝胰腺可以为性腺的发育提供大量的能量[23-24]。由此我们推测,Cg-Fem-1b在闭壳肌中的高表达可能暗示了在肌肉为精巢的发育提供了能量。此外,Cg-Fem-1c除了在精巢中表达量显著高于其他组织外,其他组织的表达量之间不存在显著性差异。池蝶蚌Fem-1c在精巢中表达量最高,此外在肝胰腺、肠和卵巢中也有较高表达量而在肾脏中则几乎不表达[17]。在已有的长牡蛎组织表达谱中发现,长牡蛎的Fem-1b和Fem-1c都在血淋巴中具有较高的表达水平,除了血淋巴之外,Fem-1b在唇瓣和卵巢中具有较高表达量而Fem-1c在闭壳肌中具有较高的表达[13]。这与我们的研究结果有一定差别,我们推测这种差别可能是样品所处的不同海域的海区环境条件不同造成的。综上,在长牡蛎中Cg-Fem-1b和Cg-Fem-1c两个基因都具有精巢中的高表达模式,我们推测它们很可能都参与了长牡蛎精巢的发育和精子的形成。此外,Cg-Fem-1b在闭壳肌以及唇瓣中也具有相对较高的表达模式,我们推测Cg-Fem-1b在闭壳肌中的高表达可能与精巢发育过程中的能量供应有关。
在秀丽隐杆线虫的研究中发现Fem-1在性别决定和分化过程中发挥了关键的作用[1-2]。在很多物种的研究中都发现了Fem-1基因,并且呈现出与性别相关的特征。研究者在大马哈鱼虱(Caligus rogercresseyi)的研究中通过对雌雄个体的转录组测序结果进行筛选得到了Fem-1b,发现其具有性别表达差异的模式[25]。同样是通过雌性性腺的转录组分析,斑马鱼中发现Fem-1c基因在卵巢中具有高表达的特征,然而其具体的功能还不明确[26]。在斑胸草雀(Zebra finch)发现Fem-1c基因存在2个拷贝,分别位于Z和W两条性染色体中[27]。斑节对虾的研究中发现Fem-1基因上存在一个与性别决定有着明显关联的SNP位点[28]。在长牡蛎中,Fem-1b和Fem-1c在精巢中表达量显著高于其他组织,而且在各个繁殖期精巢的表达量都显著高于休止期。2个基因不同的是,Cg-Fem-1b在6~9月(成熟期和排放期)的表达量显著高于其他时期,这暗示了Cg-Fem-1b基因可能在精巢功能的维持以及诱发精子排放等发面发挥了重要的作用。反观Cg-Fem-1c基因则是在3和4月的(增殖期和生长期)表达量显著高于其他组,这种在精巢发育初期高表达的模式说了Cg-Fem-1c可能参与了精巢早期的发育以及精子的形成。在东亚飞蝗的研究中发现,随着精巢的发育Fem-1三个基因表达量都有显著的上调,相比Fem-1c在第3天有显著性上调,Fem-1a和Fem-1b基因的上调发生在第12天[18]。我们推测长牡蛎的Fem-1基因的表达可能与东亚飞蝗类似,即Fem-1c表达量的显著性提高的时期与Fem-1b有所不同,这暗示了2个目的基因在精巢发育中可能起到了不同的作用。然而具体作用如何,还需要进一步的分析。在小鼠的研究中发现通过外源干扰的手段降低了Fem-1c基因的表达,并没有导致雌性幼崽数量的显著提高,研究者得出结论Fem-1c可能与小鼠的性别分化和性别决定没有关系[29]。这暗示了Fem-1c基因在性别分化作用中不保守的特点,因此其基因的具体功能还需要进一步的探究。针对不同胚胎幼虫发育阶段的荧光定量实验结果显示,Cg-Fem-1c在胚胎发育期的表达模式与Cg-Fem-1b类似,即都在2细胞和4细胞期显著上调,在之后的囊胚期继续上调至最高值,后随着胚胎幼虫的发育表达量逐渐降低。两者不同的是Cg-Fem-1c在囊胚期表达量是其在受精卵时期表达量的18.83倍,而Cg-Fem-1b只有5.56倍。随着幼虫发育,Cg-Fem-1b和Cg-Fem-1c表达量持续降低并在壳顶幼虫和眼点幼虫时达到最低水平。在毛蟹的研究中也有类似的表达模式,Fem-1基因从受精卵开始表达,持续上调至囊胚期后剧烈下降并保持在低水平[20]。囊胚期是细胞分化的关键时期,Cg-Fem-1b和Cg-Fem-1c在胚胎发育早期,特别是囊胚的高表达模式暗示了其在组织器官早期发育中的作用以及可能参与了性别决定和分化的过程。
4 结论对不同胚胎发育时期的样品分析显示,Cg-Fem-1b和Cg-Fem-1c具有类似的表达模式,即都是在胚胎发育早期表达量上升并在囊胚期达到最大值。囊胚期是组织器官形成以及细胞分化的重要时期,我们推测Cg-Fem-1b和Cg-Fem-1c可能参与了性别决定和性别分化的过程。研究还发现,Cg-Fem-1b和Cg-Fem-1c两基因虽然同源性较低,但是它们的表达模式相像。究竟Cg-Fem-1b和Cg-Fem-1c在性腺发育中起到怎样的作用以及Cg-Fem-1b和Cg-Fem-1c之间的相互作用关系如何还有待进一步研究。
| [1] |
Doniach T, Hodgkin J. A sex-determining gene, fem-1, required for both male and hermaphrodite development in Caenorhabditis elegans[J]. Developmental Biology, 1984, 106(1): 223-235. DOI:10.1016/0012-1606(84)90077-0
(  0) 0) |
| [2] |
Hodgkin J. Sex determination and dosage compensation in Caenorhabditis elegans[J]. Annual Review of Genetics, 1987, 21(1): 133-154. DOI:10.1146/annurev.ge.21.120187.001025
(  0) 0) |
| [3] |
Kimble J, Edgar L, Hirsh D. Specification of male development in Caenorhabditis elegans: The fem genes[J]. Developmental Biology, 1984, 105(1): 234-239. DOI:10.1016/0012-1606(84)90279-3
(  0) 0) |
| [4] |
Spence A M, Coulson A, Hodgkin J. The product of fem-1, a nematode sex-determining gene, contains a motif found in cell cycle control proteins and receptors for cell-cell interactions[J]. Cell, 1990, 60(6): 981-990. DOI:10.1016/0092-8674(90)90346-G
(  0) 0) |
| [5] |
Zarkower D, Hodgkin J. Molecular analysis of the C. elegans sex-determining gene tra-1: A gene encoding two zinc finger proteins[J]. Cell, 1992, 70(2): 237-249. DOI:10.1016/0092-8674(92)90099-X
(  0) 0) |
| [6] |
Kuwabara P E. A complex solution to a sexual dilemma[J]. Developmental Cell, 2007, 13(1): 6-8. DOI:10.1016/j.devcel.2007.06.004
(  0) 0) |
| [7] |
Barnes T M, Hodgkin J. The tra-3 sex determination gene of Caenorhabditis elegans encodes a member of the calpain regulatory protease family[J]. The EMBO Journal, 1996, 15(17): 4477.
(  0) 0) |
| [8] |
Krakow D, Sebald E, King L M, et al. Identification of human FEM1A, the ortholog of a C. elegans sex-differentiation gene[J]. Gene, 2001, 279(2): 213-219. DOI:10.1016/S0378-1119(01)00756-9
(  0) 0) |
| [9] |
Ventura-Holman T, Maher J F. Sequence, organization, and expression of the human FEM1B gene[J]. Biochemical and Biophysical Research Communications, 2000, 267(1): 317-320. DOI:10.1006/bbrc.1999.1942
(  0) 0) |
| [10] |
Ventura-Holman T, Seldin M F, Li W, et al. The MurineFem1Gene Family: Homologs of the Caenorhabditis elegans Sex-Determination Protein FEM-1[J]. Genomics, 1998, 54(2): 221-230. DOI:10.1006/geno.1998.5569
(  0) 0) |
| [11] |
Ventura-Holman T, Lu D, Si X, et al. The Fem1c genes: conserved members of the Fem1 gene family in vertebrates[J]. Gene, 2003, 314: 133-139. DOI:10.1016/S0378-1119(03)00712-1
(  0) 0) |
| [12] |
Chan S L, Yee K S Y, Tan K M L, et al. The Caenorhabditis elegans sex determination protein FEM-1 is a CED-3 substrate that associates with CED-4 and mediates apoptosis in mammalian cells[J]. Journal of Biological Chemistry, 2000, 275(24): 17925-17928. DOI:10.1074/jbc.C000146200
(  0) 0) |
| [13] |
Zhang N, Xu F, Guo X. Genomic analysis of the Pacific oyster (Crassostrea gigas) reveals possible conservation of vertebrate sex determination in a mollusc[J]. G3 (Bethesda, Md.), 2014, 4(11): 2207. DOI:10.1534/g3.114.013904
(  0) 0) |
| [14] |
Guo X M, Hedgecock D, Hershberger W K, et al. Genetic determinants of protandric sex in the Pacific oyster, Crassostrea gigas Thunberg[J]. Evolution, 1998, 52(2): 394-402. DOI:10.1111/evo.1998.52.issue-2
(  0) 0) |
| [15] |
Hedrick P W, Hedgecock D. Sex determination: genetic models for oysters[J]. Journal of Heredity, 2010, 101(5): 602-611. DOI:10.1093/jhered/esq065
(  0) 0) |
| [16] |
Du Y, Zhang L, Xu F, et al. Validation of housekeeping genes as internal controls for studying gene expression during Pacific oyster (Crassostrea gigas) development by quantitative real-time PCR[J]. Fish & Shellfish Immunology, 2013, 34(3): 939-945.
(  0) 0) |
| [17] |
熊文芳. 池蝶蚌性别决定相关基因feminization-1基因的分子特征及表达分析[D]. 南昌: 南昌大学, 2014. Xiong W F. Molecular characteristics and expression analysis of feminization-1 gene relatedto sex-detemination from fresh water pearl mussel, Hyriopsis schlegelii[D]. Nanchang: Nanchang University, 2014. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D554390 (  0) 0) |
| [18] |
时红, 郝友进, 陈斌, 等. 东亚飞蝗fem-1基因的克隆与表达分析[J]. 昆虫学报, 2013, 56(7): 729-737. Shi H, Hao Y J, Chen B, et al. Cloning and expression analysis of fem-1 genes from the oriental migratory locust, Locusta migratoria manilensis (Orthoptera: Locustidae)[J]. Acta Entomologica Sinica, 2013, 56(7): 729-737. (  0) 0) |
| [19] |
侍建涛, 李志, 桂建芳, 等. 斑马鱼fem-1c cDNA克隆与表达分析[J]. 水生生物学报, 2015, 39(3): 459-467. Shi J T, Li Z, Gui J F, et al. The cloning and expression analysis of zebrafish fem-1c, a member of fem-1 family[J]. Acta Hydrobiologica Sinica, 2015, 39(3): 459-467. DOI:10.7541/2015.61 (  0) 0) |
| [20] |
Song C, Cui Z, Hui M, et al. Molecular characterization and expression profile of three Fem-1 genes in Eriocheir sinensis provide a new insight into crab sex-determining mechanism[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 189: 6-14. DOI:10.1016/j.cbpb.2015.07.003
(  0) 0) |
| [21] |
Kang L, Chen X Y, Zhou Y, et al. The analysis of large-scale gene expression correlated to the phase changes of the Migratory locust[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(51): 17611-17615. DOI:10.1073/pnas.0407753101
(  0) 0) |
| [22] |
Ma Z, Yu J, Kang L. LocustDB: A relational database for the transcriptome and biology of the migratory locust (Locusta migratoria)[J]. BMC Genomics, 2006, 7(1): 11. DOI:10.1186/1471-2164-7-11
(  0) 0) |
| [23] |
Jiang H, Yin Y, Zhang X, et al. Chasing relationships between nutrition and reproduction: A comparative transcriptome analysis of hepatopancreas and testis from Eriocheir sinensis[J]. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2009, 4(3): 227-234. DOI:10.1016/j.cbd.2009.05.001
(  0) 0) |
| [24] |
Wang W, Wu X, Liu Z, et al. Insights into hepatopancreatic functions for nutrition metabolism and ovarian development in the crab Portunus trituberculatus: Gene discovery in the comparative transcriptome of different hepatopancreas stages[J]. PloS One, 2014, 9(1): 84921. DOI:10.1371/journal.pone.0084921
(  0) 0) |
| [25] |
Farlora R, Araya-Garay J, Gallardo-Escárate C. Discovery of sex-related genes through high-throughput transcriptome sequencing from the salmon louse Caligus rogercresseyi[J]. Marine Genomics, 2014, 15: 85-93. DOI:10.1016/j.margen.2014.02.005
(  0) 0) |
| [26] |
Sreenivasan R, Cai M, Bartfai R, et al. Transcriptomic analyses reveal novel genes with sexually dimorphic expression in the zebrafish gonad and brain[J]. PloS One, 2008, 3(3): 1791. DOI:10.1371/journal.pone.0001791
(  0) 0) |
| [27] |
Itoh Y, Kampf K, Arnold A P. Disruption of FEM1C-W gene in zebra finch: Evolutionary insights on avian ZW genes[J]. Chromosoma, 2009, 118(3): 323-334. DOI:10.1007/s00412-008-0199-8
(  0) 0) |
| [28] |
Robinson N A, Gopikrishna G, Baranski M, et al. QTL for white spot syndrome virus resistance and the sex-determining locus in the Indian black tiger shrimp (Penaeus monodon)[J]. BMC Genomics, 2014, 15(1): 731. DOI:10.1186/1471-2164-15-731
(  0) 0) |
| [29] |
Schlamp C L, Thliveris A T, Li Y, et al. Insertion of the βGeo promoter trap into the Fem1c gene of ROSA3 mice[J]. Molecular and Cellular Biology, 2004, 24(9): 3794-3803. DOI:10.1128/MCB.24.9.3794-3803.2004
(  0) 0) |
 2018, Vol. 48
2018, Vol. 48


