2. 中国海洋大学海洋生命学院,海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003;
3. 广西精工海洋科技有限公司,广西 北海 536000
海豚链球菌(Streptococcus iniae)是一种主要的鱼类病原体,最初分离自亚马逊海豚“高尔夫球”样的皮下脓肿病灶,目前已危害我国的鲟鱼(Acipenser sturio)、斑点叉尾鮰(Ictalurus punctatus)、卵形鲳鲹(Trachinotus ovatus)、银鼓鱼(Selenosis multifasciata)、罗非鱼(Oreochromis niloticus)等多种养殖鱼类[1-4]。海豚链球菌感染一般发生于7~9月高温时期,有研究表明当水温在25~28 ℃时,海豚链球菌造成的死亡率达到峰值[5],养殖环境污染、网箱养殖密度过大、饲料残余量大以及鱼体自身免疫力下降等都极易引发该病的爆发[6-7]。此外,海豚链球菌具有人畜共患的风险,人类感染病例通常与处理受感染鱼类有关,海豚链球菌已危及世界各地的水产养殖和食品安全[8]。研究者们运用分子生物学技术对海豚链球菌进行了深入的研究,目前已鉴定到10余种与致病性密切相关的毒力因子:M样蛋白和C5a肽酶有助于细胞黏附[9],葡萄糖磷酸变位酶与糖代谢有关[10],溶血素S用于合成SLS结构肽和SLS修饰蛋白[11],CAMP因子阻断补体级联的结合和激活[12],α-烯醇化酶与纤溶酶原形成复合体,协助微生物在宿主体内播散[13]。PCR技术在鉴定海豚链球菌中得到了良好的应用,Berridge B R等[14]建立的PCR方法可特异性扩增海豚链球菌的16S—23S rRNA转录间隔区片段,Goh S H等[15]使用Cpn 60基因特异性PCR引物确定分离株为海豚链球菌,但PCR技术需通过凝胶电泳或基因测序对结果进行检测和验证,同时无法对组织样本中的海豚链球菌进行定量分析。
卵形鲳鲹(Trachinotus ovatus)俗称金鲳,在中国主要养殖区域为海南、广东、广西和福建,养殖模式主要以深水网箱养殖和池塘养殖为主,具有生长快、肉质鲜美等优点。由于其宝贵的营养和经济价值,卵形鲳鲹被列为海南省“三鱼一螺一虾”热带苗种发展产业之一。然而病害问题一直困扰着该物种产量的增长,有许多关于卵形鲳鲹病害的研究报道,例如:由诺卡氏菌(Nocardia seriolae)、发光杆菌属(Photobacterium)、弧菌属(Vibrio)、链球菌属(Streptococcus)等引起的细菌性疾病,由车轮虫(Trichodina)、刺激隐核虫(Cryptocaryon irritans)、淀粉卵涡鞭虫(Amyloodinium ocellatum)等引起的寄生虫病,病毒性神经坏死病和肝胆综合征等[16]。其中,感染海豚链球菌的卵形鲳鲹常出现脊椎弯曲、体表发黑、尾鳍出血等症状,并伴有大规模死亡[17];该菌能够进入卵形鲳鲹的中枢神经系统造成神经性损伤,而药物难以通过血脑屏障杀灭组织中的病原体,从而大大降低了治疗效果[18]。当前尚不清楚海豚链球菌在卵形鲳鲹各组织的分布情况和动态变化,因此,建立一种能够快速确定病原体类型和菌体载量的海豚链球菌绝对定量方法,对在引起卵形鲳鲹大规模感染前检测出海豚链球菌并采取相应的防治措施具有重要意义。
本研究以卵形鲳鲹源的海豚链球菌特异性SimA基因作为目的基因,建立准确、灵敏的实时荧光绝对定量方法,检测海豚链球菌的拷贝数,并基于该方法研究海豚链球菌浸泡感染卵形鲳鲹后不同时间点鱼体各组织的菌体载量和分布变化,为有效防控海豚链球菌提供技术和理论依据。
1 材料与方法 1.1 实验菌株海豚链球菌TO-2021GX由本实验室从患病卵形鲳鲹中分离鉴定并保存。坎氏弧菌(Vibrio campbellii)、溶藻弧菌(Vibrio alginolyticus)、哈维氏弧菌(Vibrio harveyi)、副溶血弧菌(Vibrio parahaemolyticus)、轮虫弧菌(Vibrio rotiferianus)、表皮葡萄球菌(Vibrio rotiferianus)、金黄色葡萄球菌(Staphylococcus aureus)、肺炎克雷伯菌(Klebsiella pneumoniae)、格式乳球菌(Lactococcus garvieae)、停乳链球菌(Streptococcus dysgalactiae)由中国海洋大学三亚海洋研究院莫照兰教授课题组馈赠。
1.2 实验动物及取样本实验所用的卵形鲳鲹体长为(14.0±0.9) cm,体质量为(45.1±7.0)g,由广西精工海洋科技有限公司负责养殖,鱼体在养殖池内饲养2周后,将鱼体表面划伤后转移到300 L的养殖桶内,浸泡于海豚链球菌浓度为1×109 CFU/mL的海水中,海水温度为25 ℃,3 h后更换为无菌海水。在浸泡感染后12、24、48和72 h各随机选取3尾鱼,使用麻醉剂MS-222麻醉后进行解剖,解剖后采集心、肝、脾、肾、脑、鳃、肠样品,并将样品迅速投入到液氮中保存。
1.3 引物设计根据NCBI中公布的海豚链球菌SimA序列(GenBank: JF330100.1),使用Primer Premier 6软件设计荧光定量引物,通过NCBI中blast工具验证引物的特异性,引物设计为:SimA-F:5 ’-GCACGAGAATTAGATACGCTTCA-3’、SimA-R:5’-CAAGTTCCGCAATCTTAGCATCT-3’,用于扩增SimA序列766~871位点之间105 bp的片段,引物交由生工生物工程(广州)有限公司合成。
1.4 制备质粒标准品按照细菌DNA提取试剂盒的说明提取海豚链球菌的基因组DNA,以DNA为模板,采用引物SimA-F、SimA-R扩增SimA基因片段,PCR反应体系为95 ℃、5 min;95 ℃、30 s,55 ℃、30 s,72 ℃、1 min,共30个循环;72 ℃、10 min。PCR产物纯化后连接到Blunt-zero载体,转化到大肠杆菌Trans T1中,使用通用引物M13(M13-F:5 ’-GTAAAACGACGGCCAGT-3 ’、M13-R:5’-CAGGAAACAGCTATGAC-3’)鉴定并扩大培养阳性克隆Blunt-SimA。用Not Ⅰ进行Blunt-SimA质粒单酶切,根据酶切产物胶回收产物浓度(C0)和质粒相对分子量(M)计算质粒的拷贝数,计算公式为C(拷贝数)=(C0×6.03×1023)/M。
1.5 绝对定量方法建立及标准曲线绘制将质粒标准品以10倍比例梯度连续稀释用作标准曲线的模板,按照ChamQ Universal SYBR qPCR Master Mix 10 μL,模板2 μL,正反引物(10 mmol/L)各0.4 μL,双蒸水7.2 μL配制共20 μL的荧光定量PCR反应体系。反应程序为:95 ℃、5 min;95 ℃、10 s,60 ℃、30 s,进行30个循环;60~90 ℃温度梯度生成熔断曲线,每组设置三个重复和阴性对照。根据熔断曲线检验引物特异性和产物纯度,以实时荧光定量数据的Ct值(Y)和各浓度拷贝数的对数值(X)绘制标准曲线。计算三次重复实验各组Ct值的变异系数来确定此绝对定量方法的重复性。
1.6 绝对定量方法特异性检测以海豚链球菌和坎氏弧菌、溶藻弧菌、哈维氏弧菌、副溶血弧菌、轮虫弧菌、表皮葡萄球菌、金黄色葡萄球菌、肺炎克雷伯菌、格式乳球菌、停乳链球菌的基因组DNA作为模板,按照绝对定量的反应体系和程序进行荧光定量PCR,每组设置三个重复和阴性对照,检验此方法是否特异性检测海豚链球菌。
1.7 海豚链球菌感染卵形鲳鲹后组织菌体载量检测用平衡酚-氯仿方法分别提取海豚链球菌浸泡感染卵形鲳鲹后心、肝、脾、肾、脑、鳃、肠组织的DNA,使用蛋白酶K和RNA酶(RNaseA)去除DNA中残留的蛋白质和RNA,使用紫外分光光度计测量DNA浓度,通过1%琼脂糖凝胶电泳检测DNA质量,将提取的DNA浓度调整至100 ng/μL。按照已建立的绝对定量方法检测各组织中的菌体载量,分析海豚链球菌的动态分布,每组设置三个重复和阴性对照。
1.8 数据处理使用SPSS 20软件对实验数据进行统计学分析,采用单因素方差分析来分析统计学差异,p<0.05认为差异显著并具有统计学意义,使用GraphPad Prism 9软件作图。
2 实验结果 2.1 质粒标准品的构建以海豚链球菌基因组为模板进行SimA基因片段的PCR扩增,产物纯化后连接到Blunt-zero载体中并验证阳性克隆,结果如图 1 A所示,经M13引物PCR扩增的条带大小在250~500 bp之间,与预期大小一致,测序结果的Blast比对显示质粒序列与海豚链球菌SimA基因序列同源性达100%,表明Blunt-SimA质粒构建成功。使用Not Ⅰ对Blunt-SimA进行单酶切,呈现单一条带(见图 1 B),产物回收后,根据拷贝数计算公式,得到1.93×109拷贝数/μL的线性化质粒,其可作为绘制标准曲线的质粒标准品。
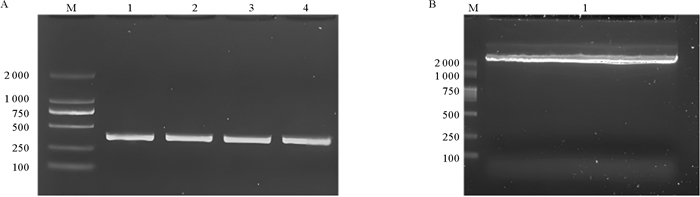
|
( A:阳性克隆质粒鉴定,1—4:M13引物PCR产物检测;B:阳性质粒Blunt-SimA线性化,1:质粒Blunt-SimA单酶切产物检测。M:DL2000 DNA标记。A: Identification of positive cloning plasmid, 1—4: Identification of M13 primer PCR product; B: Linearization of positive plasmid Blunt-SimA, 1: Single enzyme digestion product detection of plasmid Blunt-SimA. M: DL2000 DNA marker. ) 图 1 质粒Blunt-SimA PCR鉴定及线性化 Fig. 1 PCR identification and linearization of plasmid Blunt-SimA |
将质粒标准品依次稀释成1.93×102~1.93×109拷贝数/μL共8个拷贝数梯度,作为荧光定量PCR的模板进行扩增,结果如图 2 A所示,各质粒拷贝数对数值与循环数(Ct值)之间成反比例关系,如图 2 B所示,回归方程为Y=-3.289X+39.921,线性关系R2=0.999,扩增效率E=101.4%,这表示标准曲线中各拷贝数梯度与Ct值的相关性较好,扩增效率高,该方法可以用于海豚链球菌TO-2021GX的绝对定量检测。该绝对定量方法最低检测1.93×102拷贝数/μL的SimA基因。
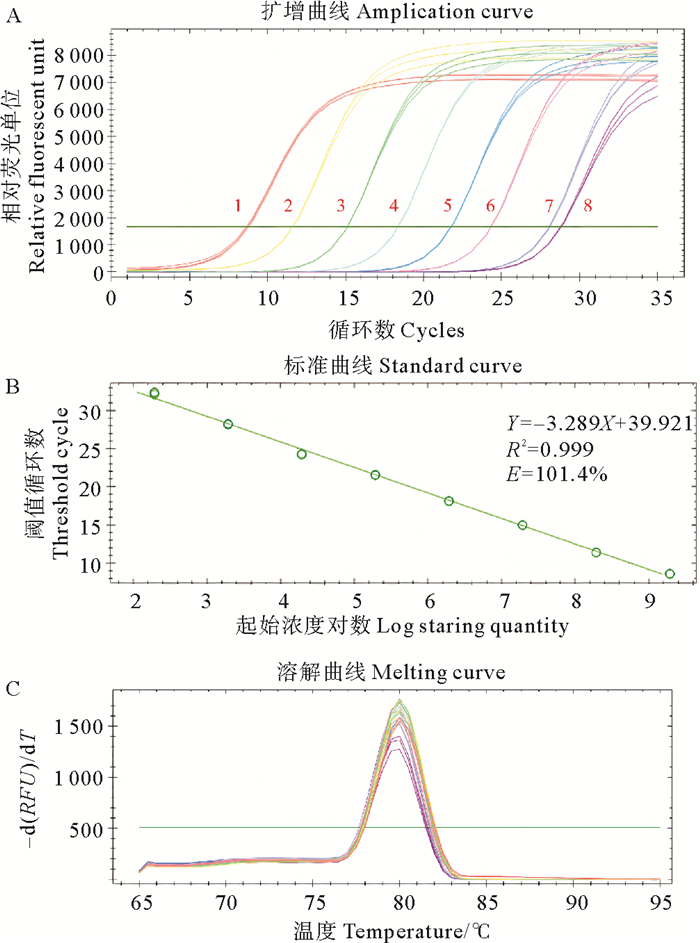
|
( A:荧光定量PCR扩增曲线,红色数字代表不同质粒浓度:1:1.93×109拷贝数/μL;2:1.93×108拷贝数/μL;3:1.93×107拷贝数/μL;4:1.93×106拷贝数/μL;5:1.93×105拷贝数/μL;6:1.93×104拷贝数/μL;7:1.93×103拷贝数/μL;8:1.93×102拷贝数/μL。B:荧光定量PCR标准曲线。C:荧光定量PCR熔断曲线, -d(RFU)/dT表示相对荧光单位随温度变化的变化率。A: Amplification curve of fluorescence quantitative PCR, the red number represents different plasmid concentrations: 1.93×109 copies/μL; 2:1.93×108 copies/μL; 3:1.93×107 copies/μL; 4:1.93×106 copies/μL; 5:1.93×105 copies/μL; 6:1.93×104 copies/μL; 7:1.93×103 copies/μL; 8:1.93×102 copies/μL. B: Standard curve of fluorescence quantitative PCR. C: Melting curve of fluorescent quantitative PCR, -d(RFU)/dT indicates the rate of change of the relative fluorescence unit as a function of temperature. ) 图 2 质粒Blunt-SimA荧光定量PCR标准曲线的建立 Fig. 2 Establishment of plasmid Blunt-SimA fluorescence quantitative PCR standard curve |
根据荧光定量PCR产物的熔断曲线(见图 2 C)确定扩增产物Tm值为80 ℃,曲线仅有单一峰,未出现其他杂峰,说明没有引物二聚体产生,引物SimA-F、SimA-R符合荧光定量PCR的引物要求,且能够特异性地扩增目的片段。
根据建立的绝对定量方法对1.93×102~1.93×109拷贝数/μL的稀释质粒进行三次重复实验,结果表明,重复实验的标准差为0.05~0.19,变异系数为0.13%~0.84%,变异系数值小于1%,具体如表 1所示,说明该绝对定量方法重复性较好,实验数据稳定。
|
|
表 1 质粒Blunt-SimA荧光定量PCR重复性检验 Table 1 Repeatability test of plasmid Blunt-SimA fluorescence quantitative PCR |
以海豚链球菌和坎氏弧菌、溶藻弧菌、哈维氏弧菌、副溶血弧菌、轮虫弧菌、表皮葡萄球菌、金黄色葡萄球菌、肺炎克雷伯菌、格式乳球菌、停乳链球菌的基因组DNA为模板,按照已建立的绝对定量方法进行荧光定量PCR,图 3的扩增曲线显示仅海豚链球菌DNA发生扩增,扩增曲线到达了平台期,其他10种菌株DNA未被扩增,说明该方法能够特异性地检测海豚链球菌。
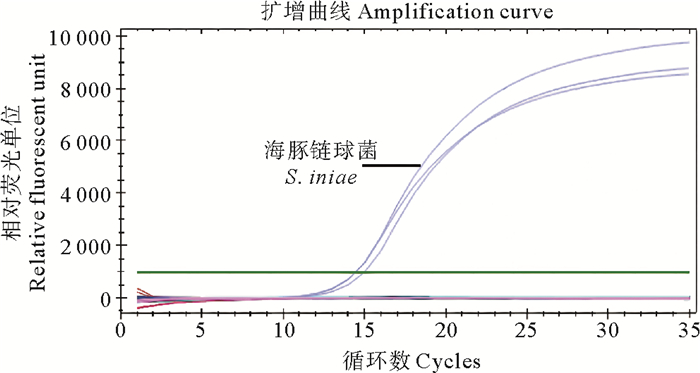
|
图 3 海豚链球菌绝对定量检测方法的特异性验证 Fig. 3 Specificity verification of absolute quantitative method for S. iniae |
以感染海豚链球菌不同时间点的卵形鲳鲹各组织DNA为模板进行绝对定量PCR检测,计算每微克DNA样品中含有的菌体拷贝数,以此判断海豚链球菌对组织嗜性的强弱以及动态变化。
结果如图 4所示:感染后12 h(见图 4 A),脑中的菌体载量显著高于其他组织(p<0.01),每微克DNA中菌体拷贝数为(7.99±0.54)×103,心和肠的菌体载量最少,菌体载量达到102水平,脾、肝、肾、鳃的菌体载量达到较高水平。感染24 h后(见图 4 B),脑中的菌体载量仍显著高于其他组织(p<0.05),肠的菌体载量显著低于其他组织(p<0.05),脾和心的菌体载量的涨幅最大,每微克DNA中菌体拷贝数分别为(2.77±0.84)× 103、(1.11±0.20)×104,是12 h菌体载量的3.43和3.05倍,脑和脾的菌体载量达到104水平,而心、肝、肾、鳃、肠的菌体载量达到103水平。感染48 h后(见图 4 C),各组织的菌体载量均有所增加,肾中菌体载量最高,达到(1.67±0.22)×104拷贝数,菌体载量是24 h的20.32倍,显著高于脾、心、肝、鳃、肠的菌体载量(p<0.05),其次肠的菌体载量是24 h时的15.55倍,每微克DNA中菌体拷贝数达到(1.80±0.10)×104,脾、肝、肠、肾、脑的菌体载量达到104水平,而心、鳃的菌体载量达到103水平。感染72 h后(见图 4 D),肾为菌体载量最高的组织,脑和肾的菌体载量显著高于鳃(p<0.05),心的菌体载量是48 h的3.30倍,各组织的菌体载量均达到104水平,鳃中每毫克DNA的菌体载量为(1.68±0.17)×104拷贝,是菌体载量最低的组织。
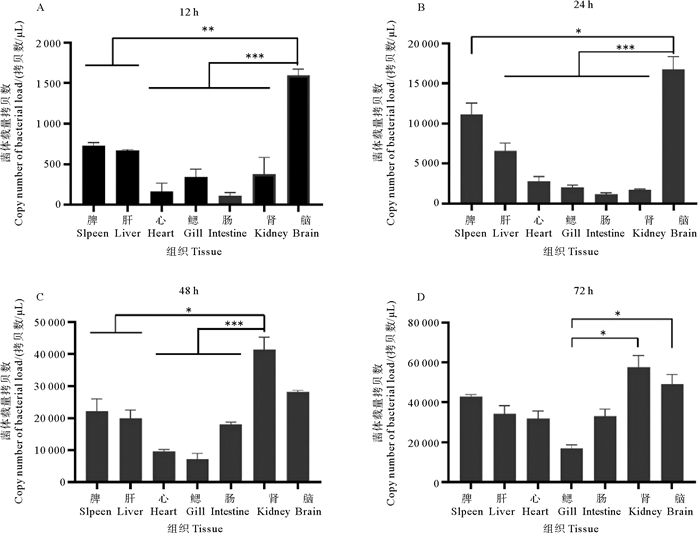
|
( A:感染后12 h菌体载量;B:感染后24 h菌体载量;C:感染后48 h菌体载量;D:感染后72 h菌体载量;*、**、***表示差异显著,*:p<0.05,**:p<0.01,***:p<0.001。A: Bacterial load for 12 h after infected; B: Bacterial load for 24 h after infected; C: Bacterial load for 48 h after infected; D: Bacterial load for 72 h after infected; *、**、*** indicates significant difference; *: p < 0.05, **: p < 0.01, ***: p < 0.001. ) 图 4 海豚链球菌浸泡感染卵形鲳鲹后各组织菌体载量拷贝数 Fig. 4 Copy number of bacterial load in each tissue of T. ovatus after immersion infected by S. iniae |
在检测的组织中,菌体载量随着感染时间的推移而呈现上升趋势。脑为感染后12、24 h菌体载量最高的组织,肾为感染后48、72 h菌体载量最高的组织。肠为感染后12、24 h菌体载量最低的组织,鳃的菌体载量在感染后12 h高于心和肠,鳃是感染后48、72 h菌体载量最低的组织。在感染后12~72 h期间,其他组织中菌体载量拷贝数的从高到低依次为脾、肝、心。综上,海豚链球菌对脑和肾的嗜性最强,对脾、肝、心、肠和鳃的嗜性相对较弱。
3 讨论在全球范围内,海豚链球菌严重影响养殖鱼类的健康,经常导致高发病率和高死亡率,并对人类健康构成潜在风险。因此,快速准确地检测海豚链球菌是实施防控措施的关键一步。定量PCR具有高灵敏度、高特异性和高重现性等优点,是目前核酸检测和定量分析各种微生物病原体的首选方法[19]。为了使用荧光定量PCR测定样本中微生物病原体的绝对数量,需要根据含有已知拷贝数或浓度的质粒、基因组DNA或其他携带靶基因的核酸分子的系列稀释定量标准品建立标准曲线[20]。研究证明环状质粒的分子结构与线性质粒存在差异,这可能影响到目的基因的扩增效率,李丽等[21]验证使用内切酶单酶切质粒后,线状质粒定量PCR的Ct值显著降低,达到检测阈值时间更短。目前检测海豚链球菌的方法大多是基于16S rRNA基因[22]、16S—23S rDNA基因转录间隔区[14]、伴侣蛋白60基因(Cpn60)[15]的常规PCR技术。由于海豚链球菌与无乳链球菌、副乳房链球菌之间的遗传相似性较高,基于这些基因设计的引物无法根据PCR扩增条带进行特异性鉴定,通常需要与基因测序技术结合使用[23]。由emm基因编码的M蛋白是一种A族链球菌的主要毒力因子,Baiano J C等[24]根据多种链球菌M蛋白编码序列,确定了海豚链球菌中emm样基因SimA和SimB编码与纤维蛋白原结合的M样蛋白。本文作者建立的荧光定量PCR技术,使用绝对定量方法计算海豚链球菌的拷贝数,根据海豚链球菌特异性基因SimA的保守序列片段设计引物,以线性化的含有SimA基因片段重组质粒作为标准品系列稀释后绘制标准曲线,一个拷贝的基因对应到一个细菌。所得回归方程为Y=-3.289X+39.921,Ct值与拷贝数lg值的线性关系较好,扩增效率为101.4%介于90%~110%之间,检测最低限值为1.93×102拷贝数/μL,在1.93×102~1.93×109拷贝数/μL之间有良好的区分。此绝对定量技术三次重复实验的标准差和变异系数均小于1,可以特异性扩增海豚链球菌的SimA基因,且与坎氏弧菌等10种常见病原菌不发生交叉反应,该方法重复性与特异性较好,能够作为一种快速检测海豚链球菌拷贝数的定量方法。
海豚链球菌的致病机制尚不明确,前期研究认为海豚链球菌的感染是一种多级进程,感染起始于海豚链球菌在鱼体外和体内的定植和增殖,随后侵入内部组织和血液,导致全身性菌血症,并由细菌毒素引起败血症[18]。海豚链球菌通过血液循环系统或者侵入单核细胞和巨噬细胞后进入中枢神经系统(CNS)引起脑膜炎,CNS感染率与细菌浓度和菌血症的持续时间相关[25]。Evans J J等[26]使用菌落平板计数法检测海豚链球菌鼻孔注射感染鲈鱼后的组织分布,海豚链球菌在鳃、心、肾、小脑、大脑中的含量较高,这证明海豚链球菌可以穿过血脑屏障传递给其他组织。本文作者发现使用海豚链球菌浸泡感染卵形鲳鲹后在不同组织中菌体载量随着感染进程而持续增长,同时不同组织中的菌体载量存在较大差异,该菌能够在鱼体内定值和繁殖,且对各组织的嗜性存在差异。海豚链球菌TO-2021GX对卵形鲳鲹脑和肾的嗜性最强,对脾、肝、肠、心和鳃组织的嗜性相对较弱。
结合海豚链球菌在卵形鲳鲹中的组织分布和动态变化,海豚链球菌对脑具有较高的嗜性,并在感染早期定植于卵形鲳鲹脑中,这可能与脑炎、脑膜炎及食欲不振等典型症状相关。鳃中的菌体载量在感染后12 h高于心和肠,在感染后期成为菌体载量最低的组织,这可能与采用浸泡感染的方式有关,环境中高浓度的海豚链球菌大量附着到鳃上。硬骨鱼的先天免疫反应主要发生在淋巴器官,如肾、肝和脾,以及各种黏膜相关部位[27],肾是高等脊椎动物应激反应的中枢器官,能够进行体液免疫和细胞免疫应答[28];脾是鱼类中与免疫应答相关的淋巴造血器官,含有多种免疫细胞,在应对病原菌感染时产生趋化因子和细胞因子[29];肝不仅参与宿主防御机制,还与应对病原感染的代谢调节密切相关。肾中的菌体载量在48 h大幅增长,成为菌体载量最高的组织,此时脾和肝的菌体载量与肾的水平相当,推测肾、脾和肝在感染早期通过调节免疫应答抵御病原菌的感染,而海豚链球菌逐步产生效应因子以适应宿主环境,凭借自身的毒力因子侵染细胞,实现免疫逃逸后在鱼体各组织内大量繁殖,致使鱼体表现出临床症状或死亡。
本文建立了针对海豚链球菌SimA基因的实时荧光绝对定量PCR检测技术,以绝对定量的方法确定海豚链球菌的拷贝数,该方法灵敏度高、重复性好,可以有效地检测卵形鲳鲹组织中海豚链球菌的载量和分布变化,为后续开发海豚链球菌防控技术和研究海豚链球菌感染卵形鲳鲹的分子机制奠定基础。
| [1] |
Miao C, Zhang H J, Li J J, et al. Differential PCR detection of Streptococcus agalactiae and Streptococcus iniae in tilapia (Oreochromis niloticus) by a single primer pair[J]. Journal of Fish Diseases, 2019, 42(8): 1211-1216. DOI:10.1111/jfd.13014 (  0) 0) |
| [2] |
Song G, Mo Z Q, Wang Z, et al. Isolation and pathogenicity of Streptococcus iniae in offshore cagecultured Trachinotus ovatus in China[J]. Aquaculture, 2018, 492: 247-252. DOI:10.1016/j.aquaculture.2018.04.015 (  0) 0) |
| [3] |
Chen D F, Wang K Y, Geng Y, et al. Pathological changes in cultured channel catfish Ictalurus punctatus spontaneously infected with Streptococcus iniae[J]. Diseases of Aquatic Organisms, 2011, 95(3): 203-208. DOI:10.3354/dao02354 (  0) 0) |
| [4] |
杨林狄, 贾新蕾, 黄增朝, 等. 银鼓鱼海豚链球菌的分离、鉴定及毒力基因检测[J]. 大连海洋大学学报, 2022, 38(2): 233-241. Yang L D, Jia X L, Huang Z C, et al. Isolation, identification and virulence gene detection of Streptococcus iniae in Selenotoca multifasciata[J]. Journal of Dalian Ocean University, 2022, 38(2): 233-241. (  0) 0) |
| [5] |
Bromage E, Leigh O. Environmental factors affecting the susceptibility of barramundi to Streptococcus iniae[J]. Aquaculture, 2009, 290(3-4): 224-228. DOI:10.1016/j.aquaculture.2009.02.038 (  0) 0) |
| [6] |
Mugetti D, Silvia C, Paolo P, et al. Episode of mortality associated with isolation of Streptococcus iniae in Adriatic sturgeon (Acipenser naccarii Bonaparte, 1836) reared in Northern Italy[J]. Journal of Fish Diseases, 2022, 45(6): 939-942. DOI:10.1111/jfd.13608 (  0) 0) |
| [7] |
Colussi S, Pastorino P, Mugetti D, et al. Isolation and genetic characterization of Streptococcus iniae virulence factors in Adriatic sturgeon (Acipenser naccarii)[J]. Microorganisms, 2022, 10(5): 883. DOI:10.3390/microorganisms10050883 (  0) 0) |
| [8] |
Lau S K P, Woo P C Y, Luk W K, et al. Clinical isolates of Streptococcus iniae from Asia are more mucoid and β-hemolytic than those from North America[J]. Diagnostic Microbiology and Infectious Disease, 2006, 54(3): 177-181. DOI:10.1016/j.diagmicrobio.2005.09.012 (  0) 0) |
| [9] |
Locke J B, Aziz R K, Vicknair M R, et al. Streptococcus iniae M-like protein contributes to virulence in fish and is a target for live attenuated vaccine development[J]. PLoS One, 2008, 3(7): e2824. DOI:10.1371/journal.pone.0002824 (  0) 0) |
| [10] |
Buchanan J T, Stannard J A, Lauth X, et al. Streptococcus iniae phosphoglucomutase is a virulence factor and a target for vaccine development[J]. Infection and Immunity, 2005, 73(10): 6935-6944. DOI:10.1128/IAI.73.10.6935-6944.2005 (  0) 0) |
| [11] |
Dale J B, Chiang E Y, Hasty D L, et al. Antibodies against a synthetic peptide of SagA neutralize the cytolytic activity of streptolysin S from group A streptococci[J]. Infection and Immunity, 2002, 70(4): 2166-2170. DOI:10.1128/IAI.70.4.2166-2170.2002 (  0) 0) |
| [12] |
Sheng X, Gao J, Liu H, et al. Recombinant phosphoglucomutase and CAMP factor as potential subunit vaccine antigens induced high protection against Streptococcus iniae infection in flounder (Paralichthys olivaceus)[J]. Journal of Applied Microbiology, 2018, 125(4): 997-1007. DOI:10.1111/jam.13948 (  0) 0) |
| [13] |
Wang J, Wang K Y, Chen D F, et al. Cloning and characterization of surface-localized α-enolase of Streptococcus iniae, an effective protective antigen in mice[J]. International Journal of Molecular Sciences, 2015, 16(12): 14490-14510. DOI:10.3390/ijms160714490 (  0) 0) |
| [14] |
Berridge B R, Fuller J D, De Azavedo J, et al. Development of specific nested oligonucleotide PCR primers for the Streptococcus iniae 16S—23S ribosomal DNA intergenic spacer[J]. Journal of Clinical Microbiology, 1998, 36(9): 2778-2781. DOI:10.1128/JCM.36.9.2778-2781.1998 (  0) 0) |
| [15] |
Goh S H, Driedger D, Gillett S, et al. Streptococcus iniae, a human and animal pathogen: Specific identification by the chaperonin 60 gene identification method[J]. Journal of Clinical Microbiology, 1998, 36(7): 2164-2166. DOI:10.1128/JCM.36.7.2164-2166.1998 (  0) 0) |
| [16] |
Harikrishnan R, Balasundaram C, Heo M S. Impact of plant products on innate and adaptive immune system of cultured finfish and shellfish[J]. Aquaculture, 2011, 317(1-4): 1-15. DOI:10.1016/j.aquaculture.2011.03.039 (  0) 0) |
| [17] |
丁泽昊, 赵子晨, 李桂华, 等. 1株卵形鲳鲹病原菌的分离、鉴定及其药敏试验[J]. 海南大学学报(自然科学版), 2021, 39(2): 153-159. Ding Z H, Zhao Z C, Li G H, et al. Isolation, identification and antibiotic sensitivity of a pathogenic bacterium from Trachinotus ovatus[J]. Natural Science Journal of Hainan University, 2021, 39(2): 153-159. (  0) 0) |
| [18] |
Zlotkin A, Chilmonczyk S, Eyngor M, et al. Trojan horse effect: Phagocyte-mediated Streptococcus iniae infection of fish[J]. Infection and Immunity, 2003, 71(5): 2318-2325. DOI:10.1128/IAI.71.5.2318-2325.2003 (  0) 0) |
| [19] |
Bustin S A, Benes V, Garson J A, et al. The MIQE guidelines: Minimum information for publication of quantitative real-time PCR experiments[J]. Clinical Chemistry, 2009, 55(4): 611-622. DOI:10.1373/clinchem.2008.112797 (  0) 0) |
| [20] |
Brankatschk R, Bodenhausen N, Zeyer J, et al. Simple absolute quantification method correcting for quantitative PCR efficiency variations for microbial community samples[J]. Applied and Environmental Microbiology, 2012, 78(12): 4481-4489. DOI:10.1128/AEM.07878-11 (  0) 0) |
| [21] |
李丽, 赵成萍, 李宏, 等. 质粒制备绝对定量PCR标准曲线方法的建立[J]. 农业生物技术学报, 2011, 19(6): 1157-1162. Li L, Zhao C P, Li H, et al. Establishment of the plasmid standard curve generation method for absolute quantification PCR[J]. Journal of Agricultural Biotechnology, 2011, 19(6): 1157-1162. (  0) 0) |
| [22] |
Bachrach G, Zlotkin A, Hurvitz A, et al. Recovery of Streptococcus iniae from diseased fish previously vaccinated with a streptococcus vaccine[J]. Applied and Environmental Microbiology, 2001, 67(8): 3756-3758. DOI:10.1128/AEM.67.8.3756-3758.2001 (  0) 0) |
| [23] |
Nguyen T L, Lim Y J, Kim D H, et al. Development of real-time PCR for detection and quantitation of Streptococcus parauberis[J]. Journal of Fish Diseases, 2016, 39(1): 31-39. DOI:10.1111/jfd.12322 (  0) 0) |
| [24] |
Baiano J C, Tumbol R A, Umapathy A, et al. Identification and molecular characterisation of a fibrinogen binding protein from Streptococcus iniae[J]. BMC Microbiol, 2008, 8: 1-16. DOI:10.1186/1471-2180-8-1 (  0) 0) |
| [25] |
Eyngor M, Chilmonczyk S, Zlotkin A, et al. Transcytosis of Streptococcus iniae through skin epithelial barriers: An in vitro study[J]. FEMS Microbiology Letters, 2007, 277(2): 238-248. DOI:10.1111/j.1574-6968.2007.00973.x (  0) 0) |
| [26] |
Evans J J, Shoemaker C A, Klesius P H. Distribution of Streptococcus iniae in hybrid striped bass (Morone chrysops×Morone saxatilis) following nare inoculation[J]. Aquaculture, 2001, 194(3): 233-243. (  0) 0) |
| [27] |
Gomez D, Sunyer J O, Salinas I. The mucosal immune system of fish: The evolution of tolerating commensals while fighting pathogens[J]. Fish & Shellfish Immunology, 2013, 35(6): 1729-1739. (  0) 0) |
| [28] |
Verleih M, Borchel A, Krasnov A, et al. Impact of thermal stress on kidney-specific gene expression in farmed regional and imported rainbow trout[J]. Marine Biotechnology, 2015, 17(5): 576-592. DOI:10.1007/s10126-015-9640-1 (  0) 0) |
| [29] |
Hua Y, Xiao S J, Wang X Q, et al. Characterization of spleen transcriptome of Schizothorax prenanti during Aeromonas hydrophila infection[J]. Marine Biotechnology, 2018, 20(2): 246-256. DOI:10.1007/s10126-018-9801-0 (  0) 0) |
2. Key Laboratory of Marine Genetics and Breeding, Ministry of Education, College of Marine Life Sciences, Ocean University of China, Qingdao 266003, China;
3. Guangxi Jinggong Marine Technology Limited Liability Company, Beihai 536000, China
 2024, Vol. 54
2024, Vol. 54


