2. Department of Microbiology, University of Veterinary and Animal Sciences, Lahore 54660, Pakistan
链状亚历山大藻(Alexandrium pacificum)作为重要的赤潮甲藻,在全球范围内分布广泛,对由其引发的赤潮的机理的研究受到了研究者的广泛关注。研究表明,氮、磷浓度的升高对该藻的生长有促进作用[1]。铁、锰等微量元素是触发塔玛亚历山大藻爆发性增殖的重要因子之一,同样在链状亚历山大藻中,锰浓度的提升(18~36 μg/L),使得其生长速率也随之上升。Laabir等的研究发现光照强度从10~90 μmol photons·m-2·s-1的提高,对链状亚历山大藻的生长有促进作用[2-3]。由于甲藻基因组巨大且复杂,目前的研究发现链状亚历山大藻基因组在60 G左右[4],因此在研究基因调控机制方面对转录组测序和研究就显得更为重要。
表达谱及转录组通过筛选不同条件下差异表达基因进行后续分析,所获得的信息往往是碎片化的,可能会忽略不同处理或不同组织间基因的表达存在的内在联系或相关性。WGCNA以比较转录组或表达谱数据为基础,是一种从高通量数据中挖掘模块信息的算法。与传统的转录组分析重点关注差异表达基因不同,WGCNA分析采用皮尔逊相关系数或其他统计学方法衡量基因间的表达关系,从而构建基因共表达模块(Module),其分析可以覆盖整个转录组,并将复杂的数据进行归纳整理,从而对传统转录组分析进行补充并获取新的发现。在高等植物中,通过转录组测序找到了羽衣甘蓝不同组织的芥子油苷的代谢的相关基因,随后对转录组测序得到的相关合成基因进行WGCNA分析发现,根以及衰老的叶子可能是芥子油合成发生的主要组织[5]。WGCNA理论最早在2005年提出,随后在理论的基础上完成了基于R语言完成相关软件包的开发[6]。在WGCNA的分析过程中,首先构建基因共表达模块(Module),基因模块中含有一组具有似表达模式的基因。模块中的枢纽基因(Hub gene)作为模块中与其他基因连接最紧密的基因往往决定了一个模块的特性,更容易发现与性状相关的生物学意义。
WGCNA被广泛应用在医学研究方面。在胶质母细胞瘤的研究中,通过对100多位患者的基因表达数据的WGCNA分析,发现了一个癌症相关的基因模块,而该模块的枢纽基因也成为了治疗胶质母细胞瘤的关键靶基因[7]。此外,在阿尔兹海默症、骨质疏松症等的研究中[8-9],也找到了与治病相关联的基因及治疗的靶点。在植物中,通过WGCNA分析在拟南芥中找到了参与中种子萌发的基因模块,并由此对种子萌发过程中基因调控机制进行了深入的研究[10]。在苹果中,利用WGCNA分析找到了与苹果酸味产生相关的基因模块。
本研究通过模拟赤潮爆发的条件对链状亚历山大藻进行表达谱测序,利用表达谱测序后得到的基因表达量数据,计算基因间的相关性,从而构建基因共表达模块,并将模块与链状亚历山大藻爆发性生长(EG)相关联,对显著性相关的模块内的枢纽基因进行筛选并进行功能富集分析,以探索链状亚历山大藻爆发性生长的分子机制。
1 链状亚历山大藻表达谱测序 1.1 藻种来源及培养链状亚历山大藻藻株来源于中国海洋大学海洋生物遗传与育种教育部重点实验室。具体培养条件如下:培养基:f/2培养基(不加硅);光照强度:30 μmol·m-2·s-1;温度:(20±1)℃;光暗周期:12 h︰12 h。
1.2 链状亚历山大藻的处理及藻细胞收集链状亚历山大藻于f/2培养基中培养至对数生长期,将其中生长活力较好的藻在黑暗条件下进行48 h同步化处理,使其处于同一生长时期。同步化完成后以2×106个/L的浓度将藻接种于高磷高锰(M)、高光(G)、低氮(N)、低磷(P)以及正常培养(C)这五个处理条件下进行培养(见表 1),每个处理条件设置3个平行。
|
|
表 1 表达谱所需样品的处理条件 Table 1 Samples used for DGE analysis |
随后在链状亚历山大藻生长对数期(第12天)用除菌方法收集藻细胞,方法如下:在20 ℃条件下,用经过无菌处理的10 μm孔径筛绢过滤藻液,每过滤100 mL藻液用300 mL相应的无菌培养基进行冲洗,对冲洗后的藻液用50 mL无菌培养基(含有0.05%Tween-80和0.01 mol/L EDTA)悬浮30 min,超声破碎1 min(50 w,超声时间5 s,间隔时间5 s),随后用溶菌酶(0.5 mg/mL)在20 ℃下处理10 min,加入0.25% SDS处理10 min,用相应的无菌培养基冲洗,去除试剂残留。随后除菌后的藻液经0.01%的吖啶橙染色2 min后,在荧光显微镜下进行观察[11],筛选其中的无菌样品进入后续实验。
收集无菌处理后的链状亚历山大藻,每个处理条件的1个平行即为1个样品,每个样品中约含5×106个藻细胞,共计15个样品保存在液氮中,用于后续RNA文库的构建。
1.3 总RNA的提取及文库构建 1.3.1 总RNA的提取及检测采用RNAiso Plus (Trizol) (TaKaRa)法进行对15个样品的总RNA进行提取,得到的RNA用DNase I(TaKaRa)进行DNA消化,并用1.5%的琼脂糖凝胶电泳150 V,10 min进行检测,用Nano Drop2000进行RNA浓度和纯度检测。
1.3.2 文库构建使用NEBNext Ultra RNA Library Prep Kit for Illumina(NEB)试剂盒按照说明书步骤进行文库的构建。
用Oligo(dT)磁珠对总RNA中的mRNA进行富集,用Fragmentation buffer将mRNA随机打断成短片段,利用六碱基随机引物合成第一条cDNA链,随后加入dNTPs、DNA polymerase I、RNaseH合成第二条链。连接测序接头,经过PCR扩增,增加模板量,构建cDNA测序文库。
1.4 测序数据质控对构建的15个cDNA文库用Illumina Genome Analyzer进行单末端100 bp测序。采用FastQC(v0.11.5)软件对测序数据进行质量评估,随后用FASTX-Toolkit(v0.0.6)去除接头序列,去除N碱基所占比例大于10%的序列,去除低质量序列,得到Clean reads。
2 加权基因共表达网络分析 2.1 WGCNA分析流程分析流程见图 1。

|
图 1 加权基因共表达分析流程 Fig. 1 The process of WGCNA |
数据预处理以实验室已经测序得到的比较转录组数据(SRX368254)为参考序列,通过软件RSEM(v1.3.0,以期望最大值算法为基础),计算表达谱所有reads的表达量情况[12]。
利用WGCNA软件包中的pickSoftThreshold函数进行软阈值的计算,软阈值的标准是使共表达网络要符合无尺度分布[13-14]。
基因共表达模块构建通过拓扑重叠来对基因间的关联程度进行计算[15]。以单个基因与剩余其他所有基因的关系为基础,利用WGCNA软件包中的TOMsimilarity函数将邻接矩阵转换为拓扑矩阵Ω=[Wxy],节点间的相异度的计算:dxyw=1-Wxy。对基因以节点间的相异度dxyw为基础,利用R语言中的hclust函数进行层次聚类,利用cutreeDynamic函数使用动态剪切法修整聚类树,随后利用R中的moduleEigengenes函数计算模块的特征值(Module epigengene,ME),利用mergeCloseModules函数,设置cutheight为0.2,依据模块特征值将聚类树中相似性80%以上的模块进行融合完成聚类数的构建,具有高拓扑重叠的一组基因构成一个基因模块,聚类树中的每个分支即代表不同的基因模块。
利用R语言的moduleEigengenes函数计算基因模块的特征向量基因,模块特征向量基因代表了模块内所有基因的一种表达模式,再引入样品的特征,计算特征向量基因与样品特征之间的相关性,相关系数越高代表模块与样品特征越关联,对其中显著相关模块进行后续的模块基因表达和功能富集分析。利用plotME和barplot函数将显著相关模块中的基因依据其在不同处理条件下的表达量进行聚类分析,并绘制各个模块的特征向量基因在不同处理条件下的柱状图以分析其表达情况。
对与性状显著相关的每个模块利用R语言包ClusterProfiler(v3.6.0)中的enrichKEGG函数进行KEGG富集分析。利用基于基因显著性和模块身份的函数NetworkScreening计算基因与模块的相关性,以相关性q值为基础进行枢纽基因的筛选。采用Cytoscape软件中的BinGO插件,对与性状显著相关模块的枢纽基因进行GO富集分析[16],富集分析的显著性水平p < 0.05。
2.3 统计学处理实验中的显著性水平的标准为p < 0.001,以错误发现率(False discovery rate,FDR)对p值进行验证得到q值,用q < 0.05来排除假阳性。
3 结果 3.1 不同处理条件下链状亚历山大藻的生长对五个处理条件下的链状亚历山大藻进行隔天计数并绘制生长曲线(见图 2),以观察其生长状态,确定取样时间。通过绘制的生长曲线可以看出,在高磷高锰以及高光的诱导条件下,链状亚历山大藻的生长速率以及细胞数量都高于其他处理条件,表现出了爆发性生长的特征,而在低氮和低磷处理条件下,该藻的生长速率较慢,通过显微镜观察在15天后,该藻开始逐渐衰亡,出现了大量的死细胞,通过生长曲线确定选取第12天作为取样时间进行后续实验。

|
图 2 不同培养条件下链状亚历山大藻生长曲线 Fig. 2 Growth curve of A. pacificum under five treatments |
将15个样品的表达谱数据进行质控,去除低表达量(RPKM值低于3.57)的基因,并筛选不同处理条件下,基因表达量方差较大的前25%的基因(表明在不同处理间变异性较高),随后利用R语言中的WGCNA软件包gsg数据过滤去除离群值,共得到16 326个基因用于网络基因的构建。
对15个样品进行聚类分析,绘制聚类图,以检验样品组间的相关性,去除离群样品,并依据样品的处理条件,标注其生物学特征。通过模拟赤潮爆发的条件,在培养过程中,链状亚历山大藻在高磷高锰以及高光的诱导条件下的生长速率和细胞总数要显著高于其他3个处理条件,表现出了爆发性生长的特性,因此将高磷高锰诱导以及高光诱导两个处理条件作为爆发性生长的样品特征(Explosive growth,EG)。聚类分析显示,没有离群样本的存在(见图 3),15个样品组间的相关性较好,均可以用于后续的分析。图中红色代表高光和高磷高锰处理的爆发性生长条件。

|
图 3 15个样品聚类结果及样品特征 Fig. 3 The phylogenetic tree and the information of the 15 samples |
在确定软阈值后,依据确定值进行基因模块的构建。首先计算两两基因间的相似性系数以及相异度,从而构建层次聚类树。最终得到35个相关性基因模块(见图 4),不同的颜色代表不同的基因共表达模块,灰色模块为未被分配的基因。

|
(Dynamic Tree Cut代表初步识别的基因模块;Merged Dynamic为融合相似模块后的基因表达模块。Dynamic Tree Cut represent the initial identification of the gene module, merged dynamic represent merged similar modules.) 图 4 基因共表达网络聚类树 Fig. 4 WGCNA cluster tree for the samples |
统计得到的各个基因模块的基因个数,在这35个基因模块中,基因数目从32到2 812个不等(见图 5)。

|
图 5 各模块基因数分布 Fig. 5 Number of genes in the 35 modules |
将每个基因模块的特征向量基因与样品的特征相关联,从而找到与样品特征密切相关的基因模块。图中颜色越红代表显著相关性越高,以p < 0.001为显著性相关的筛选标准,分析得到了与链状亚历山大藻爆发性生长(EG)显著性相关的6个模块,分别是blue4(0.79,p=8e-05)、blue(0.74,p=4e-04)、darkseagreen3(0.7,p=0.001)、firbrick3(0.79,p=8e-05)、black(0.92,p=5e-08)、darkgrey(0.82,p=3e-05)(见图 6)。

|
图 6 各模块特征向量与样品特征间的相关性分析 Fig. 6 Diagram of correlation between modules and sample treatment information |
基于基因模块的特征向量基因与样品特征关联分析结果,对在高磷高锰诱导、高光诱导条件下6个显著性相关模块(P < 0.001)的基因数进行了统计(见表 2)。由表 2可见,模块blue4中包含的基因最多有2 812个,而darkgrey模块最少有360个。对这6个模块的基因及特征向量基因在表达谱15个处理条件下的相对表达量分别进行热图层次聚类和柱状图分析,结果显示这六个模块在高磷高锰诱导以及高光诱导条件下均上调表达(见图 7)。
|
|
表 2 6个显著性相关模块的基因数统计 Table 2 The numbers of genes in 6 modules |

|
(15个处理条件为低氮(N1、N2、N3),低磷(P1、P2、P3),正常培养(C1、C2、C3),高磷高锰诱导(M1、M2、M3)和高光诱导(G1、G2、G3)。15 treatments contain low nitrogen(N1、N2、N3), low phosphorus(P1、P2、P3), normal condition(C1、C2、C3), high phosphorus(M1、M2、M3)and hight light(G1、G2、G3).) 图 7 显著性相关模块的基因在表达谱15个处理条件下的聚类分析 Fig. 7 Cluster analysis of the genes in 6 modules under 15 different treatments of DGE analysis |
对6个模块的基因分别进行KEGG富集分析,其中模块blue4中的基因显著富集到核糖体的生物合成、细胞骨架调节途径(q < 0.05),而模块black显著富集到了光合作用中的碳固定途径、磷酸戊糖途径(q < 0.05)(见图 8)。

|
(Rich factor是该模块枢纽基因中位于该通路的基因数目与所有该通路的基因数的比值。a. Blue4模块;b. Black模块。Rich factor represent the ratio of the hub genes in this pathway and all the genes of this pathway. a. Blue4 module; b. Black module.) 图 8 模块blue4和black的KEGG富集分析 Fig. 8 KEGG enrichment analysis of module black |
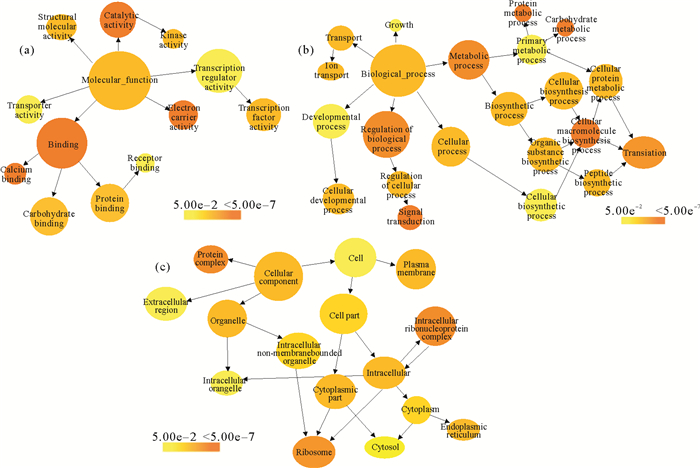
以基因与性状的相关性q值为基础对各个模块中的基因进行排序,每个模块中选取排序后前10%的基因作为枢纽基因,以6个显著性相关模块中的枢纽基因为研究对象,进行GO富集分析(见图 9),图中黄色越深代表富集越显著,圆圈大小表示GO条目的层级关系(p < 0.05)。在分子功能方面,枢纽基因显著性富集在了结合活性(p=2.70e-6),电子转运活性(p=5.20e-6)以及催化活性(p=3.20e-5);在生物过程方面,枢纽基因富集在信号转导(p=6.10e-6),蛋白质代谢(p=6.60e-6),碳水化合物代谢(p=7.30e-6)以及大分子物质的合成过程(p=8.20e-6);在细胞组分方面,枢纽基因主要富集在核糖体(p=4.70e-5)、蛋白复合体(p=6.20e-5)以及核糖核蛋白复合体(p=8.20e-5)等。
|
(a.分子功能;b.生物过程;c.细胞组分。a. Molecular function; b. Biological process; c. Cellular component.) 图 9 枢纽基因GO富集分析图 Fig. 9 GO enrichment analysis of hub genes |
对富集到的GO条目中显著性p < 5e-4的基因进行统计(见表 3),在生物过程基因包括涉及蛋白代谢的伴侣蛋白和预测蛋白,涉及碳水化合物代谢的6磷酸葡萄糖酸内酯酶,涉及信号传导的磷酸二酯酶(PDE)(同时富集在分子功能的催化活性)、钙离子依赖性激酶(同时富集在分子功能钙离子结合活性),以及涉及翻译过程的40s及60s核糖体蛋白(同时富集在细胞组分的核糖体上);在分子功能方面基因涉及电子转运活性的光系统1的铁硫中心,铁氧还蛋白还原酶和红素氧还蛋白,涉及蛋白结合活性的类泛素蛋白。
|
|
表 3 爆发性生长条件下枢纽基因GO富集结果(部分) Table 3 GO enrichment of hub genes in response to explosive growth of A. pacificum |
对6个显著相关模块中q值排序前3的hub基因进行统计(见表 4),模块blue4中包括60s核糖体蛋白亚基、核糖体蛋白,说明该模块可能与核糖体相关。模块Firebrick3中包括类形成蛋白、端锚聚合酶等。Darkgrey模块中基因包括转录起始因子、泛素激活酶以及粘蛋白。Darkseagreen3模块可能与蛋白质的合成加工相关,基因包括多聚泛素、热激蛋白(HSP)。Blue模块包含光捕获蛋白、细胞色素复合体b6f等,该模块可能与电子转运相关。模块Black中的基因包括转酮醇酶、甘油醛3磷酸脱氢酶以及辅酶A转移酶蛋白家族。
|
|
表 4 与爆发生长显著相关模块前3个枢纽基因信息 Table 4 The top 3 hub genes of modules significant correlate to explosive growth |
在链状亚历山大藻爆发性生长条件下,枢纽基因中的光捕获蛋白以及细胞色素复合体b6f参与到了光合作用的电子传递过程,光捕获蛋白捕获光能并将其转移到光系统中以开始光合作用的光反应,同时光捕获蛋白可以维持类囊体膜的结构[17]。基于GO富集分析(见表 3),红素氧还蛋白和铁氧还蛋白氧化还原酶显著富集在电子转运活性。红素氧还蛋白属于铁硫蛋白家族,在光合作用和非生物胁迫方面发挥重要作用,最初植物中的红素氧还蛋白由拟南芥类囊体上分离得到[18],随后在聚球藻的研究中发现该蛋白可能参与光系统I的组装,它的缺失将导致光系统I功能丧失[19]。在植物光合作用中,铁氧还蛋白还原酶接受铁氧还蛋白传递的电子使NADP+还原为NADPH,同时基于KEGG富集分析,显著相关模块的基因富集在光合作用碳固定途径,而光合电子传递为其提供了所需的NADPH和ATP,保证了碳固定途径的顺利进行。基于对模块基因的GO及KEGG富集分析,以及对枢纽基因的分析,说明光合作用在链状亚历山大藻爆发性生长方面起到重要作用。
枢纽基因中的40s以及60s核糖体蛋白亚基,在GO富集分析(见图 9)中显著富集在生物过程中的翻译过程以及细胞组分的核糖体中,基于KEGG分析(见图 8)参与了核糖体的生物合成,说明了核糖体的生物合成与细胞生长息息相关。
通过GO富集分析(见表 3),发现了与钙离子结合活性相关的基因钙离子依赖性激酶(CaMK)以及与信号传导相关的PDE,这两个基因均受到钙离子/钙调蛋白信号通路的调控,从而参与到细胞周期调控中。PDE是一类催化水解环核苷酸(cAMP)的关键酶,通过调节细胞内cAMP的水平,影响细胞的生理功能[20],研究发现cAMP的含量随着细胞周期的进行而改变,在S期之前,其含量的变化与DNA的合成相关,在纤细裸藻中,cAMP含量的增加会抑制DNA的合成,使细胞分裂停在G2期,而其含量下降,细胞周期也会随之恢复。钙离子及钙调蛋白信号通路参与了细胞内众多的生理过程,同时在细胞周期调控方面也起到了重要作用,受其调控的与细胞周期相关的CaMK和PDE的发现,说明了相关的信号通路在链状亚历山大藻细胞周期调控方面也有重要作用。
热激蛋白常以分子伴侣的形式存在,作为保护蛋白与其他蛋白结合从而稳定结合蛋白的结构,在细胞中参与蛋白的合成、蛋白的翻译后修饰以及蛋白质的跨膜运输等过程。其中Hsp90是真核生物中含量最丰富的蛋白之一,在真核生物中普遍存在,其活性受到ATP酶的调控,参与蛋白折叠、降解过程,与植物发育过程中参与多种酶以及激素受体的蛋白成熟过程,能促进信号肽的成熟,促进肽链的正确折叠,维持和稳定蛋白构象[21]。目前研究发现Hsp90参与到细胞周期调控,并受到钙调蛋白的作用参与信号转导过程[22]。Hsp70参与新生肽链的合成,调控新生蛋白质的折叠以及蛋白的跨膜运输等过程[23],在细胞快速生长阶段,需要大量蛋白质的合成,而Hsp90和Hsp70在蛋白质的合成加工方面的作用有助于保证蛋白质的正确折叠。
通过对hub基因进行统计后发现,大量的hub基因未被注释功能或注释为预测蛋白(Hypothetical protein),如在blue4模块中,对q值前20的hub基因进行统计并分析后发现,其中有11个基因未被注释。虽然有未注释蛋白存在,但同属于一个模块的基因在表达量上存在相关性,意味着这些基因在功能方面有相似性,可以由此来推测未注释基因的功能。
通过分析比较表达谱数据,基于WGCNA分析,核糖体的合成、磷酸戊糖途径、光合作用的碳固定途径起到了重要的调控作用,同时在表达谱测序筛选得到的差异基因外,枢纽基因中与蛋白质合成加工相关的热激蛋白以及参与光合作用光反应的荧光素结合蛋白以及电子传递蛋白的发现,更有助于完善链状亚历山大藻光合作用以及蛋白质代谢的调控机制,为进一步阐明这些调控机制对细胞生长的影响提供基础。
5 结语本研究以链状亚历山大藻模拟赤潮爆发条件下的表达谱数据为基础,首次将WGCNA分析用于赤潮爆发的机制的研究中。依据WGCNA分析的流程构建了基因的共表达网络,共得到了35个基因共表达模块。将基因模块与链状亚历山大藻爆发性生长特征相关联。通过功能富集分析以及对模块中枢纽基因的功能分析,与爆发性生长相关的枢纽基因涉及核糖体的生物合成、碳水化合物代谢(碳固定途径、磷酸戊糖途径)、光合作用光反应的电子传递过程以及蛋白质的合成和加工等,这些途径为细胞的快速增殖提供了物质基础,同时,枢纽基因中包含了大量的未知功能的基因,虽功能未知,但通过分析基因共表达模块,这些基因在调控细胞生长方面同样存在重要作用,对这些基因的进一步研究将有助于挖掘链状亚历山大藻爆发性生长的潜在的分子机制。
| [1] |
Leong S C Y, Mai M, Taguchi S. Carbon and nitrogen acquisition by the toxic dinoflagellate Alexandrium tamarense in response to different nitrogen sources and supply modes[J]. Harmful Algae, 2010, 9(1): 48-58. DOI:10.1016/j.hal.2009.07.003
(  0) 0) |
| [2] |
Laabir M, Collos Y, Masseret E, et al. Influence of environmental factors on the paralytic shellfish toxin content and profile of Alexandrium catenella (Dinophyceae) isolated from the Mediterranean Sea[J]. Marine Drugs, 2013, 11(5): 1583-1601. DOI:10.3390/md11051583
(  0) 0) |
| [3] |
Laabir M, Jauzein C, Genovesi B, et al. Influence of temperature, salinity and irradiance on the growth and cell yield of the harmful red tide dinoflagellate Alexandrium catenella colonizing Mediterranean waters[J]. Journal of Plankton Research, 2011, 33(10): 1550-1563. DOI:10.1093/plankt/fbr050
(  0) 0) |
| [4] |
Du Q, Sui Z, Chang L, et al. Genome size of Alexandrium catenella and Gracilariopsis lemaneiformis estimated by flow cytometry[J]. Journal of Ocean University of China, 2016, 15(4): 704-710. DOI:10.1007/s11802-016-2988-7
(  0) 0) |
| [5] |
Wu S, Lei J, Chen G, et al. De novo transcriptome assembly of Chinese kale and global expression analysis of genes involved in glucosinolate metabolism in multiple tissues[J]. Frontiers in Plant Science, 2017, 8: 92.
(  0) 0) |
| [6] |
Barabási A L. Scale-free networks: A decade and beyond[J]. Science, 2009, 325(5939): 412. DOI:10.1126/science.1173299
(  0) 0) |
| [7] |
Zhang J, Xiang Y, Ding L, Keen-Circle K, et al. Using gene co-expression network analysis to predict biomarkers for chronic lymphocytic leukemia[J]. Bmc Bioinformatics, 2010, 11(S9): S5. DOI:10.1186/1471-2105-11-S9-S5
(  0) 0) |
| [8] |
Horvath S, Zhang B, Carlson M, et al. Analysis of oncogenic signaling networks in glioblastoma identifies ASPM as a novel molecular target[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(46): 17402. DOI:10.1073/pnas.0608396103
(  0) 0) |
| [9] |
Yang B, Dougherty L, Cheng L, et al. A co-expression gene network associated with developmental regulation of apple fruit acidity[J]. Molecular Genetics and Genomics, 2015, 290(4): 1247-1263. DOI:10.1007/s00438-014-0986-2
(  0) 0) |
| [10] |
Bassel G W, Lan H, Glaab E, et al. Genome-wide network model capturing seed germination reveals coordinated regulation of plant cellular phase transitions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(23): 9709-9714. DOI:10.1073/pnas.1100958108
(  0) 0) |
| [11] |
Hobbie J E, Daley R J, Jasper S. Use of nuclepore filters for counting bacteria by fluorescence microscopy[J]. Applied and Environmental Microbiology, 1977, 33(5): 1225-1228.
(  0) 0) |
| [12] |
Li B, Dewey C N, Li B, et al. RSEM: Accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1): 323. DOI:10.1186/1471-2105-12-323
(  0) 0) |
| [13] |
Barabasi A L, Albert R. Emergence of scaling in random networks[J]. Science, 1999, 286(5439): 509-512. DOI:10.1126/science.286.5439.509
(  0) 0) |
| [14] |
Jeong H, Tombor B, Albert R, et al. The large-scale organization of metabolic networks[J]. Nature, 2000, 407.
(  0) 0) |
| [15] |
Li A, Horvath S. Network Neighborhood Analysis with the Multi-node Topological Overlap Measure[M]. Oxford: Oxford University Press, 2007: 222-231.
(  0) 0) |
| [16] |
Maere S, Heymans K, Kuiper M. BiNGO: A Cytoscape plugin to assess overrepresentation of gene ontology categories in biological networks[J]. Bioinformatics, 2005, 21(16): 3448-3449. DOI:10.1093/bioinformatics/bti551
(  0) 0) |
| [17] |
Kohorn B D, Harel E, Chitnis P R, et al. Functional and mutational analysis of the light-harvesting chlorophyll a/b protein of thylakoid membranes[J]. Journal of Cell Biology, 1986, 102(3): 972-981. DOI:10.1083/jcb.102.3.972
(  0) 0) |
| [18] |
Dal C B, Lezhneva L, Biehl A, et al. Inactivation of the chloroplast ATP synthase gamma subunit results in high non-photochemical fluorescence quenching and altered nuclear gene expression in Arabidopsis thaliana[J]. Journal of Biological Chemistry, 2004, 279(2): 1060-1069. DOI:10.1074/jbc.M308435200
(  0) 0) |
| [19] |
Shen G, Antonkine M L, Van dEA, et al. Assembly of photosystem Ⅰ. Ⅱ. Rubredoxin is required for the in vivo assembly of F(X) in Synechococcus sp. PCC 7002 as shown by optical and EPR spectroscopy[J]. Journal of Biological Chemistry, 2002, 277(23): 20355. DOI:10.1074/jbc.M201104200
(  0) 0) |
| [20] |
Butcher R W, Sutherland E W. Adenosine 3′, 5′-phosphate in biological materials. Ⅰ. Purification and properties of cyclic 3′, 5′-nucleotide phosphodiesterase and use of this enzyme to characterize adenosine 3′, 5′-phosphate in human urine[J]. Journal of Biological Chemistry, 1962, 237(237): 1244.
(  0) 0) |
| [21] |
Rutherford S L, Lindquist S. Hsp90 as a capacitor for morphological evolution[J]. Nature, 1998, 396(6709): 336-342. DOI:10.1038/24550
(  0) 0) |
| [22] |
Whitesell L, Lindquist S L. HSP90 and the chaperoning of cancer[J]. Nature Reviews Cancer, 2005, 5(10): 761-772. DOI:10.1038/nrc1716
(  0) 0) |
| [23] |
Mayer M P, Bukau B. Hsp70 chaperones: Cellular functions and molecular mechanism[J]. Cellular and Molecular Life Sciences Cmls, 2005, 62(6): 670-684. DOI:10.1007/s00018-004-4464-6
(  0) 0) |
2. Department of Microbiology, University of Veterinary and Animal Sciences, Lahore 54660, Pakistan
 2019, Vol. 49
2019, Vol. 49


