弧菌科(Vibrionaceae)隶属于细菌域变形菌门γ-变形菌纲弧菌目,由Véron[1]在1965年首次提出。弧菌科是弧菌目中唯一的科,目前其包括以弧菌属(Vibrio)为代表的8个属,共计159个种,主要分布在海洋环境中,某些种类是水产动物与人类重要的病原菌,具有潜在的环境效应[2]。
弧菌科成员是一类遗传与代谢多样性很高的异养细菌。其生活方式包含自由生活与附着生活[3];其分布区域涵盖了边缘海与大洋、表层海水与深层海水、热液口与冷泉等几乎所有的海洋环境[4-5];在与其它生物的关系中也扮演了共生菌[6]、病原菌[7]、益生菌[8]等多种角色。另外,弧菌科成员广泛参与到几丁质[9]、褐藻胶[10]等大分子有机物降解的过程中,是生物地球化学循环的重要环节。早期对弧菌科的研究关注其致病性[11],最近弧菌科的物种多样性与生态作用,以及测序技术发展带来的弧菌科遗传学特征与分子进化的研究引起了广泛的关注[12]。
弧菌科自确立一来,一直处于细菌分类学研究的前端[13-15]。其多样化的表型特点和较高的16S rRNA基因序列相似性,使得准确判断该科成员的分类学地位十分困难[2]。然而,弧菌科成员的这一特点也恰恰使其成为人们研究分类学、改进分类方法的绝佳模型,而且对弧菌科的分类研究是了解其诸多生物学特性的基础条件。因此,通过回顾弧菌科分类方法在近50年间的进展,总结目前在弧菌科成员分类地位中已取得的成果,进而对弧菌科的分类学研究提出展望,从而促进未来的科研工作者更进一步加强对弧菌科的研究。
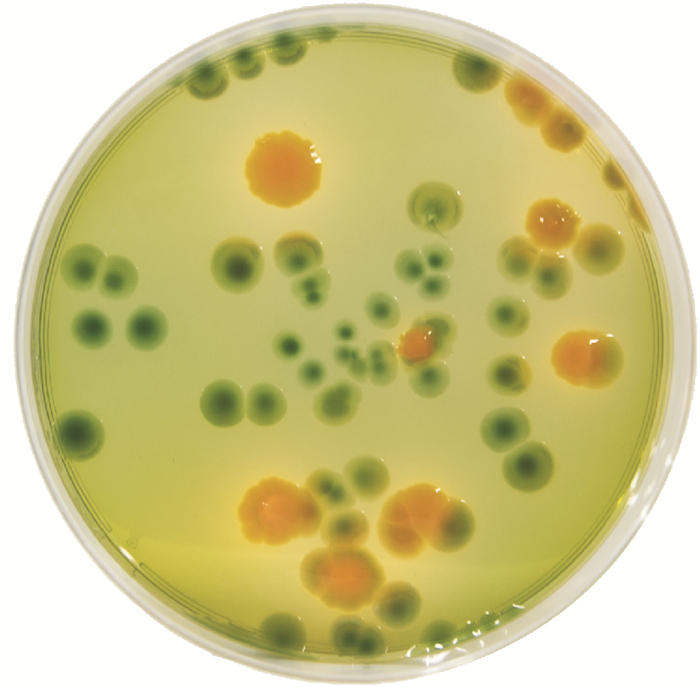
1 弧菌科细菌特征概述弧菌科细菌大多数具有如下特征:革兰氏阴性,菌体呈弯曲杆状或直杆状结构,可以通过极生鞭毛运动,化能异养,能够进行兼性厌氧代谢,具有发酵D-葡萄糖的能力且可将其作为唯一碳源,能够利用NH4+作为唯一氮源。大多数成员为水生,且需要Na+才能正常生长。氧化酶一般为阳性,还能够将硝酸盐还原为亚硝酸盐,不能进行反硝化。可以发酵D-果糖、麦芽糖和甘油。多数弧菌科细菌能够在不产气的条件下代谢多种碳水化合物,能够在TCBS(Thiosulfate Citrate Bile Salts Sucrose,硫代硫酸钠柠檬酸盐胆汁盐蔗糖)培养基上生长,并根据其不同的代谢产物性质产生不同颜色的菌落(见图 1)。另外,弧菌科中的有些种类可以发光(luminescence),如费氏另类弧菌(A. fischeri)、哈维氏弧菌(V. harveyi)、坎贝氏弧菌(V. campbellii)等,但是这些能够发光的种类中并非所有菌株都能发光[16]。
|
图 1 不同种类弧菌在TCBS培养基上的菌落特征 Fig. 1 Colony characteristics of different types of vibrios cultured on TCBS agar medium |
由于弧菌科成员的数目在不断扩增,目前有一些种类可能缺少如上所述的一个或多个共同特性,已有多个能够产气、固氮、光合营养或者不具有运动性的种类作为特例被报道[17]。弧菌科细菌主要广泛分布在海洋与河口环境中,在淡水环境中偶尔也有发现,经常与水生动植物相关。有些种类是人类的病原菌,另外还有一些种类是鱼类、贝类、珊瑚等水生生物的病原菌。
2 弧菌科分类学研究方法的递进 2.1 经典分类直到20世纪中期,弧菌科细菌的分类主要是通过“经典分类”法,即表型鉴定来完成的,其试图根据少数几种表型特征(如菌体特征、菌落形态或生理学特征等)来对细菌进行分类[18]。随着弧菌科成员的不断增多,仅仅通过有限的表型特征来确定上百个成员的分类地位显然已经不太可能,而且有时生物体的表型在其不同的生命时期或者不同的生活条件下会表现出相异的特征,这无疑给仅仅基于形态学来进行分类的手段提出了很多难题。例如,第八版《伯杰氏鉴定细菌学手册》中将哈维氏弧菌归入了“射光杆菌属(Lucibacterium)”,其分类标准是弧菌属细菌具有极生鞭毛,而射光杆菌属细菌具有侧生鞭毛(或称周生鞭毛)。而事实上,在以后的研究中人们发现弧菌属细菌在液体环境中生长时为极生鞭毛,而在固体中时基本都能够形成侧生鞭毛,因而在1980年又将射光杆菌属归并到弧菌属中[19]。另外,弧菌科细菌的成员在不断扩充,事实上人们已经发现了该科许多细菌根本不具有其原来被认为应该具有的特征。如王燕等[20]报道的非典型弧菌(V. atypicus)呈过氧化氢酶阴性,并不符合以前我们对于弧菌属细菌的认识。因此,仅仅基于表型的细菌分类学在如今已经不能满足确定弧菌科成员分类学地位的要求。
2.2 多相分类与分子分类1970年代左右,在弧菌科细菌中应用的DDH(DNA-DNA hybridization,DNA-DNA杂交)[21-22]方法,通过分子操作手段来考察其物种间的DNA组成相似性,从而确定遗传距离。这种方法规定,在退火温度ΔTm低于5 ℃时DNA间的杂交率大于70%,可作为判断物种属于同一种的黄金标准。然而这种方法在实际操作中却存在诸多问题,比如确定DNA-DNA杂交率的方法不同会得到不同的结果,而且对于70%这个标准有时也会根据情况而改变[23]。
多相分类(Polyphasic taxonomy)方法由Colwell[24]在1970年首次提出并应用于弧菌属中,其宗旨是考察物种的基因型、表型和系统发育等全部信息来对物种进行分类。后来,随着1977年Sanger测序技术的发明,利用核糖体RNA基因相似性的分类方法逐渐兴起,并在弧菌科中得到大量应用[25]。DNA测序连同分子指纹图谱方法的出现又进一步补充了当时多相分类学的不足,在弧菌科细菌分类中应用的分子指纹图谱方法包括rep-PCR(细菌基因组重复序列PCR技术)[26]、AFLP(Amplified fragment length polymorphism,扩增片段长度多态性)[27]等,这些分子生物学技术的加入使得多相分类学得到了很大的补充,其中16S rRNA基因测序法迄今仍为物种多相分类学鉴定的重要组成部分[18]。多相分类学的出现为弧菌科成员分类地位的确立提供了重要手段,纠正了很多以前仅仅利用少数几个表型特征就确立的物种分类地位。例如以前存在于弧菌科中的异单胞菌属(Allomonas)就是通过多相分类学的方法被重新判定了分类学地位[28]。
2.3 基因组分类进入21世纪后,全基因组测序(Whole Genome Sequencing,WGS)的兴起将细菌的分类学推入全新的时代。基因组分类法包括MLSA(Multilocus Sequence Analysis,多位点序列分析)、核心基因组树(超级树)、ANI(Average Nucleotide Identity,平均核苷酸一致性)、AAI(Average Amino Acid Identity,平均氨基酸一致性)、基因组特征相异性(genome signature dissimilarity)等类型[14],这些方法具有信息量大、可重复性强、稳定性强等特点。很多研究证明,这些方法在弧菌的分类中具有较高的分辨率,能够用于精确估计弧菌科成员的分类地位。尤其是MLSA方法已经得到了广泛的应用,因为这种方法事实上并不需要得到完整的细菌基因组,只需利用一些基因的组合即可得到分辨率高、可信度强的结果。例如“费氏弧菌(V. fischeri)”等几个原来弧菌属种类的分类学地位和系统发育关系一直存在疑问,2007年Urbanczyk等[29]利用MLSA的方法将包括费氏弧菌在内的4个种归并为弧菌科内的新属另类弧菌属。再比如,Sawabe等[15]利用8个管家基因(gapA、gyrB、ftsZ、mreB、pyrH、recA、ropA和ropA)建立了弧菌科中58个种的高质量系统发育树,而此种方法也与基于全基因组水平的核心基因组树具有相似的树形。多项研究表明,MLSA与核心基因组树为弧菌科细菌的分类提供了更好的分类学标准[14, 17]。基于基因组分类的方法把弧菌科细菌中的种定义为:在MLSA和核心基因组的基因序列中享有>95%的DNA一致性、>96%的AAI、≤10的基因组特征相异性和>61%的蛋白组一致性,且同种不同株和同属不同种的物种会在MLSA和核心基因组树中聚为单系群(Monophyletic group)[17]。随着近年来全基因组测序便利性的增高,传统的多相分类学方法必然将会向基因组分类方法转变[30]。
3 弧菌科细菌的分类地位与系统发育关系的发展 3.1 弧菌科分类地位的历史变化弧菌科在1965年被Véron[1]首次提出,其成员的分类地位历经多次调整,并不断有新属被添加进该科中。现存的弧菌属与发光杆菌属存在于最初定义的弧菌科中。首次提出的弧菌科成员中还包括气单胞菌属(Aeromonas)和邻单胞菌属(Plesiomonas),这两个属后来被归并到其它科中[31]。2002年出版的《伯杰氏系统细菌学手册》中收录了弧菌科的7个属[17],除了原有的弧菌属和发光杆菌属外,新添加了异单胞菌属、链状球菌属(Catenococcus)、肠弧菌属(Enterovibrio)、利斯顿氏菌属(Listonella)和盐弧菌属(Salinivibrio)。随后根据分子生物学研究,发现异单胞菌属和利斯顿氏菌属在16S rRNA基因序列、DNA杂交率和表型性状等特征上与弧菌属内某些种的相似性很高,因此又被重新归于弧菌属[28, 32],而新属格里蒙氏菌属[33]和另类弧菌属[29]则被提议纳入弧菌科。最后,2013年又报道了分离自海胆的新种成为弧菌科的新属海胆单胞菌属[34],该属至今仍然只包含一个种。至此,弧菌科形成了目前包括8个有效属的状态。
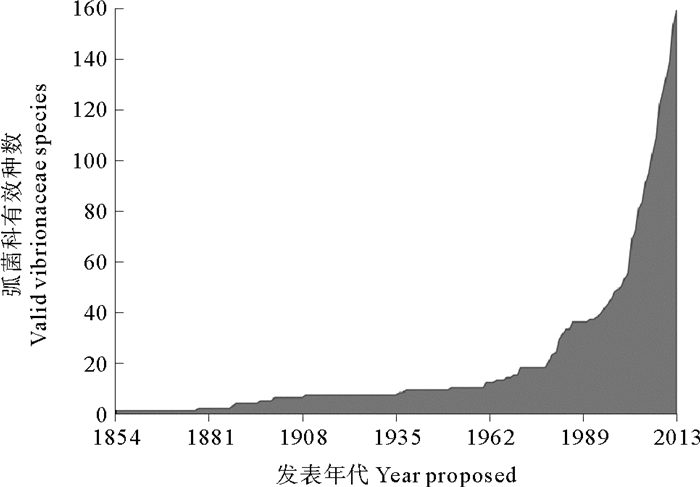
弧菌科中新种的发表近些年呈爆发趋势(见图 2)。回顾弧菌科新种的报道历史,第一株发表的弧菌科成员为霍乱弧菌(V. cholerae),于1854年由意大利著名科学家Filippo Pacini在席卷全球的第三次霍乱大流行中从一个感染霍乱病的病人身上分离得到[35]。虽然局限于当时的条件而仅仅是对细菌进行了显微镜下观察,但这一发现为后来霍乱病的防治提供了重要依据。然而,在其后的近100年中,弧菌科成员的新种鲜有报道,直到1960年弧菌科中仅有10个种被报道。在1970年以后,弧菌科内的新种数开始逐步增加,尤其是在2000年以后,弧菌科的新种报道出现激增,仅2003年报道的弧菌新种就达到14种。近10年来(2006—2016)弧菌科共新增78个新种,几乎占目前弧菌科所有种数目的一半。
|
图 2 弧菌科细菌有效种数随发表年代的累积曲线 Fig. 2 The cumulative number of valid Vibrionaceae species against the year in which they were proposed |
弧菌科属于γ-变形菌纲,与肠杆菌科(Enterobacteriaceae)、巴斯德氏菌科(Pasteurellaceae)、气单胞菌科(Aeromonadaceae)和交替单胞菌科(Alteromonadaceae)在系统发育关系中紧邻[36]。根据最新的LPSN(具有命名地位的原核生物名录,http://www.bacterio.net),弧菌科中现共包括有效的8个属,共计159个种,分别是另类弧菌属(Aliivibrio,6个种)、链状球菌属(Catenococcus,1个种)、海胆单胞菌属(Echinimonas,1个种)、肠弧菌属(Enterovibrio,5个种)、格里蒙氏菌属(Grimontia,3个种)、发光杆菌属(Photobacterium,25个种)、盐弧菌属(Salinivibrio,4个种)和弧菌属(Vibrio,114个种)。其中发光杆菌属、盐弧菌属和弧菌属中都有亚种存在(见表 1)。
|
|
表 1 弧菌科细菌所有有效种的基本信息 Table 1 Basic information of all the valid species of Vibrionaceae |
通过对这些有效的弧菌科成员进行总结,并对相关文献充分研究,本文列出了其分离地位的拉丁学名、标准菌株保藏号、分离地点与时间、生物体来源、16S rRNA基因登录号和新种首次发表年代等信息,还对近些年来已发表但未有中文译名的新种参照其拉丁学名语义进行了中文翻译(见表 1)。
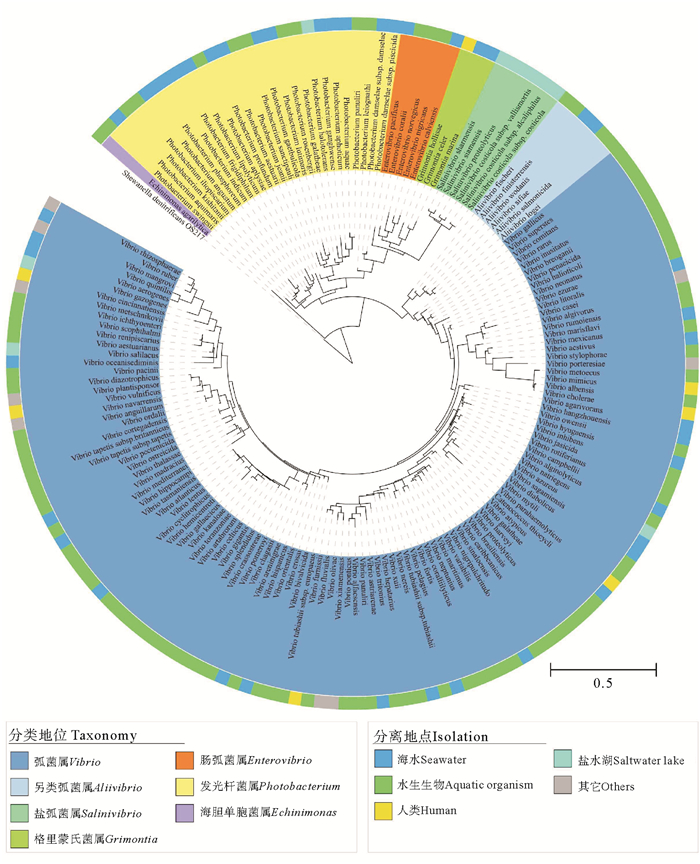
根据弧菌科成员的16S rRNA基因序列构建的系统发育树(见图 3)显示,弧菌属与另类弧菌属关系最为紧密而聚为一簇;盐弧菌属、格里蒙氏菌属、肠弧菌属聚为一簇,其共同祖先与发光杆菌属聚为一簇;海胆单胞菌属作为以上两个大簇的旁系群(Sister group)单独存在。值得一提的是,硫循环链状球菌(C. thiocycli)作为链状球菌属中的唯一种,其在进化树中的位置处于弧菌属之中,事实上在有些报道中已经建议该属不应该与弧菌属区分开来[37]。
|
(比例尺代表每位点的替换率为0.5。Bar 0.5 substitutions per site.) 图 3 基于最大似然法构建的弧菌科标准菌株16S rRNA基因的系统发育树 Fig. 3 Phylogenetic reconstruction of the family Vibrionaceae based on 16S rRNA genes of the type strains of each species by using the maximum likelihood algorithm |
我国对弧菌科细菌新种的分类鉴定研究在国际上起步较晚。1980年代由Yang等[38]报道了中国首个弧菌科新种——东方弧菌(V. orientalis)。目前由我国学者参与的在中国及其周边地区分离的弧菌科新种有12个,其中有1个种属于发光杆菌属(P. aquae),其它11个种均属于弧菌属(见表 1)。自我国分离的弧菌科细菌来源多种多样,其中以海水和水产动物来源的菌株较多,也有来自于盐水湖和红树林土壤等环境的菌株。除去新种报道,我国对其他弧菌科细菌的分离鉴定大多来自病人和经济水产动物的病原菌,我国在这一方面的研究起步时间很早,可查到的记录最早可追溯到1976年由辽宁省盘锦地区卫生防疫站[39]报道的一次有69例食物中毒群发事件的病原菌是副溶血弧菌(V. parahaemolyticus);其它还有很多水产动物病原菌的分离鉴定,如王保坤等[40]通过64项形态和生理生化指标鉴定出青岛胶南海区一次花鲈(Lateolabrax japonicus)幼鱼的传染病主要是由哈维氏弧菌引起;柯韶文[41]对我国南海西沙海域的珊瑚白化现象进行调查,发现溶藻弧菌(V. alginolytics)的入侵是引起这一现象的病原菌株。
值得一提的是,分离自我国山东省一处苗期对虾养殖水体的徐氏弧菌(V. xuii)的命名是为了纪念我国著名海洋微生物科学家徐怀恕先生而确立的[42]。另外,本实验室还于2010年报道分离自中国对虾(Penaeus chinensis O'sbeck)消化道的非典型弧菌(V. atypicus)[20],于2011年报道分离自黄海冷水团表层海水的黄海弧菌(V. marisflavi)[43]。
5 总结与展望自弧菌科被确立的50余年间,弧菌科细菌的分类学研究一直在不断发展。从1965开始的经典分类法,到1970年以后的多相分类与分子分类法相结合,再到现在的基因组分类法,弧菌科细菌一直作为模式物种被最新的分类学方法所检验,也见证了整个细菌分类学的发展历程。在这几十年里,细菌分类学变得可靠性越来越强、可重复性越来越高、操作方法越来越简单便利且易于标准化。弧菌科成员的分类学地位与系统发育关系也被一步步更清晰准确地揭示。然而,弧菌科细菌种类近些年来爆发式的增长也必然向分类学手段提出新的挑战,而如何利用有限的数据资源建立模型,从而准确评估其分类地位是解决问题的关键。另外,弧菌科细菌不仅在海水环境中无处不在,其某些种类也是人类和多种水产动物的致病菌。由弧菌引起的疾病已在我国有非常多的报道,准确快速地对病原体进行分类学地位的鉴定是进行疾病防治的首要任务。MLSA已经为我们打开了一扇大门,接下来将对用于MLSA分析的基因进行优化,建立MLSA比对的数据库,从而创建快速进行MLSA检测的方法,将是这一工作的重点。比起国外对弧菌科细菌的研究,我国虽早年对其研究甚少,但近些年来关于弧菌科细菌分类鉴定的报道越来越多,这将有助于我们更加重视弧菌科在生态学、致病性和生物多样性等研究领域中的作用,引导我们开启对弧菌科更多方面的科学研究。
| [1] |
Véron M. La position taxonomique des Vibrio et de certaines bactéries comparables[J]. Comptes Rendus Hebdomadaires Des Seances de L'Academie Des Sciences, 1965, 261(23): 5243-5246.
(  0) 0) |
| [2] |
Thompson F L, Iida T, Swings J. Biodiversity of vibrios[J]. Microbiology and Molecular Biology Reviews, 2004, 68(3): 403-431. DOI:10.1128/MMBR.68.3.403-431.2004
(  0) 0) |
| [3] |
McCarter L. The multiple identities of Vibrio parahaemolyticus[J]. Journal of Molecular Microbiology and Biotechnology, 1999, 1(1): 51-57.
(  0) 0) |
| [4] |
Takemura A F, Chien D M, Polz M F. Associations and dynamics of Vibrionaceae in the environment, from the genus to the population level[J]. Frontiers in Microbiology, 2014, 5: 38.
(  0) 0) |
| [5] |
Seckbach J, Grube M. Symbioses and Stress: Joint Ventures in Biology[M]. Dordrecht: Springer Netherlands, 2010.
(  0) 0) |
| [6] |
Verma S, Miyashiro T. Quorum sensing in the squid-Vibrio symbiosis[J]. International Journal of Molecular Sciences, 2013, 14(8): 16386. DOI:10.3390/ijms140816386
(  0) 0) |
| [7] |
Huehn S, Eichhorn C, Urmersbach S, et al. Pathogenic vibrios in environmental, seafood and clinical sources in Germany[J]. International Journal of Medical Microbiology, 2014, 304(7): 843-850. DOI:10.1016/j.ijmm.2014.07.010
(  0) 0) |
| [8] |
Balcázar J L, Blas I d, Ruiz-Zarzuela I, et al. The role of probiotics in aquaculture[J]. Veterinary Microbiology, 2006, 114(3-4): 173-186. DOI:10.1016/j.vetmic.2006.01.009
(  0) 0) |
| [9] |
Pruzzo C, Vezzulli L, Colwell R R. Global impact of Vibrio cholerae interactions with chitin[J]. Environmental Microbiology, 2008, 10(6): 1400-1410. DOI:10.1111/emi.2008.10.issue-6
(  0) 0) |
| [10] |
Badur A H, Jagtap S S, Yalamanchili G, et al. Alginate lyases from alginate-degrading Vibrio splendidus 12B01 are endolytic[J]. Applied and Environmental Microbiology, 2015, 81(5): 1865-1873. DOI:10.1128/AEM.03460-14
(  0) 0) |
| [11] |
Jones M K, Oliver J D. Vibrio vulnificus: Disease and pathogenesis[J]. Infection and Immunity, 2009, 77(5): 1723-1733. DOI:10.1128/IAI.01046-08
(  0) 0) |
| [12] |
Ceccarelli D, Colwell R R. Vibrio ecology, pathogenesis, and evolution[J]. Frontiers in Microbiology, 2014, 5: 256.
(  0) 0) |
| [13] |
Thompson F L, Gevers D, Thompson C C, et al. Phylogeny and molecular identification of vibrios on the basis of multilocus sequence analysis[J]. Applied and Environmental Microbiology, 2005, 71(9): 5107-5115. DOI:10.1128/AEM.71.9.5107-5115.2005
(  0) 0) |
| [14] |
Thompson C C, Vicente A C P, Souza R C, et al. Genomic taxonomy of vibrios[J]. BMC Evolutionary Biology, 2009, 9(1): 258. DOI:10.1186/1471-2148-9-258
(  0) 0) |
| [15] |
Sawabe T, Ogura Y, Matsumura Y, et al. Updating the Vibrio clades defined by multilocus sequence phylogeny: Proposal of eight new clades, and the description of Vibrio tritonius sp. nov[J]. Frontiers in Microbiology, 4: 414.
(  0) 0) |
| [16] |
Farmer J J, Michael Janda J. Bergey's Manual of Systematics of Archaea and Bacteria[M]. USA: John Wiley and Sons, Ltd, 2015.
(  0) 0) |
| [17] |
Rosenberg E, DeLong F E, Stackebrandt E, et al. The Prokaryotes: Gammaproteobacteria[M]. 4 edn. London: Springer, 2014.
(  0) 0) |
| [18] |
Thompson C C, Chimetto L, Edwards R A, et al. Microbial genomic taxonomy[J]. BMC Genomics, 2013, 14(1): 913. DOI:10.1186/1471-2164-14-913
(  0) 0) |
| [19] |
Baumann P, Baumann L, Bang S S, et al. Reevaluation of the taxonomy of Vibrio, Beneckea, and Photobacterium: A bolition of the genus Beneckea[J]. Current Microbiology, 1980, 4(3): 127-132. DOI:10.1007/BF02602814
(  0) 0) |
| [20] |
Wang Y, Zhang X-H, Yu M, et al. Vibrio atypicus sp nov, isolated from the digestive tract of the Chinese prawn (Penaeus chinensis O'sbeck)[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(11): 2517-2523. DOI:10.1099/ijs.0.016915-0
(  0) 0) |
| [21] |
Reichelt J L, Baumann P, Baumann L. Study of genetic relationships among marine species of the genera Beneckea and Photobacterium by means of in vitro DNA/DNA hybridization[J]. Archives of Microbiology, 1976, 110(1): 101-120. DOI:10.1007/BF00416975
(  0) 0) |
| [22] |
Sakazaki R, Gomez C Z, Sebald M. Taxonomical studies of the so-called NAG vibrios[J]. Japanese Journal of Medical Science and Biology, 1967, 20(4): 265-280. DOI:10.7883/yoken1952.20.265
(  0) 0) |
| [23] |
Vandamme P, Pot B, Gillis M, et al. Polyphasic taxonomy, a consensus approach to bacterial systematics[J]. Microbiological Reviews, 1996, 60(2): 407-438.
(  0) 0) |
| [24] |
Colwell R R. Polyphasic taxonomy of the genus Vibrio: Numerical taxonomy of Vibrio cholerae, Vibrio parahaemolyticus, and related Vibrio species[J]. Journal of Bacteriology, 1970, 104(1): 410-433.
(  0) 0) |
| [25] |
Rehnstam A S, Norqvist A, Wolf-Watz H, et al. Identification of Vibrio anguillarum in fish by using partial 16S rRNA sequences and a specific 16S rRNA oligonucleotide probe[J]. Applied and Environmental Microbiology, 1989, 55(8): 1907-1910.
(  0) 0) |
| [26] |
Shangkuan Y H, Lin H C, Wang T M. Diversity of DNA sequences among Vibrio cholerae O1 and non-O1 isolates detected by whole-cell repetitive element sequence-based polymerase chain reaction[J]. Journal of Applied Microbiology, 1997, 82(3): 335-344. DOI:10.1046/j.1365-2672.1997.00365.x
(  0) 0) |
| [27] |
Janssen P, Coopman R, Huys G, et al. Evaluation of the DNA fingerprinting method AFLP as a new tool in bacterial taxonomy[J]. Microbiology, 1996, 142(7): 1881-1893. DOI:10.1099/13500872-142-7-1881
(  0) 0) |
| [28] |
Farmer J J. International committee on systematic bacteriology subcommittee on the taxonomy of Vibrionaceae[J]. International Journal of Systematic and Evolutionary Microbiology, 1989, 39(2): 210-212.
(  0) 0) |
| [29] |
Urbanczyk H, Ast J C, Higgins M J, et al. Reclassification of Vibrio fischeri, Vibrio logei, Vibrio salmonicida and Vibrio wodanis as Aliivibrio fischeri gen nov, comb nov, Aliivibrio logei comb nov, Aliivibrio salmonicida comb nov and Aliivibrio wodanis comb nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(12): 2823-2829. DOI:10.1099/ijs.0.65081-0
(  0) 0) |
| [30] |
Gevers D, Cohan F M, Lawrence J G, et al. Re-evaluating prokaryotic species[J]. Nature Reviews Microbiology, 2005, 3(9): 733-739. DOI:10.1038/nrmicro1236
(  0) 0) |
| [31] |
Ruimy R, Breittmayer V, Elbaze P, et al. Phylogenetic analysis and assessment of the genera Vibrio, Photobacterium, Aeromonas, and Plesiomonas deduced from small-subunit rRNA sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 1994, 44(3): 416-426.
(  0) 0) |
| [32] |
Thompson F L, Thompson C C, Dias G M, et al. The genus Listonella MacDonell and Colwell 1986 is a later heterotypic synonym of the genus Vibrio Pacini 1854 (Approved Lists 1980) - a taxonomic opinion[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(12): 3023-3027. DOI:10.1099/ijs.0.030015-0
(  0) 0) |
| [33] |
Thompson F L, Hoste B, Vandemeulebroecke K, et al. Reclassification of Vibrio hollisae as Grimontia hollisae gen nov, comb. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(5): 1615-1617. DOI:10.1099/ijs.0.02660-0
(  0) 0) |
| [34] |
Nedashkovskaya O I, Stenkova A M, Zhukova N V, et al. Echinimonas agarilytica gen nov, sp nov, a new gammaproteobacterium isolated from the sea urchin Strongylocentrotus intermedius[J]. Antonie van Leeuwenhoek, 2013, 103(1): 69-77. DOI:10.1007/s10482-012-9787-y
(  0) 0) |
| [35] |
Pacini F. Osservazione microscopiche e deduzioni patologiche sul cholera asiatico[J]. Gazette Medicale de Italiana Toscano Firenze, 1854, 6: 405-412.
(  0) 0) |
| [36] |
Thompson F L, Austin B, Swings J. The Biology of Vibrios[M]. Washington, DC: ASM Press, 2006.
(  0) 0) |
| [37] |
Dikow R B. Systematic relationships within the Vibrionaceae (Bacteria: Gammaproteobacteria): Steps toward a phylogenetic taxonomy[J]. Cladistics, 2011, 27(1): 9-28. DOI:10.1111/cla.2010.27.issue-1
(  0) 0) |
| [38] |
Yang Y, Yeh L P, Cao Y, et al. Characterization of marine luminous bacteria isolated off the Coast of China and description of Vibrio orientalis sp. nov[J]. Current Microbiology, 1983, 8(2): 95-100. DOI:10.1007/BF01566965
(  0) 0) |
| [39] |
辽宁省盘锦地区卫生防疫站. 一次69例副溶血性弧菌食物中毒调查报告[J]. 卫生研究, 1976(2): 167-170. Health and Epidemic Prevention Station of Panjin. Investigation on an incident of 69 food poisoning cases caused by Vibrio parahaemolyticus[J]. Journal of Hygiene Research, 1976(2): 167-170. (  0) 0) |
| [40] |
王保坤, 余俊红, 李筠, 等. 花鲈弧菌病病原菌(哈维氏弧菌)的分离与鉴定[J]. 中国水产科学, 2002(1): 52-55. Wang B K, Yu J H, L Y, et al. The isolation and identification of a pathogen (Vibrio harveyi) from sea perch which had vibriosis[J]. Journal of Fishery Sciences of China, 2002(1): 52-55. (  0) 0) |
| [41] |
柯韶文. 海南西沙两种滨珊瑚主要疾病的调查、病原鉴定及其IGS序列分析[D]. 海南: 海南大学, 2013. Ke S W. The survey, Pathogen Identification of Two Diseases of Species of Porites in Xisha Archipelago and analysis of the IGS of the Pathogens[D]. Hainan: Hainan University, 2013. (  0) 0) |
| [42] |
Thompson F L, Li Y, Gomez-Gil B, et al. Vibrio neptunius sp. nov., Vibrio brasiliensis sp. nov. and Vibrio xuii sp. nov., isolated from the marine aquaculture environment (bivalves, fish, rotifers and shrimps)[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(1): 245-252. DOI:10.1099/ijs.0.02447-0
(  0) 0) |
| [43] |
Wang H, Liu J, Wang Y, et al. Vibrio marisflavi sp nov, isolated from seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(3): 568-573. DOI:10.1099/ijs.0.022285-0
(  0) 0) |
 2018, Vol. 48
2018, Vol. 48

