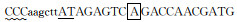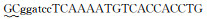2. 中国海洋大学海洋生物多样性与进化研究所,山东 青岛 266003
先天免疫系统是抵御微生物入侵的古老宿主防御系统。模式识别受体(Pattern recognition receptors,PRRs)是先天性免疫系统的重要组成部分,负责识别病原体的病原相关分子模式(Pathogen associated molecular pattern,PAMPs),激活宿主免疫反应[1-2]。Toll样受体(Toll-like receptors,TLRs)属于Ⅰ型跨膜蛋白,是一类最早被鉴定出来的PRRs,含有3个保守的结构域:胞外富含亮氨酸的重复结构域(Leucine-rich repeat,LRR)、跨膜结构域和胞内胞内Toll/白细胞介素-1受体结构域(Toll/interleukin-1 receptor,TIR) [3]。LRR结构域负责识别PAMPs,并通过序列变化和LRR的数量决定着TLR的特异性[4-5]。TIR结构域通过MyD88依赖或非依赖信号通路传递信号[6]。在脊椎动物中,已发现20多个TLR,并将其分为6个亚家族:TLR1(TLR1、TLR2、TLR6、TLR10、TLR14、TLR15、TLR16、TLR18、TLR25、TLR27和TLR28)、TLR3、TLR4、TLR5(TLR5和可溶类型TLR5S)、TLR7(TLR7、TLR8和TLR9)和TLR11(TLR11、TLR12、TLR13、TLR19、TLR20、TLR21、TLR22、TLR23、TLR24和TLR26)亚家族[7]。
TLR14是非哺乳动物受体,是TLR1家族的新成员。目前,已在河豚(Takifugu rubripes)[8]、非洲爪蟾(Xenopus tropicalis)[9]、七鳃鳗(Lampetra japonica)[10]、牙鲆(Paralichthys olivaceus)[11]、斜带石斑鱼(Epinephelus coioides)[12]、鮸鱼(Miichthys miiuy)[13]、大刺鳅(Mastacembelus armatus)[14]、金鲳鱼(Trachinotus ovatus)[15]、黄姑鱼[16](Nibea albiflora)和鳜鱼(Siniperca chuatsi)[17]中被鉴定出来。研究表明,硬骨鱼TLR14在用爱德华氏菌(Edwardsiella tarda)或刺激隐核虫(Cryptocaryon irritans)刺激后转录水平上调[11-12],可以识别细菌组分,触发宿主的炎症反应[18]。在哺乳动物中,TLR2可以与TLR1、TLR6、TLR10形成同二聚体或异二聚体,来识别病原体[19]。TLR2在硬骨鱼和哺乳动物之间具有功能保守性[20],但在硬骨鱼中,TLR1家族中没有TLR6和TLR10的直系同源物。因此,TLR14可能是哺乳动物TLR6和TLR10的功能替代品[11]。然而,还没有研究表明TLR14特异性识别的配体有哪些。
大菱鲆(Scophthalmus maximus)是中国北方重要的海水养殖经济鱼类。随着海水养殖业的迅速发展,由细菌和病毒引起的疾病已使大菱鲆养殖产业产生了严重的经济损失,因此,如何预防和控制大菱鲆疾病仍然是一个养殖难题。之前,本研究团队已经从大菱鲆中鉴定了TLR2、TLR3、TLR5M,TLR21和TLR22基因并研究了它们的免疫应答表达模式[20-24]。在本研究中,我们从大菱鲆中克隆并鉴定了TLR14基因,通过实时荧光定量PCR对其在正常组织和被脂多糖(Lipopolysaccharide,LPS)、肽聚糖(Peptidoglycan,PGN)感染的组织中表达模式进行了研究。将SmTLR14的编码序列(Coding sequence,CDS)连接到绿色荧光蛋白载体中,使用共聚焦显微镜确定其亚细胞定位。分析了SmTLR14过表达对下游免疫相关因子TNF-α,IL-1β,IL-8,IFN-1,IRF-1和IRF-3表达的影响。
1 材料和方法 1.1 实验材料及处理大菱鲆幼鱼(平均体质量(54.7±3.1) g,n=140)购自青岛市台东南山市场,采购的大菱鲆在实验室50 L的水族箱中暂养一周,每个水族箱养10条,水族箱中的水不断循环、充气,水温维持在18 ℃。
LPS (L2880, Escherichia coli 055:B5) 购自美国Sigma公司,PGN (S11184, Staphylococcus aureus) 购自上海源叶生物技术有限公司。2个实验组的大菱鲆分别从腹膜内注射LPS(10 mg/mL,每条鱼100 μL)或PGN(0.4 mg/mL,每条鱼100 μL),对照组的大菱鲆注射相同体积的PBS。分别在注射后0 h,3 h,6 h,12 h,1 d,2 d,3 d,5 d时,从2个实验组及对照组中各选3条鱼,用0.1%(w/v)的MS-222麻醉,解剖取出鳃、头肾、脾脏和肌肉,于-80 ℃储存,直至用于基因表达分析。
从3条未处理的健康鱼中取出脑、鳃、胃、肠、心脏、头肾、体肾、脾脏、肝、性腺、肌肉和皮肤,于-80 ℃储存,直至用于组织分布研究。
所有动物实验均已获得中国疾病预防控制中心实验动物护理伦理委员会的批准(实验批准号:120;批准日期:2011-09-03)。
1.2 提取RNA及基因组DNA使用Isogen试剂(Nippon Gene, 日本)提取各组织总RNA。使用Turbo DNA-free Kit(Ambion, 美国)处理RNA样品以去除基因组DNA污染。使用NanoDrop 2000分光光度计(Thermo Scientific,美国)在260 nm处测量吸光度来确定RNA浓度,样品合格标准是A260/A280在1.8~2.0之间且A260/A230>2.0。使用1.2%甲醛变性的琼脂糖凝胶电泳来检测RNA样品的完整性。使用苯酚-氯仿提取方法从肌肉中提取基因组DNA。
1.3 SmTLR14 cDNA和基因的克隆从大菱鲆头肾中提取1 μg总RNA,使用SMART cDNA Library Construction Kit(Clontech, 美国)转录出双链cDNA文库。根据已知鱼类TLR14的保守序列,设计简并引物(见表 1),通过同源克隆得到SmTLR14的1 517 bp核心cDNA序列,通过Genome Walking Kit (TaKaRa,大连)和3′-RACE法得到5′-和3′-末端片段。使用Genetyx 6.0软件编辑以上3个序列,将其拼接为连续编码序列。设计基因特异性引物TLR14-gF1,TLR14-gR1和TLR14-gF2,TLR14-gR2(见表 1),以基因组DNA为模板,通过PCR反应扩增出对应于已知cDNA区域的两个部分基因组序列。转录起始位点(Transcriptional start site,TSS)由Neural Network Promoter Prediction program (www.fruitfly.org/seq_tools/promoter.html)预测。随后,通过PCR扩增cDNA库来鉴定5′-末端片段中包含的cDNA序列。然后编辑cDNA和基因序列。使用Genetyx 6.0软件比对cDNA与基因组序列,确定外显子和内含子的结构。
|
|
表 1 本研究中所用的引物序列 Table 1 Sequences of primers used in the present study |
使用NCBI BLAST程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi),将SmTLR14的测序结果与GenBank数据库进行比较。使用ExPASy网站中的Translate工具(http://web.expasy.org/translate/)将核苷酸序列翻译为蛋白质序列。使用SignalP 4.1服务器(http://www.cbs.dtu.dk/services/SignalP)预测信号肽序列。根据Matsushima等描述的方法鉴定LRR[25]。使用SMART软件(http://smart.embl-heidelberg.de/)预测TM结构域和TIR结构域。使用Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行蛋白质序列的多重排列。在MEGA X中,利用邻接(Neighbor-joining,NJ)法和1 000次自举测试构建系统进化树。
1.5 实时荧光定量PCR用实时荧光定量PCR(Quantitative real-time PCR)研究SmTLR14 mRNA的组织分布,研究在LPS或PGN刺激的情况下该基因在免疫器官(鳃、头肾和脾脏)和非免疫器官(肌肉)中的表达情况。使用Superscript First Strand Synthesis System (Invitrogen,美国)将每个组织的1.0 μg总RNA反转录为cDNA。反应体系:10 μL荧光染料,0.2 μmol/L引物(正向/反向),1.0 μL cDNA模板(10 ng/μL),ddH2O补至20 μL。反应程序:95 ℃、10 min; 95 ℃、10 s,60 ℃、30 s,72 ℃、25 s,进行40个循环。在40个循环结束后进行熔解曲线分析,以确定PCR产物的特异性。所有样品均设置3个重复实验。在组织分布分析中,将RPSD基因作为内参基因,在基因表达分析中,将ACTB基因作为内参基因,根据2-ΔΔCT方法计算出相对于对照组中表达水平的倍数变化[26-27]。
1.6 pEGFP-C1-SmTLR14真核重组表达质粒及pcDNA3.1-SmTLR14重组过表达质粒的构建设计含有BamHⅠ(Takara,大连)和HindⅢ(Takara,大连)限制性酶切位点的引物(见表 1),通过PCR扩增SmTLR14的开放阅读框(Open reading frame,ORF)。扩增产物经琼脂糖凝胶回收试剂盒回收纯化,将PCR产物及pEGFP-C1载体(丰晖生物,长沙)用限制性内切酶BamHⅠ和HindⅢ进行双酶切,琼脂糖凝胶电泳检测并回收。随后将带有酶切位点的SmTLR14序列用T4连接酶连接到pEGFP-C1载体上,转化后,用菌落PCR及双酶切验证,送出测序,最终得到pEGFP-C1-SmTLR14真核重组表达质粒。过表达质粒是pcDNA3.1(+)(丰晖生物,长沙),酶切位点选的也是BamHⅠ和HindⅢ,所以用上述同样的方法构建出pcDNA3.1-SmTLR14重组过表达质粒。
1.7 TK细胞的培养大菱鲆肾(Turbot kindey,TK)细胞由中国水产科学研究院黄海水产研究所陈松林研究员赠送,细胞培养在含20%的胎牛血清(Gibco,美国)、1%的双抗(Solarbio,北京)及5 ng/mL的碱性成纤维细胞生长因子(STEMCELL,加拿大)的L-15培养基(Solarbio,北京)中,在24 ℃生化培养箱中培养,转染时,将细胞培养在24 ℃、5% CO2的培养箱中。
1.8 共聚焦实验在24孔板中放入爬片,将TK细胞按照每孔(2~3)×105个的标准接种在24孔培养板中。待细胞密度长到80%~90%时,用Lipofectamine 2000(Invitrogen,美国)进行转染,实验组转染0.5 μg pEGFP-C1-SmTLR14质粒,对照组转染0.5 μg pEGFP-C1空质粒。转染24 h后,将细胞用PBS漂洗两次,在室温下用4%多聚甲醛固定15~20 min,再用PBS漂洗2次。将爬片取出倒扣在滴有抗荧光淬灭封片液的载玻片上,封片后,用Leica SP8共聚焦显微镜观察。
1.9 SmTLR14过表达对下游免疫相关基因表达的影响提前一天将TK细胞按照每孔(1~2)×106个的标准接种在6孔培养板中,待细胞密度达到80%时转染,实验组转染2.5 μg pcDNA3.1-SmTLR14质粒,对照组转染2.5 μg pcDNA3.1空质粒。转染6 h后,用终浓度为50 μg/mL的LPS刺激细胞。刺激24 h后,参照1.2及1.3的方法提取RNA,合成cDNA。选取ACTB作为内参基因,并使用基因特异性引物(见表 1)对SmTLR14及其下游TNF-α、IL-1β、IL-8、IFN-1、IRF-1和IRF-3进行实时荧光定量PCR分析(方法参照1.5)。
2 结果 2.1 SmTLR14基因序列分析SmTLR14 cDNA(GenBank登录号:MH396410)长3 364 bp,包括165 bp的5′-非翻译区(UTR),ORF全长2 634 bp,编码877个氨基酸(Amino acid,aa),3′-UTR为565 bp,具有2个聚腺苷酸加尾序列(ATTAAA)。SmTLR14基因(GenBank登录号:MH396409)长12 708 bp,由4个外显子和3个内含子组成(见图 1(a))。TLR14和TLR18是同一分子,在低等真骨鱼中,TLR14通常称为TLR18。SmTLR14的基因结构与亚马逊帆鱼(Poecilia formosa)和新月鱼(Xiphophorus maculatu)的TLR18相同,与牙鲆和河豚的TLR14相同(见图 2)。推导的SmTLR14蛋白具有一个信号肽,长度为17 aa(1~17 aa),其后是细胞外LRR域(34~606 aa)、TM域(610~632 aa)和细胞内TIR域(665~810 aa)(见图 1(a)和(b))。在LRR域中,有23个LRR基序,其中有5个是典型的LRR(LRR-TYP)。LRR域的N端和C端分别含有一个LRRNT和LRRCT。TIR域含有3个保守的基序:box 1(YxxFxSY),box 2(LC-RD-PG)和box 3(FWxxL)(见图 3)。序列比对显示,SmTLR14与其它鱼类的相似性(见图 3)如下:牙鲆为84.2%,金鲳鱼为83.6%,斜带石斑鱼为83.5%,花鲈(Lateolabrax maculatus)为83.2%,日本银鱼(Argyrosomus japonicus)为82.1%,中国鱚(Sillago sinica)为82.0%,鮸鱼为81.7%。

|
( 外显子用大写字母表示,内含子用小写字母表示。点表示省略的序列。预测的信号肽(残基1~17)用粗体下划线表示。细胞外LRR域(残基34~606)中的LRR基序用灰色底色表示, N端和C端LRR基序中的保守半胱氨酸用单个方框表示。LRR域的N端和C端的保守半胱氨酸用方框表示。跨膜(TM)区(残基610~632)用虚下划线表示。TIR域(残基665~810)用方框表示,其中三个保守的基序用灰色底色表示。两个多聚腺苷酸化信号(ATTAAA)用下划波浪线表示。LRR通过Matsushima等描述的方法预测[25],TM域和TIR域由SMART程序预测。Exons are shown in uppercase, while introns in lowercase. Dots represent omitted sequences. The predicted signal peptide (residues 1~17) is shown with a bold underline. The LRR motifs in the extracellular LRR domain (residues 34~606) are indicated by a gray base color and the conservative cysteines at the N- and C- terminal LRR motiffs indicated by a single box. The transmembrane (TM) region (residues 610~632) is shown with a dashed underline. The TIR domain (residues 665~810) is boxed, in which three conserved motifs are noted with gray underlay. Two polyadenylation signals (ATTAAA) are underlined with wavy lines. The LRRs are predicted by the method described by Matsushima et al. [25], while the TM region and TIR domain by SMART program. ) 图 1 SmTLR14基因的核苷酸序列、氨基酸序列(a)以及结构域(b)的示意图 Fig. 1 Nucleotide and deduced amino acid sequences of SmTLR14 gene(a)and schematic representation of SmTLR14 domains(b) |
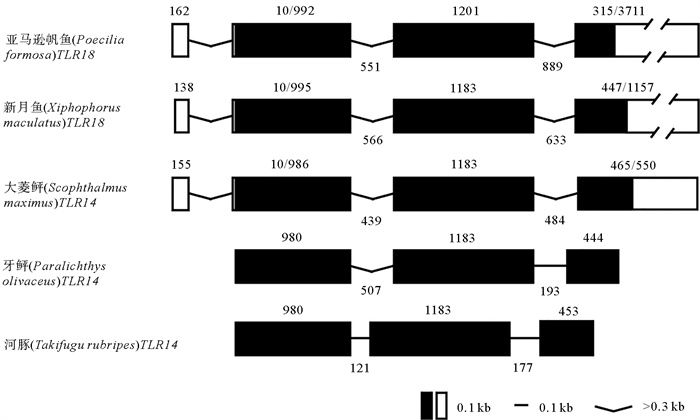
|
( 外显子用方框表示,内含子用直线或折线表示,数字表示外显子和内含子的长度,单位为bp。登录号:亚马逊帆鱼,ENSPFOG00000018309;新月鱼,ENSXMAG00000013367;河豚,AC156431.1;牙鲆,AB576806.1。Exons are indicated with boxes and introns with straight or concave lines. Numbers indicate the lengths of exons and introns in bp. Accession numbers: Poecilia formosa, ENSPFOG00000018309; Xiphophorus maculatus, ENSXMAG00000013367; Takifugu rubripes, AC156431.1; Paralichthys olivaceus, AB576806.1. ) 图 2 鱼类中已知的TLR14s和TLR18s的外显子/内含子结构比较 Fig. 2 Comparison of the exon/intron structures of known TLR14s and TLR18s in fish |
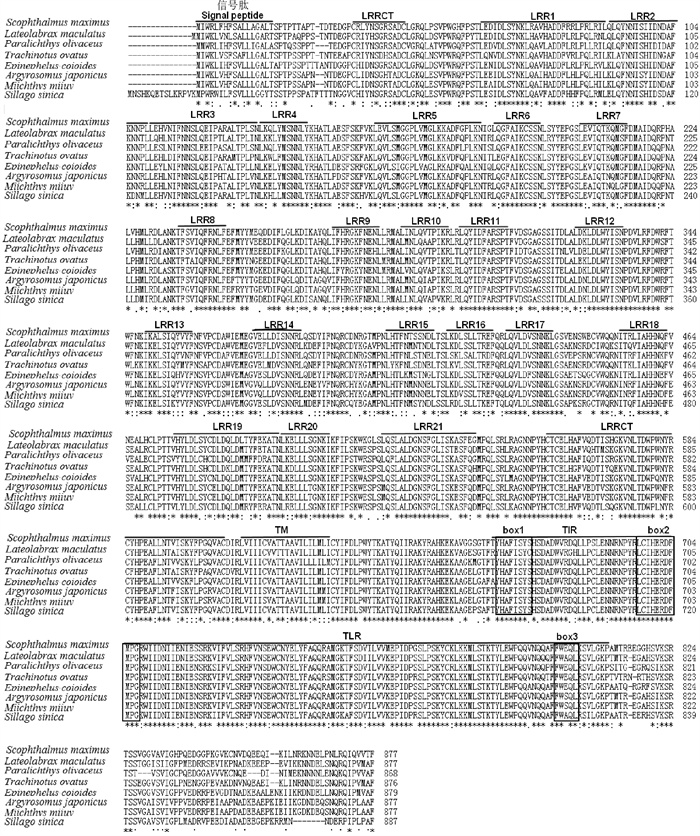
|
( 信号肽、LRRNT、21个LRR基序、LRRCT、TM域和TIR域用上划线标出。box结构用框标出。整个序列中完全保守的(100%)氨基酸残基用星号(*)表示,半保守的残基用双点(:)和点(.)表示。序列的登录号见表 2。The signal peptide, LRRNT, 21 LRR motifs, LRRCT, TM region and TIR domain are shown by overbars. Box structure is marked with a box. Completely conserved (100%) amino acid residues throughout sequences are depicted by asterisks (*), and semi-conserved residues are denoted by semicolons (: ) and dots (.). The accession numbers of the sequences are shown in Table 2. ) 图 3 SmTLR14与其他TLR14s的氨基酸多序列比对 Fig. 3 Multiple sequence alignment of SmTLR14 with other TLR14s |
|
|
表 2 本研究中所用到的蛋白登录号 Table 2 The protein accession number used in this study |
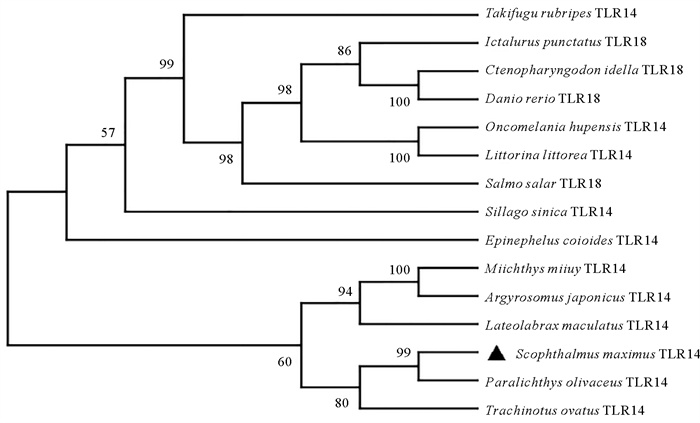
TLR14和TLR18是同一分子,采用MEGA X软件构建了不同物种TLR14/TLR18系统进化树(见图 4),结果显示SmTLR14蛋白与牙鲆、金鲳鱼、鮸鱼、日本银鱼及花鲈聚为一支,它们之间的同源性较高,其中和牙鲆的同源性最高。
|
(SmTLR14用黑色三角形表示。序列登录号见表 2。The SmTLR14 is highlighted with black triangle. The accession numbers of the sequences are shown in Table 2. ) 图 4 不同物种TLR14s/TLR18s的系统进化树 Fig. 4 Phylogenetic tree of TLR14s/TLR18s in different species |
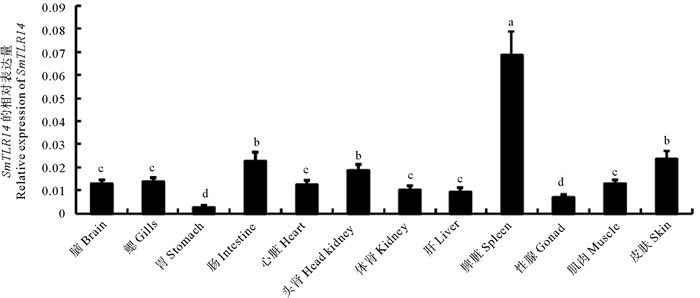
通过实时荧光定量PCR检测了SmTLR14 mRNA的组织特异性,选取RPSD作为内参基因,因为它是正常生理条件下跨组织类型最稳定的基因[26]。如图 5所示,SmTLR14在被检测的12种组织中都有表达,在脾脏中的表达量最高,在肠、头肾和皮肤中含量中等,在性腺和胃中含量较低。
|
( RPSD基因作为内参基因。值是平均值±标准误差(n=3)。不同小写字母标记表示差异显著(P<0.05)。RPSD gene as an internal reference gene. Values are means±S.E. (n=3). Values marked by different lowercase letters are significantly different from each other (P<0.05) ) 图 5 SmTLR14基因在健康大菱鲆不同组织中的表达情况 Fig. 5 Expression of SmTLR14 gene in different tissues of healthy turbot |
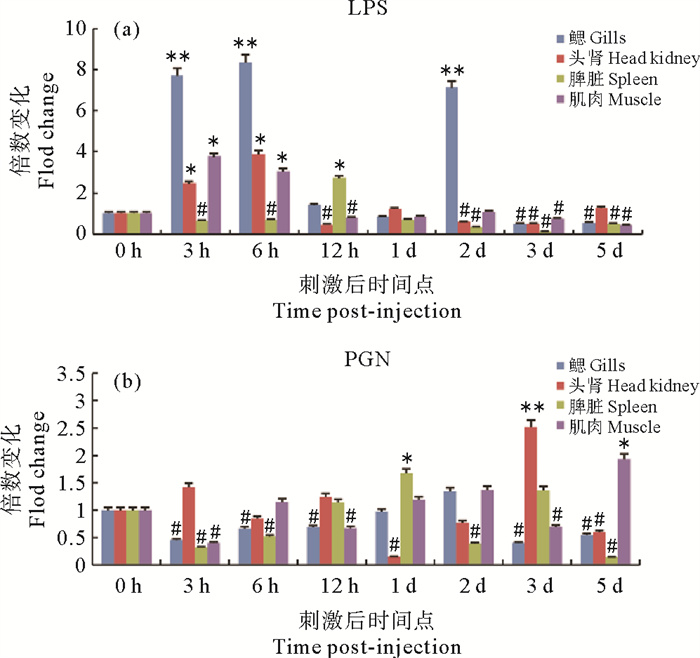
为了探究SmTLR14在大菱鲆机体免疫方面发挥的作用,用LPS及PGN刺激大菱鲆,用实时荧光定量PCR研究了SmTLR14在3个免疫器官(鳃、头肾和脾脏)和1个非免疫器官(肌肉)中不同时间点的表达。如图 6(a)所示,在受到LPS刺激后,鳃中SmTLR14 mRNA的表达在3 h(7.7倍)、6 h(8.3倍)和2 d(7.7倍)被显著上调。在头肾和脾脏中,分别在6 h(3.8倍)和12 h(2.7倍)处检测到最显著的诱导作用。在肌肉中,SmTLR14在3 h(3.7倍)和6 h(3.0倍)被显著诱导。大多数被检测的器官中,LPS在后期(3~5 d)下调了SmTLR14的表达。在PGN刺激的情况下(见图 6(b)),在实验的早期阶段(3和6 h)观察到鳃和脾脏中SmTLR14的表达下调,在1 d检测到脾脏中的第一个诱导表达峰(1.6倍),在鳃、头肾和肌肉中分别在2 d(1.3倍)、3 d(2.5倍)和5 d(1.9倍)检测到最高的表达。
|
( 每个时间点表示为三个重复的平均值±标准误差,n=3。与对照组相比,显著增加用*(P<0.05)和**(P<0.01)表示,显著减少用#(P<0.05)表示。Each time point is expressed as the mean of three replicates± standard error, n=3. The significant increase in expression level compared to the control is expressed by * (P<0.05) or ** (P<0.01)while the significant decrease in expression level compared to the control is expressed by #(P<0.05) ) 图 6 LPS(a)和PGN(b)刺激大菱鲆后SmTLR14在鳃、头肾、脾脏和肌肉中的倍数变化 Fig. 6 Fold changes of SmTLR14 expression in the gills, head kidney, spleen and muscle of turbots stimulated with LPS (a), and PGN (b) |
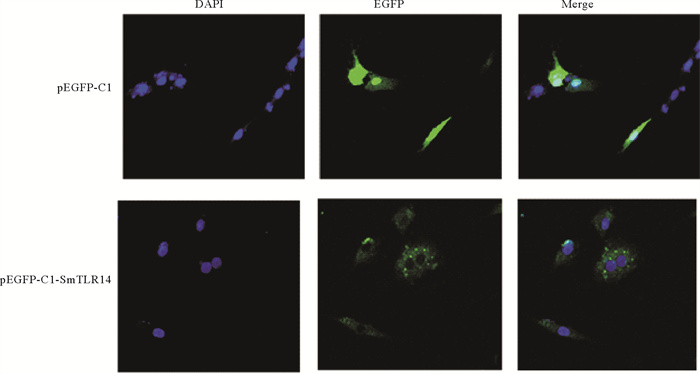
为确定SmTLR14的定位,将pEGFP-C1-SmTLR14表达载体和pEGFP-C1空载体分别转染到TK细胞中,结果如图 7所示,SmTLR14蛋白分布在细胞质中。
|
(pEGFP-C1是空载体阴性对照,pEGFP-C1-SmTLR14是目的基因融合蛋白。DAPI是染色细胞核(蓝色荧光),EGFP是标记蛋白(绿色荧光),Merge是二色合并。pEGFP-C1 is an empty vector negative control, pEGFP-C1-SmTLR14 is the target gene fusion protein, DAPI stains the nucleus (blue fluorescence), EGFP labeled protein (green fluorescence), Merge stands for two-color merge. ) 图 7 SmTLR14在TK细胞中的定位 Fig. 7 Localization of SmTLR14 in TK cells |
为了探究大菱鲆TLR14基因在免疫途径中的功能,将pcDNA3.1-SmTLR14过表达质粒及pcDNA3.1空质粒分别转染TK细胞。用实时荧光定量PCR检测SmTLR14过表达对下游免疫相关基因的影响。结果发现,过表达SmTLR14后,其表达水平与对照组相比显著上调了13倍(见图 8(a))。与对照组相比,炎性因子TNF-α(见图 8(b))、IL-1β(见图 8(c))和IL-8(见图 8(d))分别显著上调了16、176和407倍。抗病毒因子IFN-1(见图 8(e))、IRF-1(见图 8(f))和IRF-3(见图 8(g))分别轻微上调了1.3、1.5和1.2倍,远低于炎症因子的上调水平。

|
(pcDNA3.1表示空载体组,pcDNA3.1-SmTLR14表示过表达组。(a) SmTLR14;(b) TNF-α; (c) IL-1β; (d) IL-8;(e) IFN-1;(f) IRF-1;(g) IRF-3。ACTB作为内参基因,空载体组(对照组)的表达水平设为1。每个基因的值表示为3个重复的平均值±标准误差,n=3。**(p<0.01)表示实验组与对照组的差异显著。pcDNA3.1 represents the empty vector group. pcDNA3.1-SmTLR14 represents the overexpression group. (a) SmTLR14; (b) TNF-α; (c) IL-1β; (d) IL-8; (e) IFN-1; (f) IRF-1; (g) IRF-3. ACTB is used as an internal reference gene, the expression level of the empty vector group (control group) is set to 1. The value of each gene is expressed as the mean of three replicates ± standard error, n=3. **(P<0.01)was used to indicate significant differences between the experimental group and the control group. ) 图 8 SmTLR14过表达对下游免疫相关因子转录水平的影响 Fig. 8 Effect of SmTLR14 overexpression on the transcription level of downstream immune-related factors |
TLR14是非哺乳动物受体,是TLR1家族的成员。本研究克隆了大菱鲆TLR14基因:SmTLR14 cDNA(GenBank登录号:MH396410)长3 364 bp,ORF全长2 634 bp,编码877个氨基酸。SmTLR14蛋白结构包括胞外LRR域、TM域和细胞内TIR域。LRR可以形成马蹄状,在识别PAMPs时起关键作用[28]。不同TLRs胞外区LRRs数量不同,识别的PAMP也不同。SmTLR14胞外含有23个LRR,通过α螺旋和β折叠形成马蹄形,负责识别PAMPs。LRR和LRR-TYP基序的高度保守片段(Highly conserved sequence,HCS)分别是“LxxLxLxxNxL”和“LxxLxLxxNxLxxLxxxxF/LxxLxx”[25]。与其他TLR1家族成员相同,
SmTLR14还显示LRR结构域的中心部分具有更不规则的LRR,其HCS 9位上的天冬酰胺被疏水残基替代(见图 3)[25]。保守的天冬酰胺对于维持胞外域的整体形状很重要,因此缺乏天冬酰胺可能会导致LRR域中间结构发生改变[29]。先前对TLR1-TLR2-脂肽复合物晶体结构的研究表明,LRR结构域的中间结构转变形成疏水口袋,这是TLR1-TLR2异二聚体的主要配体结合区[30-31]。因此,SmTLR14 LRR域的中间部分可能在该功能中起关键作用。另外,位于N端和C端的LRRNT和LRRCT也含有保守的半胱氨酸基序,分别为Cx10C和CxCx25Cx20C(见图 1和3)[25]。胞内TIR结构域对于信号传导至关重要,SmTLR14含有3个保守的基序,分别标记为box 1(YxxFxSY)、box 2(LC-RD-PG)和box 3(FWxxL)。box 1、box 2涉及信号转导,而box 3与受体的定位有关[32]。系统进化分析显示SmTLR14和牙鲆TLR14进化关系最近。在哺乳动物中,TLR2可以与TLR1、TLR6、TLR10形成异源二聚体以识别病原体[33]。但是,在鱼类中未发现TLR6和TLR10基因[8],因此TLR14可能与TLR2结合,发挥哺乳动物TLR6和TLR10的类似功能。但是,要全面了解TLR14的功能,还需要进一步研究。
SmTLR14的组织分布呈现泛在性表达的特点,在脾脏中的表达量最高,因为脾脏是富含淋巴样细胞的主要免疫组织之一,所以SmTLR14可能在脾脏中发挥特定功能。细菌是鱼类疾病的病原体之一,TLR在抗菌宿主防御中起重要作用[34]。在本研究中,使用LPS和PGN刺激大菱鲆模仿革兰氏阴性菌和革兰氏阳性菌的感染。在LPS刺激3和6 h后,大菱鲆鳃中SmTLR14出现显著上调。在斜带石斑鱼TLR14的研究中发现,在受到白点虫(Ichthyophthirius multifiliis)感染1 d后,斜带石斑鱼鳃中TLR14 mRNA的表达也出现了显著增加(约10倍)[12]。鳃是硬骨鱼的主要黏膜组织,TLR14在鳃中的快速高表达,表明其在LPS和寄生虫感染的免疫反应中起着重要的作用。从时间上看,在LPS刺激之后的早期,SmTLR14会出现显著上调,到了后期则不变或受到抑制。TLR14受到抑制的原因有很多。研究表明,在受到鳗弧菌感染的鮸鱼体内,miRNA-122可以下调TLR14 mRNA的表达[13]。在PGN刺激后,在早期SmTLR14并没有出现显著上调,而是保持不变或受到抑制。先前的研究还显示,在牙鲆中,与其他病原体感染相比,TLR14在针对革兰氏阴性细菌感染的免疫反应中参与得更多[11],这与本文结果一致。由此可以得出,SmTLR14可能识别LPS,并且在革兰氏阴性菌感染的免疫反应中发挥着重要的功能。
本研究发现,SmTLR14定位在细胞质中。在硬骨鱼中,TLR14和TLR25都是TLR1亚家族的成员。研究发现尼罗罗非鱼(Oreochromis niloticus)中的TLR25也位于细胞质中[35]。研究表明,TLR的亚细胞定位会在刺激后发生变化,例如TLR5在人的肠细胞系HT-29中定位在细胞质,但在大肠杆菌(Escherichia coli)感染后,TLR5则定位在细胞表面,这说明在大肠杆菌的刺激下,TLR5的定位会发生变化[36]。据报道,鱼类的TLR14可以识别细菌的成分,介导鱼类机体的免疫反应[11, 13, 15]。在细菌感染下,SmTLR14的定位会不会发生改变需要进一步探究。
TNF-α、IL-1β、IL-8和TNF-α是在炎症反应过程中最早出现发挥作用的炎性因子,它们能激活免疫细胞使其发挥功能,增加血管内皮细胞的通透性,并调节其他组织的生化过程,从而促进活性因子的产生和释放。在IL-1家族中,最关键的前体因子是IL-1β,而IL-1有强大的促炎作用[37]。IL-8在消除中性粒细胞中存活的细菌上发挥着重要的作用[38]。IFN-1、IRF-1和IRF-3是抗病毒因子。IFN-1是干扰素家族中第一个被发现的,它具有广泛的功能,例如抗病毒活性、调节免疫细胞的成熟和分化以及诱导细胞凋亡[39]。IRF的主要功能是在机体受到病毒侵袭时刺激干扰素的产生[40]。IRF-3可以诱导IFN-1,IRF-1可以与IRF-3、IRF-7细胞因子相互作用来参与抗病毒反应[39, 41]。在LPS刺激后,TK细胞中SmTLR14的过表达显著上调TNF-α、IL-1β和IL-8,但对IFN-1、IRF-1和IRF-3的诱导不明显,表明大菱鲆TLR14可能通过识别细菌组分,从而激活相关的免疫通路来诱导炎症相关因子的表达,介导机体的抗细菌天然反应,但具体机制还需要进一步研究。
4 结语本研究克隆了大菱鲆TLR14基因的全长序列。组织表达分析表明,SmTLR14在组织中呈现泛在性表达,并在脂多糖感染后出现显著诱导; 亚细胞定位表明,SmTLR14定位大菱鲆肾细胞的细胞质; 过表达实验表明,SmTLR14在抗菌免疫中起着重要作用。
| [1] |
Beutler B A. TLRs and innate immunity[J]. Blood, 2008, 113(7): 1399-1407. (  0) 0) |
| [2] |
Ray A, Cot M, Puzo G, et al. Bacterial cell wall macroamphiphiles: Pathogen-/microbe-associated molecular patterns detected by mammalian innate immune system[J]. Biochimie, 2013, 95(1): 33-42. DOI:10.1016/j.biochi.2012.06.007 (  0) 0) |
| [3] |
Masahiro Y, Kiyoshi T. Current views of Toll-like receptor signaling pathways[J]. Gastroenterology Research and Practice, 2010, 2010: 1-8. (  0) 0) |
| [4] |
Bell J K, Mullenb G, Leifer C A, et al. Leucine-rich repeats and pathogen recognition in Toll-like receptors[J]. Trends in Immuno-logy, 2003, 24(10): 528-533. DOI:10.1016/S1471-4906(03)00242-4 (  0) 0) |
| [5] |
Werling D, Jann O C, Offord V, et al. Variation matters: TLR structure and species-specific pathogen recognition[J]. Trends in Immunology, 2009, 30(3): 124-130. DOI:10.1016/j.it.2008.12.001 (  0) 0) |
| [6] |
Liew F, Xu D, Brint E, et al. Negative regulation of Toll-like receptor mediated immune responses[J]. Nature Reviews Immunology, 2005, 5(6): 446-458. DOI:10.1038/nri1630 (  0) 0) |
| [7] |
Wang Y J, Li J R, Han J J, et al. Identification and characteristic analysis of TLR28: A novel number of the TLR1 family in teleost[J]. Developmental and Comparative Immunology, 2016, 62: 102-107. DOI:10.1016/j.dci.2016.05.001 (  0) 0) |
| [8] |
Roach J C, Glusman G, Rowen L, et al. The evolution of vertebrate Toll-like receptors[J]. Proceedings of the National Academy of Sciences, 2005, 102(27): 9577-9582. DOI:10.1073/pnas.0502272102 (  0) 0) |
| [9] |
Ishii A, Kawasaki M, Matsumoto M, et al. Phylogenetic and expression analysis of amphibian Xenopus Toll-like receptors[J]. Immunogenetics, 2007, 59(4): 281-293. DOI:10.1007/s00251-007-0193-y (  0) 0) |
| [10] |
Ishii A, Matsuo A, Sawa H, et al. Lamprey TLRs with properties distinct from those of the variable lymphocyte receptors[J]. Journal of Immunology, 2007, 178(1): 397-406. DOI:10.4049/jimmunol.178.1.397 (  0) 0) |
| [11] |
Hwang S D, Kondo H, Hirono I. Molecular cloning and characterization of Toll-like receptor 14 in Japanese flounder, Paralichthys olivaceus[J]. Fish & Shellfish Immunology, 2011, 30(1): 425-429. (  0) 0) |
| [12] |
Li Y W, Xu D D, Xia L, et al. Identification and characterization of three TLR1 subfamily members from the orange-spotted grouper, Epinephelus coioides[J]. Developmental and Comparative Immunology, 2016, 61: 180-189. DOI:10.1016/j.dci.2016.03.028 (  0) 0) |
| [13] |
Cui J X, Chu Q, Xu T J. miR-122 involved in the regulation of Toll-like receptor signaling pathway after Vibrio anguillarum infection by targeting TLR14 in miiuy croaker[J]. Fish & Shellfish Immunology, 2016, 58: 67-72. (  0) 0) |
| [14] |
韩崇. 大刺鳅线粒体基因组及基于转录组的免疫基因发掘[D]. 广州: 中山大学生命科学学院, 2018. Han C. The Complete Mitochondrial Genome of Mastacembelus armatus and Development of Immune Genes Based on Transcriptome[D]. Guangzhou: School of Life Sciences, Sun Yat-sen University, 2018. (  0) 0) |
| [15] |
Meng W, Guo L, Zhu K C, et al. Molecular characterization of Toll-like receptor 14 from golden pompano Trachinotus ovatus (Linnaeus, 1758) and its expression response to three types of pathogen-associated molecular patterns[J]. Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2019, 232: 1-10. (  0) 0) |
| [16] |
向军喜. 黄姑鱼经杀香鱼假单胞菌和Poly Ⅰ: C刺激后的病理特征及免疫相关基因表达特征分析[D]. 舟山: 浙江海洋大学海洋科学与技术学院, 2020. Xiang J X. Characterization of Pathological Changes and Immune-Related Genes' Expression in Yellow Drum (Nibea albiflora) in Response to Pseudomonas plecoglossicida and Poly Ⅰ: C Challenges[D]. Zhoushan: School of Marine Science and Technology, Zhejiang Ocean University, 2020. (  0) 0) |
| [17] |
Wang K L, Chen S N, Huo H J, et al. Identification and expression analysis of sixteen Toll-like receptor genes, TLR1, TLR2a, TLR2b, TLR3, TLR5M, TLR5S, TLR7-9, TLR13a-c, TLR14, TLR21-23 in mandarin fish Siniperca chuatsi[J]. Develo-pmental and Comparative Immunology, 2021, 121: 104100. DOI:10.1016/j.dci.2021.104100 (  0) 0) |
| [18] |
Brodská H, Malíčková K, Adámková V, et al. Significantly higher procalcitonin levels could differentiate Gram-negative sepsis from Gram-positive and fungal sepsis[J]. Clinical and Experimental Medicine, 2013, 13(3): 165-170. DOI:10.1007/s10238-012-0191-8 (  0) 0) |
| [19] |
Buwitt-Bec  0) 0) |
| [20] |
Zhang H Y, Hu G B, Liu Q M, et al. Cloning and expression study of a Toll-like receptor 2 (tlr2) gene from turbot, Scophthalmus maximus[J]. Fish & Shellfish Immunology, 2016, 59: 137-148. (  0) 0) |
| [21] |
Hu G B, Li X P, Liu D H, et al. A Toll-like receptor 3 homolog that is up-regulated by poly Ⅰ: C and DNA virus in turbot, Scophthalmus maximus[J]. Journal of Fish Biology, 2015, 86(2): 431-447. DOI:10.1111/jfb.12559 (  0) 0) |
| [22] |
Liu D H, Chen J J, Li S, et al. Molecular cloning and expression study on Toll-like receptor 5M in turbot, Scophthalmus maximus[J]. Developmental and Comparative Immunology, 2018, 85: 44-50. DOI:10.1016/j.dci.2018.03.020 (  0) 0) |
| [23] |
Li S, Wang G J, Liu D H, et al. Cloning and expression analysis of a Toll-like receptor 21 (TLR21) gene from turbot, Scophthalmus maximus[J]. Developmental and Comparative Immunology, 2017, 73: 163-168. DOI:10.1016/j.dci.2017.03.021 (  0) 0) |
| [24] |
Hu G B, Zhang S F, Yang X, et al. Cloning and expression analy-sis of a Toll-like receptor 22 (TLR22) gene from turbot, Scophthalmus maximus[J]. Fish & Shellfish Immunology, 2015, 44: 399-409. (  0) 0) |
| [25] |
Matsushima N, Tanaka T, Enkhbayar P, et al. Comparative sequence analysis of leucine-rich repeats (LRRs) within vertebrate Toll-like receptors[J]. BMC Genomics, 2007, 8(1): 124. DOI:10.1186/1471-2164-8-124 (  0) 0) |
| [26] |
Dang W, Sun L. Determination of internal controls for quantitative real time RT-PCR analysis of the effect of Edwardsiella tarda infection on gene expression in turbot (Scophthalmus maximus)[J]. Fish & Shellfish Immunology, 2011, 30(2): 720-728. (  0) 0) |
| [27] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2002, 25: 402-408. (  0) 0) |
| [28] |
Jin M S, Lee J O. Structures of the toll-like receptor family and its ligand complexes[J]. Immunity, 2008, 29(2): 182-191. DOI:10.1016/j.immuni.2008.07.007 (  0) 0) |
| [29] |
Wang J L, Zhang Z, Liu J, et al. Structural characterization and evolutionary analysis of fish-specific TLR27[J]. Fish & Shellfish Immunology, 2015, 45(2): 940-945. (  0) 0) |
| [30] |
Kang J Y, Lee J O. Structural biology of the Toll-like receptor family[J]. Annual Review of Biochemistry, 2011, 80(1): 917-941. DOI:10.1146/annurev-biochem-052909-141507 (  0) 0) |
| [31] |
Jin M S, Kim S E, Heo J Y, et al. Crystal structure of the TLR1-TLR2 heterodimer induced by binding of a tri-acylated lipopeptide[J]. Cell, 2007, 130(6): 1071-1082. DOI:10.1016/j.cell.2007.09.008 (  0) 0) |
| [32] |
Slack J L, Schooley K, Bonnert T P, et al. Identification of two major sites in the type Ⅰ interleukin-1 receptorcytoplasmic region responsible for coupling to pro-inflammatory signaling pathways[J]. Journal of Biological Chemistry, 2000, 275(7): 4670-4678. DOI:10.1074/jbc.275.7.4670 (  0) 0) |
| [33] |
Guan Y, Ranoa D R, Jiang S, et al. Human TLRs 10 and 1 share common mechanisms of innate immune sensing but notsignaling[J]. Journal of Immunology, 2010, 184(9): 5094-5103. DOI:10.4049/jimmunol.0901888 (  0) 0) |
| [34] |
Medzhitov R. Toll-like receptors and innate immunity[J]. Nature Reviews Immunology, 2001, 1(2): 135-145. DOI:10.1038/35100529 (  0) 0) |
| [35] |
Lee P T, Ho T H, Bao T N, et al. Expression profile, subcellular localization and signaling pathway analysis of fish-specific TLR25 in Nile tilapia (Oreochromis niloticus)[J]. Fish & Shellfish Immunology, 2020, 104: 141-154. (  0) 0) |
| [36] |
Salazar-Gonzalez H, Navarro-Garcia F. Intimate adherence by enteropathogenic Escherichia coli modulates TLR5 localization and proinflammatory host response in intestinal epithelial cells[J]. Scandinavian Journal of Immunology, 2011, 73(4): 268-283. DOI:10.1111/j.1365-3083.2011.02507.x (  0) 0) |
| [37] |
Wang Y, Wang Q, Baoprasertkul P, et al. Genomic organization, gene duplication, and expression analysis of interleukin-1 beta in channel catfish (Ictalurus punctatus)[J]. Molecular Immunology, 2006, 43(10): 1653-1664. DOI:10.1016/j.molimm.2005.09.024 (  0) 0) |
| [38] |
Harada A, Sekido N, Akahoshi T, et al. Essential involvement of interleukin-8 (IL-8) in acute inflammation[J]. Journal of Leukocyte Biology, 1994, 56(5): 559-564. DOI:10.1002/jlb.56.5.559 (  0) 0) |
| [39] |
金丽, 苏强, 周建华, 等. Ⅰ型和Ⅲ型干扰素抗病毒作用机制的研究进展[J]. 中国生物制品学杂志, 2021, 34(4): 481-486. Jin L, Su Q, Zhou J H, et al. Research progress on the antiviral mechanism of typeⅠand type Ⅲ interferons[J]. Chinese Journal of Biologicals, 2021, 34(4): 481-486. (  0) 0) |
| [40] |
Fenton S E, Saleiro D, Platanias L C. Type Ⅰ and Ⅱ interferons in the anti-tumor immune response[J]. Cancers, 2021, 13(5): 1037. DOI:10.3390/cancers13051037 (  0) 0) |
| [41] |
Honda K, Takaoka A, Taniguchi T. Type Ⅰ interferon gene induction by the interferon regulatory factor family of transcription factors[J]. Immunity, 2006, 25: 349-360. DOI:10.1016/j.immuni.2006.08.009 (  0) 0) |
2. Institute of Evolution & Marine Biodiversity, Ocean University of China, Qingdao 266003, China
 2022, Vol. 52
2022, Vol. 52