2. 青岛海洋科学与技术国家试点实验室,海洋生物学与生物技术功能实验室,山东 青岛 266237
对于大多数生物而言,先天免疫系统是抵御病原体入侵的第一道防线。先天性免疫的关键步骤是识别传染原所产生病原体的相关分子模式(PAMP)[1],该过程中,TLR、TNFR等是保守的参与先天免疫反应的关键跨膜受体,不同的受体可特异性识别不同的PAMP,并激活相应的信号转导途径启动免疫应答[2]。肿瘤坏死因子受体相关因子TRAFs是广泛的受体家族的信号转导因子,可与TLRs,TNFRs,以及IL-1,IL-17,NF-κB,MAPKs,TGFβ等通路的跨膜受体相结合,在调控先天性免疫信号转导、炎症反应、细胞凋亡等方面发挥着重要作用[3-9]。
TRAFs最初是在哺乳动物中鉴定出来的,后来被发现存在于其他多个物种中,包括鱼类、两栖类以及无脊椎动物如昆虫和线虫等[10-12]。TRAFs蛋白进化上高度保守,其主要特征是N端具有RING指和锌指结构域,这两个结构域在许多E3泛素连接酶被发现,构成了泛素连接酶催化结构域的核心,是参与信号转导的重要结构域。研究表明,大多数TRAF能够作为E3泛素连接酶活化的下游信号通路蛋白,参与调控NF-κBs、丝裂原活化蛋白激酶(MAPK)或干扰素调节因子(IRF)的活化[13-14]。不同TRAF成员之间存在功能差异,例如,TRAF1被发现是一种重要的衔接蛋白,与免疫力调节、炎症和细胞死亡等相关[15]。TRAF2则参与调控NF-κB、JNK等通路激活,在细胞增殖中发挥了重要作用[16],缺乏TRAF2的小鼠表现出免疫器官的萎缩,其造血谱系细胞对TNF诱导的细胞死亡高度敏感[17]。TRAF3蛋白可发挥B细胞肿瘤抑制剂的作用,参与调控B细胞的稳态与存活[18]。TRAF4-TRAF7在TLR等免疫相关信号传导中均发挥着重要作用,可参与抗细菌免疫应答;其中,TRAF6不仅是TNFR和Toll /白细胞介素1受体家族的信号传导的调节剂,还可通过IKK复合物激活NF-κB通路[19-22]。综上,每个TRAF家族成员都发挥着不可或缺的非冗余生理作用、在先天性和适应性免疫、胚胎发育、组织稳态、应激反应等过程中发挥着关键调控作用[7, 23-24]。
目前,仅在少数软体动物中对TRAFs中的某些成员或TRAF基因家族进行了鉴定,如太平洋牡蛎的TRAF6[25],珍珠贝的TRAF3,虾夷扇贝的TRAF家族[26-27]。虾夷扇贝的研究发现,TRAF基因可发挥保守的参与激活下游NF-κB等信号转导的免疫调节作用[27]。侏儒蛤(M. lateralis)是一种小型双壳贝类,发育、生长快速、繁殖周期短且存活力强,对该物种开展TRAF基因家族免疫相关研究可以为更好的理解双壳贝类的免疫调控提供参考。因此,本研究对侏儒蛤进行了全基因组TRAF基因筛查,共鉴定出五个TRAF基因,分别为MlTRAF2、MlTRAF3、MlTRAF4、MlTRAF6和MlTRAF7。通过亲缘关系比对、系统发生分析以及时空表达模式研究,并利用成体健康侏儒蛤进行鳗弧菌侵染实验,进一步探索各TRAF成员在贝类生长发育中的作用以及免疫应答模式,为更好的理解贝类TRAF基因家族的进化、功能差异以及免疫调控提供参考。
1 材料和方法 1.1 MlTRAFs基因的全基因组筛查与鉴定利用NCBI(http://www.ncbi.nlm.nih.gov)数据库和Ensembl(http://useast.ensembl.org)数据库,下载包括人(Homo sapiens)、鼠(Mus musculus)、虾夷扇贝(Mizuhopecten yessoensis)和栉孔扇贝(Azumapecten farreri)等在内的13个物种的同源TRAF序列。利用这些TRAF同源蛋白序列,对侏儒蛤的转录组和基因组(未发表的数据库)进行检索,筛查和鉴定MlTRAFs基因。首先通过TBLASTN获得侏儒蛤转录组MlTRAFs序列[28],然后通过BLASTN比较转录组与全基因组序列,对所获得TRAF基因的cDNA序列进行验证,利用侏儒蛤基因组注释使用IBS作图来确定MlTRAFs基因结构;使用ORF(开放阅读框)探测器(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)和DNAstar(7.1版)预测氨基酸序列,并通过BLASTP从NCBI蛋白质序列数据库确认预测氨基酸序列。使用简单的模块化架构研究工具(SMART)识别MlTRAF蛋白的保守结构域(http://smart.embl.de/),并进一步使用Compute pI/Mw工具(http://web.expasy.org/compute_pi/)计算MlTRAF蛋白的等电点(pI)和分子量;使用Geneious 7.1.7 (http://www.geneious.com/)预测侏儒蛤TRAF蛋白的二级结构;使用Phyer2(http://www.sbg.bio.ic)预测蛋白三级结构。
1.2 系统发生分析从NCBI和Ensembl Genome Browser数据库中将来自人(H. sapiens)、鼠(M. musculus)、虾夷扇贝(M. yessoensis)和栉孔扇贝(A. farreri)等13个物种的72条序列下载获得,结合侏儒蛤MlTRAFs氨基酸序列进行系统发生分析,利用MEGA7的NJ法构建进化树,Bootstrap法进行重复检验,重复1 000次[29],比较不同物种的TRAF亚家族数目。
1.3 MlTRAFs的时空表达分析利用侏儒蛤各发育时期(受精卵、2~4细胞期、8~16细胞期、多细胞期、囊胚、原肠、担轮、D型幼虫、壳顶幼虫、眼点幼虫、稚贝)以及各成体组织(肝胰腺、雌雄性腺、足、闭壳肌、外套膜)的转录组数据计算出MlTRAFs基因表达量TPM(Trans Per Kilobase of exon model per Million mapped reads),公式为TPM=(Ni/Li×106)/sum(N1/L1+N2/L2+…+Nn/Ln), 式中:Ni:比对到第i个exon的reads数,Li:第i个exon的长度,sum(N1/L1+N2/L2+…+Nn/Ln):所有(n个)exon按长度进行标准化之后数值的和。利用R软件作图,分析MlTRAFs各成员的时空表达模式。
1.4 内参基因筛查和鳗弧菌侵染实验参考本实验前期已发表的虾夷扇贝的内参基因筛选[30],选取包含ACTB、CTC、TBP、RPL13a、TUB、His3.3、GAPDH、UBQ、CYP、RS23、EF1b、EIF3F、SELR1、NDUS4在内的14个基因作为侏儒蛤的候选内参基因。利用侏儒蛤鳃、性腺、足、肝胰腺、闭壳肌、外套膜、全组织等成体组织转录组数据计算出候选内参基因表达量(TPM)。最终,根据候选内参基因表达水平以及在不同组织的表达变化筛选出5个最优内参候选基因,分别是TBP、GAPDH、RS23、EF1B、NDUS4,进一步通过qPCR在外套膜、肝胰腺、肉柱、鳃、足中验证(引物序列见表 1),利用geNorm、NormFinder和delta-Ct这3种统计算法验证内参的稳定性[31-33]。
|
|
表 1 侏儒蛤MlTRAFs以及候选内参引物序列 Table 1 MlTRAFs and candidate primer sequences in Mulinia lateralis |
利用3月龄健康成体侏儒蛤进行革兰氏阴性细菌鳗弧菌(V. anguillarum)侵染实验。如Cong等[34]所述,将鳗弧菌终浓度调节至1×107 CFU/mL用于进行侵染实验。挑选健康侏儒蛤成体150只,随机分成对照组与实验组,在利用鳗弧菌侵染后0、3、6、12、24 h进行取样。取样时,从每组中随机选取10个个体,5个个体用于解剖后收集肝胰腺、雌性腺、雄性腺样品,其余5个个体直接摘取全组织,组织分离后立即利用液氮冷冻,-80 ℃冷冻保存至RNA提取等[35]。
1.5 RNA提取和定量实时PCR分析按照本实验室前期发表的方法提取总RNA [36]后,使用莫洛尼鼠白血病病毒(MMLV)逆转录酶(Thermo,USA)合成第一链cDNA用作实时PCR的模板,引物序列见表 1。利用LightCycler480进行实时定量PCR分析(Roche Diagnostics公司,曼海姆,德国)[37],选取最优内参RS23作为鳗弧菌侵染实验中qPCR分析的内参基因,所有引物设计均使用Primer Premier5.0软件,所设计引物首先通过BLASTN(1×10-10)对比侏儒基因组(未发表的数据)来评估特异性,并进一步通过溶解曲线验证。使用相对表达软件工具(REST)2009版本分析实时PCR的数据,然后基于对照组计算鳗弧菌侵染后各实验样品的相对表达水平,使用SPSS进行数据的统计分析(16.0版本),使用独立t检验的统计软件包,进而分析各MlTRAF各成员的表达变化。
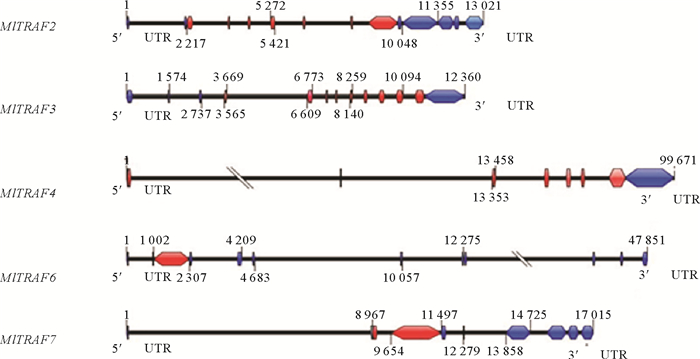
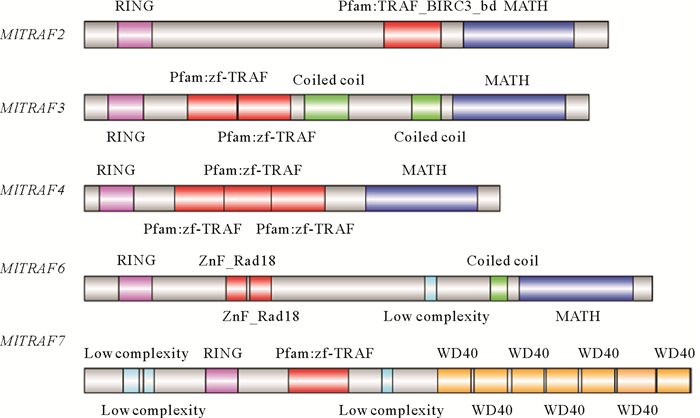
2 结果 2.1 侏儒蛤MlTRAFs全基因组鉴定及其系统发生分析侏儒蛤中成功鉴定出5个TRAF基因,分别为MlTRAF2、MlTRAF3、MlTRAF4、MlTRAF6和MlTRAF7(见表 2),其开放阅读框分别为1 758、1 686、1 392、1 305和2 031 bp,分别含有7、8、7、1、3个外显子并分别编码586、562、464、435和677个氨基酸(见图 1)。这些MlTRAFs的预测分子量范围:54.033~75.606 kDa,预测的等电点(pI)范围:5.63~7.15,通过Geneious7.0.6推测MlTRAF2、MlTRAF3、MlTRAF4、MlTRAF6和MlTRAF7的二级结构,这些蛋白分别含有27、26、18、27和27个α螺旋,30、32、33、30和42个β链,44、30、32、44和40个线圈(见图 2、表 2)。虽然侏儒蛤TRAF基因的结构多样,但均含有进化上保守的结构域,这与其他物种中发现的一致。例如,MlTRAF2、MlTRAF3、MlTRAF4、MlTRAF6在C末端均含有一个经典的MATH结构域,而MlTRAF7的C末端则含有7个重复的WD40结构域。在5个MlTRAF成员中,MlTRAF6、MlTRAF7含有低复杂性区域(LCRs),这与其他家族成员不同(见图 3)。
|
|
表 2 侏儒蛤MlTRAFs基因家族总结 Table 2 Summary of MlTRAFs gene family in Mulinia lateralis |
|
( 蓝色框表示3′UTR和5′UTR; 红色框表示外显子,单杠表示内含子。The blue boxes indicate 3′UTR and 5′UTR. The red box indicates the exon, and the horizontal bar indicates the intron. ) 图 1 侏儒蛤MlTRAFs基因结构 Fig. 1 Structure of MlTRAFs gene in Mulinia lateralis |

|
( 粉色的圆柱代表α螺旋; 橙色的直线箭头代表β螺旋; 波浪线表示无规则卷曲数;弯曲的箭头代表转角数。The pink cylinder represents the alpha helix; the orange straight arrow represents the beta chain; the wavy line represents the coil; and the curved arrow represents the turn. ) 图 2 侏儒蛤MlTRAFs二级结构以及三级结构预测 Fig. 2 Secondary and tertiary structure prediction of MlTRAFs in Mulinia lateralis |
|
( 水平的灰色条表示没有预测功能域的氨基酸序列,彩色框表示具有成功预测域的区域。带注释的域:RING: RING域(粉色); TRAF _BIRC3_bd:TNF受体相关因子BIRC3结合域;zf-TRAF:锌指结构域(红色);ZnF-TRAF:类似于Rad18的CCHC锌指结构域;Colled coil:缠绕线圈(绿色);MATH: TRAF结构域(蓝色);Low complexity: 低复杂序列(青色)和WD40: WD40结构域(橘黄色)。The horizontal gray bar indicates the amino acid sequence without prediction domain, and the color box indicates the region with successful prediction domain. Annotated fields: RING: RING (pink); TRAF_ BIRC3_ BD: the binding domain of TNF receptor related factor birc3; ZF_TRAF: zinc finger domain (red); ZNF_Rad18: CCHC zinc finger domain similar to Rad18; Colled coil: winding coil (green); MATH: TRAF Domain (blue); low complexity: low complexity sequence(cyan) and WD40: WD40 domain (orange). ) 图 3 侏儒蛤MlTRAFs保守结构域 Fig. 3 Conserved domains of MlTRAFs in Mulinia lateralis |
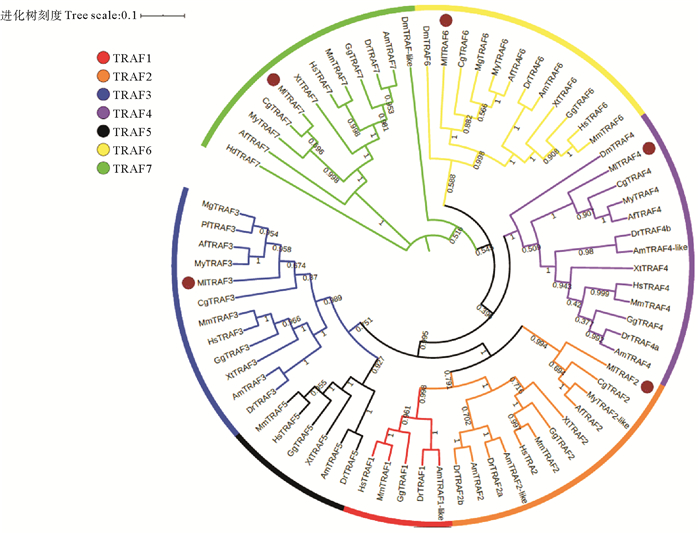
通过检索文献以及公共数据库发现(见表 3),在人、鸡、鼠、非洲爪蟾中存在TRAF1-TRAF7共7个成员,在斑马鱼中发现TRAF2和TRAF4分别有2个拷贝,在虾夷扇贝、栉孔扇贝、太平洋牡蛎中发现缺少TRAF1、TRAF5[38-39]。与扇贝和牡蛎类似,侏儒蛤中共鉴定出5个TRAF同源基因,成员TRAF1、TRAF5缺失。为了确定TRAFs的系统发生关系,本研究构建了NJ系统发生树, 结果表明,TRAF蛋白进化关系明确,其中双壳类TRAF2形成独立进化枝,并与TRAF3聚在一起;而TRAF6与TRAF7亲缘关系较近,并首先与TRAF4聚在一起;TRAF1和TRAF5仅在脊椎动物中发现,参考TRAF2、TRAF3、TRAF4和TRAF6聚类发现脊椎动物类群的TRAF蛋白亲缘关系明显更近(见图 4)。
|
|
表 3 不同物种TRAF序列亚家族比较分析 Table 3 Comparative analysis of TRAF subfamilies in different species |
|
( Hs: Homo sapiens; Mm: Mus musculus; Gg: Gallus gallu; Xt: Xenopus tropicalis; Dr: Danio rerio; Am: Astyanax mexicanus; Dm: Drosophilidae melanogaster; Hd: Haliotis discus discus; Cg: Crassostrea gigas; Pf: Pinctada fucata; Mg: Mytilus galloprovincialis; My: Mizuhopecten yessoensis; Af: Azumapecten farreri; Ml: Mulinia lateralis ) 图 4 TRAF蛋白系统发生分析 Fig. 4 Phylogenetic analysis of TRAF proteins |
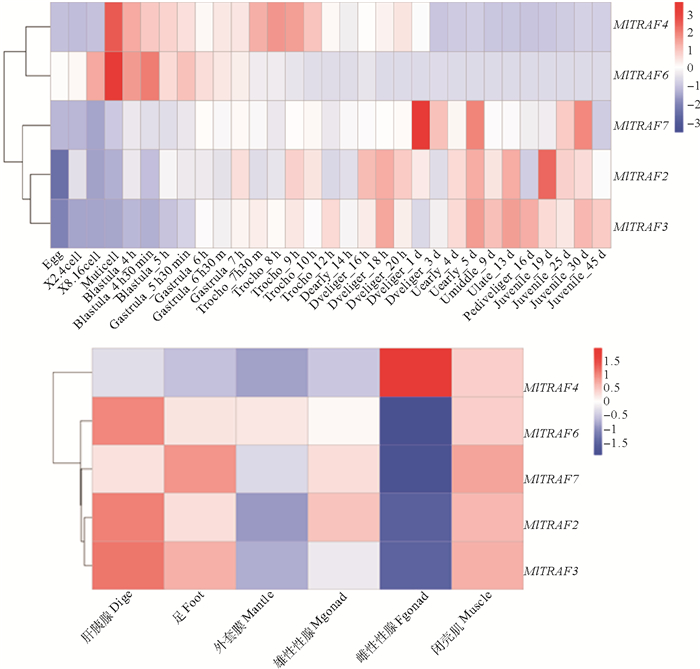
利用侏儒蛤发育时期转录组数据,分析MlTRAF各成员从卵母细胞到稚贝各发育阶段的表达变化(见图 5A),发现MlTRAF4与MlTRAF6主要在胚胎发育早期高表达,而MlTRAF2、MlTRAF3与MlTRAF7主要在幼虫发育后期高表达。MlTRAF4基因的高表达起始于多细胞期,并持续至担轮幼虫后期;D型幼虫期该基因表达呈下降趋势,并在发育至壳顶幼虫之后表达明显降低。MlTRAF6基因同样在发育早期高表达,起始于8~16细胞期,但与MlTRAF4不同,其表达水平在担轮幼虫期已经明显下降,高表达时期仅覆盖多细胞期、囊胚期和原肠期。MlTRAF4基因的高表达有两个明显的阶段,D型幼虫后期至壳顶幼虫前期以及稚贝期,形成原肠胚后的其他阶段该基因的表达处于中等水平。MlTRAF2和MlTRAF3基因的胚胎表达模式比较相近,均起始高表达于原肠胚后期,其表达水平有随着胚胎发育逐渐增高的趋势,前者在早期稚贝中达到表达高峰期,后者则在壳顶幼虫期之后呈持续高表达状态。
|
( Egg: 卵母细胞期 Oocyte phase; X2.4cell: 2~4细胞期 2~4 Cell phase;X8.16cell: 8~16细胞期 8~16 Cell phase;Muticell:多细胞期 Multicellular phase;Blastula_4 h,Blastula_4 h 30 m,Blastula_5 h:囊胚期 Blastocyst stage;Gastrula_5 h 30 m,Gastrula_6 h,Gastrula_6 h 30 m,Gastrula_7 h:原肠胚 Gastrula stage;Trocho_7 h 30 m,Trocho_8 h,Trocho_9 h,Trocho_10 h,Trocho_12 h:担轮幼虫时期 Trochanter stage;Dearly_14 h,Dveliger_16 h, Dveliger_18 h, Dveliger_20 h, Dveliger_1 d,Dveliger_3 d:D型幼虫时期 D-shaped larval stage;Uearly_4 d,Uearly_5 d,Umiddle_9 d,Ulate_13 d:壳顶幼虫时期 Umbo larva stage;Pediveliger_16 d: 匍足面幼虫时期 Pediveliger larva stage;Juvenile_19 d,Juvenile_25 d,Juvenile_30 d,Juvenile_45 d:稚贝 Juvenile shellfish。) 图 5 侏儒蛤MlTRAFs的表达 Fig. 5 Expression of MlTRAFs in Mulinia lateralis |
分析健康侏儒蛤成体所测定的6个组织(肝胰腺、足、外套膜、雄性腺、雌性腺、闭壳肌)的表达谱数据,发现MlTRAFs具有多样的组织表达(见图 5B)。闭壳肌组织中5个MlTRAF成员均呈现中高表达状态,而除了MlTRAF6之外,其余4个成员在肝胰腺,足以及雄性性腺中均呈现中高表达状态。外套膜组织中的表达量相对较高的成员是MlTRAF4,而雌性性腺中可以检测到MlTRAF6基因的特异性高表达(与其余组织相比高出2.12~10.41倍),且该组织中其余4个基因表达极低,暗示雌性性腺的免疫相关调控可能与MlTRAF6有关。此外,各个MlTRAF成员相比,MlTRAF2、MlTRAF3、MlTRAF4的表达在肝胰腺组织中相对较高,同时MlTRAF7在足中的表达相对较高,这些发现均提示不同的MlTRAF成员可能具有不同的功能分化。
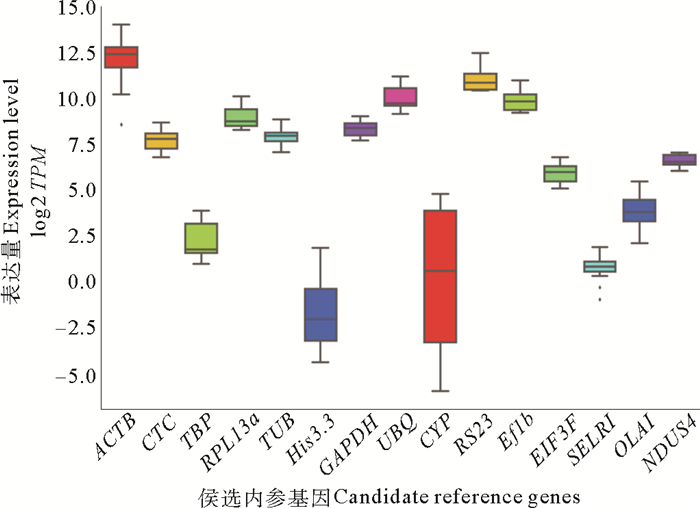
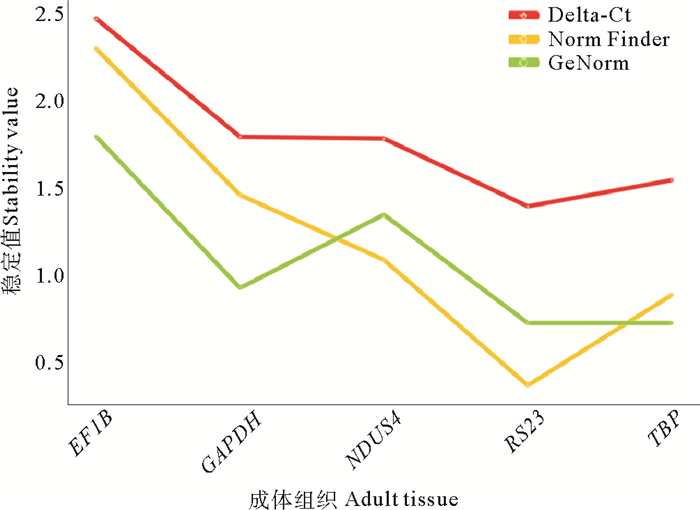
2.3 侏儒蛤转录组水平内参基因筛查利用侏儒蛤胚胎各时期以及成体组织的转录组数据,对包含ACTB、CTC、TBP、RPL13a、TUB、His3.3、GAPDH、UBQ、CYP、RS23、EF1b、EIF3F、SELR1、OLA1、NDUS4在内的15个基因进行初步分析(见图 6)。进一步,选择表达相对稳定的TBP、GAPDH、RS23、EF1B、NDUS4这5个候选内参基因进行qPCR验证,并使用geNorm、NormFinder和delta-Ct 3种统计算法评估了5个候选内参基因的表达稳定性。最终,3种分析方法所获得的评估结果一致,综合排名显示RS23是进行下一步实验最佳内参基因(见图 7)。
|
图 6 侏儒蛤中15个候选内参基因物log 2 TPM分析 Fig. 6 log 2 TPM values of 15 candidate reference genes in Mulinia lateralis |
|
图 7 基于GeNorm,NormFinder和delta-Ct评估侏儒蛤中5个候选内参基因的表达稳定性 Fig. 7 Stability evaluation of the five candidate reference Genes in Mulinia lateralis based on GeNorm, NormFinder and delta-Ct |
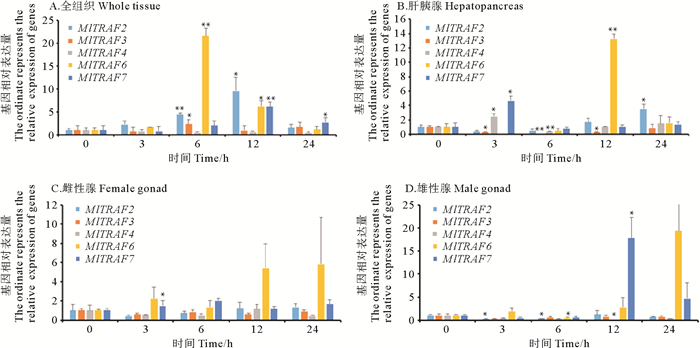
为研究MlTRAFs基因响应鳗弧菌(V. anguillarum)侵染的表达模式,本研究采用RT-qPCR法对侵染24 h内的5个时间点进行MlTRAFs表达变化监测。结果显示,5个MlTRAF基因的显著表达上调(P < 0.05)均可在至少一个时间点被检测到(见图 8A~D)。其中,3 h的MlTRAFs急性表达变化可在肝胰腺和雌雄性腺中被检测到,如MlTRAF7在肝胰腺、雌性性腺中表现出急性上调表达,而MlTRAF2、MlTRAF3则分别在雄性性腺和肝胰腺中急性下调表达。侵染6 h后,MlTRAF3、MlTRAF4和MlTRAF2、MlTRAF6分别在肝胰腺和雄性性腺中显著下调,而全组织混合样品中MlTRAF2、MlTRAF3、MlTRAF6均显著上调。12 h后,肝胰腺和雄性性腺中MlTRAF6、MlTRAF7的分别表达上调,而MlTRAF3、MlTRAF4的分别表达下调,同时全组织混合样品中MlTRAF2、MlTRAF6、MlTRAF7均显著上调。24 h后,MlTRAF2、MlTRAF7在肝胰腺和全组织混合样品中分别被检测到显著上调。表达相关性分析结果显示,全组织混合样品中,MlTRAF2和MlTRAF7的应答变化显著正相关,且肝胰腺中,MlTRAF4和MlTRAF7的应答变化显著正相关(见表 4、5),暗示这些基因的免疫应答可能存在级联调控。此外,本研究观察到不同MlTRAF成员存在不同的组织应答模式,如MlTRAF6仅在肝胰腺和全组织混合样品中表达显著上调,而MlTRAF7在所有样品中均可被检测到显著表达上调,提示MlTRAF7可作为候选免疫调控指示基因。此外,雄性性腺和肝胰腺组织中还可以分别检测到MlTRAF2和MlTRAF3的组织特异性表达下调,提示可能与其组织特异性功能有关。
|
( 竖线表示平均值±SE(n=3),星号表示显着差异(*P < 0.05,**P < 0.01)。The vertical bar represents the mean±SE (n=3). The asterisk indicates significant difference (*P < 0.05, **P < 0.01). ) 图 8 鳗弧菌感染后MlTRAFs基因表达 Fig. 8 MlTRAFs responses after challenge with V. anguillarum |
|
|
表 4 鳗弧菌侵染后,侏儒蛤全组织混合样品和肝胰腺中各MlTRAFs的表达相关性 Table 4 Correlation of MlTRAFs expression in mixed-tissue sample and in hepatopancreas of M. lateralis after infection with V. anguillarum |
|
|
表 5 鳗弧菌侵染后,侏儒蛤雌雄性腺中各MlTRAFs的表达相关性 Table 5 Correlation of MlTRAFs expression in female and male gonad of M. lateralis after infection with V. anguillarum |
本研究在侏儒蛤中共鉴定出5个TRAF成员,且不同TRAF成员在其不同发育阶段和不同成体组织表现出多样的表达模式(见图 4、5)暗示其功能的分化。在不同双壳贝类中TRAF表达具有相似的表达特征,例如侏儒蛤和牡蛎中TRAF2均在幼虫发育后期高表达[40],侏儒蛤和虾夷扇贝TRAF3均在幼虫发育后期高表达[27]。MlTRAF4与MlTRAF6主要在胚胎发育早期高表达,特别是多细胞期至囊胚形成期,暗示这两个基因可能参与了卵裂调控和三胚层的分化过程。MlTRAF2、MlTRAF3与MlTRAF7主要在幼虫发育后期高表达,其中MlTRAF7主要在D型幼虫发育早期、壳顶幼虫早期以及稚贝中高表达,猜测该基因可能参与幼虫壳的发育过程。在雌性生物中TRAF4过表达增加细胞增殖和迁移[4],而儒蛤蜊成体雌性性腺组织中可以检测到MlTRAF4基因的特异性高表达,因此推测雌性性腺的细胞增殖和迁移与MlTRAF4有关。此外,发现闭壳肌组织是唯一一个所有MlTRAF成员均呈现中高表达状态的组织,这可能与双壳类的血淋巴体外循环有关。双壳类动物闭壳肌组织中的血细胞含量丰富,对细菌侵染防御发挥着重要作用。多数MlTRAFs在闭壳肌和肝胰腺高水平表达,进一步提示这些基因可能在先天免疫反应中起重要作用[42]。
进一步,利用RT-qPCR对鳗弧菌侵染24 h内各MlTRAF基因进行免疫应答分析,前期研究表明,TRAF基因的功能非常保守,推测MlTRAFs基因家族成员在不同时间点的组织表达显著上调可能与促进NF-κB信号通路激活,从而促进机体免疫应答有关[43]。在雄性性腺和肝胰腺组织中分别检测到的MlTRAF2和MlTRAF3的组织特异性表达抑制,猜测这两个基因可能通过参与调控细胞迁移、细胞凋亡等发挥组织特异性免疫,与炎症反应等相关[22, 44-46]。此外,全组织混合样品和肝胰腺中分别检测到的MlTRAF2与MlTRAF7以及MlTRAF4与MlTRAF7表达正相关,该结果暗示MlTRAF不同成员间可能存在基因协同表达模式,提示这些基因在防御鳗弧菌侵染过程中可能具有协同的生理功能。还发现,在鳗弧菌胁迫下,各MlTRAF成员中MlTRAF6基因表达水平相对较高,作者推测这可能与MlTRAF6基因N端的低复杂结构域(LCR)有关。有研究显示,含有LCR的蛋白质往往会与其他蛋白质发生更多的相互作用[47],据此推测MlTRF6的LCR可能会促进该基因与不同受体的结合能力,进而促进下游通路的信号转导。
4 结语综上所述,本研究首次在双壳贝类侏儒蛤中鉴定了完整的MlTRAFs基因家族,包括MlTRAF2,MlTRAF3,MlTRAF4,MlTRAF6和MlTRAF7在内共5个成员,系统分析了MlTRAFs的基因结构,保守功能域,蛋白特性并对其进行了系统发生分析。利用转录组数据和RT-qPCR分析,深入探讨了不同MlTRAF成员在胚胎各发育时期和各个成体组织的表达规律以及鳗弧菌侵染24 h内的表达变化,发现不同成员可能存在功能分化。本研究以侏儒蛤为研究对象,对先天免疫防御重要调控因子TRAF进行基因家族鉴定分析,通过亲缘关系比对、系统发生分析以及时空表达模式研究,并利用成体健康侏儒蛤进行鳗弧菌侵染实验,进一步探索各TRAF成员在贝类生长发育中的作用以及免疫应答模式,为更好的理解贝类TRAF基因家族的进化、功能差异以及免疫调控提供参考。
| [1] |
Costa M M, Prado-Alvarez M, Gestal C, et al. Functional and molecular immune response of Mediterranean mussel (Mytilus galloprovincialis) haemocytes against pathogen-associated molecular patterns and bacteria[J]. Fish & Shellfish Immunology, 2009, 26(3): 515-523.
(  0) 0) |
| [2] |
Roach J C, Glusman G, Rowen L, et al. The evolution of vertebrate Toll-like receptors[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(27): 9577-9582. DOI:10.1073/pnas.0502272102
(  0) 0) |
| [3] |
Bulatov E, Valiullina A, Sayarova R, et al. Promising new therapeutic targets for regulation of inflammation and immunity: RING-type E3 ubiquitin ligases[J]. Immunology Letters, 2018, 202: 44-51. DOI:10.1016/j.imlet.2018.08.001
(  0) 0) |
| [4] |
Swaidani S, Liu C N, Zhao J J, et al. TRAF Regulation of IL-17 Cytokine Signaling[J]. Frontiers in Immunology, 2019, 10: 1293. DOI:10.3389/fimmu.2019.01293
(  0) 0) |
| [5] |
Xie T, Chen T, Li C S, et al. RACK1 attenuates RLR antiviral signaling by targeting VISA-TRAF complexes[J]. Biochemical and Biophysical Research Communications, 2019, 508(3): 667-674. DOI:10.1016/j.bbrc.2018.11.203
(  0) 0) |
| [6] |
Bishop G A, Abdul-Sater A A, Watts T H. TRAF proteins in health and disease[J]. Frontiers in Immunology, 2019, 10: 326. DOI:10.3389/fimmu.2019.00326
(  0) 0) |
| [7] |
Li K M, Li M, Wang N, et al. Genome-wide identification, characterization, and expression analysis of the TRAF gene family in Chinese tongue sole (Cynoglossus semilaevis)[J]. Fish & Shellfish Immunology, 2020, 96: 13-25.
(  0) 0) |
| [8] |
Hacker H, Tseng P H, Karin M. Expanding TRAF function: TRAF3 as a tri-faced immune regulator[J]. Nature Reviews Immunology, 2011, 11(7): 457-468. DOI:10.1038/nri2998
(  0) 0) |
| [9] |
Bouraoui Y, Achour M, Royuela M, et al. Immune profiling of human prostate epithelial cells determined by expression of p38/TRAF-6/ERK MAP kinases pathways[J]. Kaohsiung Journal of Medical Sciences, 2018, 34(3): 125-133. DOI:10.1016/j.kjms.2017.10.002
(  0) 0) |
| [10] |
Regnier C H, Tomasetto C, Mooglutz C, et al. Presence of a new conserved domain in Cart1, a novel member of the Tumor-Necrosis-Factor receptor-associated protein family, which is expressed in breast-carcinoma[J]. Journal of Biological Chemistry, 1995, 270(43): 25715-25721. DOI:10.1074/jbc.270.43.25715
(  0) 0) |
| [11] |
Wajant H, Muhlenbeck F, Scheurich P. Identification of a TRAF (TNF receptor-associated factor) gene in Caenorhabditis elegans[J]. Journal of Molecular Evolution, 1998, 47(6): 656-662. DOI:10.1007/PL00006423
(  0) 0) |
| [12] |
Liu H, Su Y C, Becker E, et al. A Drosophila TNF-receptor-associated factor (TRAF) binds the ste20 kinase misshapen and activates Jun kinase[J]. Current Biology, 1999, 9(2): 101-104. DOI:10.1016/S0960-9822(99)80023-2
(  0) 0) |
| [13] |
Zhu S N, Jin J, Gokhale S, et al. Genetic alterations of TRAF proteins in human cancers[J]. Frontiers in Immunology, 2018, 9: 2111. DOI:10.3389/fimmu.2018.02111
(  0) 0) |
| [14] |
Gupta I, Varshney N K, Khan S. Emergence of members of TRAF and DUB of ubiquitin proteasome system in the regulation of hypertrophic cardiomyopathy[J]. Frontiers in Genetics, 2018, 9: 336. DOI:10.3389/fgene.2018.00336
(  0) 0) |
| [15] |
Xu W, Zhang L, Zhang Y, et al. TRAF1 exacerbates myocardial ischemia reperfusion injury via ASK1-JNK/p38 signaling[J]. Journal of the American Heart Association, 2019, 8(21): e012575.
(  0) 0) |
| [16] |
Cabal-Hierro L, Rodriguez M, Artime N, et al. TRAF-mediated modulation of NF-kB and JNK activation by TNFR2[J]. Cellular Signalling, 2014, 26(12): 2658-2666. DOI:10.1016/j.cellsig.2014.08.011
(  0) 0) |
| [17] |
Ye H, Park Y C, Kreishman M, et al. The structural basis for the recognition of diverse receptor sequences by TRAF2[J]. Molecular Cell, 1999, 4(3): 321-330. DOI:10.1016/S1097-2765(00)80334-2
(  0) 0) |
| [18] |
Muro I, Fang G, Gardella K A, et al. The TRAF3 adaptor protein drives proliferation of anaplastic large cell lymphoma cells by regulating multiple signaling pathways[J]. Cell Cycle, 2014, 13(12): 1918-1927. DOI:10.4161/cc.28895
(  0) 0) |
| [19] |
Yoon J H, Cho Y J, Park H H. Structure of the TRAF4 TRAF domain with a coiled-coil domain and its implications for the TRAF4 signalling pathway[J]. Acta Crystallographica(Section D), 2014, 70(1): 2-10.
(  0) 0) |
| [20] |
Buchta C M, Bishop G A. TRAF5 negatively regulates TLR signaling in B lymphocytes[J]. Journal of Immunology, 2014, 192(1): 145-150. DOI:10.4049/jimmunol.1301901
(  0) 0) |
| [21] |
Li L, Yang R L, Feng M J, et al. Rig-I is involved in inflammation through the IPS-1/TRAF(6) pathway in astrocytes under chemical hypoxia[J]. Neuroscience Letters, 2018, 672: 46-52. DOI:10.1016/j.neulet.2018.02.035
(  0) 0) |
| [22] |
Fu D, Zhang Y, Xiao S, et al. The first homolog of a TRAF7 (TNF receptor-associated factor 7) gene in a mollusk, Crassostrea hongkongensis[J]. Fish & Shellfish Immunology, 2011, 31(6): 1208-1210.
(  0) 0) |
| [23] |
Boyce B F, Li J, Xing L, et al. Bone Remodeling and the role of TRAF3 in osteoclastic bone resorption[J]. Frontiers in Immunology, 2018, 9: 2263. DOI:10.3389/fimmu.2018.02263
(  0) 0) |
| [24] |
Zhu L J, Yang T C, Wu Q, et al. Tumor necrosis factor receptor-associated factor (TRAF) 6 inhibition mitigates the pro-inflammatory roles and proliferation of rheumatoid arthritis fibroblast-like synoviocytes[J]. Cytokine, 2017, 93: 26-33. DOI:10.1016/j.cyto.2017.05.001
(  0) 0) |
| [25] |
Mao F, Li J, Zhang Y, et al. Molecular cloning and functional analysis of tumor necrosis factor receptor-associated factor 6 (TRAF6) in Crossastrea gigas[J]. Fish & Shellfish Immunology, 2017, 68: 37-45.
(  0) 0) |
| [26] |
Huang X D, Liu W G, Guan Y Y, et al. Molecular cloning, characterization and expression analysis of tumor necrosis factor receptor-associated factor 3 (TRAF3) from pearl oyster Pinctada fucata[J]. Fish & Shellfish Immunology, 2012, 33(3): 652-658.
(  0) 0) |
| [27] |
Wang J, Wang R J, Wang S Y, et al. Genome-wide identification and characterization of TRAF genes in the Yesso scallop (Patinopecten yessoensis) and their distinct expression patterns in response to bacterial challenge[J]. Fish & Shellfish Immunology, 2015, 47(1): 545-555.
(  0) 0) |
| [28] |
Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2
(  0) 0) |
| [29] |
Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054
(  0) 0) |
| [30] |
Li Y, Zhang L, Li R, et al. Systematic identification and validation of the reference genes from 60 RNA-Seq libraries in the scallop Mizuhopecten yessoensis[J]. BMC Genomics, 2019, 20(1): 288. DOI:10.1186/s12864-019-5661-x
(  0) 0) |
| [31] |
Van Zeveren A M, Visser A, Hoorens P R, et al. Evaluation of reference genes for quantitative real-time PCR in Ostertagia ostertagi by the coefficient of variation and geNorm approach[J]. Molecular and Biochemical Parasitology, 2007, 153(2): 224-227. DOI:10.1016/j.molbiopara.2007.03.005
(  0) 0) |
| [32] |
Wang Q, Ishikawa T, Michiue T, et al. Stability of endogenous reference genes in postmortem human brains for normalization of quantitative real-time PCR data: Comprehensive evaluation using geNorm, NormFinder, and BestKeeper[J]. International Journal of Legal Medicine, 2012, 126(6): 943-952. DOI:10.1007/s00414-012-0774-7
(  0) 0) |
| [33] |
Yu R, Chen H P, Yan X F. Application of real-time fluorescence quantitative PCR accompanied with comparison of Delta CT for diagnosis of Down's syndrome from a single cell[J]. Chinese Journal of Medical Genetics, 2007, 24(2): 200-202.
(  0) 0) |
| [34] |
Cong M, Song L, Wang L, et al. The enhanced immune protection of Zhikong scallop Chlamys farreri on the secondary encounter with Listonella anguillarum[J]. Comp Biochem Physiol B Biochem Mol Biol, 2008, 151(2): 191-196. DOI:10.1016/j.cbpb.2008.06.014
(  0) 0) |
| [35] |
Gao Q, Song L, Ni D, et al. cDNA cloning and mRNA expression of heat shock protein 90 gene in the haemocytes of Zhikong scallop Chlamys farreri[J]. Comparative Biochemistry and Physiology Part B Biochemistry & Molecular Biology, 2007, 147(4): 704-715.
(  0) 0) |
| [36] |
Hu X, Bao Z, Hu J, et al. Cloning and characterization of tryptophan 2, 3-dioxygenase gene of Zhikong scallop Chlamys farreri (Jones and Preston 1904)[J]. Aquaculture Research, 2006, 37(12): 1187-1194. DOI:10.1111/j.1365-2109.2006.01546.x
(  0) 0) |
| [37] |
Pfaffl M W, Horgan G W, Dempfle L. Relative expression software tool (REST) for group-wise comparison and statistical analysis of relative expression results in real-time PCR[J]. Nucleic Acids Research, 2002, 30(9): e36. DOI:10.1093/nar/30.9.e36
(  0) 0) |
| [38] |
Li Y, Li Y, Cao X, et al. Pattern recognition receptors in zebrafish provide functional and evolutionary insight into innate immune signaling pathways[J]. Cellular & Molecular Immunology, 2017, 14(1): 80-89.
(  0) 0) |
| [39] |
Zhang H, Tan K, Li S, et al. Genome-wide analysis of TRAF gene family and its response to bacterial infection in noble scallop Chlamys nobilis with different carotenoids content[J]. Aquaculture, 2020, 535(5625): 736309.
(  0) 0) |
| [40] |
Huang B, Zhang L, Du Y, et al. Molecular characterization and functional analysis of tumor necrosis factor receptor-associated factor 2 in the Pacific oyster[J]. Fish & Shellfish Immunology, 2016, 48: 12-19.
(  0) 0) |
| [41] |
Xie P, Wang X, Kong M, et al. TRAF4 promotes endometrial cancer cell growth and migration by activation of PI3K/AKT/Oct4 signaling[J]. Experimental and Molecular Pathology, 2019, 108: 9-16. DOI:10.1016/j.yexmp.2019.03.003
(  0) 0) |
| [42] |
Li Y, Song X, Wang W, et al. The hematopoiesis in gill and its role in the immune response of Pacific oyster Crassostrea gigas against secondary challenge with Vibrio splendidus[J]. Developmental & Comparative Immunology, 2017, 71: 59-69.
(  0) 0) |
| [43] |
Li C, Wei J, Zhang X, et al. Fish TRAF2 promotes innate immune response to RGNNV infection[J]. Fish & Shellfish Immunology, 2020, 102(1050-4648): 108-116.
(  0) 0) |
| [44] |
Grech A P, Amesbury M, Chan T, et al. TRAF2 differentially regulates the canonical and noncanonical pathways of NF-kappaB activation in mature B cells[J]. Immunity, 2004, 21(5): 629-642. DOI:10.1016/j.immuni.2004.09.011
(  0) 0) |
| [45] |
Xu Z Y, Sun B J, Chang M X, et al. Characterization and expression analysis of TNFR-associated factor 1 (TRAF1) in grass carp Ctenopharyngodon idella[J]. Veterinary Immunology and Immunopathology, 2008, 121(1-2): 44-57. DOI:10.1016/j.vetimm.2007.08.001
(  0) 0) |
| [46] |
Liu X, Zhang L, Qin H, et al. Inhibition of TRAF3 expression alleviates cardiac ischemia reperfusion (IR) injury: A mechanism involving in apoptosis, inflammation and oxidative stress[J]. Biochem Biophys Res Commun, 2018, 506(1): 298-305. DOI:10.1016/j.bbrc.2018.10.058
(  0) 0) |
| [47] |
Maria Velasco A, Becerra A, Hernandez-Morales R, et al. Low complexity regions (LCRs) contribute to the hypervariability of the HIV-1 gp120 protein[J]. Journal of Theoretical Biology, 2013, 338: 80-86. DOI:10.1016/j.jtbi.2013.08.039
(  0) 0) |
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
 2021, Vol. 51
2021, Vol. 51


