2. 青岛海洋科学与技术试点国家实验室 海洋渔业科学与食物产出过程功能实验室,山东 青岛 266237
DNA甲基化,即胞嘧啶的5号碳原子上连接的氢原子被替换成为一个甲基,是真核生物类群中的重要的表观遗传修饰,广泛存在于植物,动物和真菌中。DNA甲基化在基因表达调控,基因组稳定性维持,哺乳动物X染色体失活等生物学现象中起到十分重要的作用[1-2]。在生物的生长发育过程中,细胞的DNA甲基化水平并不是一成不变的,在哺乳动物生长发育中就存在两次大规模的DNA甲基化的重编程过程[3]。第一次DNA甲基化的重编程发生在配子发生过程中,原始生殖细胞首先去除来源于外胚层的DNA甲基化模式,在形成精子和卵子的过程中,分别形成配子特异的DNA甲基化模式,这种DNA甲基化模式与配子内基因表达,精子DNA的包装以及基因印迹有着密切关系[4-6]。第二次DNA甲基化的重编程发生在精卵结合之后,精子来源的DNA会迅速发生DNA去甲基化,在受精卵第一次分裂之前精子来源的DNA去甲基化就已经完成;在随后的细胞分裂过程中,卵子来源的DNA会逐渐去甲基化,在囊胚时期达到DNA甲基化的最低水平。早期发育时期DNA的去甲基化对胚胎细胞的全能性具有重要意义[3]。
DNA甲基化的重编程包括DNA的去甲基化和重新甲基化,其中DNA的去甲基化过程分为两种类型:第一种是DNA的被动去甲基化,在细胞分裂的过程中,随着DNA的复制,如果不形成新的DNA甲基化,DNA的甲基化水平就会逐渐降低,如胚胎时期卵子来源DNA的去甲基化现象。第二种是DNA的主动去甲基化,即在不发生DNA复制的情况下,通过一系列反应脱去胞嘧啶五号碳原子上所连接的甲基基团的过程[7]。该过程主要由TET(ten-eleven translocation)蛋白催化,TET先将5甲基胞嘧啶5mC催化生成5羟甲基胞嘧啶5hmC [8], 然后脱氢生成5甲酰基胞嘧啶5fC,并可进一步氧化形成5羧基胞嘧啶5caC [9]。5fC和5caC都可以被胸腺嘧啶DNA糖基化酶(TDG)识别并脱去对应的甲酰基和羧基,使碱基恢复为没有修饰的胞嘧啶,从而完成DNA的主动去甲基化过程[10]。在哺乳动物中,TET基因家族有三个成员TET1,TET2和TET3,它们分别在不同的生物学过程中发挥DNA去甲基化的功能。TET3基因在卵和受精卵中表达量较高,缺失TET3基因会导致早期胚胎中的5hmC含量下降,表明TET3蛋白可能参与受精卵中父本来源DNA的去甲基化[11-12]。TET1和TET2基因在小鼠的原始生殖细胞中检测到表达[13],小鼠中的基因敲除实验也表明这两个基因对配子发生过程中基因印迹的擦除具有重要作用,并且TET1和TET2蛋白分别作用于不同的目标基因[14-15]。
虾夷扇贝是中国重要的冷水性养殖贝类,开展对其科学、有效的育种和养殖工作是水产领域的重点工作之一。对虾夷扇贝功能基因,包括生长[16]、生殖[17]和免疫[18]相关基因的研究是产业发展的基础,并在近年来得到了一系列成果。特别最近虾夷扇贝基因组的完成[19],更是大大推动了虾夷扇贝功能基因的研究。虾夷扇贝每年经历一个生殖周期,包括休止期,增殖期,生长期和成熟期四个时期[20]。笔者近期结合DNA甲基化水平和DNMT基因家族的表达模式,分析了虾夷扇贝配子发生和早期发育过程中DNA甲基化的产生和维持[21]。本研究将从DNA去甲基化酶TET的角度,进一步探讨虾夷扇贝配子发生和早期发育中DNA去甲基化过程的发生。
1 材料方法 1.1 虾夷扇贝TET基因的鉴定人(Hs), 小鼠(Mm), 野猪(Ss), 牛(Bt), 美洲河狸(Cc), 单峰驼(Cd),原鸽(Cl), 安氏蜂鸟(Ca),鬃狮蜥(Pv), 海龟(Cm),非洲爪蟾(Xl), 斑马鱼(Dr), 青鳉(Nf), 菜粉蝶(Pr), 果蝇(Dm), 太平洋牡蛎(Cg), 美洲牡蛎(Cv), 紫贻贝(Mg), 帽贝(Lg)的TET蛋白序列下载自NCBI和Uniprot,其Accession Number见表 1。用SMART在线软件(http://smart.emblheidelberg.de/)预测这些蛋白的结构域。用blast软件将这些TET蛋白的保守结构域序列比对到虾夷扇贝的蛋白组数据库(GenBank assembly accession: GCA_002113885.2),比对参数为“blastall -p blastp -a 16 -v 10 -b 20 -e 1e-6”,获得虾夷扇贝候选TET基因,并用SMART预测虾夷扇贝TET蛋白的结构域。用ClustalX使用默认参数进行结构域的比对,确定虾夷扇贝TET蛋白序列中的保守残基。使用TET蛋白全长构建系统发生树,先用Protest预测出合适的氨基酸替换模型和参数(-m JTT+I+G+F -v 0.034 -a 1.414),然后用PhyML软件构建系统发生树。
|
|
表 1 各物种TET蛋白的序列信息 Table 1 Accession number of TET protein in different species |
本研究中虾夷扇贝TET基因的表达量由RNA-Seq数据计算得到。其中,配子发生过程中的表达数据来自本实验室前期工作[22]。首先通过石蜡切片鉴定获得休止期、增殖期、生长期和成熟期的精巢和卵巢,用异硫氰酸胍法提取性腺组织总RNA,然后使用VAHTS stranded mRNA-seq library prep kit for Illumina试剂盒构建RNA-Seq文库并测序。早期发育时期的表达数据来自虾夷扇贝基因组文章[19],包括2~8细胞期、囊胚、原肠胚、担轮幼虫、D型幼虫、早期壳顶幼虫、中期壳顶幼虫、后期壳顶幼虫和稚贝的RNA-Seq数据。
表达量计算方法如下:先用STAR将RNA-Seq数据比对到虾夷扇贝基因组[23],之后用HTSeq-count统计每个基因上覆盖的reads数[24],基因的表达量(TPM)通过自行编写的perl脚本计算获得。
1.3 数据分析用单因素方差分析检测各性腺发育时期之间TET基因的表达差异。用t检验进行同一时期两种性别的基因差异表达分析。P值小于0.05视为差异显著。
1.4 组织原位杂交为探究虾夷扇贝性腺中表达PyTET基因的细胞类型,本研究开展了原位杂交实验。用含有Sp6启动子序列的正向引物和含有T7启动子序列的反向引物扩增cDNA片段,以纯化后的PCR产物为模板,分别用Sp6和T7聚合酶合成地高辛标记的正义探针和反义探针。原位杂交引物序列见表 2。
|
|
表 2 原位杂交引物序列 Table 2 Sequences of primers used for in situ hybridization |
将性腺组织切片在PBST(磷酸盐缓冲盐水,0.1% Tween-20)中连续再水化,并用2 μg/mL蛋白酶K在37 ℃下消化15 min。60 ℃预杂交4 h后,再用含1 μg/mL变性RNA探针的杂交缓冲液(50%甲酰胺,5×SSC,100 μg/mL酵母tRNA,1.5%封闭试剂,0.1% Tween-20)在60 ℃条件下杂交16 h。然后洗去探针,并在含有地高辛抗体的封闭缓冲液(1:2 000稀释)中4 ℃孵育16 h。用马来酸缓冲液(0.1 mol/L马来酸,0.15 mol/L NaCl,0.1% Tween-20,pH=7.5)充分洗涤后,将切片置于NBT/BCIP显色液中避光显色,最后用1%中性红溶液复染观察。
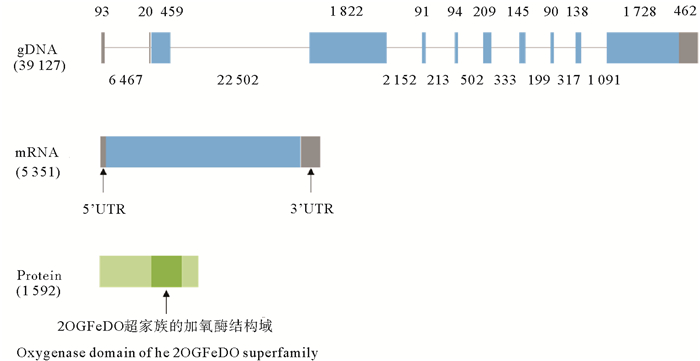
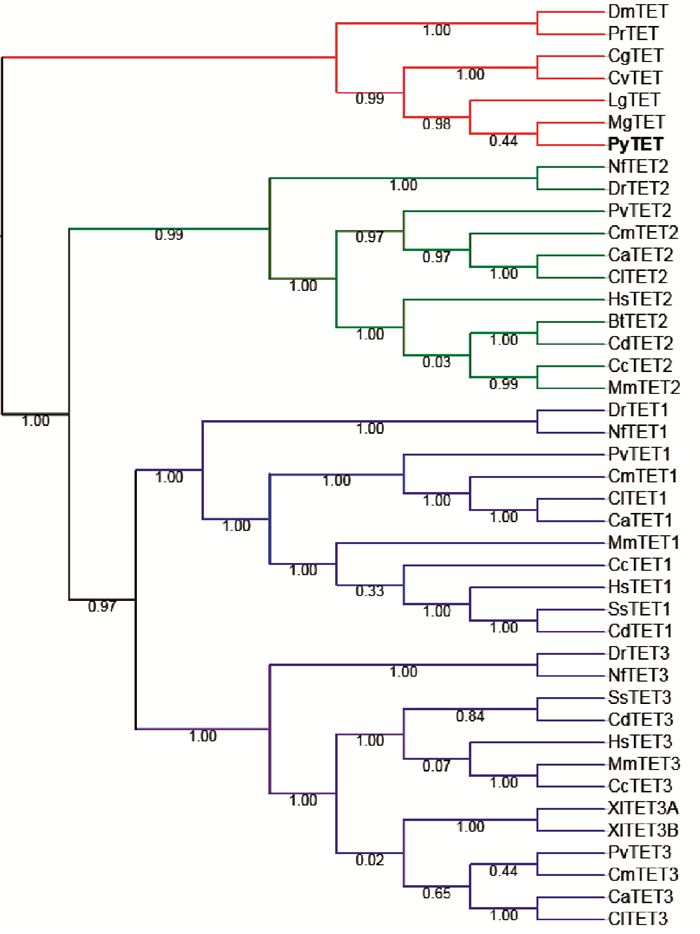
2 实验结果 2.1 虾夷扇贝TET基因的鉴定用其他物种TET蛋白的加氧酶结构域比对到虾夷扇贝蛋白组,发现虾夷扇贝中存在一个TET同源蛋白,命名为PyTET。该蛋白由1 591个氨基酸组成,分子量为175 668 Do,等电点为8.57。PyTET基因由10个外显子组成,其基因结构见图 1。蛋白结构域的预测结果显示PyTET包含一个完整的2OGFeDO超家族的加氧酶结构域(见图 1),该结构域与其他物种TET蛋白同源序列的相似度为46.59%~74.85%,并且和其他物种TET蛋白一样,PyTET的保守结构域中包含铁离子结合位点和2-氧代戊二酸结合残基(见图 2)。TET蛋白序列的系统发生分析显示,PyTET与软体动物TET聚在一起,然后与节肢动物(果蝇和菜粉蝶)TET聚在一起,最后和脊椎动物的3个TET聚在一起(见图 3)。
|
(蓝色表示编码区,灰色表示非翻译区,深绿色代表 2OGFeDO超家族的加氧酶结构域。The blue and grey regions represent CDS and UTR, respectively. The oxygenase domain of the 2OGFeDO superfamily is highlighted in deep green. ) 图 1 PyTET基因结构图 Fig. 1 Gene structure of PyTET |

|
(代表铁离子结合位点,●代表 2-氧代戊二酸结合残基。for iron ion binding site, ● for 2-oxoglutarate binding residues. ) 图 2 2OGFeDO超家族加氧酶结构域中的保守残基 Fig. 2 Conserved amino acid residues in oxygenase domain of the 2OGFeDO superfamily |
|
图 3 TET蛋白的系统发生分析 Fig. 3 Phylogenetic analysis of TET |
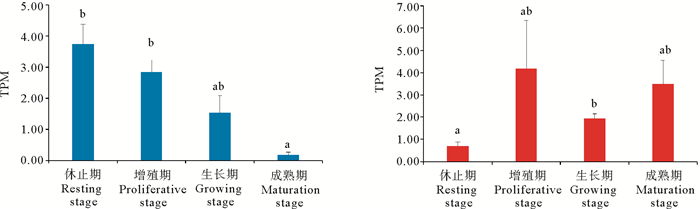
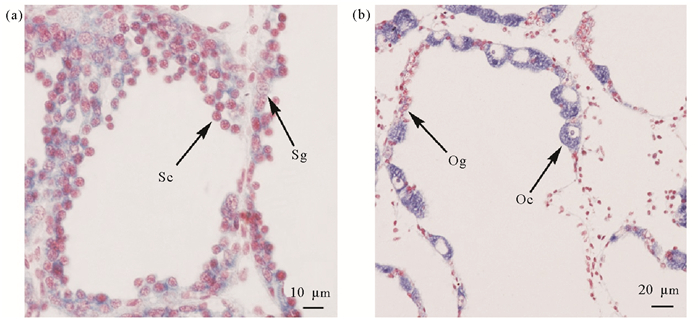
PyTET在精巢和卵巢中的表达呈现不同的趋势。如图 4所示,在精巢中,从休止期到成熟期,PyTET基因的表达量呈现逐步下降的趋势,在休止期最高,成熟期最低,休止期和增殖期的表达量显著高于成熟期。原位杂交结果表明,PyTET基因在精原细胞、精母细胞中均有表达(见图 5a)。在卵巢中,PyTET基因在增殖期表达量最高,在生长期的表达量显著高于休止期。原位杂交结果表明,PyTET基因主要在卵母细胞中表达,在卵原细胞中有微弱表达(见图 5b)。
|
(不同字母表示差异显著(P < 0.05)。Different letters indicate significant differences (P < 0.05). ) 图 4 PyTET基因在配子发生过程中的表达模式 Fig. 4 Expression profiles of PyTET during gametogenesis |
|
((a) PyTET在精巢中的定位;(b) PyTET在卵巢中的定位。Sg:精原细胞;Sc:精母细胞;Og:卵原细胞;Oc:卵母细胞。(a) Localization of PyTET with an anti-sense probe in testes; (b) Localization of PyTET with an anti-sense probe in ovaries. Sg, spermatogonium; Sc, spermatocyte; Og, oogonium; Oc, oocyte. ) 图 6 PyTET在增殖期精巢和卵巢的细胞学定位 Fig. 6 Localization of PyTET in testes and ovaries at the proliferative stage by in situ hybridization |
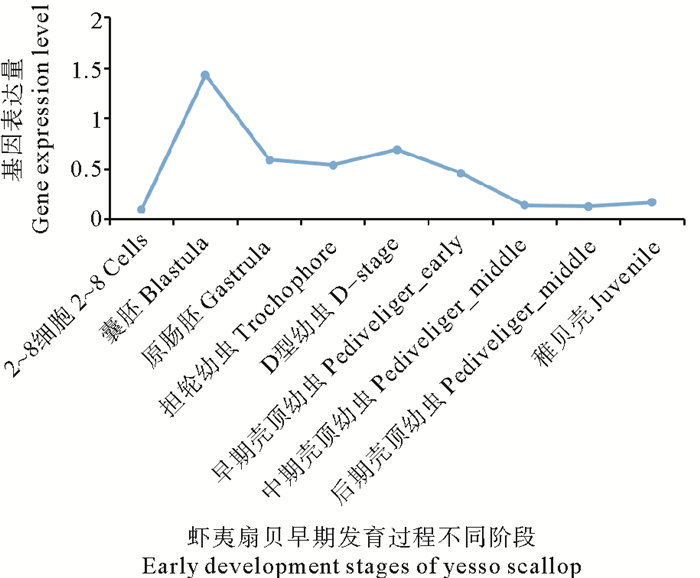
PyTET基因在早期发育过程中的表达量如图 6所示,在多细胞期PyTET基因的表达量接近0,随后表达量急剧升高,在囊胚期达到峰值;从原肠胚到D型幼虫期表达量较低且趋于稳定;进入壳顶期后PyTET基因表达量又开始逐渐下降,直至中期壳顶幼虫;之后表达无明显变化,一直维持到稚贝。
|
图 5 PyTET基因在虾夷扇贝早期发育过程中的表达模式 Fig. 5 Expression profiles of PyTET geneduring early development of yesso scallop |
DNA甲基化现象广泛存在于真核生物的各个类群,并在真核生物的生长发育过程中发挥重要的调控作用,因此DNA甲基化水平的调节对真核生物的生长发育十分关键。DNA甲基化水平的变化包括DNA去甲基化,DNA从头甲基化,DNA甲基化的维持等过程,每个过程都有相应的酶发挥功能。在哺乳动物中,DNMT1/2/3[25-26]和TET/1/2/3[8, 11, 27]都已经被鉴定,对它们的功能也做了比较深入的研究。但在海洋软体动物中,只有少数生物的DNMT基因被报道[28],还未见关于TET基因的研究报道。本研究发现虾夷扇贝只存在一个TET的同源基因,与蜕皮动物和其它冠轮动物类似。而脊椎动物基因组大多含有3个TET基因,各成员间的功能有所不同,TET1和TET2参与配子发生过程中的DNA去甲基化[13],TET3参与胚胎发育过程中的DNA去甲基化[11-12]。PyTET基因在配子发生和胚胎发育过程中都有明显的表达量变化,提示PyTET蛋白可能同时参与两个发育过程中DNA的去甲基化。以上结果暗示,PyTET可能接近于TET基因家族的祖先形式,并同时具有脊椎动物不同TET蛋白的功能。
配子发生过程中DNA甲基化模式的重建包括DNA去甲基化和重新甲基化的两个过程,在哺乳动物研究的比较深入,但在无脊椎动物,特别是海洋软体动物中研究还比较欠缺。最近的研究报道了虾夷扇贝配子发生过程中性腺整体DNA甲基化水平的变化和PyDNMT在该过程中的作用。但PyDNMT基因的表达仅能解释DNA甲基化的形成和维持,对PyTET基因的表达分析有助于理解在虾夷扇贝配子发生过程中DNA去甲基化的过程。在精子发生过程中,PyTET基因在休止期和增殖期的表达量较高,随着精子的形成过程,其表达量逐步降低,在成熟期达到最低。由于休止期和增殖期的生殖细胞类型主要为精原细胞和精母细胞[20],原位杂交结果也表明PyTET基因在这两种细胞中均有表达,表明在虾夷扇贝精子发生过程中,DNA的主动去甲基化过程可能发生在精原细胞和精母细胞中。在卵子发生过程中,PyTET基因表达量在休止期最低,增殖期开始升高,并一直维持到成熟期。与原位杂交结果观察到的PyTET基因在卵原细胞有微弱表达,而在卵母细胞中有大量表达的结果一致。这里可能涉及到两个DNA去甲基化的过程,第一个过程是卵原细胞中的PyTET参与了卵子发生过程中的DNA主动去甲基化过程,第二个过程是在初级卵母细胞中的PyTET,在精卵结合后,与哺乳动物TET3的相似,发挥着受精卵中DNA主动去甲基化的功能[11-12]。
虾夷扇贝受精卵的DNA来源于精子和成熟卵,其DNA甲基化模式也同时被继承,初级卵母细胞中表达大量PyTET基因可能与受精卵时期的DNA去甲基化过程有关,这一过程在哺乳动物中已有报道[29-31]。由于PyTET基因在2~8细胞期的表达量接近于0,与哺乳动物中的情况相同,推测这一过程可能在受精卵分裂之前已经完成。但不同的是在囊胚期,PyTET基因的表达突然升高,在随后的原肠胚时期又降低,使囊胚期形成了PyTET基因的表达量高峰,这一现象表明,虾夷扇贝可能在囊胚期发生了与哺乳动物不同的DNA去甲基化过程,这或许是海洋软体动物中特有的现象。前期研究显示,PyDNMT3在囊胚期的表达量也突然升高,基因组整体DNA甲基化水平在囊胚期也突然升高然后降低[21]。综合以上结果,本研究者推测在虾夷扇贝囊胚期可能发生了剧烈的DNA甲基化的重建,既包括DNA从头甲基化,也包括DNA去甲基化,PyDNMT3和PyTET在其中发挥着重要作用。
| [1] |
Jones P A. Functions of DNA methylation: Islands, start sites, gene bodies and beyond[J]. Nature Reviews Genetics, 2012, 13(7): 484-492. DOI:10.1038/nrg3230
(  0) 0) |
| [2] |
Suzuki M M, Adrian B. DNA methylation landscapes: Provocative insights from epigenomics[J]. Nature Reviews Genetics, 2008, 9(6): 465-476. DOI:10.1038/nrg2341
(  0) 0) |
| [3] |
Seisenberger S, Julian R P, Timothy A, et al. Reprogramming DNA methylation in the mammalian life cycle: Building and breaking epigenetic barriers[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1609): 20110330. DOI:10.1098/rstb.2011.0330
(  0) 0) |
| [4] |
Hajkova P, Erhardt S, Lane N, et al. Epigenetic reprogramming in mouse primordial germ cells[J]. Mechanisms of Development, 2002, 117(1-2): 15-23. DOI:10.1016/S0925-4773(02)00181-8
(  0) 0) |
| [5] |
Kurimoto K, Yabuta Y, Ohinata Y, et al. Complex genome-wide transcription dynamics orchestrated by Blimp1 for the specification of the germ cell lineage in mice[J]. Genes & Development, 2008, 22(12): 1617-1635.
(  0) 0) |
| [6] |
Hajkova P, Ancelin K, Waldmann T, et al. Chromatin dynamics during epigenetic reprogramming in the mouse germ line[J]. Nature, 2008, 452(7189): 877-881. DOI:10.1038/nature06714
(  0) 0) |
| [7] |
Kohli R M, Zhang Y. TET enzymes, TDG and the dynamics of DNA demethylation[J]. Nature, 2013, 502(7472): 472-479. DOI:10.1038/nature12750
(  0) 0) |
| [8] |
Tahiliani M, Koh K P, Shen Y, et al. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1[J]. Science, 2009, 324(5929): 930-935. DOI:10.1126/science.1170116
(  0) 0) |
| [9] |
Zhao H B, Chen T P. Tet family of 5-methylcytosine dioxygenases in mammalian development[J]. Journal of Human Genetics, 2013, 58(7): 421-427. DOI:10.1038/jhg.2013.63
(  0) 0) |
| [10] |
He Y F, Li B Z, Li Z, et al. Tet-mediated formation of 5-carboxylcytosine and its excision by TDG in mammalian DNA[J]. Science, 2011, 333(6047): 1303-1307. DOI:10.1126/science.1210944
(  0) 0) |
| [11] |
Gu T P, Guo F, Yang H, et al. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes[J]. Nature, 2011, 477(7366): 606-610. DOI:10.1038/nature10443
(  0) 0) |
| [12] |
Wossidlo M, Nakamura T, Lepikhov K, et al. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming[J]. Nature Communications, 2011, 2: 241. DOI:10.1038/ncomms1240
(  0) 0) |
| [13] |
Molyneaux K A, Stallock J, Schaible K, et al. Time-lapse analysis of living mouse germ cell migration[J]. Developmental Biology, 2001, 240(2): 488-498. DOI:10.1006/dbio.2001.0436
(  0) 0) |
| [14] |
Dawlaty M M, Breiling A, Le T, et al. Combined deficiency of Tet1 and Tet2 causes epigenetic abnormalities but is compatible with postnatal development[J]. Developmental Cell, 2013, 24(3): 310-323. DOI:10.1016/j.devcel.2012.12.015
(  0) 0) |
| [15] |
Yamaguchi S, Hong K, Liu R, et al. Tet1 controls meiosis by regulating meiotic gene expression[J]. Nature, 2012, 492(7429): 443-447. DOI:10.1038/nature11709
(  0) 0) |
| [16] |
Feng L Y, Li X, Yu Q, et al. A scallop IGF binding protein gene: Molecular characterization and association of variants with growth traits[J]. Plos One, 2014, 9(2).
(  0) 0) |
| [17] |
Li Y P, Zhang L L, Sun Y, et al. Transcriptome sequencing and comparative analysis of ovary and testis identifies potential key sex-related genes and pathways in scallop Patinopecten yessoensis[J]. Marine Biotechnology, 2016, 18(4): 1-13.
(  0) 0) |
| [18] |
Li R J, Zhang R, Zhang L L, et al. Characterizations and expression analyses of NF-κB and Rel genes in the Yesso scallop (Patinopecten yessoensis) suggest specific response patterns against Gram-negative infection in bivalves[J]. Fish & Shellfish Immunology, 2015, 44(2): 611-621.
(  0) 0) |
| [19] |
Wang S, Zhang J B, Jiao W Q, et al. Scallop genome provides insights into evolution of bilaterian karyotype and development[J]. Nature Ecology & Evolution, 2017, 1(5): 120.
(  0) 0) |
| [20] |
Yu J C, Zhang L L, Li Y P, et al. Genome-wide identification and expression profiling of the SOX gene family in a bivalve mollusc Patinopecten yessoensis[J]. Gene, 2017, 627: 530. DOI:10.1016/j.gene.2017.07.013
(  0) 0) |
| [21] |
Li Y P, Zhang L L, Li Y J, et al. Dynamics of DNA methylation and DNMT expression during gametogenesis and early development of callop Patinopecten yessoensis[J]. Marine Biotechnology, 2019, 1-10.
(  0) 0) |
| [22] |
李若佼.虾夷扇贝性别分化的分子基础[D].青岛: 中国海洋大学, 2018. Li R J. Molecular Basis of Sex Differentiation in Patinopecten yessoensis[D]. Qingdao: Ocean University of China, 2018. (  0) 0) |
| [23] |
Dobin A, Davis C A, Schlesinger F, et al. STAR: Ultrafast universal RNA-seq aligner[J]. Bioinformatics, 2013, 29: 15-21. DOI:10.1093/bioinformatics/bts635
(  0) 0) |
| [24] |
Anders S, Pyl P T, Huber W. HTSeq--a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166-169. DOI:10.1093/bioinformatics/btu638
(  0) 0) |
| [25] |
Uysal F, Ozturk S, Akkoyunlu G. DNMT1, DNMT3A and DNMT3B proteins are differently expressed in mouse oocytes and early embryos[J]. Journal of Molecular Histology, 2017, 48(5-6): 417-426. DOI:10.1007/s10735-017-9739-y
(  0) 0) |
| [26] |
Goll M G, Kirpekar F, Maggert K A, et al. Methylation of tRNAAsp by the DNA methyltransferase homolog Dnmt2[J]. Science, 2006, 311(5759): 395-398. DOI:10.1126/science.1120976
(  0) 0) |
| [27] |
Ko M, Bandukwala H S, An J, et al. Ten-Eleven-Translocation 2 (TET2) negatively regulates homeostasis and differentiation of hematopoietic stem cells in mice[J]. Proceedings of the National Academy of Sciences, 2011, 108(35): 14566-14571. DOI:10.1073/pnas.1112317108
(  0) 0) |
| [28] |
Wang X, Li Q, Lian J, et al. Genome-wide and single-base resolution DNA methylomes of the Pacific oyster Crassostrea gigas provide insight into the evolution of invertebrate CpG methylation[J]. BMC Genomics, 2014, 15(1): 1119. DOI:10.1186/1471-2164-15-1119
(  0) 0) |
| [29] |
Monk M, Boubelik M, Lehnert S. Temporal and regional changes in DNA methylation in the embryonic, extraembryonic and germ cell lineages during mouse embryo development[J]. Development, 1987, 99(3): 371-82.
(  0) 0) |
| [30] |
Howlett S K, Reik W. Methylation levels of maternal and paternal genomes during preimplantation development[J]. Development, 1991, 113(1): 119-127.
(  0) 0) |
| [31] |
Mayer W, Niveleau A, Walter J, et al. Embryogenesis: Demethylation of the zygotic paternal genome[J]. Nature, 2000, 403(6769): 501-502.
(  0) 0) |
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
 2020, Vol. 50
2020, Vol. 50


