2. 青岛海洋科学与技术国家实验室,海洋渔业科学与食物产出过程功能实验室,山东 青岛 266237;
3. 中国水产科学研究院黄海水产研究所,农业部海洋渔业可持续发展重点实验室,山东 青岛 266071
生长性状是水产动物育种研究过程中重要的衡量指标,也是直接的育种目标。周期性蜕皮跳跃式生长是甲壳动物特殊的生长发育方式,甲壳动物的一生中要进行多次脱壳才能完成其全部的生长发育,所以,甲壳动物的生长发育主要依赖于蜕皮过程中肌肉的重塑和生长速率[1-3]。脊尾白虾(Exopalaemon carinicauda)是我国重要的小型经济虾类,以黄、渤海产量最高,具有生长速度快、繁殖周期短(一年可繁殖2~3代)、适应性广等优点[4],因而研究脊尾白虾的生长发育的调控,不仅具有理论价值,还具有重要的实际意义。
甲壳动物的肌肉结构与哺乳动物等其他脊椎动物一样,肌纤维是由肌球蛋白、肌动蛋白、原肌球蛋白和肌钙蛋白等构成的肌节单元组成[5]。其中,肌球蛋白是一类高度保守且广泛存在于真核细胞中的多功能蛋白,是构成骨骼肌和心肌的主要结构和功能蛋白之一。在脊椎动物和无脊椎动物肌肉中的肌球蛋白基本结构是一致的,一个完整的常规肌球蛋白(也称为Ⅱ型肌球蛋白)分子是由2条肌球蛋白重链(Myosin heavy chain, MHC)、2条必需轻链(Myosin light chain, MLC)和2条的调节性轻链组成的六聚体[6-7]。肌球蛋白重链具有ATP酶活性,且能够与肌动蛋白结合,这两个特性使其在肌球蛋白分子间的相互作用和肌肉的收缩中起主导作用;肌球蛋白轻链是调控链,而且其对肌球蛋白重链有某种调控作用[8]。肌球蛋白既是肌肉细胞的重要组成成分,同时在肌肉运动、肌细胞的物质转运、胞质流动、能量供应和信号传导等生理活动中发挥重要的作用[9]。
有关肌球蛋白的研究主要涉及到功能特性、理化性质、肌肉类型和生物学效应等诸多领域[10]。相对于脊椎动物MHC和MLC基因的研究[11-13],甲壳动物的研究相对较少,李文兰等[14]通过抑制消减杂交的方法获得凡纳滨对虾(Litopenaeus vannamei)的MLC基因,发现MLC的表达量在生长速度较快的时期显著高于其它时期;Koyama等[15]克隆凡纳滨对虾和斑节对虾(Penaeus monodon)的2种MHC的亚型,并分析其在整个生长发育过程中的表达情况,发现凡纳滨对虾的MHCa基因在仔虾期开始表达,而斑节对虾的MHCa基因只在成虾中表达。
在脊椎动物中,利用海量的基因组、转录组序列信息来挖掘、鉴定与生长发育的相关的基因及其分子机制的研究已经取得了长足进展,并成为基因组学、分子遗传学的重要发展方向。然而,由于目前缺乏甲壳动物基因组信息,有关十足目甲壳动物生长相关基因的研究还很薄弱,多集中于与蜕皮相关基因的研究[16]。本研究通过克隆获得脊尾白虾EcMHC2、EcMLC1全长序列,并分析其在脊尾白虾不同组织、不同幼体发育时期和不同生长时期的大小差异个体组的表达模式,以期阐明EcMHC2、EcMLC1在脊尾白虾生长发育过程中的作用,为完善脊尾白虾生长发育调控机理提供基础资料,也为进一步研究该类基因在脊尾白虾生长和发育中的作用奠定基础。
1 材料与方法 1.1 实验样品采集实验用脊尾白虾取自日照,体长(44.15±3.02)mm,体重(1.36±0.42)g,暂养一周。养殖水温(25±1)℃,盐度31,pH=8.2,持续充氧,每天换水清污一次,换水量为1/3,投喂配合饲料。取10尾健康的脊尾白虾各组织,包括肌肉、眼柄、甲壳(不带表皮)、胃、心脏、肠道、血细胞、卵巢、鳃和肝胰脏于液氮中研磨,用于RNA的提取。
梁象秋等[17]对脊尾白虾幼体发育特征进行描述,其中溞状幼体Ⅱ期时,其头胸甲出现眼上刺和颊刺,眼柄延长,并与头胸甲分离,可以转动。在溞状幼体时期,脊尾白虾在水中倒向运动,转为仔虾后开始腹部向下的向前运动。本实验选取溞状幼体Ⅰ期至Ⅵ期的脊尾白虾,转为仔虾时期第1天、第5天和第10天的脊尾白虾各6尾,置于液氮中研磨,用于RNA的提取。脊尾白虾孵化为溞状幼体的第1天记为1日龄,在仔虾60、80、100和120日龄分别选择极大个体和极小个体各10尾,测量其体长体重后(见表 1),分为极大个体组(H组)和极小个体组(L组),分组取腹部肌肉组织混合后置于液氮中研磨,用于RNA的提取。
|
|
表 1 脊尾白虾大小差异个体组生长性状比较 Table 1 Comparison on body length and weight traits of E. carinicauda between heavier group and lighter group |
通过Trizol法提取脊尾白虾各组织的总RNA,利用紫外分光光度计与琼脂糖凝胶电泳等方式对提取的总RNA质量进行检测。选择质量及完整性较好的肌肉组织的总RNA,使用SMARTTMRACEcDNA Amplification Kit分别合成3’和5’RACE的cDNA第一链。从脊尾白虾转录组文库中获得EcMHC2、EcMLC1基因部分EST序列,利用PrimerPremier 6.0软件设计中间片段引物验证其准确性后,设计3’和5’RACE特异性引物(见表 2)。3’和5’RACE引物分别与UPM配对进行cDNA的3’和5’末端扩增。参照核酸凝胶回收试剂盒说明书,经1.5%琼脂糖凝胶电泳后通过切胶回收得到扩增目的片段,将目的片段与pEASY-T1载体连接,然后转入到感受态细胞TransT1中,过夜培养后,通过蓝白斑筛选挑出阳性单克隆,经PCR检测合格后送测序。
|
|
表 2 本研究所用引物序列 Table 2 Primers used in the study |
利用DNAStar软件SeqMan程序进行拼接序列的拼接与组装,得到2个基因的cDNA全长。NCBI ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)确定其开放阅读框并预测氨基酸序列,利用BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性比对。用ProtParam工具(http://web.expasy.org/protparam/)对推测蛋白进行基本理化参数分析;根据PredictProtein工具(https://www.predictprotein.org/)分析蛋白二级结构及功能;InterProScan工具(http://www.ebi.ac.uk/interpro/search/sequence-search)分析结构域。利用MEGA 6.0软件构建NJ系统进化树(Bootstraps设为1 000次)。
1.4 反转录实时荧光定量PCR(RT-qPCR)将脊尾白虾肌肉、眼柄、甲壳、胃、心脏、肠道、血细胞、卵巢、鳃和肝胰脏等组织及不同幼体发育时期、不同生长阶段的大个体组和小个体组的肌肉组织提取总RNA,按照TakaraPrime ScriptTMRTreagentKit说明书反转录合成cDNA。
Duan等[18]研究发现脊尾白虾18SrRNA作为内参基因具有稳定性,本研究根据脊尾白虾18SrRNA(登录号:GQ369794)序列设计荧光定量引物并利用geNorm程序,并对18SrRNA内参基因的表达稳定性进行统计学分析,计算出基因表达稳定性的平均值M,通常M<1.5为选择阈值,认为内参基因表达稳定性较好。本实验中结果显示不同组织间的M值为0.435,不同幼体生长发育阶段的M值为0.600,大小差异个体不同生长阶段的M值为0.479,表明选择脊尾白虾18SrRNA作为内参基因具有较高的稳定性。
根据脊尾白虾EcMHC2、EcMLC1和内参基因18SrRNA的开放阅读框cDNA序列,由上海生工生物工程股份有限公司设计并合成荧光定量引物(见表 2)。参照TaKaRa SYBR Premix Ex TaqⅡ说明书,利用ABI 7500 Real Time PCR仪器进行荧光定量分析。10 μL反应体系包括: 5 μL SYBR Premix Ex TaqTM Ⅱ (2×)、0.2 μL ROX Reference Dye Ⅱ (50×)、0.4 μL 10 μmol/L的正反向引物、3.0 μL去离子水、1.0 μL cDNA。PCR反应程序为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 34 s,收集荧光信号,共40个循环;溶解曲线分析:95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s,每个循环增加0.5 ℃,收集荧光信号。
标准曲线法计算18SrRNA、EcMHC2、EcMLC1的荧光定量引物扩增效率,根据公式E=10(-1/S)-1(S为标准曲线的斜率),计算可得其扩增效率分别为103.2%、101.3%和102.7%。每个实验组设置3次重复,采用2-ΔΔCt法进行相对表达分析,数据处理使用SPSS 22.0软件进行差异显著性等统计学分析,利用Origin Pro 9.0对统计结果进行作图,P<0.05表示差异显著。
2 结果 2.1 EcMHC2、EcMLC1的cDNA全长序列的获得及氨基酸序列分析3’RACE和5’RACE扩增产物分别测序后与中间片段进行拼接,获得脊尾白虾肌球蛋白重链、肌球蛋白轻链基因的全长cDNA序列,分别命名为EcMHC2、EcMLC1,GenBank登录号为MG545144、MG545145。EcMHC2全长5 929 bp,包括127 bp的5’端非编码区(UTR),72 bp的3’端非编码区和5 727 bp的开放阅读框,编码1909个氨基酸,预测分子量大小为216.97 kD,理论等电点为5.62。EcMLC1全长1 506 bp,包括69bp的5’端非编码区(UTR),975bp的3’端非编码区和459 bp的开放阅读框,编码153个氨基酸,预测分子量大小为17.39 kD,理论等电点为4.64。3’端含有多聚腺苷酸加尾信号AATAAA和PolyA。
采用InterProScan分析蛋白结构域发现,与其它肌球蛋白的氨基酸结构相似,EcMHC2可分为头区(81-778 aa)和尾区(844-1906 aa),N端有一种选择性结合多脯氨酸配体的结构域Src同源区3(Src homology 3, SH3)(37-71 aa)(见图 1)。编码的EcMLC1氨基酸序列经InterProScan分析发现,在8-43aa处和82-117aa处各有1个可与Ca2+结合的EF-hand结构(见图 2)。Predict Protein分析发现EcMLC1具有46.41%的LOOP环和53.59%螺旋结构。

|
(ATG起始密码子;TAA终止密码子;AATAAA多腺苷酸信号;“*”表示终止密码子;划线部分是结构域Src同源区3;第一个实线框是头区,第二个实线框是尾区。ATG is a start codon; TAA is a stop codon; AATAAA is a consensus polyadenylation signal; ‘*’represents no translation amino acid.SH3 is underlined; Head region and tail region are boxed in the first and second region, respectively.) 图 1 脊尾白虾EcMHC2的cDNA核苷酸序列和推导氨基酸序列 Fig. 1 Nucleotide and deduced amino acid sequence of EcMHC2 cDNA |

|
(ATG起始密码子;TAA终止密码子;AATAAA多腺苷酸信号;“*”终止密码子;划线区是EF-hand结构。ATG is a start codon; TAA is a stop codon; AATAAA is a consensus polyadenylation signal; ‘*’represents no translation amino acid. EF-hand domain is underlined.) 图 2 脊尾白虾EcMLC1的cDNA核苷酸序列和推导氨基酸序列 Fig. 2 Nucleotide and deduced amino acid sequence of EcMLC1 cDNA |
通过BLAST同源性比对分析,发现脊尾白虾EcMHC2氨基酸序列与斑节对虾MHC2(GenBank登录号:BAM65720.1)的同源性最高为87%,因此本实验克隆所得肌球蛋白重链基因为Ⅱ型肌球蛋白重链的MHC2亚型;EcMLC1氨基酸序列与大西洋沟虾(Palaemonvarians)MLC1(GenBank登录号:ACR 54116.1)的同源性最高为96%,与克氏原鳌虾MLC1(GenBank登录号:ACR54116.1)的同源性为94%,因此,本实验克隆所得为Ⅱ型肌球蛋白碱性轻链的MLC1亚型。
利用MEGA 6.0软件对MHC、MLC进行系统进化分析,结果表明显示肌球蛋白重链和肌球蛋白轻链在进化上聚类为两大支脊索动物和无脊椎动物。脊尾白虾EcMHC2与斑节对虾等十足目甲壳动物聚为一支,EcMLC1与大西洋沟虾等聚为一枝,然后与昆虫分支聚为一大枝;脊索动物中被囊动物、鸟类、鱼类和哺乳动物聚为一支(见图 3)。

|
图 3 脊尾白虾EcMHC2(A)、EcMLC1(B)氨基酸序列NJ系统进化树 Fig. 3 Phylogenetic analysis (Neighbor Joining) of E. carinicauda and other deduced EcMHC2(A) andEcMLC1(B) amino acid sequences |
利用荧光定量的方法检测EcMHC2、EcMLC1在脊尾白虾各个组织中的表达情况(见图 4)。结果显示,EcMHC2、EcMHC2的组织表达量情况比较相似:在肌肉组织中的表达量最高,眼柄次之,而在其他组织中表达量均比较低(P<0.05)。

|
(不同小写字母表示EcMHC2和EcMLC1在不同组织的表达量差异显著(P<0.05)。Different lowercase letters indicate significant difference of EcMHC2, EcMLC1 mRNA expressions in different tissues (P<0.05).) 图 4 脊尾白虾EcMHC2和EcMLC1在各组织中的表达情况 Fig. 4 Relative expression levels of EcMHC2 and EcMLC1 mRNA in tissues of E. carinicauda |
如图 5所示,对比幼体发育各时期,受精卵时期脊尾白虾体内的EcMHC2、EcMLC1的表达量较低(P<0.05)。随着幼体的发育,脊尾白虾体内的EcMHC2、EcMLC1表达量呈现先升高再下降的趋势,在仔虾第1天达到最大(P<0.05),然后表达量显著下降。在脊尾白虾溞状幼体Ⅱ期EcMHC2、EcMLC1的表达量显著高于溞状幼体发育的其它各时期(P<0.05);在溞状幼体Ⅵ期、Ⅴ期,EcMHC2的表达量无显著性差异,但显著高于Ⅲ期、Ⅳ期(P<0.05)。EcMLC1的表达量在蚤状发育的5个时期(Ⅰ、Ⅲ、Ⅵ、Ⅴ和Ⅳ期)无显著性差异。

|
(Oo.受精卵时期;Z1.溞状幼体Ⅰ期;Z2.溞状幼体Ⅱ期;Z3.溞状幼体Ⅲ期;Z4.溞状幼体Ⅳ期;Z5.溞状幼体Ⅴ期;Z6.溞状幼体Ⅵ期;P1.仔虾第1天;P5.仔虾第5天;P10.仔虾第10天。不同小写字母表示EcMHC2、EcMLC1表达量在受精卵和幼体不同发育时期差异显著(P<0.05).Oo. Oosperm, Z1.zoeaⅠ, Z2. zoeaⅡ, Z3.zoeaⅢ, Z4.zoeaⅣ, Z5.zoeaⅤ, Z6.zoeaⅥ, P1.postlarva-1, P5.postlarva-5, P10.postlarva-10. Different lowercase letters indicate significant difference of EcMHC2 and EcMLC1 mRNA expressions during oosperm and different early larval development stages (P<0.05).) 图 5 脊尾白虾EcMHC2和EcMLC1在受精卵和幼体不同发育时期的表达 Fig. 5 Expression profile of EcMHC2 andEcMLC1 during oosperm and different early larval development stages |
|
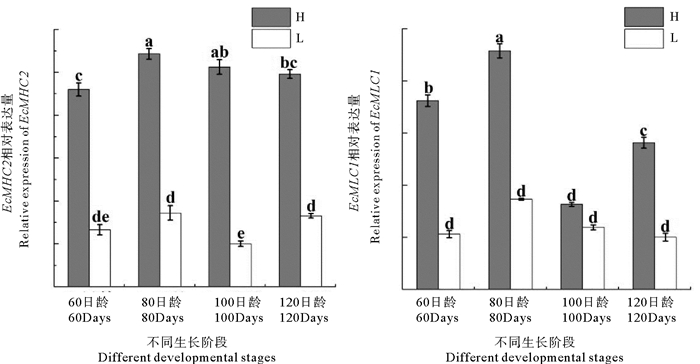
(不同小写字母表示EcMHC2、EcMLC1表达量在不同组别不同生长阶段差异显著(P<0.05)。Different lowercase letters indicate significant difference of EcMHC2 and EcMLC1 mRNA expressions during different group and stages (P<0.05).) 图 6 脊尾白虾EcMHC2和EcMLC1在大小差异个体不同生长阶段的表达 Fig. 6 Expression profile of EcMHC2 and EcMLC1 during different early development stages |
在脊尾白虾不同生长阶段,分别测定极大个体组(H组)和极小个体组(L组)的体长、体重(见表 1)。在脊尾白虾生长的四个阶段中,EcMHC2在H组的表达量均显著的高于L组;在60、80和120日龄时,H组的表达量均显著的高于L组;100日龄时,EcMLC1表达量在H组与L组无显著性差异。在H组和L组中,80日龄时EcMHC2、EcMLC1的表达量最高。
3 讨论 3.1 脊尾白虾EcMHC2、EcMLC1氨基酸序列分析本研究克隆获得脊尾白虾EcMHC2,具有Ⅱ型肌球蛋白家族蛋白的典型结构:由头部区域和尾部区域组成,具有SH3结构域、ATP结合位点等关键结构[19]。肌球蛋白重链的N-末端的头部区域保守性较强是肌球蛋白家族的主要功能域,是肌球蛋白“分子马达”功能的关键结构区,具有ATP酶活性位点及与肌动蛋白的结合位点,在肌肉收缩过程中,肌球蛋白重链和轻链相互缠绕形成肌球蛋白,通过使分子间构型变化使肌球蛋白与ATP结合,引起头部与肌动蛋白结合减弱而使ATP水解产生能量,供头部迁移、滑动[20]。对不同的氨基酸序列进行分析,发现尾部结构域差异较大,末端尾部呈卷曲螺旋形态,其主要在调节运动活动中起作用。
细胞中最广泛的存在的信号转导机制是Ca2+信号转导,一般是由具有EF-hand结构的蛋白参与执行的[21]。EF-hand是一种由螺旋区-环-螺旋(Helix-loop-helix motif,HLH)构型构成的Ca2+结合结构,通常成对出现。每个EF-hand结构域是由12个残基组成的环状结构及两侧各12个碱基组成的α螺旋组成,而位于中央的环区是肌球蛋白轻链与Ca2+的结合区域。分析EcMLC1氨基酸结构,发现与Ca2+结合的2个EF-hand结构。对比其它4种甲壳动物,包括大西洋沟虾、褐虾(Crangoncrangon)、克氏原鳌虾(Procambarusclarkii)和日本囊对虾(Marsupenaeusjaponicus)的MLC氨基酸序列发现,都存在2个EF-hand结构。EF-hands的结构是由一个钙离子结合域经过4次串联重复的方式进化而来。在高等脊椎动物中,肌球蛋白轻链和肌钙蛋白C由同一个祖先进化而来,均含有4个相似的能与Ca2+结合的结构域[22],而无脊椎动物的肌球蛋白轻链EF-hand结构,由于在进化过程中Ca2+结合所必需的氨基酸残基发生突变,导致蛋白中某些EF-hand结构失去了与Ca2+结合的能力[23-24]。
肌球蛋白是多基因家族具有不同的亚型,在哺乳动物中,已发现的肌球蛋白重链和肌球蛋白轻链分别有10余种亚型。甲壳动物中,MHC1和MHC2在多种虾类中发现,且可能与幼体的发育有关[15];MLC1和MLC3在碱性条件下容易从肌球蛋白分子上解离下来,故称碱性轻链[25],其参与肌球蛋白与肌动蛋白相互作用,而且MLC1和MLC3表达量的比值与肌肉收缩速率存在线性关系[26]。
3.2 脊尾白虾EcMHC2、EcMLC1表达分析脊尾白虾EcMHC2、EcMLC1具有组织表达的特异性,通过荧光定量PCR分析可以看出EcMHC2、EcMLC1主要在肌肉中表达,其次是眼柄、甲壳,而在其它组织的表达量较低。在其它的动物中MHC、MLC的表达模式也不尽相同,翘嘴鳜(Sinipercachuasti)MHC主要在红肌中表达量最高,在其余组织中表现为中低表达[27];凡纳滨对虾的MLC主要在肌肉中表达,其次是心脏和鳃组织,而在其它组织中的表达量较低[14];桔小实蝇(Bactroceradorsalis)MLC在胸部的肌肉表达量显著的高于其它组织[28]。MHC、MLC在不同的物种中主要的表达组织器官为肌肉组织的主要原因是肌球蛋白重链和肌球蛋白轻链是肌小节粗肌丝的组成成分,促进肌肉生长的重要基因,而次要的表达部位有所不同,可能的原因是是肌球蛋白的不同亚型的表达模式不同。
在无脊椎动物中,对于调控肌球蛋白基因表达的机制研究很少,而在哺乳动物和鱼类中的大量研究表明其主要受体内的一些重要的信号通路调节。研究表明钙调神经磷酸酶信号通路(CaN-NFAT)是一种受细胞内钙离子浓度调节的信号通路,经过一系列的信号转导后,NFAT进入细胞核内,与其他的辅助因子结合后作用到特异的靶基因,从而促使肌球蛋白基因的表达[29];MRF基因家族在调控肌球蛋白基因表达上起关键作用,在肌肉特异基因转录调控中,MyoD起着总开关的作用,特殊的结构域与肌球蛋白等基因的启动子区结合进而促进它们的转录激活[30];Myogenin作为一个转录调节因子,它的表达启动了一系列收缩蛋白如肌球蛋白重链的表达[31];研究表明不同肌球蛋白重链基因之间存在的拮抗和促进作用,MHC7b可以通过促进miR-499的表达从而抑制MHC7的表达[32],总之,肌球蛋白基因的表达受多种因子调控。
脊尾白虾幼体的发育过程主要分为6个阶段,在溞状幼体Ⅱ期脊尾白虾的特点是眼柄出现,并与头胸甲分离,可以自由的转动[17]。在脊尾白虾溞状幼体Ⅱ期EcMHC2、EcMLC1表达量显著高于溞状幼体发育的其它各时期,而在组织表达分析中EcMHC2、EcMLC1在眼柄中的表达量也较高,推测EcMHC2、EcMLC1参与了脊尾白虾眼柄发生过程。在转为仔虾的第一天,EcMHC2、EcMLC1的表达量显著的高于幼体发育时期,这是由于仔虾的形态习性与成虾相似,同溞状幼体的形态有很大的变化。变为仔虾后,脊尾白虾开始作水平游泳,游泳能力大大增强[33],此时EcMHC2、EcMLC1表达量的升高可能是由于其为肌肉生长提供动力。同时有研究发现,在斑节对虾幼体发育过程中,仔虾MHC2表达量显著高于幼体发育的其它时期[15];在脊椎动物胚胎发育过程中,MHC2表达量与肌纤维形成的过程具有一致性[34];在鳜鱼幼体发育过程中,MLC1表达呈现上升的趋势[35],暗示MHC2、MLC1可能在肌肉发育过程中起作用。
Hevroy等[36]发现大西洋鲑(Atlantic salmon)的特定生长率的高低与MHC的表达量有关;陈之航等[37]研究发现在翘嘴鳜早期生长阶段中,MHC在快长组中的表达量大于慢长组;Ren等[38]利用蛋白质学技术挖掘与三疣梭子蟹(Portunustrituberculatus)生长相关的基因,发现MHC、MLC在大个体组中表达上调;李文兰等[14]发现凡纳滨对虾MLC的表达量在高速生长时期时显著高于其它生长时期。本实验中,在脊尾白虾孵化后60、80、100和120日龄,发现EcMHC2在极大个体组的表达量均显著的高于极小个体组。在脊尾白虾的4个生长阶段中,2个组的EcMHC2、EcMLC1表达量最高均在80日龄的脊尾白虾。这些结果进一步说明了EcMHC2、EcMLC1表达水平的变化与物种的生长进程以及生长速度有极大的相关性,为肌肉的生长提供分子基础,但MHC、MLC在生长发育过程中的作用机理有待进一步的研究。
4 结语本研究克隆了脊尾白虾EcMHC2、EcMLC1基因cDNA的全长序列,并分析其在不同组织、不同幼体发育时期的表达情况,以及大小差异个体在不同生长阶段的表达变化。研究结果表明EcMHC2、EcMLC1在脊尾白虾肌肉生长发育过程中发挥重要作用,然而其作用机理有待进一步研究。本研究为脊尾白虾肌球蛋白功能的后续研究奠定基础,同时为脊尾白虾生长相关基因的挖掘提供重要参考依据。
| [1] |
Medler S, Mykles D L. Muscle Structure, Fiber Types, and Physiology[M]. Oxford: Natural History of Crustacea, 2015.
(  0) 0) |
| [2] |
Jung H, Lyons R E, Hurwood D A, et al. Genes and growth performance in crustacean species: A review of relevant genomic studies in crustaceans and other taxa[J]. Reviews in Aquaculture, 2013, 5(2): 77-110. DOI:10.1111/raq.2013.5.issue-2
(  0) 0) |
| [3] |
阎斌伦, 薛蓓, 赵莲, 等. 甲壳类生长相关基因研究进展[J]. 海洋湖沼通报, 2016(4): 97-104. Yan Bin Lun, Xue Bei, Zhao Lian, et al. Progress on the research of the growth-related genes in crustaceans[J]. Transactions of Oceanology and Limnology, 2016(4): 97-104. (  0) 0) |
| [4] |
Ge Q Q, Li J, Duan Y F, et al. Isolation of prawn (Exopalaemon carinicauda) lipopolysaccharide and β-1, 3-Glucan binding protein gene and its expressionin responding to bacterial and viral infections[J]. Journal of Ocean University of China, 2016, 15(2): 288-296. DOI:10.1007/s11802-016-2741-2
(  0) 0) |
| [5] |
Knight A E, Molloy J E. Muscle, myosin and single molecules[J]. Essays in Biochemistry, 2000, 35(35): 43.
(  0) 0) |
| [6] |
Heissler S M, Sellers J R. Various themes of myosin regulation[J]. Journal of Molecular Biology, 2016, 428(9): 1927-1946. DOI:10.1016/j.jmb.2016.01.022
(  0) 0) |
| [7] |
Santos M, Moura R S, Gonzaga S, et al. Embryonic essential myosin light chain regulates fetal lung development in rats[J]. American Journal of Respiratory Cell & Molecular Biology, 2007, 37(3): 330-338.
(  0) 0) |
| [8] |
Andruchov O, Andruchova O, Wang Y, et al. Dependence of cross-bridge kinetics on myosin light chain isoforms in rabbit and rat skeletal muscle fibres[J]. The Journal of Physiology, 2006, 571(1): 231-242. DOI:10.1113/jphysiol.2005.099770
(  0) 0) |
| [9] |
Heissler S M, Sellers J R. Kinetic adaptations of myosins for their diverse cellular functions[J]. Traffic, 2016, 17(8): 839-859. DOI:10.1111/tra.2016.17.issue-8
(  0) 0) |
| [10] |
Berg J S, Powell B C, Cheney R E. A millennial myosin census[J]. Molecular Biology of the Cell, 2001, 12(4): 780. DOI:10.1091/mbc.12.4.780
(  0) 0) |
| [11] |
Kim G D, Yang H S, Jeong J Y. Comparison of myosin heavy chain content determined by label-free quantification between porcine longissimus thoracis, psoas major and semimembranosus muscles[J]. Food Research International, 2017, 10: 504-513.
(  0) 0) |
| [12] |
Kang J H, Bang W Y, Eunjung K, et al. Expression of porcine myosin heavy chain 1 gene in Berkshire loins with a high pH24 value[J]. African Journal of Biotechnology, 2016, 9(44): 7568-7572.
(  0) 0) |
| [13] |
Asaduzzaman M, Akolkar D B, Kinoshita S, et al. The expression of multiple myosin heavy chain genes during skeletal muscle development of torafuguTakifugurubripes embryos and larvae[J]. Gene, 2013, 515(1): 144-154. DOI:10.1016/j.gene.2012.10.089
(  0) 0) |
| [14] |
李文兰, 盛小伟, 高永华, 等. 凡纳滨对虾肌球蛋白轻链基因MLC的克隆及表达分析[J]. 西南农业学报, 2011, 24(4): 1558-1563. Li W L, Sheng X W, Gao Y H, et al. Clone and expression analysis of myosin light chain in Litopenaeus vannamei[J]. Southwest China Journal of Agricultural Sciences, 2011, 24(4): 1558-1563. DOI:10.3969/j.issn.1001-4829.2011.04.070 (  0) 0) |
| [15] |
Koyama H, Akolkar D B, Piyapattanakorn S, et al. Cloning, expression, and localization of two types of fast skeletal myosin heavy chain genes from black tiger and Pacific white shrimps[J]. Journal of Experimental Zoology Part A Ecological Genetics & Physiology, 2012, 317(10): 608-621.
(  0) 0) |
| [16] |
蒲红双, 高祥刚, 侯红漫, 等. 甲壳动物蜕皮相关基因的研究进展[J]. 水产科学, 2015, 34(6): 391-398. Pu S H, Gao X G, Hou H M, et al. Advances in Genes Related to Molting in Crustacean[J]. Fisheries Science, 2015, 34(6): 391-398. (  0) 0) |
| [17] |
梁象秋, 李亚娟, 周昭曼. 脊尾白虾的幼体发育[J]. 水产学报, 1988, 12(2): 157-168. Liang X Q, Li Y J, Zhou Z M. The larval development of Exopalaemon carinicauda[J]. Journal of Fisheries of China, 1988, 12(2): 157-168. (  0) 0) |
| [18] |
Duan Y, Liu P, Li J, et al. cDNA cloning, characterization and expression analysis of peroxiredoxin 5 gene in the ridgetail white prawn Exopalaemon carinicauda[J]. Molecular Biology Reports, 2013, 40: 6569-6577. DOI:10.1007/s11033-013-2702-4
(  0) 0) |
| [19] |
Gordon A M, Homsher E, Regnier M. Regulation of contraction in striated muscle[J]. Physiological Reviews, 2000, 80(2): 853-924. DOI:10.1152/physrev.2000.80.2.853
(  0) 0) |
| [20] |
Rayment I, Holden H M, Whittaker M, et al. Structure of the actin-myosin complex and its implications for muscle contraction[J]. Science, 1993, 261(5117): 58-65. DOI:10.1126/science.8316858
(  0) 0) |
| [21] |
Kawasaki H, Kretsinger R H. Structural and functional diversity of EF-hand proteins: Evolutionary perspectives[J]. Protein Science a Publication of the Protein Society, 2017, 26(10): 1898-1920. DOI:10.1002/pro.3233
(  0) 0) |
| [22] |
陈剑清, 张耀洲. EF手图像超家族成员——肌钙蛋白C的研究进展[J]. 生物工程学报, 2007, 23(3): 375-380. Chen J Q, Zhang Y Z. The Research Progress of One Member of the EF-handSuperfamily—Troponin C[J]. Chinese Journal of Biotechnology, 2007, 23(3): 375-380. DOI:10.3321/j.issn:1000-3061.2007.03.003 (  0) 0) |
| [23] |
Kalyanasundaram A, Santiago T C. Identification and characterization of new allergen troponin C (Pen m 6.0101) from Indian black tiger shrimp Penaeus monodon[J]. European Food Research & Technology, 2015, 240(3): 509-515.
(  0) 0) |
| [24] |
Tanaka H, Takahashi H, Ojima T. Ca2+-binding properties and regulatory roles of lobster troponin C sites Ⅱ and Ⅳ[J]. Febs Letters, 2013, 587(16): 2612. DOI:10.1016/j.febslet.2013.06.042
(  0) 0) |
| [25] |
Pette D, Staron R S. Myosin isoforms, muscle fiber types, and transitions[J]. Microscopy Research & Technique, 2015, 50(6): 500-509.
(  0) 0) |
| [26] |
Lowey S, Trybus K M. Role of skeletal and smooth muscle myosin light chains[J]. Biophysical Journal, 1995, 68(4): 120S.
(  0) 0) |
| [27] |
农小献, 赵发兰, 陈敦学, 等. 翘嘴鳜肌球蛋白轻链3b基因(MLC3b)的分子克隆和特征分析[J]. 基因组学与应用生物学, 2010, 29(6): 1033-1038. Nong X X, Zhao F L, Chen D X, et al. Molecular cloning and characteristic analysis of myosin light chain 3b(MLC3b) gene from Siniperca chuasti[J]. Genomics and Applied Biology, 2010, 29(6): 1033-1038. DOI:10.3969/gab.029.001033 (  0) 0) |
| [28] |
申建梅, 胡黎明, 宾淑英, 等. 桔小实蝇肌球蛋白轻链2基因的克隆及表达分析[J]. 昆虫学报, 2011, 54(5): 508-514. Shen J M, Hu L M, Bin S Y, et al. Cloning and expression profiling of myosin light chain 2 gene in Bactrocera dorsalis[J]. Acta Entomologica Sinica, 2011, 54(5): 508-514. (  0) 0) |
| [29] |
Chen X, Luo Y, Huang Z, et al. Akirin2 promotes slow myosin heavy chain expression by CaN/NFATc1 signaling in porcine skeletal muscle satellite cells[J]. Oncotarget, 2017, 8(15): 25158-25166.
(  0) 0) |
| [30] |
Gossett L A, Kelvin D J, Sternberg E A, et al. A new myocyte-specific enhancer-binding factor that recognizes a conserved element associated with multiple muscle-specific genes[J]. Molecular & Cellular Biology, 1989, 9(11): 5022-5033.
(  0) 0) |
| [31] |
Myer A, Olson E N, Klein W H. MyoD cannot compensate for the absence of myogenin during skeletal muscle differentiation in murine embryonic stem cells[J]. Developmental Biology, 2001, 229(2): 340-350. DOI:10.1006/dbio.2000.9985
(  0) 0) |
| [32] |
Rooij E V, Quiat D, Johnson B A, et al. A family of micro RNAs encoded by myosin genes governs myosin expression and muscle performance[J]. Developmental Cell, 2009, 17(5): 662-673. DOI:10.1016/j.devcel.2009.10.013
(  0) 0) |
| [33] |
王绪峨. 脊尾白虾的幼体发育[J]. 海洋科学, 1991, 15(1): 12-14. Wang X E. The Larval Development of Exopalaemon carinicauda[J]. Marine Science, 1991, 15(1): 12-14. (  0) 0) |
| [34] |
李艳艳, 邵芳, 吴小娟, 等. 鹅肌球蛋白重链1基因MyHC1全长cDNA克隆、序列及胚胎期表达特征分析[J]. 农业生物技术学报, 2015, 23(3): 359-368. Li Y Y, Shao F, Wu X J, et al. Molecular cloning, sequence and expression characterization of myosin heavy chain 1 genein goose (Anseranser) at embryonic period[J]. Journal of Agricultural Biotechnology, 2015, 23(3): 359-368. DOI:10.3969/j.issn.1674-7968.2015.03.010 (  0) 0) |
| [35] |
周瑞雪, 黄斌, 蒙涛, 等. 鳜碱性肌球蛋白轻链基因cDNA的克隆及其发育表达分析[J]. 水生生物学报, 2009, 34(5): 927-934. Zhou R X, Shao F, Wu X J, et al. Cloning and ontogenetic expression analysis of the alkali myosin light chain gene in Siniperca chuasti[J]. Acta Hydrobiologica Sinica, 2009, 34(3): 927-934. (  0) 0) |
| [36] |
Hevroy E M, Jordal A E O, Hordvik I, et al. Myosin heavy chain mRNA expression correlates higher with muscle protein accretion than growth in Atlantic salmon, Salmosalar[J]. Aquaculture, 2006, 252(2-4): 453-461. DOI:10.1016/j.aquaculture.2005.07.003
(  0) 0) |
| [37] |
陈之航, 董浚键, 孙成飞, 等. 基于转录组测序对翘嘴鳜(Sinipercachuatsi)2种肌球蛋白重链基因的克隆与分析[J]. 渔业科学进展, 2017, 38(3): 51-61. Chen Z H, Dong J J, Sun F C, et al. cDNA cloning and analyses of Two myosin heavy chain isoforms of mandarin fish (Sinipercachuatsi) based on transcriptome sequencing[J]. Progress in Fishery Sciences, 2017, 38(3): 51-61. (  0) 0) |
| [38] |
Ren X, Yu X, Gao B, et al. iTRAQ-based identification of differentially expressed proteins related to growth in the swimming crab, Portunus trituberculatus[J]. Aquaculture Research, 2017, 48(6): 1-11.
(  0) 0) |
2. Function Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China;
3. Key Laboratory for Sustainable Utilization of Marine Fisheries Resources of Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China
 2019, Vol. 49
2019, Vol. 49


