2. 青岛海洋科学与技术试点国家实验室 海洋药物与生物制品功能实验室,山东 青岛 266237
芳基硫酸酯酶(Arylsulfatase, E.C.3.1.6.1)是一类断裂硫酸酯键、生成无机硫酸盐类、参与调节代谢的酶,广泛分布于自然界、细菌、哺乳动物中,结构较为保守[1]。目前已报道的芳基硫酸酯酶种类繁多,如来源于Pseudomonas aeruginosa[2]、Kluyveromyces lactis[3]、Fusarium proliferatum LE1[4]及来源于人的芳基硫酸酯酶[5],其在土壤环境评价、水治理、医学方面都有应用价值,具有较好的发展前景。
琼胶是红藻类细胞壁的主要组成成分,常使用江蓠、石花菜、紫菜等作为原料来进行生产。琼胶具有良好的凝胶性及稳定性,被广泛应用于食品、医药、科学等领域。琼胶品质取决于琼胶的凝胶强度,而琼胶的凝胶强度取决于其中硫酸基的含量,硫酸基会在琼胶凝胶形成的过程中阻碍分子交联,降低琼胶的凝胶强度,因此脱除硫酸基对改善琼胶品质具有重要意义[6]。目前工业上多采用碱处理法对琼胶上的硫酸基进行脱除,但是碱处理法存在反应条件剧烈、琼脂糖产率低、制备出的产品琼胶色泽差,环境污染严重等问题[7]。
芳基硫酸酯酶的反应机制可应用于脱除琼胶中的硫酸基,相较于碱处理来说酶法脱硫的反应条件更加温和、环境友好,此外芳基硫酸酯酶还可以脱除碱处理法不能脱除的琼胶中琼脂糖前体C4位上的硫酸基,用于制备更高价值的产品如低电渗的琼脂糖、色谱树脂等[8]。Wang等从Marinomonas sp. FW-1中分离纯化得到的芳基硫酸酯酶,对于琼胶上硫酸基的脱除率为89.61%[9]。Kim等从Sphingomonas sp. AS6330中分离纯化得到的芳基硫酸酯酶对于琼胶上的硫酸基的脱除效果可达97.7%,这是目前报道的酶法脱除琼胶硫酸基的最高脱除率。然而,尽管来源于Sphingomonas sp. AS6330的芳基硫酸酯酶脱硫率很高,但其以琼胶为底物时的酶活(3.93 U)仅为以对硝基苯硫酸酯(p-nitrophenyl sulfate, p NPS)为底物时酶活(97.2 U) 的4%[10]。到目前为止已报道的芳基硫酸酯酶对琼胶的酶活都很低。因此,扩大芳基硫酸酯酶基因库,寻找以琼胶为底物时酶活较高的芳基硫酸酯酶对推进酶法进行琼胶的高效脱硫,将酶法琼胶脱硫从实验室规模推向产业化,实现高品质琼胶与琼脂糖的规模化生产具有重要意义。
嗜琼胶华美菌(Formosa agariphila) KMM 3901可高效降解琼胶,其基因组含多种蛋白酶及糖苷水解酶,其中糖苷水解酶通常与转运因子、转运蛋白和硫酸酯酶共同形成独特的多糖利用位点,这说明硫酸酯酶对于硫酸化海藻多糖的降解至关重要[11]。因此,嗜琼胶华美菌KMM 3901中可能存在有重要价值的芳基硫酸酯酶。
本研究从嗜琼胶华美菌KMM 3901基因组中发掘出一段芳基硫酸酯酶目的基因,并构建大肠杆菌工程菌用于异源表达该芳基硫酸酯酶片段,对表达的芳基硫酸酯酶进行分离纯化和酶学性质研究,最后探究了其在脱除琼胶上硫酸基中的应用。
1 材料和方法 1.1 试剂材料 1.1.1 菌株及质粒嗜琼胶华美菌KMM 3901购自中国普通微生物菌种保藏管理中心(菌株编号:1.12423);大肠杆菌(Escherichia coli) DH5α、大肠杆菌BL21(DE3)、表达载体pET-28a(+)均购自北京天根生化科技有限公司。
1.1.2 试剂KOD高保真酶购自日本东洋纺生物科技有限公司;胶回收试剂盒Gel Extraction Kit (200) 购自美国OMEGA公司;PCR回收试剂盒Gel Extraction Kit (200) 购自美国OMEGA公司;Taq聚合酶混合体系2×Taq Master Mix购自诺唯赞生物科技有限公司;快速质粒小提试剂盒TIANprep Rapid Mini Plasmid Kit购自北京天根生化科技有限公司;DNA标准分子量Marker和蛋白质标准分子量Marker购自索莱宝生物科技有限公司;卡那霉素购自上海生工生物工程有限公司;琼脂粉购自福建绿新集团;其他试剂均为国产分析纯。
1.1.3 培养基(1) LB培养基:用电子天平按比例称取1%蛋白胨、1% NaCl、0.5%酵母粉,加一定蒸馏水搅拌溶解,分装,于121 ℃高温灭菌20 min,取出备用。
(2) ZYP-5052培养基:按比例称量0.2% 1 mol/L MgSO4,0.5% 酵母粉,1% 蛋白胨,5% 20×P(1 mol/L Na2HPO4,1 mol/L KH2PO4,0.5 mol/L (NH4)2SO4),加一定蒸馏水搅拌溶解,分装,于121 ℃高温灭菌20 min。在诱导目的菌种表达时,还应加入2%的已灭菌50×5052母液(25%甘油,2.5%葡萄糖,10% α-半乳糖)。
1.2 方法 1.2.1 目的基因的发掘与序列分析通过NCBI基因库从F. agariphila KMM 3901中找到一段芳基硫酸酯酶编码基因 ouc-fars6,使用Expasy在线网站预测该序列的蛋白大小,使用SignalP 5.0在线网站预测该序列是否存在信号肽,使用SMART在线网站对该序列翻译的蛋白可能存在的结构域进行预测,使用TMHMM Server v.2.0在线网站预测该序列翻译的蛋白是否存在跨膜结构域,使用MEGA 6.06软件制作进化树,使用clustalx软件对该基因序列进行多序列比对,并用ESPript 3.0在线网站将多序列比对结果输出。
1.2.2 产芳基硫酸酯酶重组质粒的构建使用2216E海水培养基培养F. agariphila KMM 3901,利用试剂盒提取菌株基因组。根据目的基因序列和pET-28a(+) 质粒图谱,分别设计上下游引物,如表 1所示,分别以F. agariphila KMM 3901和pET-28a(+)质粒为模板,用各自上下游引物进行目的基因和载体的PCR扩增和同源臂的构建。
|
|
表 1 引物名称及序列 Table 1 Primer names and sequences |
PCR结束后,用琼脂糖凝胶电泳对其进行分离验证,分别用PCR回收试剂盒和胶回收试剂盒回收条带大小正确的扩增目的基因和扩增载体。用无缝拼接试剂盒连接目的基因和载体,构建重组质粒pET-28a(+)- OUC-FARS6,并采用热激法,将连接后的重组质粒转入感受态细胞E. coli DH5α中,经抗性平板筛选后,进行阳性克隆验证及相应的序列测序。
1.2.3 产芳基硫酸酯酶重组质粒在E. coli BL21(DE3)中的表达用质粒提取试剂盒从测序结果正确的E. coli DH5α中提取重组质粒,重新转化入E. coli BL21(DE3)中,将转化成功的工程菌活化后,接种至ZYP-5052培养基中,20 ℃,220 r/min,培养48 h进行目的蛋白的诱导表达。发酵结束后,4 ℃,8 000 r/min冷冻离心15 min分离菌体和上清液。加入15 mL PBS缓冲液(pH=8.0),悬浮菌体,超声破碎30 min后,在4 ℃,12 000 r/min冷冻离心15 min去除不可溶成分,即可得到含目的蛋白的粗酶液。
1.2.4 芳基硫酸酯酶OUC-FARS6的纯化将上述步骤得到的粗酶液用镍柱进行纯化,使用含有不同浓度咪唑(10~500 mol/L)PBS缓冲液溶液进行蛋白的梯度洗脱,每个梯度洗脱至无蛋白洗下。分别收集洗脱后的溶液,进行SDS-PAGE蛋白电泳分析,并使用超滤浓缩管对有酶活的洗脱液进行浓缩脱盐,即可得到纯酶。
1.2.5 芳基硫酸酯酶OUC-FARS6的酶活测定测定芳基硫酸酯酶酶活的体系为25 μL芳基硫酸酯酶酶液、50 μL 25 mmol/L p NPS溶液以及100 μL 50 mmol/L Glycine-NaOH缓冲液(pH=9.0),总体积为175 μL。将上述混合溶液置于45 ℃反应90 min,最后加入75 μL 1 mol/L NaOH终止反应。取200 μL上述反应液于96孔板中,用酶标仪测定410 nm下的吸光值,共3组平行。其反应原理是芳基硫酸酯酶可以断裂p NPS的硫酸酯键,释放对硝基苯酚(p-nitrophenol, p NP),使溶液由无色变为黄色。芳基硫酸酯酶OUC-FARS6酶活单位(U/mg)定义为1 min产生1 μmol p NP所需的酶量。
1.2.6 芳基硫酸酯酶OUC-FARS6的酶学性质测定 1.2.6.1 最适温度和温度稳定性将按1.2.4中方法纯化后的酶液、p NPS溶液与pH=9.0的50 mmol/L Glycine-NaOH缓冲液混合,分别放置于25、30、35、40、45、50、55、60、65和70 ℃中反应90 min,最后加入1 mol/L NaOH终止反应,测定OD410 nm,具体测定方法如1.2.5中所述。
将上述纯化后的酶液与pH=9.0 50 mmol/L Glycine-NaOH缓冲液混合,置于4、25、35、45、50和55 ℃分别孵育0、2、10、24、36、48、60和72 h后,加入底物p NPS溶液后反应90 min,最后加入1 mol/L NaOH终止反应,测定OD410 nm,确定该酶在各温度下的稳定性,具体测定方法如1.2.5中所述。
1.2.6.2 最适pH和pH稳定性将上述纯化后的酶液、p NPS溶液分别与pH为3.0~6.0的50 mmol/L柠檬酸缓冲液、pH为6.0~8.0的50 mmol/L PB缓冲液、pH为7.0~9.0的50 mmol/L Tris-HCl缓冲液及pH为9.0~10.0的50 mmol/L Glycine-NaOH缓冲液混合,置于45 ℃反应90 min,最后加入1 mol/L NaOH终止反应。测定OD410 nm,具体测定方法如1.2.5中所述。
将上述纯化后的酶液分别与pH为3.0~6.0的50 mmol/L柠檬酸缓冲液、pH为7.0~8.0的50 mmol/L Tris-HCl缓冲液及pH为9.0~10.0的50 mmol/L Glycine-NaOH缓冲液混合,置于4 ℃分别孵育0、2、10、24、36、48、60和72 h后,加入底物p NPS溶液后反应90 min,最后加入1 mol/L NaOH终止反应,测定OD410 nm,确定该酶在各个pH下的稳定性,具体测定方法如1.2.5中所述。
1.2.6.3 金属离子对酶活的影响向原有反应体系中加入不同金属离子Na+(NaCl)、K+(KCl)、Mg2+(MgCl2)、Ni2+(NiCl2)、Mn2+(MnCl2)、Ba2+(BaCl2)、Ca2+(CaCl2)、Zn2+(ZnCl2)、Co2+(CoCl2)、Co2+(CoCl2)、Fe3+(FeCl3)、SDS及Na2EDTA,使其终浓度为1.4和14 mmol/L,具体测定方法如1.2.5中所述。对照组为未加金属离子、SDS及Na2EDTA(乙二胺四乙酸二钠)的反应体系,探究不同浓度、不同金属离子条件下酶活的变化。
1.2.6.4 OUC-FARS6酶促反应动力学参数测定用50 mmol/L pH=9.0的50 mmol/L Glycine-NaOH缓冲液配制浓度分别为0、0.1、0.2、0.3、0.4、0.5、1.0、1.5、2.0、2.5、5.0、7.5、10.0、12.5、15.0、17.5、20.0、22.5和25.0 mmol/L的p NPS底物,45 ℃温育10 min后,分别将10 μL纯酶与140 μL上述不同浓度的底物混合,45 ℃下反应30 min后,加入100 μL 1 mol/L NaOH终止反应,测定OD410 nm。利用米氏方程将所得数据进行非线性回归拟合,计算Km和Vmax的值。
1.2.7 芳基硫酸酯酶OUC-FARS6在脱除琼胶硫酸基上的应用称取0.2 g琼脂粉,加入19 mL pH=9.0的50 mmol/L Glycine-NaOH缓冲液形成悬浊液,并加入1 mL纯化后(纯化方法见1.2.4)的芳基硫酸酯酶OUC-FARS6(0.014 U),混合完全(总体积为20 mL),置于磁力搅拌锅中,于35 ℃分别反应6和24 h。反应完毕后,置于70 ℃保持30 min终止反应。此外还探究了经35 ℃孵育10 h后的纯酶液与0.2 g琼脂粉在pH=9.0的50 mmol/L Glycine-NaOH缓冲液中按上述过程共同反应24 h的效果。以上3组反应的对照组为灭活的芳基硫酸酯酶,其他反应条件均与实验组相同。用DNS法和TLC法测量体系中是否有还原糖生成。将酶解后的琼脂粉清洗后冻干,用电子天平称量冻干琼脂粉质量,并加入10 mL 1 mmol/L HCl形成悬浊液,置于灭菌锅中115 ℃高温高压酸解4 h。结束后,酸解液用0.22 μm滤膜过滤,用离子色谱法测定溶液中游离硫酸根的含量(由菲优特检测公司完成),可得到脱硫后琼胶上剩余的硫含量。对照组为未处理的琼脂粉,其他条件均与实验组相同,经离子色谱法测定后,可得到琼胶本身的硫含量,二者结合即可得到琼胶脱硫率。取部分作为样品用于扫描电镜(SEM),观察芳基硫酸酯酶OUC-FARS6脱除琼胶硫酸基前后琼胶的微观变化。同时进行脱硫前后琼胶的傅里叶红外变换光谱(FI-IR)测定,用干燥的溴化钾与琼胶样品粉末一起研磨压片,波长400~4 000 cm-1内扫描光谱吸收值。
2 结果与分析 2.1 序列分析上传该酶的氨基酸序列,经SignalP 5.0在线网站预测,该序列第1~21个氨基酸为一段信号肽,信号肽在胞内异源表达中无用,因此在后续质粒构建中会将信号肽去除。SMART在线网站预测结果表明该序列确实存在信号肽,这与SignalP 5.0预测一致,除此之外该序列还存在一个硫酸酯酶特定功能域,表明该酶属于硫酸酯酶并具有断裂硫酸酯键的特性。TMHMM Server v.2.0在线网站预测结果表明该序列不存在跨膜结构域。使用MEGA 6.06软件制作的进化树如图 1a所示,结果表明OUC-FARS6与已报道的来源于Pyrococcus furiosus[12]的芳基硫酸酯酶处于同一分支。多序列比对结果如图 1b所示,结果表明OUC-FARS6与已报道芳基硫酸酯酶具有相同的保守氨基酸残基位点(C/S)-X-(A/P)-X-R-X4-T-G[13]。以上结果表明,该编码序列确实具有芳基硫酸酯酶的序列特征。

|
( a:OUC-FARS6进化树分析;b:多序列比对结果。WP_097046647.1,来自Flagellimonas pacifica的芳基硫酸酯酶;WP_034043253.1,来自Flaviramulus ichthyoenteri的芳基硫酸酯酶;CDF79639.1,来自Formosa agariphila KMM 3901的芳基硫酸酯酶;WP_084182146.1,来自Maribacter ulvicola的芳基硫酸酯酶;WP_126160703.1,来自Arenibacter aquaticus的芳基硫酸酯酶;WP_084812086.1,来自Flammeovirga pacificaa的芳基硫酸酯酶。a: Phylogenetic analysis of OUC-FARS6 with other arylsulfatases; b: Results of multiple sequence alignment. WP_097046647.1, arylsulfatase of Flagellimonas pacifica; WP_034043253.1, arylsulfatase of Flaviramulus ichthyoenteri; CDF79639.1, arylsulfatase of Formosa agariphila KMM 3901; WP_084182146.1, arylsulfatase of Maribacter ulvicola; WP_126160703.1, arylsulfatase of Arenibacter aquaticus; WP_084812086.1, arylsulfatase of Flammeovirga pacificaa. ) 图 1 进化树分析和多序列比对结果 Fig. 1 Evolutionary tree and results of multiple sequence alignment |
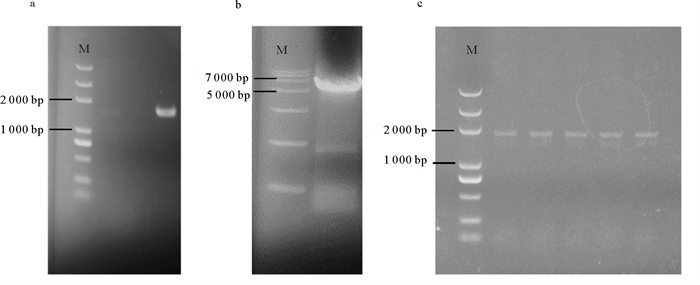
以F. agariphila KMM 3901基因组和pET-28a(+) 质粒为模板,利用表 1所述上下游引物进行扩增,去除该芳基硫酸酯酶片段上的信号肽,用琼脂糖凝胶电泳对扩增出的目的片段和载体进行验证纯化,如图 2a和图 2b所示,目的基因条带单一,大小在1 000~2 000 bp之间,载体条带也单一,大小介于5 000~7 000 bp之间。将目的基因和载体回收后,使用无缝拼接的方法构建重组质粒并转化入感受态细胞E. coli BL21(DE3)中,之后挑取抗性筛选平板上生长的单菌落进行阳性克隆验证,结果如图 2c所示,条带大小正确,且测序后序列正确,证明该目的基因已成功连接在质粒上并转入细菌体内。
|
( a:目的片段核酸电泳结果;b:载体核酸电泳结果;c:阳性克隆验证结果。a: Results of fragment nucleic acid electrophoresis; b: Results of carrier nucleic acid electrophoresis; c: Results of positive clones verification. ) 图 2 pET-28a(+)-OUC-FARS6重组质粒的PCR扩增及阳性克隆验证结果 Fig. 2 Results of PCR amplification and positive clone verification for recombinant plasmid pET-28a(+)-OUC-FARS6 |
按上述条件发酵结束后,离心收集菌体,超声破碎再次离心后,得到的上清液即是芳基硫酸酯酶OUC-FARS6的粗酶液,该粗酶液与p NPS反应后会变黄,证明OUC-FARS6成功表达且有酶活。破碎后的上清粗酶液通过镍柱纯化法分离纯化目的蛋白,其原理是表达目的蛋白时也会表达一段His-tag标签蛋白,该蛋白可以与镍柱结合,而不同浓度咪唑也可以与镍柱结合且结合亲和力不同,从而用不同浓度咪唑可将目的蛋白从镍柱上洗脱下来,实现目的蛋白的分离纯化。收集不同浓度咪唑镍柱洗脱液,并通过SDS-PAGE蛋白电泳观察条带大小,来确定目的蛋白的浓度和纯度,结果如图 3所示。当用100 mmol/L以上浓度咪唑洗脱时,蛋白条带单一且测定有酶活,为目的蛋白,分子量大小在63 kDa左右,与Expasy在线网站预测的58.7 kDa相差不大,说明该酶可以成功表达且可以成功纯化。经超滤浓缩置换后备用,用于测定其酶学性质。

|
( M:蛋白标准分子量;泳道1:粗酶液;泳道2:过Ni柱后的溶液;泳道3:10 mmol/L咪唑洗脱液;泳道4:20 mmol/L咪唑洗脱液;泳道5:50 mmol/L咪唑洗脱液;泳道6:80 mmol/L咪唑洗脱液;泳道7:100 mmol/L咪唑洗脱液;泳道8:200 mmol/L咪唑洗脱液;泳道9:500 mmol/L咪唑洗脱液。M: Molecular mass marker of proteins; Lane 1: Crude enzyme liquid; Lane 2: Through the column penetration fluid; Lane 3: 10 mmol/L imidazole eluent; Lane 4: 20 mmol/L imidazole eluent; Lane 5: 50 mmol/L imidazole eluent; Lane 6: 80 mmol/L imidazole eluent; Lane 7: 100 mmol/L imidazole eluent; Lane 8: 200 mmol/L imidazole eluent; Lane 9: 500 mmol/L imidazole eluent. ) 图 3 蛋白电泳结果 Fig. 3 Results of protein electrophoresis |
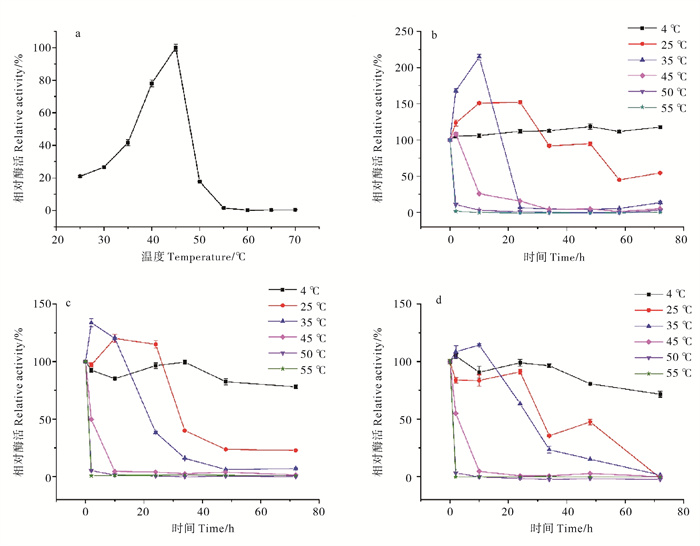
如图 4a所示,芳基硫酸酯酶以p NPS为底物时的最适温度为45 ℃(pH=9.0的50 mmol/L Glycine-NaOH缓冲液,反应90 min),温度在25~45 ℃时,酶活会随着温度的升高而逐渐升高,40~45 ℃时酶活维持在最高酶活的70%以上。之后随着温度继续升高,酶活会快速下降,60 ℃及以上温度,该酶失去活性。该酶在最适pH(pH=9.0的50 mmol/L Glycine-NaOH缓冲液)下温度对OUC-FARS6酶活稳定性影响如图 4b所示,可知该酶在4和25 ℃时,以p NPS为底物时的酶活较为稳定,孵育72 h仍存在100%和54%以上酶活。且35 ℃及以下温度,该酶会出现酶活上升的趋势,35 ℃孵育10 h后酶活可提升2倍多。为了验证是否是缓冲液中Na+对酶活的影响,分别在pH=9.0的Tris-HCl缓冲液和含1.4 mmol/L NaCl pH=9.0的50 mmol/L Tris-HCl缓冲液中测定该酶的温度稳定性,结果分别如图 4c和4d所示,可知该酶在Tris-HCl缓冲液中也会出现酶活上升的现象,加了Na+后这种效果并没有明显提升,故酶活的上升应该不是缓冲液中Na+的影响。经查阅文献[14]后发现,部分酶和芳基硫酸酯酶确实会出现与缓冲液孵育后酶活上升现象。
|
( a:最适温度的研究;b:在pH=9.0的50 mmol/L Glycine-NaOH缓冲液中的温度稳定性;c:在pH=9.0的50 mmol/L Tris-HCl的缓冲液中的温度稳定性;d:在含1.4 mmol/L NaCl pH=9.0的50 mmol/L Tris-HCl缓冲液中的温度稳定性。a: Effects of temperature on the activity of OUC-FARS6; b: Temperature stability in Glycine-NaOH buffer at pH=9.0; c: Temperature stability in a Tris-HCl buffer at pH=9.0; d: Temperature stability in a Tris-HCl buffer containing 1.4 mmol/L NaCl at pH=9.0. ) 图 4 OUC-FARS6最适温度及温度对酶活稳定性影响的探究 Fig. 4 Effects of temperature on the activity and thermostability of OUC-FARS6 |
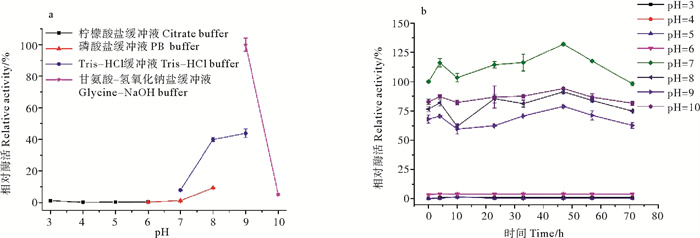
如图 5a所示,芳基硫酸酯酶以p NPS为底物时的最适pH体系为pH=9.0的50 mmol/L Glycine-NaOH缓冲液,根据pH对OUC-FARS6酶活稳定性影响(见图 5b)可看出,该酶在中性和碱性条件下(pH为8.0~10.0)稳定性很好,孵育72 h后仍能保持90%以上酶活,而在酸性条件下(pH为3.0~6.0)则快速失活。
|
( a:最适pH的研究;b:不同pH缓冲液对OUC-FARS6酶活稳定性的影响。a: Effects of pH on the activity of OUC-FARS6; b: Effects of pH on the stability of OUC-FARS6 in different pH buffers. ) 图 5 最适pH和pH对OUC-FARS6酶活稳定性的影响 Fig. 5 Effects of pH on the activity and stability of OUC-FARS6 |
如表 2所示,大部分金属离子在低浓度和高浓度下都对该酶有强烈的抑制作用,如Ca2+、Zn2+、Co2+、Cu2+、Fe3+、Ni2+、Mn2+。Ba2+和Mg2+在低浓度时对该酶活性有稍微弱的抑制作用,随金属离子浓度升高,这种抑制作用不断增强。仅Na+和K+两种一价阳离子对该酶的酶活有促进作用,在反应体系中添加1.4 mmol/L的Na+可以提高62.1%的酶活,而高浓度的Na+对酶活有抑制作用。而低浓度(1.4 mmol/L)和高浓度(14 mmol/L)的K+都对该芳基硫酸酯酶的酶活有促进作用,但是其促进效果不如低浓度Na+。此外,FARS6的活性可被Na2EDTA抑制,说明有金属离子存在于该酶催化位点附近,Na2EDTA可螯合金属离子从而进一步影响酶活。表面活性剂SDS在低浓度时可抑制94%的酶活,在高浓度时可抑制99%以上酶活。
|
|
表 2 金属离子对酶活的影响 Table 2 Effect of metal ions on OUC-FARS6 |
利用米式方程对数据进行非线性拟合,得出该酶的Km值为32.63 μmol/L,Vmax值为9.25 μmol/(L·min)。相比于来源于Pseudoalteromonas carrageenovora[15]和Sphingomonas sp. AS6330[10]的芳基硫酸酯酶的Km值来说(Km值分别为54.9和1 150 μmol/L),该酶的Km值较小,对底物p NPS的亲和力较好,但是比来源于Marinomonas sp. FW-1[9]的芳基硫酸酯酶的Km值略大(Km值为13.73 μmol/L)。经计算FARS6的比酶活为0.19 U/mg,相比于其他芳基硫酸酯酶来说比酶活偏低[16-17]。
2.5 对琼胶的脱硫作用琼胶粉末与芳基硫酸酯酶共同搅拌孵育24 h后(温度为35 ℃、pH=9.0的50 mmol/L Glycine-NaOH缓冲液),将琼胶粉末与体系分离、清洗后冷冻干燥,作为SEM和FT-IR的样品。SEM的结果如图 6所示,从不同放大倍数的图中均可看出,与空白对照组相比,经芳基硫酸酯酶作用后的样品内部空隙变小,呈现出较好的空间网状结构,通过更大的放大倍数也可以观察到其表面从光滑变得粗糙。由于OUC-FARS6在35 ℃孵育10 h后酶活会出现上升的趋势,因此也补充了一组先将芳基硫酸酯酶pH=9.0的50 mmol/L Glycine-NaOH缓冲液孵育10 h后再与琼胶共孵育酶解的实验组。通过SEM结果可以看出,直接酶解组比先孵育再酶解组琼胶样品内部空隙更小,效果更好,DNS法和TLC法测定结果表明,琼胶经OUC-FARS6孵育24 h后未有还原糖产生,因此证明琼胶微观结构的变化并不是琼胶降解,而是因为琼胶上硫酸基的脱除。

|
图 6 经芳基硫酸酯酶OUC-FARS6处理后琼胶的SEM结果 Fig. 6 SEM results of agar after treated with OUC-FARS6 |
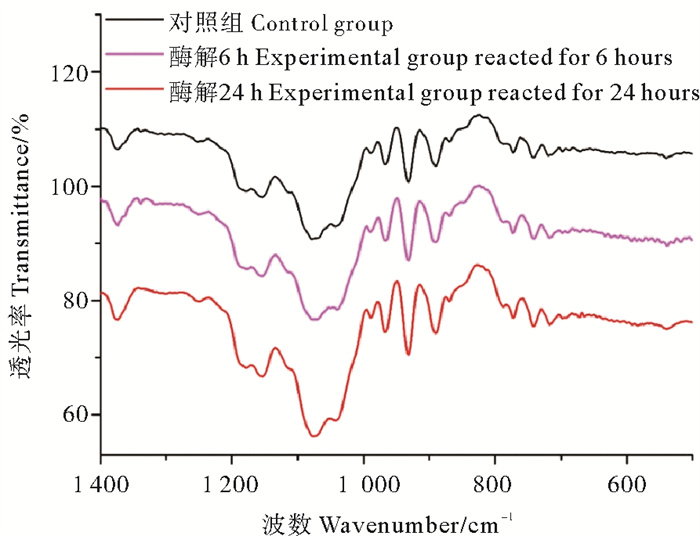
查阅文献[18]得出,850 cm-1处的吸收峰表示4-硫酸基-D-半乳糖C4位上存在硫酸基。由FT-IR的结果(见图 7)可知,芳基硫酸酯酶酶解琼胶6 h组和24 h组在850 cm-1处峰值减小,表明OUC-FARS6可用于脱除琼胶4-硫酸基-D-半乳糖C4位上的硫酸基。但由于所使用的琼胶原料本身的含硫量较低(0.66%),同时加酶量较低,反应条件还需优化等问题,使得峰值变化不是特别明显,脱除效果有待提升,经芳基硫酸酯酶酶解琼胶24 h后脱硫率仅为15.6%。
|
图 7 经芳基硫酸酯酶处理后琼胶的FT-IR图谱 Fig. 7 FT-IR spectra of agar after treated with OUC-FARS6 |
本研究通过基因工程技术构建重组质粒pET-28a(+)-OUC-FARS6,并以大肠杆菌为宿主成功异源表达出来源于嗜琼胶华美菌KMM 3901的芳基硫酸酯酶OUC-FARS6。该芳基硫酸酯酶以p NPS为底物时的最适温度为45 ℃(pH=9.0的50 mmol/L Glycine-NaOH缓冲液,反应90 min),在4 ℃和常温下酶活稳定。在pH=9.0的50 mmol/L Glycine-NaOH缓冲液中酶活最高,在中性和碱性环境(pH为8.0~10.0)中酶活稳定,在酸性条件下(pH为3.0~6.0)则快速失活。大部分金属离子、SDS和Na2EDTA都可抑制该酶酶活,低浓度的Na+和K+对酶活有促进作用。该酶的Km值为32.63 μmol/L,Vmax值为9.25 μmol/(L·min),对底物p NPS的亲和力较好。该酶可应用于脱除琼胶的硫酸基,SEM检测结果显示,经芳基硫酸酯酶孵育后的琼胶内部空隙变小,呈现出较好的空间网状结构;FT-IR检测结果显示,经芳基硫酸酯酶孵育后,琼胶上4-硫酸基-D-半乳糖上C4位硫酸基被脱除,这些都是琼胶凝胶强度增强的有利条件,能够提高琼胶的品质和价值。该酶相对于之前已经报道的芳基硫酸酯酶来说,稳定性更好,表达量较高,纯化效果更好,可用于制作酶制剂,适合应用于酶法脱除琼胶硫酸基的工业化生产。但目前仍存在酶活较低、脱硫率不高、以琼胶为底物时反应温度低于琼胶凝固温度等问题,后续应继续通过优化反应条件提高琼胶脱硫率,通过更科学的改造,获得催化活性与热稳定性均大幅提高的改造酶,将酶法琼胶脱硫从实验室推向产业化。
| [1] |
刘韩. 定向进化提高 Pseudoalteromonas carrageenovora 芳香基硫酸酯酶热稳定性的研究[D]. 厦门: 集美大学, 2016. Liu H. Thermostability Improvement of Pseudoalteromonas carrageenovora Arylsulfatase by Direct Evolution[D]. Xiamen: Jimei University, 2016. (  0) 0) |
| [2] |
Luo J, van Loo B, Kamerlin S C. Catalytic promiscuity in Pseudomonas aeruginosa arylsulfatase as an example of chemistry-driven protein evolution[J]. FEBS Letters, 2012, 586(11): 1622-1630. DOI:10.1016/j.febslet.2012.04.012
(  0) 0) |
| [3] |
Stressler T, Leisibach D, Lutz Wahl S, et al. Homologous expression and biochemical characterization of the arylsulfatase from Kluyveromyces lactis, and its relevance in milk processing[J]. Applied Microbiology and Biotechnology, 2016, 100(12): 5401-5414. DOI:10.1007/s00253-016-7366-2
(  0) 0) |
| [4] |
Shvetsova S V, Zhurishkina E V, Bobrov K S, et al. The novel strain Fusarium proliferatum, LE1(RCAM02409) produces α-L-fucosidase and arylsulfatase during the growth on fucoidan[J]. Journal of Basic Microbiology, 2015, 55(4): 471-479. DOI:10.1002/jobm.201400309
(  0) 0) |
| [5] |
Peters C, Schmidt B, Rommerskirch W, et al. Phylogenetic conservation of arylsulfatases cDNA cloning and expression of human arylsulfatase B[J]. Journal of Biological Chemistry, 1990, 265(6): 3374-3381. DOI:10.1016/S0021-9258(19)39778-9
(  0) 0) |
| [6] |
Tuvikene R, Truus K, Kollist A, et al. Gel-forming structures and stages of red algal galactans of different sulfation levels[J]. Journal of Applied Phycology, 2008, 20(5): 527-535. DOI:10.1007/s10811-007-9229-9
(  0) 0) |
| [7] |
González Leija J A, Hernández Garibay E, Pacheco Ruíz I, et al. Optimization of the yield and quality of agar from Gracilariopsis lemaneiformis (Gracilariales) from the Gulf of California using an alkaline treatment[J]. Journal of Applied Phycology, 2009, 21(3): 321-326. DOI:10.1007/s10811-008-9370-0
(  0) 0) |
| [8] |
Wang X, Duan D, Fu X. Enzymatic desulfation of the red seaweeds agar by Marinomonas arylsulfatase[J]. International Journal of Biological Macromolecules, 2016, 93(A): 600-608.
(  0) 0) |
| [9] |
Wang X, Duan D, Xu J, et al. Characterization of a novel alkaline arylsulfatase from Marinomonas sp. FW-1 and its application in the desulfation of red seaweed agar[J]. Journal of Industrial Microbiology and Biotechnology, 2015, 42(10): 1353-1362. DOI:10.1007/s10295-015-1625-6
(  0) 0) |
| [10] |
Kim J H, Byun D S, Godber J S, et al. Purification and characterization of arylsulfatase from Sphingomonas sp. AS6330[J]. Applied Microbiology and Biotechnology, 2004, 63(5): 553-559. DOI:10.1007/s00253-003-1463-8
(  0) 0) |
| [11] |
Mann A J, Hahnke B, Huang A S, et al. The genome of the alga-associated marine flavobacterium Formosa agariphila KMM 3901T reveals a broad potential for degradation of algal polysaccharides[J]. Applied and Environmental Microbiology, 2013, 79(21): 6813-6822. DOI:10.1128/AEM.01937-13
(  0) 0) |
| [12] |
Jung K T, Kim H W, You D J, et al. Identification of the first archaeal arylsulfatase from Pyrococcus furiosus and its application to desulfatation of agar[J]. Biotechnology and Bioprocess Engineering, 2012, 17(6): 1140-1146. DOI:10.1007/s12257-012-0228-6
(  0) 0) |
| [13] |
王嫱, 付晓婷, 毛相朝, 等. 一株从红藻中分离出的产芳基硫酸酯酶的海单胞菌FW-1的全基因组测序及结果分析(英文)[J]. 微生物学报, 2019, 59(4): 744-752. Wang Q, Fu X T, Mao X Z, et al. Complete genome sequence of Marinomonas sp. FW-1, an arylsulfatase-producing bacterium isolated from red seaweed[J]. Acta Microbiologica Sinica, 2019, 59(4): 744-752. (  0) 0) |
| [14] |
Bisswanger H. Enzyme assays[J]. Perspectives in Science, 2014, 1: 41-55. DOI:10.1016/j.pisc.2014.02.005
(  0) 0) |
| [15] |
Kim D E, Kim K H, Bae Y J, et al. Purification and characterization of the recombinant arylsulfatase cloned from Pseudoalteromonas carrageenovora[J]. Protein Expression and Purification, 2005, 39(1): 107-115. DOI:10.1016/j.pep.2004.09.007
(  0) 0) |
| [16] |
王志朋. 产芳基硫酸酯酶菌株的筛选及酶学性质研究[D]. 青岛: 中国海洋大学, 2014. Wang Z P. Isolation of an Arylsulfatase-Producing Marine Bacterium and Characterization of Its Arylsulfatase[D]. Qingdao: Ocean University of China, 2014. (  0) 0) |
| [17] |
Zhu Y B, Qiao C C, Li H B, et al. Improvement thermostability of Pseudoalteromonas carrageenovora arylsulfatase by rational design[J]. International Journal of Biological Macromolecules, 2018, 108: 953-959. DOI:10.1016/j.ijbiomac.2017.11.014
(  0) 0) |
| [18] |
Pereira L, Gheda S F, Ribeiro-Claro P J A. Analysis by vibrational spectroscopy of seaweed polysaccharides with potential use in food, pharmaceutical, and cosmetic industries[J]. International Journal of Carbohydrate Chemistry, 2013, 2013(22): 7.
(  0) 0) |
2. Laboratory for Marine Drugs and Bioproducts, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China
 2022, Vol. 52
2022, Vol. 52


