2. 青岛海洋科技中心海洋药物与生物制品功能实验室,山东 青岛 266237
作为第三代单分子测序技术,纳米孔测序具有序列读长长、速度快、实时、便捷等优点[1-4],因此广泛应用于大片段的基因组拼接[5]、全长转录组测序[6]、微生物快速鉴定[7]以及检测碱基修饰[8-9]等领域。但对于500 bp以下的短链DNA,其测序深度不够,且会占用大量的纳米孔蛋白,导致出现错误率较高、测定效率降低、成本升高且数据拼接处理过程繁琐等问题[10-11],这些问题限制了其在单核苷酸变异(SNV)检测[12]、microRNA[13]分析等方面的应用。实际上,纳米孔测序获得的原始数据中通常存在碱基插入、缺失、误读等错误[14],故需要提取多个测序Reads(单孔单次测序所得到的碱基序列)的数据,通过复杂的比对和计算才能获得正确序列。尤其是短链DNA混合样本的测序,如果不生成重复序列而直接测序,所得数据难以分析,甚至需要对比数以百万计的Reads,这导致数据分析效率极大降低。
为将纳米孔测序应用于短链DNA的测序中(如检测单核苷酸变异),一些研究者们采用了将短链DNA转化为长串联重复序列的策略[10, 15-16],这样可通过对比单个读长(Single read)中原始序列的多个重复获得测序结果,提高准确度的同时又可简化数据分析流程。Wilson等[10]开发了一种用纳米孔测序分析短单链DNA的方法,通过连接环化和滚环扩增的方式把短链DNA扩增成长串联重复序列。然而这一方法只适合以单链DNA为靶标,但纳米孔测序进样的要求是双链DNA,因此需要将其二次扩增成双链。Li等[16]建立了一种基于分子内连接环化双链DNA后进行滚环扩增的方法,将测序准确度提高到97%以上,但此方法难以环化500 bp以下的双链DNA[17]。所以,建立一种通用的能对短链DNA精确测序的方法以拓展纳米孔测序的应用范围是亟需解决的问题。
本研究受重组PCR(Recombinant PCR)技术中两段双链DNA经重叠杂交扩增成长序列的启发[18-20],借助一对完全互补的长引物,使几十至几百碱基对的DNA延伸成为适合纳米孔测序的含有重复序列的长双链(产物长度可达到原料DNA的5~20倍)。同时,采用本方法进行纳米孔测序后仅通过分析一个或少数几个Reads的数据就可以获得短链DNA的精确序列,这大大简化了数据分析流程,节省了分析时间,对拓宽纳米孔测序的应用范围具有重要意义。
1 实验方法 1.1 试剂与材料DNA序列由生工生物工程(上海)股份有限公司合成(见表 1);质粒PBR322购自宝日生物工程(大连)有限公司(Takara);2× Taq PCR StarMix with Loading Dye购自北京康润诚业生物科技有限公司(GenStar);DNA聚合酶Vent (exo-)、限制性内切酶Hinf Ⅰ购自美国纽英伦生物技有限公司(New England Biolabs);2× Phanta Max Master Mix购自Vazyme生物科技股份有限公司(中国南京);琼脂糖(Agarose)、2-氨基-2-羟甲基-1, 3-丙二醇(Tris)、硼酸(Boracic acid)和乙二胺四乙酸二钠盐(EDTA-2Na)等化学试剂购自台湾生工生物科技有限公司(中国);GeneRuler 1 kb DNA Ladder购自Thermo Fisher Scientific公司(美国)。
|
|
表 1 本研究中主要用到的寡核苷酸序列 Table 1 The main oligonucleotide sequences in this study |
以PBR322质粒为模板,以312-F、312-R(序列见表 1)为引物,PCR扩增得到312 bp的双链DNA。反应条件:95 ℃ 3 min;95 ℃ 15 s,58 ℃ 20 s,72 ℃ 20 s(1 000~2 060 bp延伸2 min),30个循环;72 ℃ 2 min。20 μL PCR体系各组分终浓度为:1×105 copies/μL PBR322质粒,0.3 μmol/L引物,1× Taq PCR StarMix with Loading Dye。在普适性验证实验中制备不同长度双链DNA,除所需引物不同,其他条件与上述反应相同。
1.3 短链DNA生成重复序列的反应(重叠延伸法)20 μL总反应体系中包括如下组分:1 μL 312 bp双链DNA(约5 nmol/L),10~50 nmol/L引物RF′-noA、R′F-noA(或表 1中所示的插入3个A或5个A的引物对),1× Taq PCR StarMix with Loading Dye。反应程序为:95 ℃预变性3 min;95 ℃变性15 s,72 ℃退火和延伸2 min,20个循环;95 ℃变性15 s,72 ℃退火和延伸5 min,40个循环;72 ℃终延伸10 min。对于单引物生成重复序列的实验,除加入的引物不同外(仅加入RF′),其余反应条件均与以上实验相同。
在探究引物浓度和聚合酶种类对重复序列影响的实验中,除聚合酶和反应缓冲液种类与上述条件不同外,其余均相同。以Phanta为DNA聚合酶催化的反应体系中含有1× Phanta Max Master Mix;以Vent (exo-)为DNA聚合酶催化的反应体系中,含有0.02 U/μL Vent (exo-),0.1 mmol/L dNTP,1× Thermolpol reaction buffer。制备的产物通过0.5% Agarose进行分析。
在条件优化实验中,如无特殊强调,20 μL总反应体系中包含如下组分:1 μL 312 bp双链DNA(约5 nmol/L),1 nmol/L引物RF′-noA、R′F-noA,1× Phanta Max Master Mix,反应条件同上。
对于生成重复序列普适性验证的实验,除所需引物不同外,其他条件与条件优化实验的反应条件相同。
1.4 限制性内切酶酶切验证产物结构10 μL酶切体系中各组分终浓度如下:5 μL重叠延伸产物,0.5 U/μL Hinf Ⅰ限制性内切酶,1× Cutsmart buffer。37 ℃酶切3 h,80 ℃灭活20 min。酶切产物通过10% PAGE进行分析。
1.5 纳米孔测序及序列分析用上述方法扩增得到的长链DNA样品经醇沉后,采用Nanodrop(Thermo Fisher Scientific)测定浓度后稀释至100 ng/μL,送至广州吉赛生物科技股份有限公司,用MinION纳米孔测序仪(Oxford Nanopore Technologies, Oxford, UK)对长重复序列进行测序。测序过程如下:首先,用基因组DNA连接试剂盒(SQK-LSK109)制备文库;其次,在FLO-MINTO6(R9.4.1)flow cells(牛津纳米孔技术公司)中运行;然后,使用MinKNOW软件(牛津纳米孔技术公司)中的Guppy v5.0.16进行实时的碱基调用。
测序后的数据用DNAMAN软件(LynnonBiosoft Co.)进行序列分析。分析过程如下:首先,在软件工具栏载入需要对齐的序列;其次,在菜单栏选择Sequence—Alignment—Multiple alignment sequence进行多条序列对齐,根据序列比对结果获得多条序列的一致性(共有)序列,其余位置按照多条序列出现概率最高的序列进行碱基读取,得到测得的序列结果;最后, 将得到的序列与参考序列(原始的短链DNA)进行比对,即DNAMAN软件计算出一致性(Identity),所得数值为测序结果的准确度。
2 结果与讨论 2.1 短链DNA生成重复序列的重叠延伸法策略本研究提出的将短链DNA通过重叠延伸生成重复序列的策略如图 1所示。在目的基因中以F和R为引物,通过普通PCR获得待测序的短链DNA(见图 1A)。根据F和R的序列设计一对特殊引物(R′F/RF′,图 1中仅以RF′为例,R′F以同样的原理发挥作用)。RF′主要由两部分构成,其3′端的序列与R完全相同,而5′端的序列与F互补(以F′表示)。对于R′F,序列设计是相反的(3′端的序列与F相同,5′端的序列与R互补)。短链DNA(相当于本方法扩增后产物的一个重复单元)在重叠延伸过程中同时充当模板与引物。首先,RF′与短链DNA中一条链的3′端杂交(见图 1B)。其次,引物延伸后,使得这条短链DNA两端均带有F/F′序列(见图 1C),因此短链DNA变性后两条单链DNA的3′端是完全互补的,在PCR过程中,它们的末端可以杂交重叠(见图 1D)并延伸形成短链DNA的二聚体(见图 1E)。最后,这种二聚体在接下来的循环中可以继续重叠并延伸,产生具有多个重复单元的长序列(见图 1F)。短链DNA形成二聚体后的重叠延伸参考Wang等[19, 21]构建的PEAR(Polymerase-Endonuclease Amplification Reaction)原理进行,产物长度随着热循环次数的增加而增加。由此可见,本方法虽受重组PCR的启发,但与其有很大不同。重组PCR是分别制备带有重叠序列的两条双链DNA,然后进行PCR扩增生成长DNA。而本方法是利用一个双链DNA引物(目前未见以双链DNA作为引物的报道)以PEAR原理生成重复序列。
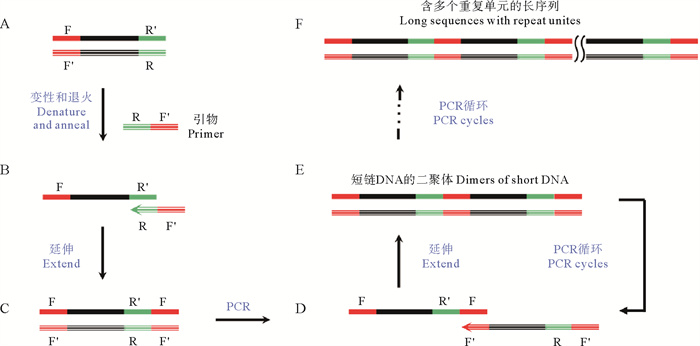
|
图 1 短链DNA生成长重复序列的策略 Fig. 1 Strategy for generating long repetitive sequences from short DNA |
用于重叠延伸的特殊引物对(R′F和RF′)是完全互补的。在通常的认知中,完全互补的引物可能无法引发DNA扩增。为探究其是否能将短链DNA扩增成长序列,我们以312 bp的双链DNA为模板,以Taq DNA聚合酶催化重叠延伸反应,分别在引物浓度为10、20和50 nmol/L(约为312 bp双链DNA模板浓度的2倍、4倍和10倍)条件下进行重叠延伸反应(见图 2A1)。为考察使用上述引物对的必要性,还与仅用特殊引物中的一条引物(RF′)进行重叠延伸(见图 2A2)的结果进行了比较(见图 2B)。值得注意的是,对于双引物引发的扩增反应,在3种浓度条件下均生成了长序列(胶孔中的产物);然而单引物对应的扩增反应却未有长序列产生。这一结果表明,双引物引发的重叠延伸扩增反应更高效。产生这一现象的原因可能是,当体系中仅存在一条特殊引物时,其与短链DNA杂交延伸后,仅能形成一条3′端与5′端含有相同序列的DNA(见图 1C),二者仅有20 bp互补,而PCR的退火温度为72 ℃,自身的3′端很难发生“滑动”以形成图 1D的结构(此时它们更倾向于自身互补);然而,当双引物存在的时候,两条引物分别能够与短链DNA杂交,形成图 2C所示的结构后发生重叠的两条单链DNA的3′端有40 bp左右的互补,因此更容易发生重叠延伸。一旦形成短链DNA的二聚体,其相当于一个“种子DNA”,满足了进行PEAR延伸扩增的所有条件,最终生成更长的重复序列。

|
( A:双引物与单引物设计示意图;B:双引物与单引物生成重复序列扩增效率的比较;C:双引物扩增可能生成的产物;D:长引物中部的短序列对重复序列生成的影响。A: Scheme of the reaction system incorporating the double primer and single primer; B: Comparison of the amplified efficiency between double primer and single primer; C: Possible products generated by double primer; D: Effect of short linker in the middle of long primers on repetitive sequence generation. ) 图 2 引物设计对生成重复序列的影响 Fig. 2 Effect of primer design on generating repetitive sequences |
重叠延伸法中“双引物”(R′F/RF′)的设计是关键,本研究设计了3种不同的引物(见图 2D)。引物对中间引入了A-A错配,期待有利于引物对的解离。以RF′为例,即在引物R和F′中间分别设计不加A、加3个A和加5个A三种情况,探究引物中间加入A-A错配是否能更容易延伸短链DNA。图 2D电泳图表明,3对引物引发的扩增反应无明显区别。这可能是因为在引物中间加入3个A或5个A与未加A的引物对的Tm值相似,难以有效避免引物对互补结合。另外,进行PEAR扩增时产物之间重叠杂交会形成T-T错配,可能使PEAR效率降低。因此,后续将选用“双引物”中间不加A的情况(即RF′-noA和R′F-noA)进行实验。
2.3 限制性内切酶酶切验证的产物结构为确定重叠延伸法得到的产物是否为重复序列,我们设计了限制性内切酶进行酶切来验证产物结构。限制性内切酶Hinf Ⅰ在312 bp的短链DNA重复单元中仅有一个酶切位点,因此产物被Hinf Ⅰ酶切后将主要生成带有黏性末端的与重复单元长度大致相同的DNA片段(见图 3A)。如图 3A所示,具有两个重复单元的产物可被酶切为3条DNA双链,其长度大致为296、309和13 bp。如果产物含有更多的重复单元,那么309 bp的DNA链为主要的酶切产物。如图 3B所示,经酶切后,产物长度集中在309 bp,这与预期结果一致。泳道上方600 bp左右的条带可能是未被酶切完全的重复单元的二聚体。以上结果表明,重叠延伸法获得的长序列确实为原双链DNA的重复序列。

|
( A:限制性内切酶酶切短链DNA二聚体的示意图;B:酶切产物的分析(10% PAGE)结果。A: Scheme of restriction endonuclease digestion of a short DNA dimer; B: Analysis of digestion products by 10% PAGE. ) 图 3 重复序列的限制性内切酶酶切验证 Fig. 3 Verification of the repetitive sequence by restriction endonuclease digestion |
为确定生成重复序列的最优条件,我们分别探究了聚合酶种类、引物浓度、退火温度、循环数以及模板浓度对重复序列生成的影响。选择Taq DNA聚合酶(PCR常用酶)、Vent (exo-) DNA聚合酶(具有较强链置换活性)和Phanta DNA聚合酶(聚合活性是Taq酶的52倍)三种性质不同的聚合酶进行重叠延伸反应。如图 4A所示,在引物浓度为1 nmol/L时,Vent (exo-)和Phanta DNA聚合酶均催化生成了重复序列,而Taq DNA聚合酶没有生成。另外,Phanta DNA聚合酶催化生成的重复序列的条带更亮,表明其催化效率更高,后续选择Phanta DNA聚合酶进行探究。
引物浓度也是决定重复序列生成的重要因素。需要强调的是,我们设计的用于重叠延伸的特殊引物是完全互补的,不同于普通的PCR引物。因此,如果引物量过高,一部分引物可能倾向于自身杂交形成无效的双链DNA;而待扩增短链DNA已经形成多聚体后,剩余的引物会不断地对这些短的重复单元进行扩增,导致短链DNA难以发生重叠延伸获得更长的重复序列(见图 1D)。如果引物浓度过低,则大部分引物可能会自身杂交而难以形成用于引发重叠延伸反应的多聚物。基于上述原因,我们在1~50 nmol/L的范围内(是模板浓度5 nmol/L的0.2~10倍)考察了浓度对于重叠延伸的影响。以图 4A中Phanta DNA聚合酶为例,结果显示引物浓度越高泳道中的弥散条带越多,说明对应的DNA链可能越短。当引物浓度为1 nmol/L时,泳道中弥散条带的长度在10 kb以上,因此后续实验中的引物浓度定为1 nmol/L。如图 1所示,本方法在扩增过程中存在多种杂交形式的竞争,因此退火温度对重复序列的生成起着重要作用。起始时引物R′F或RF′同初始短链DNA的互补部分只有20 bp左右,需要较低的退火温度;然而一旦形成可进行PEAR的多聚体后,互补部分为重复单元的长度或其整数倍,此时较高的退火温度更有利。另外,考虑到本方法中的模板浓度比一般的PCR高几百甚至上千倍,为生成更长的重复序列,本文采用了较高的退火温度(见图 4B)。当退火温度为70和72 ℃时,成功生成了较长的重复序列;当退火温度升高至75 ℃时,则几乎无产物生成。推测是过高的退火温度导致多聚物难以在最初几个循环中生成(见图 1B—D)。因此,后续实验均采用72 ℃的退火温度。此外,我们对循环数及模板浓度进行了优化(见图 4C、D),确定了循环数为60,模板浓度为5 nmol/L的扩增条件。

|
( A:DNA聚合酶种类以及引物浓度;B:退火温度;C:热循环数;D:模板浓度。A: Type of DNA polymerase and concentration of primer; B: Annealing temperature; C: Number of thermal cycles; D: Template concentration. ) 图 4 生成长重复序列的条件优化 Fig. 4 Optimization of conditions for generating long repetitive sequences |
为确定我们建立的重叠延伸法是否可适用于不同长度的DNA,我们分别以45~2 060 bp的10组双链DNA为模板,采用Phanta DNA聚合酶和各自的双引物,在72 ℃退火温度条件下进行了重叠延伸反应。如图 5所示,所有DNA链的扩增产物在电泳时得到了堵在胶孔里的条带,这表明生成了长的重复序列。以上结果表明,我们建立的重叠延伸法和所采用的反应条件具有较强的普适性,即在较宽长度范围内(45~2 060 bp)可用于短链DNA的延伸及纳米孔测序。

|
图 5 重叠延伸法的普适性验证 Fig. 5 Verification the universality of overlap-extension method |
我们将312 bp短链DNA生成的重复序列用于纳米孔测序,以验证重叠延伸法是否可实现短链DNA的精准测序。为了更快捷、简便地分析测序结果,我们建立了数据分析流程,具体步骤如图 6A所示。首先,在测序数据中找到含有目标序列的Read,并根据引物序列(作为Index)将其拆分为重复单元。然后, 使用DNAMAN软件进行多重算法的比对,通过序列对齐,获得一致性(共有)序列。在其余位置(非一致性序列),根据最大概率的原则确定碱基为A、G、C或T,并最终给出结果序列。最后, 将所得序列与原始参考序列进行比较,通过软件自动识别碱基错误,如碱基插入、缺失以及误读等,得出准确度[22]。

|
( A:生成长重复序列实现短链DNA的纳米孔测序数据分析的原理;B:从单个读长中获得序列的准确度分析;C:纳米孔测序序列分析举例,绿色、红色、黑色字体表示共有序列,蓝色字体是根据最高出现概率读取的碱基。A: Data analysis principle for nanopore sequencing of short DNA after generating its long repeats; B: Accuracy of obtained sequence from analysis of single Read; C: An example of obtained sequence by nanopore sequencing, the green, red and black fonts indicate consensus sequences, blue fonts are nucleotides based on the highest probability of occurrence. ) 图 6 纳米孔测序法测定短链DNA序列的数据分析及其准确度评价 Fig. 6 Data analysis and accuracy evaluation of nanopore sequencing for short DNA |
我们分别从重复次数为6~10,11~15,16~20的原始数据中随机选取4个Reads,通过对单个Read中重复序列的相互对比获得了精确的序列信息,并计算了测序结果的准确度(图 6B中的每个点表示单个Read所得序列的准确度)。根据每一组中4个Reads获得的测序准确度计算其平均值(用横线表示),从而获得重复次数对测序准确度的影响。结果表明,Read中目的短链DNA的重复次数越多,测序准确度越高。当单个Read重复次数达到16次以上时,其测序的准确度可达99.28%,这说明我们建立的重叠延伸法可以实现短链DNA高准确度测序。图 6C展示了原始测序数据的一个例子,长度为2 533 nt,是原料DNA的8倍。实际应用中,一般待测序列越短本方法所得长序列中的重复次数越多,所得测序结果的准确率越高。
3 结语本研究采用含有上下游引物信息的两条完全互补的长引物(约40 nt),成功构建了一种重叠延伸法,使短链DNA转变为长串联重复序列。将所得重复序列作为纳米孔测序的原料,实现了短链DNA的精准测序。本方法具有较强的普适性,且充分利用了纳米孔测序技术读长较长的优势,用于各种长度的短链DNA的测序。本方法另一个优势在于仅需选取一个或少数几个Reads的测序结果(只要保证待测序列重复次数大于15次),便可获得准确度高达99%以上的短链DNA序列信息,这极大降低了测序数据的处理难度并提高了数据分析效率(减少分析时间)。此外,还可根据需要分析更多的Reads,使测序精度接近100%。更重要的是,本方法可用于大量不同序列的短链DNA的测序,即可以将其分别延伸为长重复序列后混合,测序后以所用引物为标签(Index)进行数据分析,实现序列分析过程中多条DNA链互不干扰的测序。例如用本方法可以高效扩增并分析PCR中产生的非特异性扩增产物(数据将在另外的文章中发表)。总之,本方法可有效降低短链DNA的测序成本,拓宽纳米孔测序技术的应用范围。本方法有望应用于SNV检测、基因分型及基因突变分析等领域。
| [1] |
Jain M, Koren S, Miga K H, et al. Nanopore sequencing and assembly of a human genome with ultra-long reads[J]. Nature Biotechnology, 2018, 36(4): 338-345. (  0) 0) |
| [2] |
Michael T P, Jupe F, Bemm F, et al. High contiguity Arabidopsis thaliana genome assembly with a single nanopore flow cell[J]. Nature Communications, 2018, 9(1): 541. (  0) 0) |
| [3] |
任子林. 基于纳米孔测序的个体识别技术研究[D]. 北京: 军事科学院, 2021. Ren Z L. A Study of Human Identification Using Nanopore Sequencing[D]. Beijing: Academy of Military Sciences, 2021. (  0) 0) |
| [4] |
Lu H, Giordano F, Ning Z. Oxford nanopore MinION sequencing and genome assembly[J]. Genomics, Proteomics & Bioinforma-tics, 2016, 14(5): 265-279. (  0) 0) |
| [5] |
Shafin K, Pesout T, Lorig-Roach R, et al. Nanopore sequencing and the Shasta toolkit enable efficient de novo assembly of eleven human genomes[J]. Nature Biotechnology, 2020, 38(9): 1044-1053. (  0) 0) |
| [6] |
Jiang T, Liu Y, Jiang Y, et al. Long-read-based human genomic structural variation detection with cuteSV[J]. Genome Biology, 2020, 21(1): 189. (  0) 0) |
| [7] |
Grädel C, Terrazos Miani M A, Barbani M T, et al. Rapid and cost-efficient enterovirus genotyping from clinical samples using flongle flow cells[J]. Genes, 2019, 10(9): 659. (  0) 0) |
| [8] |
Wescoe Z L, Schreiber J, Akeson M. Nanopores discriminate among five C5-cytosine variants in DNA[J]. Journal of the American Chemical Society, 2014, 136(47): 16582-16587. (  0) 0) |
| [9] |
Schreiber J, Wescoe Z L, Abu-Shumays R, et al. Error rates for nanopore discrimination among cytosine, methylcytosine, and hydroxymethylcytosine along individual DNA strands[J]. Proceedings of the National Academy of Sciences, 2013, 110(47): 18910-18915. (  0) 0) |
| [10] |
Wilson B D, Eisenstein M, Soh H T. High-fidelity nanopore sequencing of ultra-short DNA targets[J]. Analytical Chemistry, 2019, 91(10): 6783-6789. (  0) 0) |
| [11] |
Zee A, Deng D Z Q, Adams M, et al. Sequencing Illumina libraries at high accuracy on the ONT MinION using R2C2[J]. Genome Research, 2022, 32(11-12): 2092-2106. (  0) 0) |
| [12] |
Christensen E, Nordentoft I, Vang S, et al. Optimized targeted sequencing of cell-free plasma DNA from bladder cancer patients[J]. Scientific Reports, 2018, 8(1): 1917. (  0) 0) |
| [13] |
Mitchell P S, Parkin R K, Kroh E M, et al. Circulating microRNAs as stable blood-based markers for cancer detection[J]. Proceedings of the National Academy of Sciences, 2008, 105(30): 10513-10518. (  0) 0) |
| [14] |
Chen Y, Nie F, Xie S Q, et al. Efficient assembly of nanopore reads via highly accurate and intact error correction[J]. Nature Communications, 2021, 12(1): 60. (  0) 0) |
| [15] |
Volden R, Palmer T, Byrne A, et al. Improving nanopore read accuracy with the R2C2 method enables the sequencing of highly multiplexed full-length single-cell cDNA[J]. Proceedings of the National Academy of Sciences, 2018, 115(39): 9726-9731. (  0) 0) |
| [16] |
Li C, Chng K R, Boey E J H, et al. INC-Seq: Accurate single molecule reads using nanopore sequencing[J]. Gigascience, 2016, 5(1): 34. (  0) 0) |
| [17] |
Rusche J R, Howard-Flanders P. Hexamine cobalt chloride promotes intennolecular ligation of blunt end DNA fragments by T4 DNA ligase[J]. Nucleic Acids Research, 13(6): 1997-2008. (  0) 0) |
| [18] |
Hilgarth R S, Lanigan T M. Optimization of overlap extension PCR for efficient transgene construction[J]. MethodsX, 2020, 7: 100759. (  0) 0) |
| [19] |
Wang X, Gou D, Xu S. Polymerase-endonuclease amplification reaction (PEAR) for large-scale enzymatic production of antisense oligonucleotides[J]. PLoS One, 2010, 5(1): e8430. (  0) 0) |
| [20] |
Kadkhodaei S, Memari H R, Abbasiliasi S, et al. Multiple overlap extension PCR (MOE-PCR): An effective technical shortcut to high throughput synthetic biology[J]. RSC Advances, 2016, 6(71): 66682-66694. (  0) 0) |
| [21] |
Li B, Dong S, Wu J, et al. Preparation of 5'-O-(1-Thiotriphosphate)-modified oligonucleotides using polymerase-endonuclease amplification reaction (PEAR)[J]. PLoS One, 2013, 8(7): e67558. (  0) 0) |
| [22] |
Wang C, Liu H, Wang H, et al. Robust storage of Chinese language in a pool of small single-stranded DNA rings and its facile reading-out[J]. Bulletin of the Chemical Society of Japan, 2021, 94(1): 53-59. (  0) 0) |
2. Laboratory for Marine Drugs and Bioproducts, Qingdao Marine Science and Technology Center, Qingdao 266237, China
 2024, Vol. 54
2024, Vol. 54


