2. 青岛海洋科学与技术国家实验室海洋药物与生物制品功能实验室, 山东 青岛 266235
环状DNA具有线性DNA不具备的动力学和拓扑学特点, 因此环状DNA作为DNA纳米技术的重要组成部分被人们广泛关注[1-3]。Zheng等[4]以单链环状DNA为基本单元成功制备了DNA纳米管结构; 多个课题组以单链环状DNA为原料, 制备了2个或多个DNA环相互穿套的连环结构(Catenane), 并将其制备成可控的分子开关或分子机器等[1, 3, 5-7]。相对于线性DNA, 环状DNA不易被核酸外切酶降解, 在溶液中单一分散、不易聚合, 这些特性决定了其在药物运输、基因调控、疾病诊断和基因治疗等领域的应用更具优越性[4, 8-10]。此外, 单链环状DNA还与滚环扩增技术等以环状DNA为元件的核酸检测技术密切相关[5, 11-12]。因此, 单链环状DNA在分子生物学、医学和生物技术等方面应用广泛。
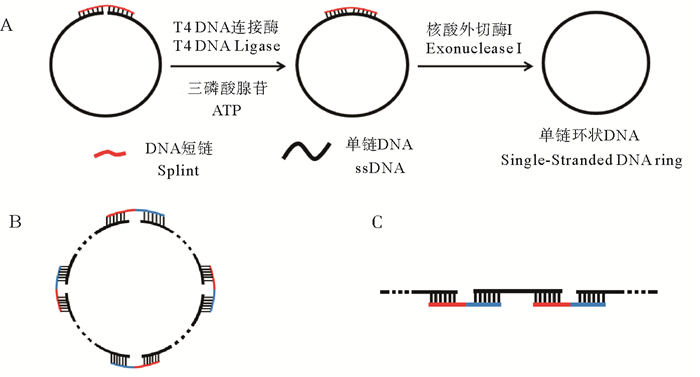
然而, 单链环状DNA难以大量制备成为了目前制约以环状DNA研究和应用的一个难题。通常, 其制备是利用一条与单链DNA两端互补的DNA短链(Splint), 使单链DNA两端靠近, 在连接酶作用下使单链封闭成环(见图 1A)。但在生成由一条单链DNA首尾相接的单倍环的同时, 也会产生由多条单链DNA首尾相接生成的副产物(见图 1B、C), 且随着单链DNA浓度的增大, 副产物的比例会急剧增加。因此为减少副产物产生, 目前在大多数研究中, 单链DNA浓度不超过0.5 μmol/L甚至为nmol/L级别[1, 8, 13-15], 这就使得单链环状DNA的产量受到制约, 从而增加了对其研究和应用的难度。
|
图 1 单链环状DNA的制备(A)及制备时产生的副产物(B和C) Fig. 1 Preparation of single-stranded DNA rings (A) and the polymers produced during the preparation (B, C) |
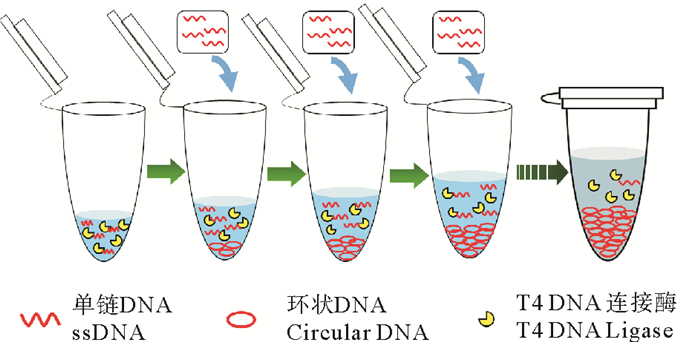
针对上述问题, 且考虑到已成环产物的存在不影响环化反应, An等[16]提出了一种新型的逐步添加法成环, 即每次向成环体系中加入少量单链DNA, 使其在低浓度下充分成环后再次加入单链DNA, 重复多次, 这样可使单链DNA的成环浓度始终处于较低水平的同时, 最终累积得到高浓度的单链环状DNA(见图 2)。然而, 该研究并未深入探讨利用逐步添加法制备单链环状DNA时的影响因素和具体制备条件的确定方法。因此本文拟围绕逐步添加法成环过程中, 连接酶Buffer浓度、单链DNA的添加浓度、添加间隔时间、反应温度等多方面因素进行探讨, 找到逐步添加法的关键因素, 以指导逐步添加法中各条件的确定方法, 为单链环状DNA的高效大量制备提供技术支持。
|
图 2 逐步添加法制备单链环状DNA示意图 Fig. 2 Preparation of single-stranded DNA rings by "Step-by-Step" method |
T4 DNA连接酶、10× T4 DNA连接酶Buffer(400 mmol/L Tris-HCl, 100 mmol/L MgCl2, 100 mmol/L DTT, 5 mmol/L ATP(pH=7.8, 25℃))购于Thermo Fisher Scientific公司; SYBR Green Ⅱ购于Sigma公司; DNA单链(见表 1)由苏州金唯智生物科技公司合成; 其他实验试剂均为国产分析纯试剂; PCR仪购于杭州博日科技有限公司; 旋转式真空浓缩机购于德国Marin Christ公司; 真空泵购于美国圣斯特公司; 聚丙烯酰胺凝胶电泳装置购于北京六一仪器厂; 凝胶成像仪购于BIO-RAD公司。
|
|
表 1 连接成环所用的单链DNA及短链Splint序列 Table 1 Single-stranded DNA and short splint sequences for ligation into circular DNA |
20 μL的总反应体系:0.1× T4 DNA连接酶Buffer(4 mmol/L Tris-HCl, 1 mmol/L MgCl2, 1 mmol/L DTT, 0.05 mmol/L ATP(pH=7.8, 25 ℃)), 0.5 μmol/L 5’端磷酸化的单链DNA, 1 μmol/L Splint短链DNA, 5 Weiss U T4 DNA连接酶。连接反应前先将上述组分混匀后置于PCR仪中, 30 ℃反应10~60 min, 65 ℃ 10 min使连接酶变性。典型的连接反应条件为:0.1× T4 DNA连接酶Buffer, 0.5 μmol/L 5’端磷酸化的单链DNA, 单链DNA与Splint的摩尔浓度比例为1:2, 5 Weiss U T4 DNA连接酶。
1.3 逐步添加法连接成环反应管1为制备液体系(20 μL):10 Weiss U T4 DNA连接酶, 8 μmol/L Splint短链DNA, 0.1× T4 DNA连接酶Buffer(4 mmol/L Tris-HCl, 1 mmol/L MgCl2, 1 mmol/L DTT, 0.05 mmol/L ATP(pH=7.8, 25 ℃)); 管2为添加液体系(20 μL):0.1× T4 DNA连接酶Buffer(4 mmol/L Tris-HCl, 1 mmol/L MgCl2, 1 mmol/L DTT, 0.05 mmol/L ATP(pH=7.8, 25 ℃)), 4 μmol/L 5’端磷酸化的单链DNA。每间隔一定时间向管1制备液体系(只含Splint)加入管2的添加液(只含成环单链DNA), 分10次添加, 以确保每次加入的单链DNA充分反应成环; 添加液添加完毕后延长反应5~10 h后结束反应; 反应温度为25 ℃。
1.4 产物分析连环产物浓度较低(如0.1~0.5 μmol/L)时, 为使产物更易检测, 需要将产物进行真空浓缩, 然后加少量水复溶至1 μmol/L。产物用10%变性聚丙烯酰胺凝胶电泳进行检测。SYBR Green Ⅱ染色10~15 min后利用凝胶成像仪成像。用Image Lab软件定量分析条带亮度, 计算每条泳道中单链环状DNA条带占所有条带的百分比, 得出连接产物中单链环状DNA的产率。
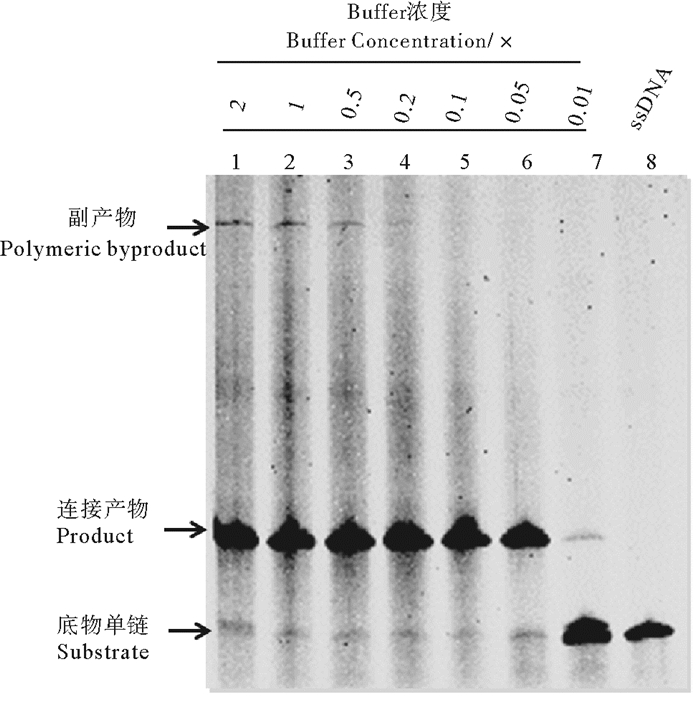
2 结果 2.1 T4 DNA连接酶Buffer的浓度对成环的影响为探究各种因素对成环效率及副产物产生的影响, 本研究选取了一条72 nt的单链DNA(L1-72)用于成环实验, 其两端不具有稳定二级结构, 不会干扰与Splint结合。Splint长度为12 nt, 各有6 nt与成环链的两端互补。由于拓扑结构不同, DNA单链连接成环后迁移速率变慢, 可与DNA单链显著区分开。An等[16]提出低浓度Buffer能减少成环副产物产生, 是逐步添加法中不可忽视的因素之一。因此为确定逐步添加法过程中Buffer浓度, 本研究探究了7种不同的T4 DNA连接酶Buffer浓度对连接成环的影响。图 3结果显示, Buffer浓度对DNA的成环影响显著, 当浓度大于0.1×时, 副产物开始出现; 当浓度为0.01×时, 连接酶效率急剧降低, 仅有极少量的环状DNA产生。因此, 适宜的T4 DNA连接酶Buffer浓度为0.1~0.05×。本实验选择0.1× T4 DNA连接酶Buffer作为最终逐步添加法的Buffer浓度。
|
(泳道1~7:T4 DNA连接酶Buffer浓度分别为2~0.01×; 泳道8:L1-72;25 ℃连接6 h。Lane 1-7: The concentration of T4 DNA Ligase Buffer is 2~0.01×; Lane 8: L1-72; Ligation at 25 ℃ for 6 h.) 图 3 不同浓度T4 DNA连接酶Buffer对成环的影响 Fig. 3 Effects of the concentration of T4 DNA Ligase Buffer on the yield of single-stranded DNA rings |
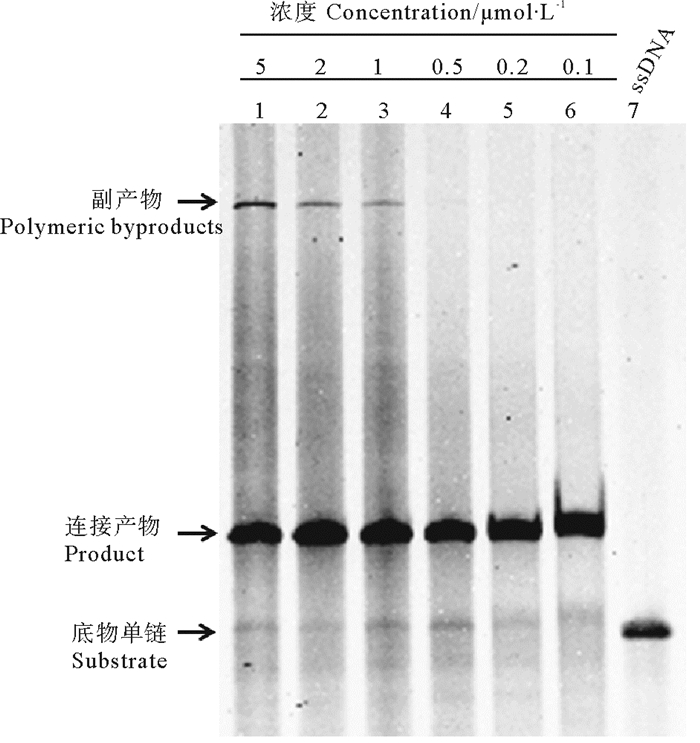
利用逐步添加法制备单链环状DNA时, DNA浓度是影响成环效率的重要因素之一, 控制成环体系中单链DNA的恒定浓度是减少副产物产生的关键, 因此本研究探究了6种不同的单链DNA浓度对成环的影响。结果显示, 当单链DNA的浓度大于0.5 μmol/L时, 连接产物中均有副产物出现; 当单链DNA的浓度小于或等于0.5 μmol/L时, 产物均只有单链环状DNA(见图 4)。也就是说, 对于L1-72链, 逐步添加法每次向制备液体系加入添加液后, 单链DNA的浓度需保持在0.5 μmol/L以下, 以避免副产物的产生与积累。
|
(泳道1~6:单链DNA终浓度分别为5~0.1 μmol/L; 泳道7:L1-72;30 ℃反应6 h。Lane 1~6: The concentration of the single-stranded DNA is 5~0.1 μmol/L; Lane 7: L1-72; Ligation at 30 ℃ for 6 h.) 图 4 不同浓度单链DNA制备环状结构的电泳结果 Fig. 4 Effects of the concentration of the single-stranded DNA on preparing of single-stranded DNA rings |
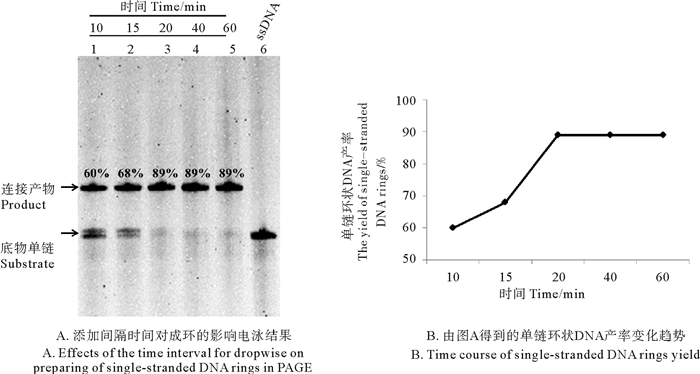
在逐步添加法中, 若2次添加单链DNA之间的间隔时间过短, 则会使剩余单链DNA较多, 累积超过一定浓度后可能导致副产物产生。图 5中表示不同反应时间下的环状DNA产率。结果显示, 反应时间在10~15 min时, 单链环状DNA的收率均低于70%, 体系中剩余成环单链DNA较多; 而当反应时间大于或等于20 min后, 单链环状DNA的收率可达89%且趋于稳定。因此, 对于成环单链L1-72, 逐步添加法的添加间隔时间应控制在20 min以上。
|
(A:泳道1~5:单链DNA L1-72分别反应10~60 min; 泳道6:L1-72。A: Lane 1~5: The time of the single-stranded DNA for ligation into circular DNA is 10~60 min; Lane 6: L1-72.) 图 5 添加间隔时间对成环效率的影响 Fig. 5 Effects of the time interval for dropwise on the yield of single-stranded DNA rings |
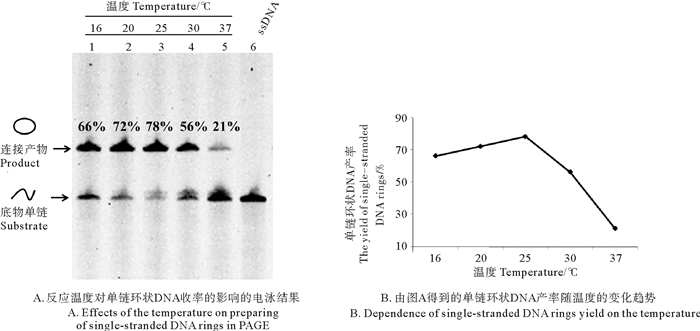
反应温度是影响单链DNA连接成环的因素之一, 温度会影响酶的活性以及Splint和单链DNA的结合从而影响环状结构的收率。本研究探究了5种不同反应温度对成环速率的影响, 不同温度下连接15 min的结果如图 6所示。结果显示, 当温度为25 ℃时, 单链环状DNA的收率最高, 达到了78%;而其他温度下单环的收率相对较小, 如37 ℃时连接成环的效率仅为21%, 成环率远低于25 ℃。因此对于单链DNA L1-72, 制备环状结构时最佳温度为25 ℃, 后续实验也均在该条件下进行。
|
(A:泳道1~5:反应温度分别为16~37 ℃; 泳道6:L1-72;反应时间15 min。A: Lane 1~5: The temperature is 16~37 ℃; Lane 6: L1-72; The time of Ligation is 15 min.) 图 6 反应温度对单链环状DNA收率的影响 Fig. 6 Effects of the temperature on the yield of single-stranded DNA rings |
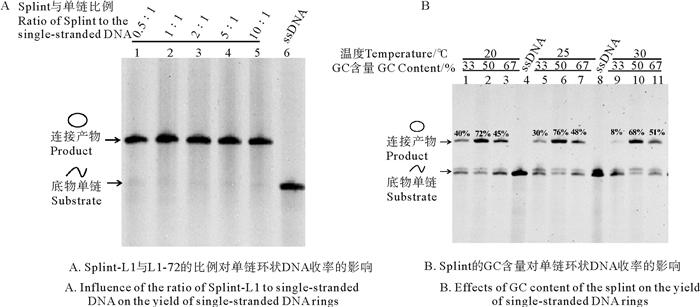
Splint与成环单链DNA的比例及Splint GC含量可能会影响Splint与单链DNA的结合效率, 从而影响单链环状DNA的产率。因此, 本研究考察了二者对于环状DNA产率的影响, 结果如图 7所示。当Splint与单链DNA比例为0.5:1~10:1时, 产物中均只有环状结构条带, 未产生副产物。这表明, 在逐步添加法中可一次性加入高浓度Splint, 不需分次添加。
|
(A:泳道1~5中L1-72与Splint-L1的比例为:0.5:1~10:1;泳道6:L1-72;25 ℃连接10 h。B:泳道1~3、5~7、9~11:20~30 ℃时, GC含量不同的Splint对单链DNA L1-33、L1-72、L1-67成环效率的影响; 连接时间15 min。A: Lane 1~5: The ratio of Splint-L1 to single-stranded DNA is 0.5:1~10:1; Lane 6: L1-72; Ligation at 25 ℃ for 10 h. B: Lane 1~3, 5~7, 9~11: Impact of GC content of the splint on the yield of single-stranded DNA rings when the temperature is 20~30 ℃; The time of ligation is 15 min.) 图 7 Splint对单链环状DNA收率的影响 Fig. 7 Influence of the Splint on the yield of single-stranded DNA rings |
当Splint的GC含量适中时(50%), 单链DNA成环效果最好; Splint的GC含量过高或过低均会降低连接效率。Splint GC含量较低时(33%), 其与单链DNA结合部分熔点低, 在较高的连接温度下二者难以结合, 如随着温度升高, Splint GC含量较低(33%)的单环得率急剧下降(30 ℃仅为8%)。因此, 在设计成环单链末端位置时, 应选择适中的GC含量(Splint的GC含量为50%左右)。
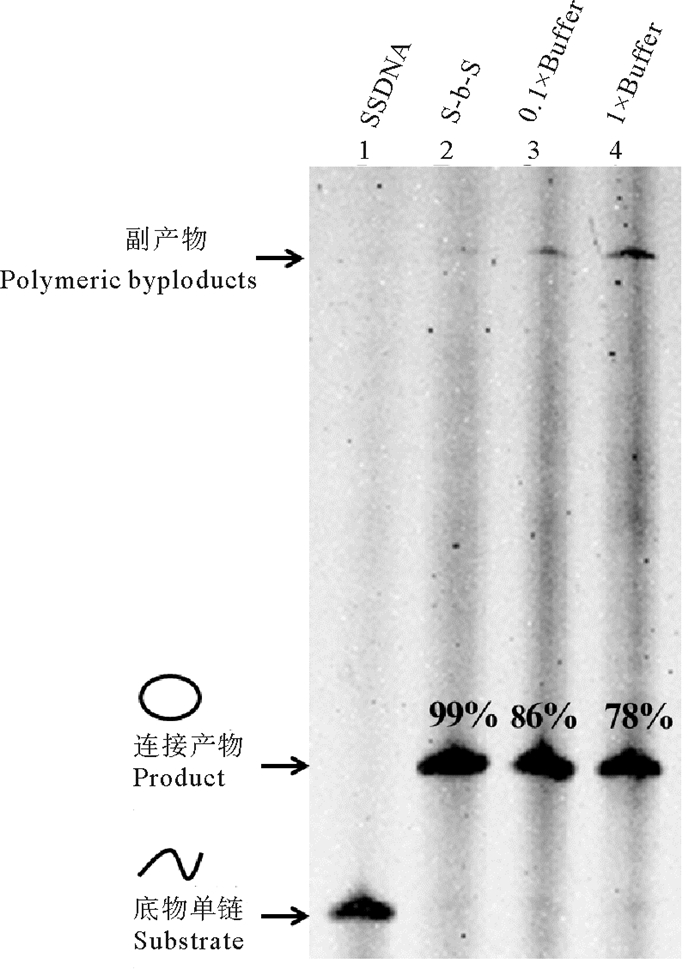
2.6 常规一步法与逐步添加法对比根据上述结果, 针对L1-72单链DNA, 逐步添加法中应考虑的关键条件为:0.1× T4 DNA连接酶Buffer、添加间隔时间为20 min、反应温度为25 ℃。根据此条件, 图 8中对比了将所有单链DNA和其他辅助试剂(包括Splint、连接酶等)一次性加入成环体系中的常规一步法和逐步添加法制备单链环状DNA的结果。可以发现, 一步法连接产物中, 均有副产物产生, 单链环状DNA得率分别为78%(1×Buffer)和86%(0.1×Buffer), 而利用逐步添加法制备单链环状结构, 环状DNA的产率达99%。
|
(泳道1:L1-72;泳道2为逐步添加法制备结果; 泳道3~4为一步法连接结果; 泳道2~4:T4 DNA连接酶Buffer浓度分别是0.1×、0.1×和1×; L1-72终浓度2 μmol/L; 25 ℃连接10 h; 逐步添加法中, 每隔20 min向制备液体系中加入一定量的添加液, 分10次添加。Lane 1: L1-72; Lane 2: The result of preparation of single-stranded DNA rings by “Step-by-Step” method; Lane 3~4: The result of conventional “One-Step” cyclization; Lane 2~4: The concentration of the T4 DNA Ligase Buffer is 0.1×, 0.1× and 1×; The concentration of the single-stranded DNA is 2 μmol/L; Ligation at 25 ℃ for 10 h; Add the additive solution into the preparation system every 20 min for 10 times in the “Step-by-Step” method.) 图 8 一步法与逐步添加法制备单链环状DNA的结果比较 Fig. 8 Comparision of single-stranded DNA rings prepared by different methods |
本文针对逐步添加法制备单链环状DNA, 探究了单链DNA浓度、添加间隔时间等因素对制备单链环状DNA的影响, 得出了影响逐步添加法制备单链环状DNA的关键因素。其中, T4 DNA连接酶Buffer浓度、逐次加入后体系中单链DNA浓度、添加间隔时间、温度、Splint GC含量是逐步添加法制备环状结构时的必需考虑条件, 是减少或避免副产物产生的关键, 而Splint与单链DNA比例(0.5:1~10:1)、酶使用量在一定范围内(1~10 Weiss U)对成环效率影响不大。
本研究发现, T4 DNA连接酶Buffer浓度对于副产物的产生影响较大, 随着T4 DNA连接酶Buffer浓度降低, 副产物急剧减少, 这与An等[16]的研究结果一致。Mg2+是T4 DNA连接酶的依赖型辅因子[17-18], 在低Mg2+浓度下, T4 DNA连接酶催化活性不受明显影响, 同时还可以降低副产物产生[16]。当使用0.1~0.05× Buffer时, Mg2+浓度处于较低水平(0.5~1 mmol/L), 由于DNA带负电荷而使得分子间斥力变大, 分子间的连接效率受到约束, 从而抑制了副产物的产生。
当L1-72浓度高于0.5 μmol/L时, 连接产物中会出现副产物, 这是因为体系中单链DNA浓度增高, 分子间距离减小, 碰撞几率增大, 分子内连接成环的同时分子间连接的几率同时增大, 产生大分子副产物; 反之则分子间接触几率降低, 以分子内连接为主, 反应更倾向于自身环化。同时, 添加间隔时间若过短, 则体系中尚有未连接的单链DNA, 再次加入单链DNA后导致未反应的底物浓度产生积累, 多次积累后浓度达到较高水平, 则同样会增加分子间反应几率, 导致副产物产生。由于在不同条件下, 连接酶效率不同, 因此在制备不同单链环状DNA时, 逐步添加法的添加间隔时间需要通过实验进行确定。
另外, 当温度为25 ℃时, L1-72连接得到单链环状DNA的收率最高, 这可能是由于在25 ℃时酶活较高, 且该温度下Splint与成环单链DNA的杂交程度较好, 因而在逐步添加法中, 温度对单链环状DNA制备效率的影响较大, 需要通过实验进行确定; 而酶活不受明显影响时, Splint的GC含量会影响成环效率。Splint的GC含量适中时, Splint与单链DNA杂交情况较好, 可能会使连接过程中的腺苷化、去腺苷化反应[17]速率更快速, 使单环收率增高; 若Splint GC含量过低或过高, 均有可能降低连接效率, 可考虑适当延长反应时间。而Splint与单链DNA的比例、连接酶使用量并不影响单链环状结构收率, 与An等[16]方法相比, Splint及T4 DNA连接酶在逐步添加法中可在初始时一次性加入制备液中, 不需分次添加, 进一步简化了逐步添加法步骤, 提高了制备效率。
以单链DNA L1-72为例, 逐步添加法可使制备2 μmol/L的单链环状DNA的产率达99%。当成环体系为1 mL时, 产量可达46.3 μg, 相比于传统的制备方法(0.5 μmol/L, 产率为81%, 见图 3泳道2), 产量可提高至4.9倍。当成环链0.5 μmol/L时, 虽然应用常规一步法在低浓度下也可制备得到产率较高的单链环状DNA, 但后续的旋蒸、醇沉等浓缩步骤不仅操作复杂, 还会造成产物的一定损失, 难以达到高效、大量制备的效果; 而使用逐步添加法制备的高浓度环状DNA, 可直接用于常规实验, 显著提高制备效率, 为以单链环状DNA为基础的研究提供极大便利。
4 结语本文研究了单链环状DNA制备过程中各主要因素对单链环状DNA制备的影响, 确定了逐步添加法制备过程中的关键条件, 能够为逐步添加法的具体过程提供指导, 从而实现大量单链环状DNA的高效制备。对于一条随机的DNA单链, 利用逐步添加法大量制备环状结构时, 应考虑的主要关键因素为:T4 DNA连接酶Buffer浓度、每次加入后单链DNA浓度、添加间隔时间、温度、Splint GC含量。本研究为单链环状DNA的制备提供了技术参考, 从而能够为以单链环状DNA为基础的纳米结构技术、核酸检测及疾病诊断治疗等研究奠定基础。
| [1] |
Wu Z S, Shen Z, Tram K, et al. Engineering interlocking DNA rings with weak physical interactions[J]. Nature Communications, 2014, 5: 4279. DOI:10.1038/ncomms5279
(  0) 0) |
| [2] |
Seeman N C. DNA in a material world[J]. Nature, 2003, 421(6921): 427-431. DOI:10.1038/nature01406
(  0) 0) |
| [3] |
Qi L, Wu G, Wu W, et al. Efficient synthesis of topologically linked three-ring DNA catenanes[J]. Chembiochem A European Journal of Chemical Biology, 2016, 17(12): 1127-1131. DOI:10.1002/cbic.201600071
(  0) 0) |
| [4] |
Zheng H, Xiao M, Yan Q, et al. Small circular DNA molecules act as rigid motifs to build DNA nanotubes[J]. Journal of the American Chemical Society, 2014, 136(29): 10194-10197. DOI:10.1021/ja504050r
(  0) 0) |
| [5] |
Wang L, Tram K, Ali M M, et al. Arrest of rolling circle amplification by protein-binding DNA aptamers[J]. Chemistry, 2014, 20(9): 2420-2424. DOI:10.1002/chem.v20.9
(  0) 0) |
| [6] |
Lu C H, Cecconello A, Elbaz J, et al. A three-station DNA catenane rotary motor with controlled directionality[J]. Nano Letters, 2013, 13(5): 2303-2308. DOI:10.1021/nl401010e
(  0) 0) |
| [7] |
Liang X, Kuhn H, Frank-Kamenetskii M D. Monitoring single-stranded DNA secondary structure formation by determining the topological state of DNA catenanes[J]. Biophysical Journal, 2006, 90(8): 2877-2889. DOI:10.1529/biophysj.105.074104
(  0) 0) |
| [8] |
Aldaye F A, Lo P K, Karam P, et al. Modular construction of DNA nanotubes of tunable geometry and single-or double-stranded character[J]. Nature Nanotechnology, 2009, 4(6): 349-352. DOI:10.1038/nnano.2009.72
(  0) 0) |
| [9] |
Darquet A M, Rangara R, Kreiss P, et al. Minicircle: an improved DNA molecule for in vitro and in vivo gene transfer[J]. Gene Therapy, 1999, 6(2): 209-218. DOI:10.1038/sj.gt.3300816
(  0) 0) |
| [10] |
Lu C H, Willner B, Willner I. DNA nanotechnology: from sensing and DNA machines to drug-delivery systems[J]. Acs Nano, 2013, 7(10): 8320-8332. DOI:10.1021/nn404613v
(  0) 0) |
| [11] |
Wang X, Li C, Gao X, et al. Preparation of small RNAs using rolling circle transcription and site-specific RNA disconnection[J]. Molecular Therapy Nucleic Acids, 2015, 4(1): 215.
(  0) 0) |
| [12] |
Christian A T, Pattee M S, Attix C M, et al. Detection of DNA point mutations and mRNA expression levels by rolling circle amplification in individual cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(25): 14238-14243. DOI:10.1073/pnas.251383598
(  0) 0) |
| [13] |
Liu Y, Kuzuya A, Sha R, et al. Coupling across a DNA helical turn yields a hybrid DNA/organic catenane doubly tailed with functional termini[J]. Journal of the American Chemical Society, 2008, 130(33): 10882-10883. DOI:10.1021/ja8041096
(  0) 0) |
| [14] |
Kuhn H, Demidov V V, Frankkamenetskii M D. Rolling-circle amplification under topological constraints[J]. Nucleic Acids Research, 2002, 30(2): 574-580. DOI:10.1093/nar/30.2.574
(  0) 0) |
| [15] |
Liu M, Zhang Q, Li Z, et al. Programming a topologically constrained DNA nanostructure into a sensor[J]. Nature Communications, 2016, 7: 12074. DOI:10.1038/ncomms12074
(  0) 0) |
| [16] |
An R, Li Q, Fan Y, et al. Highly efficient preparation of single-stranded DNA rings by T4 ligase at abnormally low Mg(Ⅱ) concentration[J]. Nucleic Acids Research, 2017, 45(15): 139. DOI:10.1093/nar/gkx553
(  0) 0) |
| [17] |
Cherepanov A V, Vries S D. Kinetic mechanism of the Mg2+-dependent nucleotidyl transfer catalyzed by T4 DNA and RNA ligases[J]. Journal of Biological Chemistry, 2002, 277(3): 1695-1704. DOI:10.1074/jbc.M109616200
(  0) 0) |
| [18] |
Cherepanov A V, De V S. Kinetics and thermodynamics of nick sealing by T4 DNA ligase[J]. Febs Journal, 2003, 270(21): 4315-4325.
(  0) 0) |
2. Laboratory for Marine Drugs and Bioproducts, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266235, China
 2019, Vol. 49
2019, Vol. 49


