龙须菜(Gracilariopsis lemaneiformis)广泛分布于中国的黄海海域[1], 是中国重要的栽培海藻, 主要作为提取琼胶的原料, 也大量用于鲍鱼养殖等。随着龙须菜养殖业的不断发展壮大, 龙须菜的种质退化现象也越来越严重, 尽管目前已有多个高产的新品种, 如981龙须菜[2]、龙须菜2008[3]、鲁龙1号[4]等, 但仍然无法完全满足生产上的需求。此外, 目前龙须菜的养殖方式为营养繁殖, 其苗种的来源与保存都存在一定风险。为了获得稳定可控的苗种来源以及更精准的品种改良方法, 都要求对其世代交替的繁殖发育调控机制有更加深入的认识。
龙须菜是典型的真红藻亚纲(Florideophycidae)的三世代型生活史, 包括二倍的四分孢子体以及附生于雌配子体藻枝上的果孢子体世代和单倍的配子体世代[5], 其四分孢子体与雌雄配子体分别萌发自果孢子和四分孢子, 但萌发的时间和空间相同, 可形成混杂的群落。在成熟之前形态大小完全相同, 无法进行区分[6]。
通过转录水平的比较分析来寻找世代差异表达的基因在红藻相关研究中已经不少的报道。例如在紫菜(Pyropia/Porphyra)属中, 曾报道在孢子体中特异表达的一种延长因子EF-1a[7]; 还有在配子体中发现的依赖于特殊启动子元件的PyKPA1基因, 可编码钠离子泵[8-9]; 此外还有研究通过抑制性消减杂交对紫菜配子体和孢子体形态差异相关候选基因进行分析注释, 发现配子体更趋向于具有生长和自我保护的趋势, 而孢子体在发育过程中更活跃[10]。在江蓠中也报道了在果孢子体中特有的泛素基因[11]和硫转运酶等基因[12]以及在雌配子体中特异表达的gmf-01基因[13]。丁弘叶等[14]还通过AFLP分子标记技术, 得到了一个在四分孢子体和雌配子体中稳定表达, 而在雄配子体中不表达的遗传标记。
本文将利用ContigExpress、Blast2GO等生物信息学软件对实验所得1 873条有效序列进行进一步的聚类和分析, 进一步整理并深入理解以上消减杂交的结果, 并利用实时荧光定量PCR的方法对部分差显序列进行转录水平的比较和验证, 为后续江蓠属红藻世代交替及发育的研究提供数据并开拓思路。
1 材料与方法 1.1 实验用材料文库构建及RT-qPCR验证所用材料为本实验室保种的一株采自青岛湛山湾的野生龙须菜四分孢子体以及由它放散的四分孢子所萌发培养的雌配子体和雄配子体各一株。以这3个样品进行实验目的是尽量减少遗传背景的差异。
1.2 序列的聚类和Blast2GO分析本实验室在前期工作中构建了5个正反向的龙须菜雌、雄配子体间以及它们分别与四分孢子体之间的抑制性消减杂交文库, 再通过斑点杂交方法在各库中进一步对差异显示序列进行筛查, 尽量去除假阳性片段。根据斑点杂交结果, 总共获得2 682个差异显示的阳性克隆(阳性克隆的定义为该克隆与本组差减产物探针杂交的信号值同与反向差减产物探针杂交的信号值的比值≥2), 测序后共获得1 873条可用序列(见表 1)[15]。将所得数据全部导入ContigExpress中进行聚类并检查聚类结果的可行性, 减少因PCR扩增或是测序过程中产生的碱基错误而造成的误差。最后将这些经过聚类的序列导入Blast2GO软件, 进行一系列的比对和注释。
|
|
表 1 斑点杂交阳性克隆的测序结果统计 Table 1 The positive clones and effective sequences for the dot-blot results |
分别称取龙须菜雌配子体、雄配子体和四分孢子体各约100 mg, 于盛有液氮的研钵中迅速研磨至粉末, 利用RNase-Free产品提取各样品的总RNA。利用1.2%琼脂糖凝胶电泳检测所提取的总RNA质量, 并用NanoDrop 2000C测定总RNA的浓度, 用以计算反转录时总RNA的用量。分别以上述步骤提取的4种总RNA为模板, 用PrimeScript 1st Strand cDNA Synthesis Kit(TaKaRa)将总RNA反转录成单链的cDNA, 将所得cDNA样品置于-20℃冰箱贮藏备用。
1.4 部分候选差显基因的实时荧光定量PCR(RT-qPCR)验证根据比对结果中的GO IDs、KEGG Pathway Map和序列所在SSH文库的代表性, 本轮实验选择了9个重叠群(contig-03、contig-11、contig-19、contig-35、contig-44、contig-52、contig-60、contig-114、contig-116)及3个单条序列(7310、8142、0125)共12条候选基因, 采用RT-qPCR的方法对SSH文库筛选的结果进行进一步的定量验证。
首先进行差异基因RT-qPCR反应的引物设计与反应条件优化, 使用RealMaster Mix (SYBR Green)试剂盒(TIANGEN)进行PCR反应, 使用仪器为ABI 7500荧光定量PCR仪, 反应步骤如下:95 ℃ 5 min, 95 ℃ 1 min, 54 ℃ 30 s, 72 ℃ 45 s, 35个循环, 最后72 ℃延长10 min。内参基因使用的是本实验筛选出两个在龙须菜不同世代中表达稳定的rbcL和Co 基因对实验数据进行分析[16]。待测基因的相对定量表达以2-ΔΔCt方法进行计算[17]。
2 结果与分析 2.1 龙须菜抑制性消减杂交差异表达基因的聚类去除载体后得到1 873条序列, 将其用Vector NTI Suite 6.0软件中的ContigExpress进行聚类分析, 最终获得118个重叠群(contig), 以及未被聚类进任何一个重叠群的247条单条序列, 共计365个基因片段序列。
2.2 生物信息学分析本实验对得到的118个重叠群及247条单条序列利用Blast2GO在线软件进行分析及注释。结果显示, 应用blastx搜索比对上的序列所对应的生物物种大多数来自植物, 但非常分散, 在比对上的3 000条序列中, 匹配数较高的物种有玉米(Zea mays)(52次)、小立碗藓(Physcomitrella patens)(53次)、水稻(Oryza sativa)(57次)、蒺藜苜蓿(Medicago tuncatula)(59次)和大豆(Glycine max)(65次)等, 前10名物种比对上的序列仅有437条。图 1中列出e值最小, 即匹配度最高的blast对应的物种。结果的指向性更加明确, 在比对上的183条序列中, 匹配次数最高的的物种为:隐藻(Guillardia theta)(3次)、条斑紫菜(Pyropia yezoensis)(3次)、极地胶球藻(Coccomyxa subellipsoidea)(4次)、龙须菜(G. lemaneiformis)(4次)、铜绿微囊藻(Microcystis aerugisona)(5次)、细基江蓠(Gracilaria tenuistipitata)(5次)、长囊水云(Ectocarpus siliculosus)(6次)、蒺藜苜蓿(Medicago tuncatula)(6次)、小立碗藓(Physcomitrella patens)(9次)等, 其中日本凋毛藻(Griffithsia japonica)(17次)最多, 共计62次, 并且物种中藻类明显增加。同时, 可以看到blast结果中显示大量没有任何序列可以匹配的待测序列, 推测为红藻或龙须菜及其近缘物种的特有基因, 这些序列也是未来研究的难点和重点。

|
图 1 Blast匹配度最高的物种分布图 Fig. 1 Top-Hit species distribution |
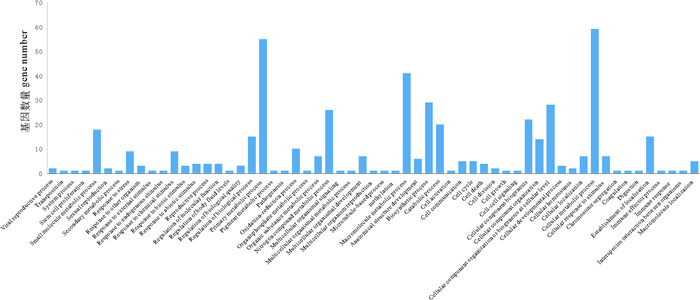
将GO注释过的基因归类到基因本体论的三大词条下, 如图 2、3、4所示。在对生物学途径注释结果的分析中发现, 其中最多的为参与机体的代谢途径的序列(210条), 另外包括有关细胞途径、生物调控、生物合成、信号传递、复制、应激反应、发育过程、细胞定位、细胞组分生源的序列等。分子功能注释结果中, 包括与转移酶活性、水解酶活性、裂合酶活性、氧化还原酶活性等酶活性相关的序列, 以及与小分子结合、蛋白质结合、核酸结合、离子结合等结合相关的序列等。细胞组件注释结果中, 包括细胞组分、蛋白质复合体组分、无膜细胞器组分、有膜细胞器组分等, 分析显示其绝大多数都在细胞(71条)、细胞器(78条)及蛋白质复合体(23条)水平进行描述。
|
(Viral reproduction process病毒繁殖过程Transposition转座System process系统进程Stem cell proliferation干细胞增殖Small molecule metabolic process小分子代谢过程Sexual reproduction有性生殖Secondary metabolic process次生代谢过程Response to stress应激反应, 胁迫应答Response to other organism对其他生物体的反应Response to external stimulus应对外部刺激Response to endogenous stimulus应对内源性刺激Response to chemical stimulus应对化学刺激Response to biotic stimulus应对生物刺激Response to abiotic stimulus应对非生物刺激Reproductive process生殖过程Regulation of molecular function分子功能的调控Regulation of body fluid levels体液水平的调控Regulation of biological quality生物质量的调控Regulation of biological process生物过程的调控Primary metabolic process初级代谢过程Pigment metabolic process色素代谢过程Pathogenesis发病机制Oxidation-reduction process氧化还原过程Organophosphate metabolic process有机磷代谢过程Organic substance metabolic process有机物质代谢过程Nitrogen compound metabolic process氮化合物代谢过程Multicellular organismal signaling多细胞有机体的生物信号Multicellular organismal metabolic process多细胞有机体的代谢过程Multicellular organismal development多细胞有机体的发育Multicellular organismal reproduction多细胞有机体的繁殖Microtubule-based process基于微管的生物过程Methylation甲基化Macromolecule metabolic process大分子代谢过程Anatomical structure development解剖结构发育Biosynthetic process生物合成过程Catabolic process分解代谢过程Cell activation细胞活化Cell communication细胞通讯Cell cycle细胞周期Cell death细胞死亡Cell division细胞分裂Cell growth细胞生长Cell-cell signaling细胞间信号Cellular component biogenesis细胞组分的生物发生Cellular component organization细胞组分的组织结构Cellular component organization and biogenesis at cellular level细胞水平上细胞组分结构和生物发生Cellular developmental process细胞发育过程Cellular homeostasis细胞稳定性Cellular localization细胞定位Cellular metabolic process细胞代谢过程Cellular response to stimulus细胞对刺激的反应Chromosome segregation染色体分离Coagulation凝结物Digestion消化Establishment of localization定位的建立Immune effector process免疫效应过程Immune response免疫反应Interspecies interaction between organisms生物体种间相互作用Macromolecule localization大分子定位) 图 2 生物过程功能分类中水平3的匹配序列数目 Fig. 2 Number of hits for biological process at Level 3 |

|
(Transmembrane transporter跨膜转运蛋白Transferase activity转移酶活性Transcription factor binding结合转录因子的Tetrapyrrole binding结合四吡咯的Substrate-specific基质特异性Structural constituent of ribosome结构组成型核糖体Small molecule binding结合小分子的Site-specific recombinase位点特异重组酶Sequence-specific DNA序列特异DNA Protein binding结合蛋白质的Cofactor binding结合辅助因子的Enzyme activator activity酶激活物活性Hydrolase activity水解酶活性Integrase activity整合酶活性Ion binding结合离子的Isomerase activity异构酶活性Ligase activity连接酶活性Lipid binding结合脂质的Lyase activity裂合酶活性Metal cluster binding结合金属原子簇的Nucleic acid binding结合核酸的Oxidoreductase activity氧化还原酶活性Peroxidase activity过氧化酶活性Phosphatase regulator磷酸酶调节物) 图 3 分子作用功能分类中水平3的匹配序列数目 Fig. 3 Number of hits for molecular function at Level 3 |

|
(Virion part病毒粒子部分Vesicle囊泡Protein-DNA complex DNA蛋白复合体Protein complex蛋白质复合体Organelle part细胞器部分Non-membrane-bounded organelle非膜系统的细胞器Apoplast质外体Cell part细胞部分Cell-cell junction细胞间连接extracellular region part细胞外区域部分Membrane part膜部分Membrane-bounded organelle膜系统的细胞器) 图 4 细胞成分功能分类中水平3的匹配序列数目 Fig. 4 Number of hits for cellular component at Level 3 |
本轮实验原本选择了9个重叠群(contig-03、contig-11、contig-19、contig-35、contig-44、contig-52、contig-60、contig-114、contig-116)及3个单条序列(7310、8142、0125)共12条候选基因。由于测序所得片段并非基因全长, 最终仅有8条序列优化出合适的反应引物, 进行了RT-qPCR验证。检测基因及内参基因所使用的引物见表 2。
|
|
表 2 候选差显基因荧光定量PCR反应的引物序列 Table 2 RT-qPCR primers for candidate differential expression genes |
以上8条候选序列选自各个SSH文库, 具有一定的代表性, 其来源和性质见表 3。这里面既有来自于单个库的多条序列拼接而成的contig及单条序列, 也有出现在多个库中的序列拼接而成的contig。因此有的基因仅仅出现在1个文库内, 如contig-03、contig-11、contig-19、contig-52、contig-60以及7310;有的基因仅出现在一种藻体世代内, 如contig-35, 尽管在2#和5#库中都找到了该序列, 但这两个文库都是成熟的雄配子体分别与成熟的雌配子体和未成熟的雄配子体杂交而来, 所以它仅在成熟的雄配子体中调高表达; 还有的基因不仅仅出现在一种藻体世代内, 如contig-44在多个库中出现, 且不属于同一个世代, 因此它们的情况更加复杂, 需要谨慎和深入的分析。
|
|
表 3 候选差显基因的来源和性质 Table 3 The original library and characters of candidate differential expression genes |
图 5是8条候选序列的在龙须菜不同世代中的相对表达量, 每个序列的结果都以3个样品中表达量最低的值作为参照, 每个样品计算与其的差值作图。这8条候选基因的实时荧光定量PCR的结果可以分为两大类, 一类是与SSH文库筛选结果基本一致的, 包括contig-03、contig-11、contig-52、contig-60以及7310;另一类则是与SSH文库筛选结果不一致的, 有contig-19、contig-35和contig-44这3条序列。

|
(①Tetrasporaphyte; ②Immature female gametophyte; ③Mature female gametophyte; ④Male gametophyte.) 图 5 8个待测序列在龙须菜不同世代中的相对表达定量分析 Fig. 5 The relative quantitation of the 8 candidate sequences in different phases of G. lemaneiformis |
Contig-03来自3#库, SSH文库筛选结果显示, 其在成熟雌配子体的表达相对于未成熟雌配子体中调高。图 5(a)显示的RT-qPCR结果同样表明, 该基因的确是在成熟雌配子体中的表达量显著高于未成熟雌配子体, 在四分孢子体和雄配子体中的表达量远低于雌配子体, 因此contig-03有可能是一条真正与雌配子体世代相关的序列。
Contig-11来自2#库, 根据SSH文库筛选结果是成熟的雄配子体相对于成熟的雌配子体中调高表达的序列。图 5(b)显示的RT-qPCR结果表明该基因在成熟雄配子体中的表达量的确显著高于成熟的雌配子体。但是, 结果还显示该基因在四分孢子体甚至是未成熟雌配子体中也有相似的调高表达, 因此contig-11看起来并不是一条与雄配子体世代相关的特异序列。
Contig-52和contig-60这2个基因非常相似, 都来自4#库, 根据SSH文库筛选结果, 未成熟雌配子体相对于成熟雌配子体调高表达。图 5(c)、(d)显示的RT-qPCR结果表明这两个基因在未成熟雌配子体中的表达量确实都高于在成熟的雌配子体中。但是, 结果还显示它们在四分孢子体中的表达量与在未成熟雌配子体中类似, 也高于成熟雌配子体, 因此这两个基因应该也都不是与雌配子体世代相关的序列。
序列7310来自7#库, 根据SSH文库筛选结果是成熟的雌配子体相对于四分孢子体中调高表达的基因。图 5(h)显示的RT-qPCR结果表明该基因在成熟与未成熟雌配子体中的表达量类似, 都高于在四分孢子体中的表达量, 并远远高于在雄配子体中的表达量, 因此7310也很可能是一条真正与雌配子体世代相关的基因。
Contig-19来自2#库, 根据SSH文库筛选结果该基因应该在雄配子体中上调表达, 但是图 5(c)显示的RT-qPCR结果表明它在未成熟雌配子体和四分孢子体的表达量均远高于雄配子体, 而在成熟雌配子体中的表达量也非常低, 因此contig-19很可能只是一条与生长相关的基因序列。
Contig-35来自2#和5#库, 根据SSH文库筛选结果, 该基因的表达量应该是雄配子体高于成熟雌配子体。而图 5(d)显示的RT-qPCR结果表明它在成熟雌配子体中的表达量最高, 这与SSH文库筛选结果相反。但该基因在成熟的雌配子体以及雄配子体中的表达量均高于在未成熟雌配子体和四分孢子体中的表达量, 暗示contig-35有可能与配子体世代相关。
Contig-44是由2#、8#和10#库的多条序列混杂拼接而成的一条序列, 因此本身就存在矛盾性, 既可能在雄配子体中调高表达, 也可能在四分孢子体中调高表达。图 5(e)显示的RT- qPCR结果表明, 该基因在未成熟雌配子体的表达量非常低, 而在其它3种成熟藻体材料中的表达量都较高, 说明contig-44可能是一条与成熟藻体生长相关的基因序列。
3 讨论通过Blast2GO进一步对龙须菜SSH文库中获得的1 873条差异显示基因的生物信息学分析可以看出, 这些所得的序列广泛分布在龙须菜生长发育的多种过程中, 参与龙须菜多种生物学功能以及组分构成, 更加说明龙须菜性别和世代的遗传是由多因子参与、多细胞共同完成的复杂生命活动。文库中大多数的序列都没有能够比对上准确的基因信息, 说明这些序列有可能是龙须菜, 或者亲缘关系很近的江蓠属物种, 甚至是红藻中所特有的基因, 还需要大量细致的工作去揭示这一过程的遗传调控。
根据Blastx的比对结果, 提示contig-03和contig-11分别与NADH脱氢酶活性和过氧化物酶的活性有关, 而contig-44可能编码α-1, 4-葡聚糖裂解酶, 7310与磷脂酰肌醇-3激酶基因有很高的相似性。其余4个序列均是功能未知的基因。
为了验证SSH文库所筛选到的差显基因的可信度, 确定真正在龙须菜不同世代及不同发育阶段特异表达或差异表达的基因, 本研究利用RT-qPCR方法对一些可能的差显序列进行了转录水平的比较。通过上节对RT-qPCR结果的分析可以得出, contig-03和序列7310有可能是真正与雌配子体世代相关的序列, contig-35也有可能与配子体世代有关, 而其余5个序列都基本只是与生长过程有关的基因。其中contig-03的部分序列与NADH脱氢酶活性有一定相似性, 可能与发育过程中能量的需求有关; 7310则显示编码磷脂酰肌醇-3激酶, 可能参与了PI3K通路, 与细胞的生长、极化和分化有关。但是contig-03与7310的RT-qPCR模式也不相同, 尽管都是在雌配子体中的表达显著高于四分孢子体和雄配子体, 但contig-03在成熟雌配子体中的表达量显著高于未成熟雌配子体, 而7310在成熟与未成熟雌配子体中的表达量类似, 这可解释为contig-03是到雌配子体发育的后期才开始行使功能, 而7310的表达从雌配子体早期发育阶段就开始了。
在RT-qPCR验证的8条序列中有5条序列与SSH文库筛选结果基本一致, 而另外3条与SSH文库筛选结果不一致, 说明SSH技术的确存在较高的假阳性率。但通过对斑点杂交结果重新的进行聚类后分析以及RT-qPCR验证, 可得到更多更准确的信息, 例如contig-44这条序列既在四分孢子体文库中出现, 又在雄配子体文库中出现, 使人对它的作用产生疑惑, 但经聚类和RT-qPCR分析, 发现该基因很可能在龙须菜成熟部分的表达高于在未成熟部分, 它的注释表明其编码的是葡聚糖裂解酶。该酶可以将储存物质——红藻淀粉降解为果糖, 参与龙须菜的糖代谢[18], 因此在藻体成熟部分该基因看起来更加活跃。
由此可以看出, 采取构建多个SSH文库共同筛选的方法可以较好过滤掉SSH文库产生的假阳性克隆, 使实验结果更加准确; 而对斑点杂交结果进行进一步的聚类分析和RT-qPCR验证可以更加有效地提高实验结果的可信度。
| [1] |
张峻甫, 夏邦美. 中国江蓠属植物地理学的初步研究[J]. 海洋与湖沼, 1962, 4(Z2): 189-198. Chang C F, Xia B M. A preliminary phytogeographical studies on Chinese species of Gracilaria[J]. Oceanologia Et Limnologia Sinica, 1962, 4(Z2): 189-198. (  0) 0) |
| [2] |
张学成, 费修绠, 王广策, 等. 江蓠属海藻龙须菜的基础研究与大规模栽培[J]. 中国海洋大学学报(自然科学版), 2009, 39(5): 947-954. ZHANG Xue-Cheng, FEI Xiu-Geng, WANG Guang-Ce, et al. Genetic studies and large scale cultivation of Gracilaria lemaneiformis[J]. Periodical of Ocean University of China, 2009, 39(5): 947-954. (  0) 0) |
| [3] |
孟琳, 徐涤, 陈伟洲, 等. 龙须菜新品系07-2的筛选及性状分析[J]. 中国海洋大学学报(自然科学版), 2009(S1): 94-98. MENG Lin, XU Di, CHEN Wei-Zhou, et al. Selection and characterization of a new strain of Gracilaria lemaneiformis[J]. Periodical of Ocean University of China, 2009(S1): 94-98. (  0) 0) |
| [4] |
周伟, 隋正红. 龙须菜"鲁龙1号"[J]. 中国水产, 2016(3): 56-57. ZHOU Wei, SUI Zheng-Hong. Gracilaria lemaneiformis "Lu Long 1"[J]. China Fisheries, 2016(3): 56-57. DOI:10.3969/j.issn.1002-6681.2016.03.026 (  0) 0) |
| [5] |
张学成. 海藻遗传学[M]. 北京: 中国农业出版社, 2005: 209. ZHANG Xue-Cheng. The Genetics of Marine Algae[M]. Beijing: China Agriculture Ture Press, 2005: 209. (  0) 0) |
| [6] |
Kain J M, Destombe C. A review of the life history, reproduction and phenology of Gracilaria[J]. Journal of Applied Phycology, 1995, 7(3): 269-281. DOI:10.1007/BF00004001
(  0) 0) |
| [7] |
Liu Q Y, Baldauf S L, Reith M E. Elongation factor 1 alpha genes of the red alga Porphyra purpurea include a novel, developmentally specialized variant[J]. Plant Molecular Biology, 1996, 31(1): 77-85. DOI:10.1007/BF00020608
(  0) 0) |
| [8] |
Uji T, Hirata R, Mikami K, et al. Molecular characterization and expression analysis of sodium pump genes in the marine red alga Porphyra yezoensis[J]. Molecular Biology Reports, 2012, 78(5): 985-991.
(  0) 0) |
| [9] |
Uji T, Mizuta H, Saga N. Characterization of the sporophyte-preferential gene promoter from the red alga Porphyra yezoensis using transient gene expression[J]. Marine Biotechnology, 2013, 15(2): 188-196. DOI:10.1007/s10126-012-9475-y
(  0) 0) |
| [10] |
Shen S, Zhang G, Li Y, et al. Comparison of RNA expression profiles on generations of Porphyra yezoensis(Rhodophyta), based on suppression subtractive hybridization (SSH)[J]. Bmc Research Notes, 2011, 4(1): 1-10. DOI:10.1186/1756-0500-4-1
(  0) 0) |
| [11] |
Ren X Y, Sui Z H, Mao Y X, et al. Cloning and characterization of two types of ubiquitin genes from Gracilariopsis lemaneiformis(Gracilariales, Rhodophyta)[J]. Journal of Applied Phycology, 2009, 21(3): 273-278. DOI:10.1007/s10811-008-9361-1
(  0) 0) |
| [12] |
Mitsunobu K, Hiroshi K, Debra M, et al. Isolation and characterization of phase-specific cDNAs from carposporophytes of Gracilariopsis andersonii(Gracilariales, Rhodophyta)[J]. European Journal of Phycology, 2011, 46(1): 27-35. DOI:10.1080/09670262.2010.548101
(  0) 0) |
| [13] |
Chen P, Shao H, Xu D. Cloning and characterization of a female gametophyte- specific gene in Gracilaria lemaneiformis (Gracilariales, Rhodophyte)[J]. African Journal of Microbiology Research, 2011, 5(18): 2590-2595. DOI:10.5897/AJMR11.133
(  0) 0) |
| [14] |
丁弘叶.青岛野生种群不同倍性龙须菜AFLP遗传多样性分析以及与性别相关SCAR标记的开发与验证[D].青岛: 中国海洋大学, 2011. DING Hong-Ye. Genetic Diversity Analysis of Gracilaria lemaneiformis with Different Phase Types of Wild Population of Qingdao and Development and Validation of a Sex-related SCAR Marker Via AFLP Technique[D]. Qingdao: Ocean University of China, 2011. (  0) 0) |
| [15] |
陈鹏.龙须菜配子体SSH文库的建立及与性别相关基因的分析[D].青岛: 青岛科技大学, 2010. CHEN Peng. Suppression Subtractive Hybridization(SSH) Libraries Construction Between Gametophytes of Gracilaria lemaneiformis and Analysis of Sex-related Genes[D]. Qingdao: Qingdao University of Science and Technology, 2010. http://med.wanfangdata.com.cn/Paper/Detail?id=DegreePaper_Y1740630 (  0) 0) |
| [16] |
王莉莉.龙须菜世代差异基因在不同世代及不同发育阶段的转录分析[D].青岛: 中国海洋大学, 2013. WANG Li-Li. Transcript Analysis of Phase-related Differential Expressed Genes of Gracilaria lemaneiformis in Different Phase and Developmental Alage[D]. Qingdao: Ocean University of China, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10423-1013368609.htm (  0) 0) |
| [17] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 2001, 25: 402-408. DOI:10.1006/meth.2001.1262
(  0) 0) |
| [18] |
Yu S, Kenne L, Pedersén M. Alpha-1, 4-glucan lyase, a new class of starch/glycogen degrading enzyme. I. Efficient purification and characterization from red seaweeds[J]. Planta, 1993, 1156(1): 137-142.
(  0) 0) |
 2019, Vol. 49
2019, Vol. 49


