2. 青岛海洋科学与技术国家实验室海洋生态与环境科学功能实验室,山东 青岛 266071;
3. 海洋环境与生态教育部重点实验室,山东 青岛 266100;
4. 中国海洋大学环境科学与工程学院,山东 青岛 266100
厌氧氨氧化过程(Anaerobic ammonium oxidation, Anammox)是在厌氧环境中,以NH4+作为电子供体,NO2-作为电子受体,将氨氮(NH4+-N)和亚硝酸氮(NO2--N)转化为氮气的生物氧化过程[1]。这一发现,改变了传统观念中反硝化为唯一氮素移除途径的认识。近几年的研究表明,厌氧氨氧化现象几乎存在于所有的含氮低氧环境中[2-4]。厌氧氨氧化细菌(Anammox bacteria)作为该过程的主要驱动者,广泛分布于自然界低氧环境以及污水处理厂等人工低氧环境中,富含有机质的厌氧或低氧沉积物是其典型生境。尽管至今尚未被纯培养,通过一系列以DNA为基础的分子生物学技术手段的研究表明,厌氧氨氧化细菌属浮霉菌门(Planctomycetes),目前已发现6个属,分别是Ca. Scalindua[5],Ca. Brocadia[6],Ca. Kuenenia[7],Ca. Anammoxglobus[8],Ca. Jettenia[9],Ca. Anammoximicrobium moscowii[10],在人工环境和自然环境等不同生态系统中均有分布。其中,Ca. Scalindua属在海洋环境中为优势属,其他5个属常发现于污水处理厂污泥以及淡水湖泊河流沉积物中[11-12]。
厌氧氨氧化现象对于海洋氮元素生物地球化学循环及海洋生态学具有重要的意义。研究者们在海洋低氧水体与沉积物、缺氧海湾盆地[2]、极地海冰[13]、深海热液喷口[14]等多种海洋低氧环境中,均发现了厌氧氨氧化细菌的存在,对于生源要素——氮的海洋生物地球化学循环具有意义重大。研究者在越来越多的海区中发现了厌氧氨氧化细菌存在的踪迹[3, 13, 15-18],通过实时荧光定量PCR技术(qPCR)对中国边缘海沉积物中厌氧氨氧化细菌的丰度进行研究,Dang等[19]在胶州湾中检测到厌氧氨氧化细菌占总微生物量的0.047%~0.21%,Fu等[20]在珠江口海域的研究发现,厌氧氨氧化细菌与总微生物量的比值仅为0.02%,结合本课题组此前在长江口及邻近海域研究厌氧氨氧化细菌比例在0.26%~5.2%之间的实验结果[21],可以看出,相较于总微生物量,海洋生态环境中厌氧氨氧化细菌的丰度较低,通过微生物16S rRNA基因通用引物来研究厌氧氨氧化细菌的群落组成尤为困难。而考虑到不同属的厌氧氨氧化细菌的16S rRNA基因序列分歧较大(< 87.1%序列相似性)[22],选择能够覆盖全部厌氧氨氧化细菌的16S rRNA基因的特异性引物来研究其群落结构和丰度就显得十分必要。
近期研究中,厌氧氨氧化细菌16S rRNA(Amx-16SrRNA)基因的两对特异性引物(Amx368F/Amx820R和Brod541F/Amx820R)在研究海洋、河口等生态环境中厌氧氨氧化细菌的丰度分布以及群落结构时应用较为广泛[23-26]。据报道,Amx368F是定向扩增厌氧氨氧化细菌所有属的前引物,之前的研究发现,Amx368F可以十分高效地覆盖厌氧氨氧化细菌[22, 27],而Brod541F更专注于Ca. Scalindua属,同时,后引物Amx820R侧重于Ca. Scalindua属、Ca. Brocadia属和Ca. Kuenenia属[3, 5](见表 1)。Hong等[23]用Amx368F/Amx820R、Brod541F/Amx820R两对引物在南海远海沉积物中检测到的厌氧氨氧化细菌均为Ca. Scalindua属。Hou等[24]用引物Amx368F/Amx820R在长江口潮浸浅滩地沉积物中检测到Ca. Scalindua, Ca. Brocadia, Ca. Kuenenia三个属的厌氧氨氧化细菌。Zheng等[25]以引物Amx368F/Amx820R在长江口及邻近东黄海海域沉积物中检测到Ca. Scalindua、Ca. Brocadia、Ca. Kuenenia和 Ca.Anammoxoglobus四个属。Chen等[26]以引物Brod541F/Amx820R在德国Scheld-Rhine Meuse三角洲盐湖Grevelingen湖泊沉积物中检测到厌氧氨氧化细菌优势类群为Ca. Scalindua属。
|
|
表 1 引物名称、序列及覆盖范围 Table 1 Primers sequences, target sites and coverage on genus level |
针对以上常用于研究海洋环境中厌氧氨氧化细菌的两对特异性引物扩增所得的群落结构差异较大这一现状,选择这两对引物对同一样品检测到的厌氧氨氧化细菌群落特征进行比较分析。本研究基于高通量测序结果对长江口及邻近海域沉积物中厌氧氨氧化细菌的群落丰度、多样性信息进行分析,着重分析2对Amx-16S rRNA基因特异性引物对研究海域沉积物中的厌氧氨氧化细菌的覆盖情况,比较2对引物在研究厌氧氨氧化细菌群落多样性、丰度分布等群落特征的差异和优劣,以期为后期实验中基因以及引物的选择提供依据,为长江口及邻近海域沉积物中厌氧氨氧化细菌的分子生态学研究提供指导性参考,从而提高对海洋沉积物中厌氧氨氧化细菌研究的准确性,促进对河口生态系统氮循环过程的全面了解。
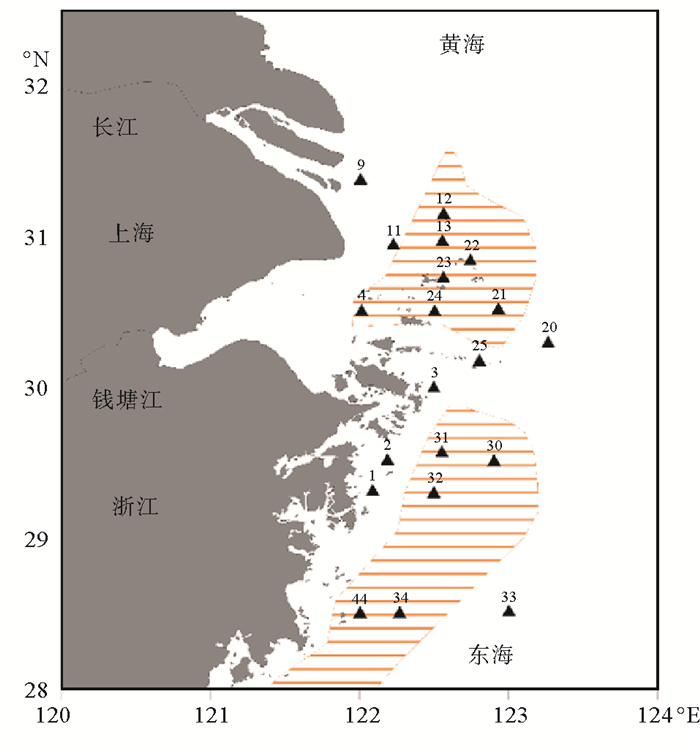
1 材料与方法 1.1 样品采集2011年7月29日—8月4日在长江口邻近海域(122°E~123.5°E,28°N~32°N)20个站位(见图 1),采集样品是深度为0~3 cm的表层沉积物。沉积物样品放置在无菌封口袋中,用锡箔纸包裹后立即置于液氮中保存,到实验室后转于-80℃冰箱中保存。
1.2 DNA提取与引物特异性验证用Power Soil DNA Isolation Kit(MO Bio,美国)提取沉积物微生物基因组DNA,于-80℃保存,用于分子生态学研究。用下列引物(见表 2)分别扩增Amx-16S rRNA基因、功能基因hzo片段,并进行回收纯化。
|
|
表 2 荧光定量PCR引物与反应程序 Table 2 Real-time fluorescence quantitative PCR primers and amplification procedures |
将目的基因片段连接、转化、测序后制备质粒标准品,建立qPCR体系,使用FastStart Universal SYBR Green Master(ROX)试剂盒(Roche, Germany)进行荧光定量PCR(qPCR)反应(ABI7500, USA),并于反应体系中加入0.2 μg·μL-1牛血清蛋白,绘制标准曲线。
用引物Amx368F/Amx820R和Brod541F/Amx820R分别定量Amx-16S rRNA基因的拷贝数,引物hzo-hzo5F/hzo5R定量hzo基因的拷贝数,反应体系为:质粒DNA 2 μL,引物各0.6 μL,ROX 10 μL,BSA 0.2 μL,ddH2O 6.6 μL,反应程序如表 2。取2 μL样品DNA按上述条件进行qPCR实验,每个DNA样品设置3个平行,同时设置阳性、阴性对照。通过标准曲线换算样品中Amx-16S rRNA基因2对引物以及功能基因hzo的拷贝数。
|
(图中阴影部分分别为长江口泥质区、浙闽沿岸泥质区(重绘自秦蕴珊等,1987)。The shaded area means the muddy area in the Changjiang Estuary and Zhejiang-Fujian offshore (repainted by Qin et al., 1987).) 图 1 长江口临近海域采样站位示意 Fig. 1 Sampling sites in the Changjiang Estuaryandits adjacent area |
用上述两对引物分别扩增Amx-16S rRNA基因,扩增时在引物5’端加入barcode以区分不同样品的序列,构建扩增子克隆文库。由于测序平台对扩增子目的基因片段长度的要求,针对引物Amx368F/Amx820R采用454测序平台对沉积物DNA进行双端(Paired-end)测序,同时对引物Brod541F/Amx820R采用Illumina Miseq PE250测序平台对沉积物DNA进行双端测序。将原始下机数据进行格式转换后,利用QIIME(Version 1.9.0, http://qiime.org/scripts/split_libraries_fastq.html)筛选原始下机序列得到高质量序列,用于后续分析。
使用Uparse软件[29]对高质量序列按97%序列相似度进行OTU聚类,选择每个OTU的一条序列作为该OTU的代表序列,对代表序列进行物种注释后,分析物种的相对丰度。利用QIIME软件分析各个高通量测序样品的Alpha多样性,绘制稀释曲线。将每个OTU代表序列用Mega5.0软件以邻接法(Neighbour Joining, NJ)进行系统发育树分析。
1.5 基因序列登录号基于2对特异性引物(Amx368F/Amx820R和Brod541F/Amx820R)的高通量测序委托上海派森诺生物科技有限公司进行。本研究获得的Amx-16S rRNA基因序列在SRA上的登录号分别为SRP112655:PRJNA394772(Amx368F/Amx820R)和SRP131428:PRJNA431658(Brod541F/Amx820R)。
2 结果与分析选取2011年7—8月在东海所采集的4个站位(S13、S20、S31、S33)的表层沉积物样品作为代表站位,其中S13站位于长江口外泥质区;S20站位于长江口外非泥质区;S31站位于浙闽沿岸泥质区;S33站位于浙闽沿岸泥质区外的外海,厌氧氨氧化细菌的丰度在考察站位中最高。通过厌氧氨氧化细菌16S rRNA基因的454、Illumina高通量测序对东海表层沉积物中厌氧氨氧化细菌群落特征进行描述。
2.1 厌氧氨氧化细菌群落多样性比较将每个沉积物样品(S13、S20、S31、S33)得到的高质量序列,使用QIIME、Uparse等生物信息软件对序列进行严格质量控制,对优质序列按序列相似度97%进行OTU聚类,选取每个OTU中的最长序列为代表序列。通过GenBank序列比对,结果表明,质控后的序列均为厌氧氨氧化细菌的序列。质控后的高质量序列条数和在相似度为97%下进行聚类,得到OTUs数及覆盖度分布情况(见表 3)。
|
|
表 3 厌氧氨氧化细菌16S rRNA基因的两对引物的多样性和丰富度参数 Table 3 Diversity Characteristics of two Anammox bacterial 16S rRNA gene primers |
对于同一沉积物样品,基于2对引物检测到的Chao1指数、Shannon指数、Simpson指数的比较可以看出,引物Brod541F/Amx820R明显高于引物Amx368F/Amx820R,从整体趋势来说,引物Brod541F/Amx820R检测到的群落丰富度和多样性都明显高于引物Amx368F/Amx820R。
在4个表层沉积物样品中,两对引物Amx368F/Amx820R(引物1)、Brod541F/Amx820R(引物2)分别获得92 007、156 931条优质序列,每个样品对应获得的序列条数区间为(见表 3)。以97%序列相似性对上述高质量序列进行OTU聚类,并采用RDP Classifier方法将OTU代表序列与NCBI数据库进行比对、物种注释,删除出现频数为1(singletons)、无法聚类到OTU的序列。经随机抽样将测序深度均一化后,每个样品两对引物所得的OTUs个数分别介于54~73、349~2 199之间(见表 3)。
Chao1、Shannon和Simpson指数分别用于表征群落的多样性和丰富度情况。Chao1指数越大,表明群落的丰富度越高;Shannon和Simpson指数综合考虑了群落的丰富度和均匀度,其指数值越高,表明群落的多样性越高,一般而言,Shannon指数对群落丰富度以及稀有OTU较为敏感,而Simpson指数对均匀度和群落中的优势OTU更为敏感。由表 3可知,两对引物的Chao1、Shannon和Simpson指数有较大差别,Brod541F/Amx820R引物的Chao1指数较大,说明检测到的厌氧氨氧化细菌菌群的丰富度较高;Brod541F/Amx820R引物的Simpson指数较大,说明该引物检测到的细菌菌群的多样性较高且均匀度较好。在4个样品每个基因测得序列的覆盖度均在99%以上,说明本研究所获得的序列条数足以覆盖样品中厌氧氨氧化细菌的绝大多数物种信息,而不同站位采用不同引物测得的多样性和丰富度不同。
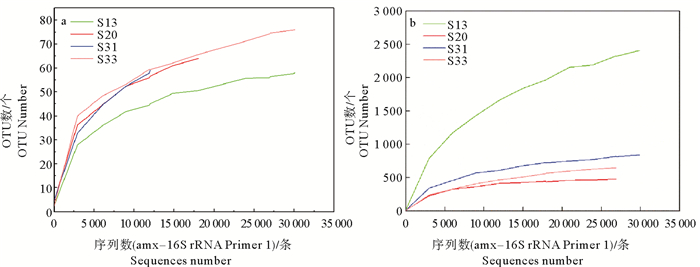
如图 2所示,4个样品的两对引物的稀释曲线除引物1的S31站外,均趋于平缓,说明本研究测序条数足够,测序深度选取合理。引物1的稀释曲线是S13站首先趋于平缓,S31站是最后出现平缓趋势;而对于引物2的情况是,S20站首先趋于平缓,S13站最后出现平缓趋势,这与表 2中的多样性指数和丰富度指数的高低变化相一致。
|
(a、b分别对应引物Amx368F/Amx820R、Brod541F/Amx820R的稀释曲线。a and b represent the rarefaction curves of Primer Amx368F/Amx820R and Brod541F/Amx820R, respectively.) 图 2 不同引物所得厌氧氨氧化细菌群落的稀释曲线 Fig. 2 Rarefaction curves of Anammox bacterial community obtained from high-throughput sequencing |
将引物1通过454平台和引物2通过Illumina Miseq平台高通量测序所得OTU代表序列分别与NCBI数据库比对并进行物种注释,结果表明,在门分类水平上,引物1测得序列均属浮霉菌门(Planctomycetes)(见表 4)。引物2可以检测到浮霉菌门(Planctomycetacia)、放线菌门(Actinobacteria)、硝化螺旋菌门(Nitrospirae)、绿弯菌门(Chloroflexi)等近40个门类的细菌,而不仅仅是浮霉菌门(Planctomycetacia)(见表 4),厌氧氨氧化细菌所在的浮霉菌门(Planctomycetacia)的序列条数仅占总序列条数的44.1%。为便于分析,选取丰度最高的前30个OTUs进行后续的相对丰度与系统发育分析。
|
|
表 4 两对引物在不同分类水平对厌氧氨氧化细菌的覆盖情况 Table 4 Results of the two pairs of primers detected the Anammox bacteria at different levels |
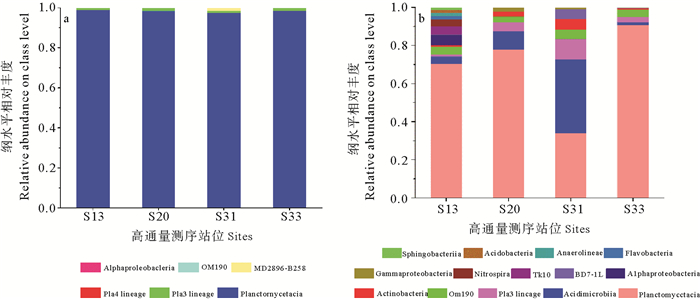
在纲分类水平上,浮霉菌纲(Planctomycetacia)在引物1测序所得序列中占绝对优势(>99%),其他序列约占0~1%,分别属于α-变形杆菌纲(Alphaproteobacteria)、Pla3、Pla4和MD2896-B258等(见表 4、图 3a)。引物2可以覆盖到浮霉菌纲(Planctomycetacia)、放线菌纲(Actinobacteria)、α-变形杆菌纲(Alphaproteobacteria)、硝化螺旋菌纲(Nitrospira)、黄杆菌纲(Flavobacteria)、γ-变形菌纲(Gammaproteobacteria)、酸杆菌纲(Acidobacteria)、厌氧绳菌纲(Anaerolineae)、Pla3、OM190等(见表 4、图 3b)。
|
图 3 引物1(a)、引物2(b)扩增序列在纲分类水平上的相对丰度 Fig. 3 Quantitated sequences by Amx-16S rRNA gene primer 1 (a) and primer 2 (b) at class level |
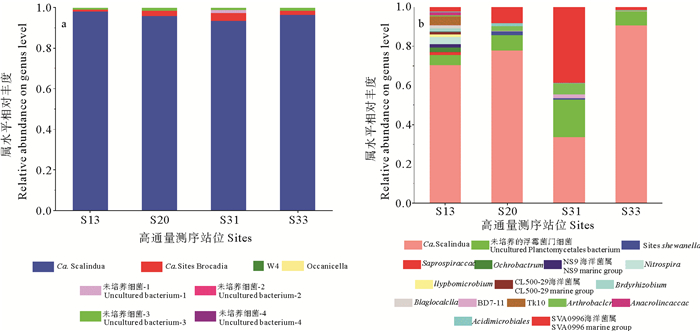
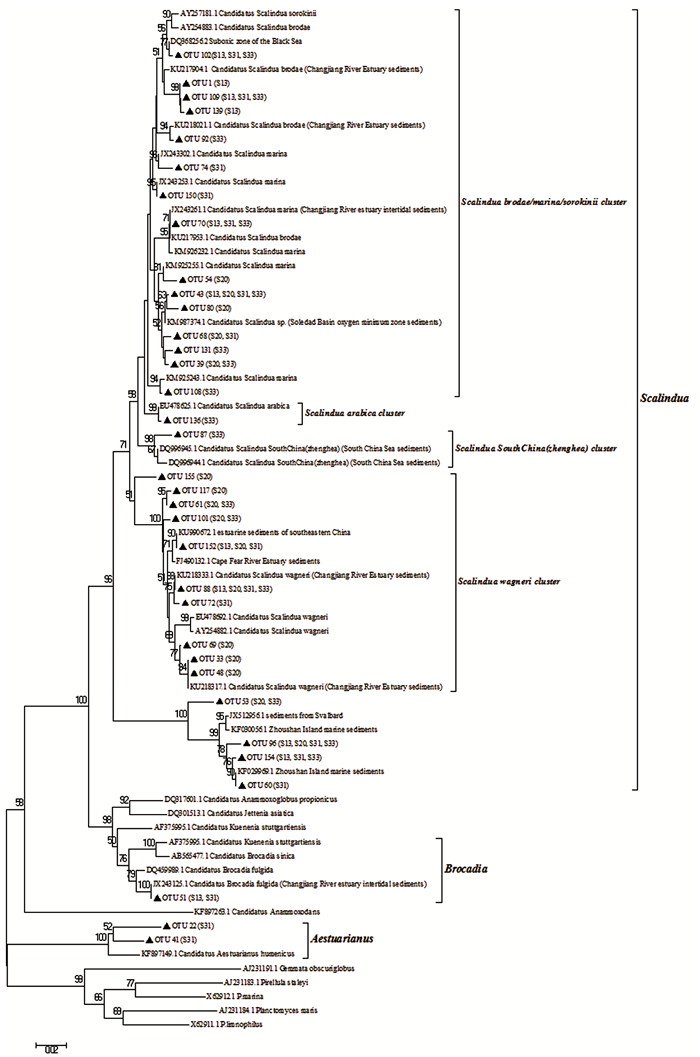
在属分类水平,引物1可以检测到Ca. Scalindua、Ca.Brocadia两个已知厌氧氨氧化细菌属,和Oceanicella、W4以及浮霉菌纲的其它未培养厌氧氨氧化细菌(见表 4、图 4a)。Ca. Scalindua在每个站位的相对丰度都在90%以上,为测序站位的优势属,其他属的厌氧氨氧化细菌相对丰度很低(见表 4、图 4b)。
|
图 4 引物1(a)、引物2(b)扩增序列在属分类水平上的相对丰度 Fig. 4 Quantitated sequences by Amx-16S rRNA gene primer 1 (a) and primer 2 (b) at genus level |
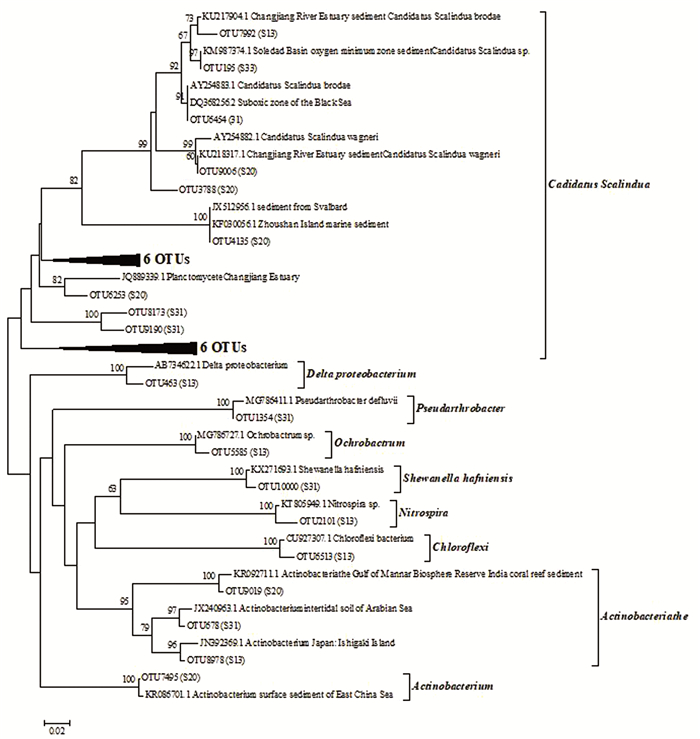
从排名前30的OTUs属水平上看,引物2可以检测到的优势菌群中,仍有较多非厌氧氨氧化细菌的菌群。系统发育分析表明,已经检测到的厌氧氨氧化细菌有3个已知属,分别为Ca. Scalindua、Ca. Brocadia、Ca. Kuenenia以及一些未知的厌氧氨氧化细菌,但该对引物还检测到了许多非厌氧氨氧化菌群的其他微生物类群(见图 5、6),通过引物1检测到的厌氧氨氧化细菌优势属Ca.Scalindua仅占引物2测序所得总序列条数的43.7%,Ca. Brocadia、Ca. Kuenenia的相对丰度约为0.4%。在引物2覆盖的丰度排名前30的OTUs中,存在大量非厌氧氨氧化细菌类群。因而认为,引物1较适合厌氧氨氧化细菌的分子生态学研究,而引物2不适合。
|
图 5 长江口及邻近海域表层沉积物中厌氧氨氧化细菌16S rRNA基因(引物1)的系统发育分析 Fig. 5 Phylogenetic tree of Amx-16S rRNA gene (Primer 1) in surface sediments from the Changjiang Estuary and its adjacent area |
|
图 6 长江口及邻近海域表层沉积物中厌氧氨氧化细菌16S rRNA基因(引物2)的系统发育分析 Fig. 6 Phylogenetic tree of Amx-16S rRNA gene (Primer 2) in surface sediments from the Changjiang Estuary and its adjacent area |
将含有目的基因片段的质粒进行PCR扩增,经电泳检验、测序、GenBank比对,证实所得质粒标准品中已插入目的基因片段。qPCR实验分析结果表明,3对引物的熔解曲线仅有1个显著峰值,即在解链温度处,排除引物二聚体及其他非特异性扩增对qPCR结果的影响。对具有浓度梯度的质粒标准品进行qPCR反应,获得3对引物对应基因的标准曲线。qPCR实验制备标准曲线过程中,所得Ct值与3对引物相对应质粒拷贝数的常用对数值有很好的线性负相关关系(RAmx-16S-12=0.997,RAmx-16S-22= 0.999,Rhzo2= 0.997)。
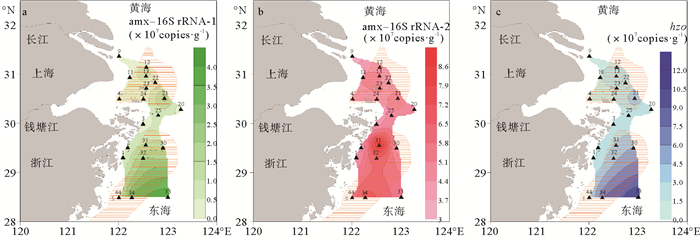
通过上述3对引物研究表层沉积物中厌氧氨氧化细菌丰度分布趋势,对采样站位的amx-16S rRNA基因和功能基因hzo进行定量分析,结果表明,3对引物的分布趋势一致,厌氧氨氧化细菌丰度分布趋势大致表现为南部海域大于北部海域,近海海域大于远海海域(见图 7)。
|
(图a、b、c分别对应引物Amx368F/Amx820R、Brod541F/ Amx820R、hzo5F/hzo5R覆盖的厌氧氨氧化细菌的水平分布趋势。a, b and c represent the horizontal distribution of Anammox bacterial abundance covered by Primer Amx368F/Amx820R, Brod541F/Amx820R and hzo5F/hzo5R, respectively.) 图 7 表层沉积物中厌氧氨氧化细菌丰度(cells/g(湿重))的水平分布 Fig. 7 Horizontal distribution of Anammox bacterial abundance (cells/g(Wet weight)) in surface sediments |
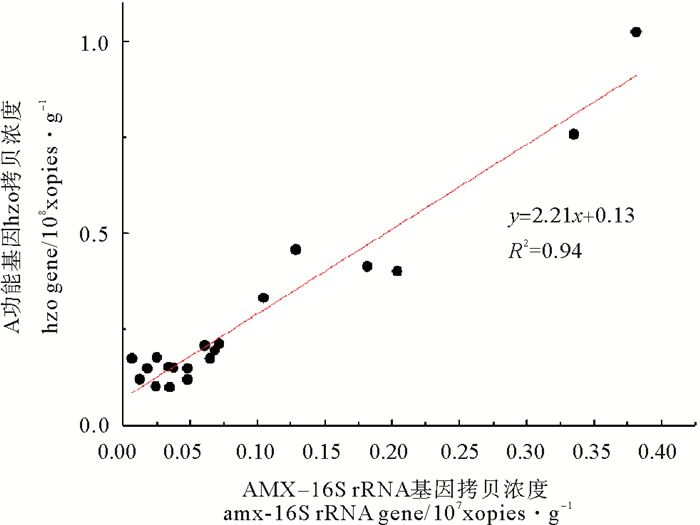
Amx-16S rRNA基因引物1检测的厌氧氨氧化细菌的丰度最高出现在S33站和最低是S22站;Amx-16S rRNA基因引物2检测的厌氧氨氧化细菌的丰度最高出现在S31站和最低是S9站;功能基因hzo的丰度结果是S33站厌氧氨氧化细菌数最高,S1站最低。由qPCR结果可知,通过功能基因hzo定量厌氧氨氧化细菌的平均丰度明显高于Amx-16S rRNA基因2对引物的结果,且由hzo基因量化的厌氧氨氧化细菌是Amx-16S rRNA基因引物1的2倍(P=0.000 < 0.01, r= 0.969)(见图 8),而与Amx-16S rRNA基因引物2的量化结果无明显相关关系(P=0.052 > 0.05)。Shimamura等[30]通过富集培养后宏基因测序发现,厌氧氨氧化细菌功能基因hzo有2个基因编码hzoA和hzoB,厌氧氨氧化细菌基因组中功能基因hzo与16SrRNA基因数量上的2倍关系。引物1符合厌氧氨氧化细菌本身的基因数量关系。因而引物1更适合应用于相关分子生态学研究。
|
图 8 Amx-16S rRNA基因(引物1)和hzo基因 Fig. 8 The quantitative relationship between Amx-16S rRNA gene(Primer 1) and hzo gene copy numbers |
自上1990年代厌氧氨氧化过程被发现[1, 31]以来,研究者在多种海洋低氧环境中[2, 13-14]均发现厌氧氨氧化细菌。以PCR为基础的克隆文库技术为主流的分子生物学技术被广泛应用于厌氧氨氧化细菌群落信息的研究,从一定程度上揭示了厌氧氨氧化菌群的多样性及其在各种生态环境中分布的广泛性。但由于克隆文库技术的实验操作时挑选克隆数量的限制其测序深度,此种技术手段所得有效目的片段的序列条数通常在几百条,对于环境样品中菌群的覆盖度较低,难以全面阐述菌群的群落多样性信息。同时,克隆文库技术自身的限制可能会使实验反映的群落信息存在一定的偏向性和偶然性,使获得的厌氧氨氧化菌群多样性信息的真实性降低。Dang等[19]、Hou等[24]、Zheng等[25]采用克隆文库的技术手段先后对辽东湾及渤海表层沉积物、长江口潮浸浅滩、长江口及邻近区域沉积物中厌氧氨氧化菌群分布及多样性的研究表明,Amx-16S rRNA基因的有效序列条数在100条以下,覆盖度在90%左右;Li等[32]研究南海红树林沉积物中厌氧氨氧化群落结构多样性信息时,应用克隆文库技术检测到的hzo基因序列的有效条数在20~60条之间,覆盖度在80%~100%之间不等,由此可能导致对厌氧氨氧化菌群群落结构信息的分析产生一定的偶然性。
通过不同引物对研究海域沉积物中的厌氧氨氧化细菌丰度分布进行检测,将所得结果对数处理后用单样本k-s检验验证了3对引物检测的厌氧氨氧化细菌丰度数值的正态性,结果表明Amx-16S rRNA基因引物1(P=0.958>0.05)、Amx-16S rRNA基因引物2(P=0.718>0.05)和功能基因hzo引物(P=0.299>0.05)检测到的厌氧氨氧化细菌在研究海域的丰度分布趋势具有空间异质性。用皮尔森(Pearson)相关性检验验证了不同引物覆盖厌氧氨氧化细菌丰度分布的相关性,结果表明3对引物拷贝数对数值具有显著相关关系(P < 0.01, r1-2=0.613, r1-hzo=0.842, r2-hzo=0.566),3对引物覆盖的厌氧氨氧化细菌在研究区域的分布趋势大体一致。One-Way ANOVA检验结果也表明厌氧氨氧化细菌的丰度趋势是一致的(P>0.05)。在所考察区域,整体表现为长江口处丰度较低,长江口外较远区域较高;近岸较低,远岸较高(见图 7)。对于个别站位,如位于外海非泥质区的S33站位的厌氧氨氧化菌群丰度,Amx-16S rRNA基因引物1和功能基因hzo引物hzo5F/hzo5R表现为所有站位中厌氧氨氧化细菌的丰度最高,Amx-16S rRNA基因引物2则表现为S33站位较泥质区略有降低,引物2检测到的厌氧氨氧化细菌的分布趋势与其它2对引物略有不同。且由于Amx-16S rRNA基因引物2扩增的占总量50%左右的序列不是厌氧氨氧化细菌,该对引物较其他2对引物来说不适合用于研究厌氧氨氧化菌群的丰度分布趋势。
以往研究者大多采用克隆文库技术对环境样品中的厌氧氨氧化菌群多样性信息进行分析,覆盖度之间的不均性,存在一定的不完整性与偶然性。本研究采用454、Illumina高通量测序技术研究中国边缘海沉积物中厌氧氨氧化细菌的群落分布情况,高通量测序可获得的序列条数在几万条。Yang等[33]通过Illumina高通量测序技术以Amx-16S rRNA基因为目标对淡水湖沉积物进行厌氧氨氧化细菌群落多样性分析,获得10 000~30 000条序列,覆盖度在99.9%~100%之间。Qin等[34]通过Illumina高通量测序技术以hzsβ基因为目的基因对湖泊沉积物中厌氧氨氧化细菌进行分子生态学研究,获得18 000~35 000条序列,较为全面地揭示了湖泊生态系统中厌氧氨氧化细菌的组成及其对生态环境的作用。结合本研究可知,高通量测序所得序列条数远远高于克隆文库技术所得数值,覆盖度都能到99%以上,较克隆文库而言,高通量测序更能全面真实地反映厌氧氨氧化细菌群落的多样性信息。
目前,已知的厌氧氨氧化细菌都属于浮霉菌门(Planctomycetes),共有6个属,分别是Ca. Scalindua、Ca. Kuenenia、Ca. Brocadia、Ca. Anammoxoglobus、Ca. Jettenia[27]以及Ca. Anammoximicrobiummoscowii[10]。海洋环境中最常见的属是Ca. Scalindua。常用于检测环境中厌氧氨氧化菌群群落结构的基因是16S rRNA基因,本研究采用Amx-16S rRNA基因的2对引物过去广泛被克隆文库技术利用。引物Amx368F/Amx820R广泛应用于检测淡水[35]和滨海湿地[36],水稻田[37-38]以及河口、海洋沉积物[24, 40]中的厌氧氨氧化细菌。就以上研究成果可知,该引物可以覆盖不同环境中的Ca. Scalindua、Ca. Brocadia和Ca. Kuenenia三个属。厌氧氨氧化细菌在不同生态环境中的优势属随外界环境因素变化而改变。Dale等[39]应用该对引物对Cape Fear河口沉积物中厌氧氨氧化细菌的群落结构研究表明,河口沉积物中厌氧氨氧化菌群优势属随盐度升高由Ca. Brocadia过渡为Ca. Kuenenia,继续向外延伸的海洋沉积物中则主要以Ca. Scalindua属为主。引物Brod541F/Amx820R在过去研究中,更多地应用于滨海[40]、海洋[41]沉积物中厌氧氨氧化菌群的研究,可以覆盖Ca. Scalindua、Ca. Brocadia和Ca. Kuenenia属。本章通过3对不同引物检测到的厌氧氨氧化细菌优势属也均为Ca. Scalindua,通过Amx-16S rRNA基因引物1检测到的序列全都是浮霉菌门,覆盖到厌氧氨氧化细菌的3个已知属,Amx-16S rRNA基因引物2检测到浮霉菌门序列所占比例不到50%,有一半以上是其他门类菌群,覆盖到厌氧氨氧化细菌的3个已知属。
总体而言,两对引物检测到的厌氧氨氧化菌群优势属均为Ca. Scalindua,能较完整的覆盖研究区域的厌氧氨氧化菌群,引物1扩增的序列均为厌氧氨氧化菌群,而引物2扩增结果中有50%以上的序列不是厌氧氨氧化菌群,因此,Amx-16S rRNA基因引物Amx368F/Amx820R更适合该研究海域的厌氧氨氧化细菌的丰度分布和多样性特征的分析。
4 结语本研究采用454、Illumina高通量测序和qPCR技术,利用厌氧氨氧化细菌16S rRNA基因的两对特异性引物(Amx368F/Amx820R、Brod541F/Amx820R)对长江口及邻近海域沉积物中厌氧氨氧化细菌的群落组成、多样性及丰度进行比较分析,结果表明,虽然两对特异性引物均覆盖到研究海域中厌氧氨氧化细菌的优势属Ca. Scalindua,但引物Amx368F/Amx820R的特异性优于引物Brod541F/Amx820R,该引物更适用于海洋沉积物中厌氧氨氧化细菌的分子生态学研究。
致谢: 感谢中国海洋大学环境科学与工程学院张玉博士在软件使用中提供的帮助!
| [1] |
Mulder A, Graaf A A V D, Robertson L A, et al. Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor[J]. FEMS Microbiology Ecology, 1995, 16(3): 177-184. DOI:10.1111/fem.1995.16.issue-3
(  0) 0) |
| [2] |
Kuypers M MM, Lavik G, Thamdrup B. Anaerobic ammonium oxidation in the marine environment[J]. Past and Present Water Column Anoxia, 2006, 64: 311-335. DOI:10.1007/1-4020-4297-3
(  0) 0) |
| [3] |
Penton C R, Devol A H, Tiedje J M. Molecular evidence for the broad distribution of anaerobic ammonium-oxidizing bacteria in freshwater and marine sediments[J]. Applied and Environmental Microbiology, 2006, 72(10): 6829-6832. DOI:10.1128/AEM.01254-06
(  0) 0) |
| [4] |
Francis C A, Beman J M, Kuypers M MM. New processes and players in the nitrogen cycle: The microbial ecology of anaerobic and archaeal ammonia oxidation[J]. ISME Journal, 2007, 1(1): 19-27. DOI:10.1038/ismej.2007.8
(  0) 0) |
| [5] |
Schmid M, Walsh K, Webb R I, et al. Candidatus′ Scalinduabrodaea′, spec. nov.Candidatus ′Scalinduawagneri′, spec. nov. two new species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2003, 26(4): 529-538. DOI:10.1078/072320203770865837
(  0) 0) |
| [6] |
Jetten M S M, Wagner M, Fuerst J, et al. Microbiology and application of the anaerobic ammonium oxidation('Anammox') process[J]. Current Opinion in Biotechnology, 2001, 12(3): 283-288. DOI:10.1016/S0958-1669(00)00211-1
(  0) 0) |
| [7] |
Schmid M, Twachtmann U, Klein M, et al. Molecular evidence for genus level diversity of bacteria capable of catalyzing anaerobic ammonium oxidation[J]. Systematic and Applied Microbiology, 2000, 23(1): 93-106. DOI:10.1016/S0723-2020(00)80050-8
(  0) 0) |
| [8] |
Kartal B, Rattray J, van Niftrik L A, et al. Candidatus "Anammoxoglobuspropionicus" a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2007, 30(1): 39-49. DOI:10.1016/j.syapm.2006.03.004
(  0) 0) |
| [9] |
Quan Z X, Rhee S K, Zuo J E, et al. Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox) reactor[J]. Environmental Microbiology, 2008, 10(11): 3130-3139. DOI:10.1111/emi.2008.10.issue-11
(  0) 0) |
| [10] |
Khramenkov S V, Kozlov M N, Krevbona M V, et al. A novel bacterium carrying out anaerobic ammonium oxidation in a reactor for biological treatment of the filtrate of wastewater fermented residue[J]. Microbiology, 2013, 82(5): 628-636. DOI:10.1134/S002626171305007X
(  0) 0) |
| [11] |
Kalvelage T, Jensen M M, Contreras S, et al. Oxygen sensitivity of anammox and coupled N-cycle processes in oxygen minimum zones[J]. Plos One, 2011, 6(12): e29299. DOI:10.1371/journal.pone.0029299
(  0) 0) |
| [12] |
Han P, Gu J D. More refined diversity of anammox bacteria recovered and distribution in different ecosystems[J]. Applied Microbiology and Biotechnology, 2013, 97(8): 3653-3663. DOI:10.1007/s00253-013-4756-6
(  0) 0) |
| [13] |
Rysgaard S, Glud R N. Anaerobic N2 production in Arctic Sea ice[J]. Limnology and Oceanography, 2004, 49(1): 86-94. DOI:10.4319/lo.2004.49.1.0086
(  0) 0) |
| [14] |
Byrne N, Strous M, Crépeau V, et al. Presence and activity of anaerobic ammonium-oxidizing bacteria at deep-sea hydrothermal vents[J]. ISME Journal, 2009, 3(1): 117-123. DOI:10.1038/ismej.2008.72
(  0) 0) |
| [15] |
Kuypers M MM, Sliekers A O, Lavik G, et al. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J]. Nature, 2003, 422(6932): 608-611. DOI:10.1038/nature01472
(  0) 0) |
| [16] |
Amano T, Yoshinaga I, Okada K, et al. Detection of anammoxactivity and diversity of anammox bacteria-related 16S rRNA genes in coastal marine sediment in Japan[J]. Microbes and Environments, 2007, 22(3): 232-242. DOI:10.1264/jsme2.22.232
(  0) 0) |
| [17] |
Hamersley M R, Lavik G, Woebken D, et al. Anaerobic ammonium oxidation in the Peruvian oxygen minimum zone[J]. Limnology and Oceanography, 2007, 52(3): 923-933. DOI:10.4319/lo.2007.52.3.0923
(  0) 0) |
| [18] |
Dalsgaard T, Canfield D E, Petersen J, et al. N2 production by the anammox reaction in the anoxic water column of Golfo Dulce, Costa Rica[J]. Nature, 2003, 422(6932): 606-608. DOI:10.1038/nature01526
(  0) 0) |
| [19] |
Dang H, Chen R, Wang L, et al. Environmental factors shape sediment anammox bacterial communities in hypernutrifiedJiaozhou Bay, China[J]. Applied and Environmental Microbiology, 2010, 76(21): 7036-7047. DOI:10.1128/AEM.01264-10
(  0) 0) |
| [20] |
Fu B B, Liu J W, Yang H M, et al. Shift of anammox bacterial community structure along the Pearl Estuary and the impact of environmental factors: Anammox bacteria along the Pearl Estuary[J]. Journal of Geophysical Research Oceans, 2015, 120(4): 2869-2883. DOI:10.1002/2014JC010554
(  0) 0) |
| [21] |
付璐璐, 甄毓, 贺惠, 等. 长江口邻近海域沉积物中厌氧氨氧化细菌分布特征研究[J]. 环境科学, 2016, 37(10): 3914-3922. (  0) 0) |
| [22] |
Fu LL, Zhen Y, He H, et al. Distribution characteristics of anaerobic ammonia oxidation bacteria in sediments from the adjacent seas of Yangtze Estuary[J]. Environmental Sciences, 2016, 37(10): 3914-3922.
(  0) 0) |
| [23] |
Han P, Huang Y T, Lin J G, et al. A comparison of two 16S rRNA gene-based PCR primer sets in unraveling anammox bacteria from different environmental samples[J]. Applied Microbiology and Biotechnology, 2013, 97(24): 10521-10529. DOI:10.1007/s00253-013-5305-z
(  0) 0) |
| [24] |
Hong Y G, Li M, Cao H, et al. Residence of habitat-specific anammox bacteria in the deep-sea subsurface sediments of the South China Sea: Analyses of marker gene abundance with physical chemical parameters[J]. Microbial Ecology, 2011, 62(1): 36-47.
(  0) 0) |
| [25] |
Hou L, Zheng Y, Liu M, et al. Anaerobic ammonium oxidation(Anammox) bacterial diversity, abundance, and activity in marsh sediments of the Yangtze Estuary[J]. Journal of Geophysical Research Biogeosciences, 2014, 118(3): 1237-1246.
(  0) 0) |
| [26] |
Zheng Y, Jiang X, Hou L, et al. Shifts in the community structure and activity of anaerobic ammonium oxidation bacteria along an estuarine salinity gradient[J]. Journal of Geophysical Research Biogeosciences, 2016, 121(6): 1632-1645. DOI:10.1002/2015JG003300
(  0) 0) |
| [27] |
Chen J, Zhou H C, Pan Y, et al. Effects of polybrominated diphenyl ethers and plant species on nitrification, denitrification and anammox in mangrove soils[J]. Science of the Total Environment, 2016, 553(4): 60-70.
(  0) 0) |
| [28] |
Sonthiphand P, Hall M W, Neufeld J D. Biogeography of anaerobic ammonia-oxidizing(Anammox) bacteria[J]. Frontiers in Microbiology, 2014, 5(399): 1-14.
(  0) 0) |
| [29] |
Li M, Cao H, Hong Y G, et al. Using the variation of Anammox bacteria community structures as a bio-indicator for anthropogenic/terrestrial nitrogen inputs in the Pearl River Delta(PRD)[J]. Applied Microbiology and Biotechnology, 2013, 97(22): 9875-9883. DOI:10.1007/s00253-013-4990-y
(  0) 0) |
| [30] |
Edgar R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604
(  0) 0) |
| [31] |
Shimamura M, Nishiyama T, Shigetomo H S, et al. Isolation of a multiheme protein with features of a hydrazine-oxidizing enzyme from an anaerobic ammonium-oxidizing enrichment culture[J]. Applied and Environmental Microbiology, 2007, 73(4): 1065-1072. DOI:10.1128/AEM.01978-06
(  0) 0) |
| [32] |
Mulder A. Anoxic ammonia oxidation[P]: US, 1992, 5078884.
(  0) 0) |
| [33] |
Li M, Cao H, Hong Y G, et al. Seasonal dynamics of anammox bacteria in estuarial sediment of the Mai Po Nature Reserve revealed by analyzing the 16S rRNA and hydrazine oxidoreductase(hzo) genes[J]. Microbes and Environments, 2011, 26(1): 15-22. DOI:10.1264/jsme2.ME10131
(  0) 0) |
| [34] |
Yang Y, Dai Y, Li N, et al. Temporal and spatial dynamics of sediment anaerobic ammonium oxidation(Anammox) bacteria in freshwater lakes[J]. Microbial Ecology, 2017, 73(2): 1-11.
(  0) 0) |
| [35] |
Qin H, Han C, Jin Z, et al. Vertical distribution and community composition of anammox bacteria in sediments of a eutrophic shallow lake[J]. Journal of Applied Microbiology, 2018. DOI:10.1111/jam.13758
(  0) 0) |
| [36] |
Wang J, Gu J D. Dominance of Candidatus, Scalindua species in anammox community revealed in soils with different duration of rice paddy cultivation in Northeast China[J]. Applied Microbiology and Biotechnology, 2013, 97(4): 1785-1798. DOI:10.1007/s00253-012-4036-x
(  0) 0) |
| [37] |
Wang Y F, Feng Y Y, Ma X, et al. Seasonal dynamics of ammonia/ammonium-oxidizing prokaryotes in oxic and anoxic wetland sediments of subtropical coastal mangrove[J]. Applied Microbiology and Biotechnology, 2013, 97(17): 7919-7934. DOI:10.1007/s00253-012-4510-5
(  0) 0) |
| [38] |
Wang Y F, Gu J D. Higher diversity of ammonia/ammonium-oxidizing prokaryotes in constructed freshwater wetland than natural coastal marine wetland[J]. Applied Microbiology and Biotechnology, 2013, 97(15): 7015-7033. DOI:10.1007/s00253-012-4430-4
(  0) 0) |
| [39] |
Zhu G, Wang S, Wang Y, et al. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. ISME Journal, 2011, 5(12): 1905-1912. DOI:10.1038/ismej.2011.63
(  0) 0) |
| [40] |
Dale O R, Tobias C R, Song B. Biogeographical distribution of diverse anaerobic ammonium oxidizing(Anammox) bacteria in Cape Fear River Estuary[J]. Environmental Microbiology, 2009, 11(5): 1194-1207. DOI:10.1111/emi.2009.11.issue-5
(  0) 0) |
| [41] |
Li M, Cao H, Hong Y G, et al. Seasonal dynamics of anammox bacteria in estuarial sediment of the maipo nature reserve revealed by analyzing the 16s rRNA and hydrazine oxidoreductase(hzo) genes[J]. Microbes and Environments, 2011, 26(1): 15-22. DOI:10.1264/jsme2.ME10131
(  0) 0) |
| [42] |
Li M, Hong Y, Cao H, et al. Community structures and distribution of anaerobic ammonium oxidizing and nirs -encoding nitrite-reducing bacteria in surface sediments of the South China Sea[J]. Microbial Ecology, 2013, 66(2): 281-296. DOI:10.1007/s00248-012-0175-y
(  0) 0) |
2. Key Laboratory of Marine Chemical Theory and Technology, Ministry of Education, Qingdao 266100, China;
3. Laboratory for Marine Ecologyand Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;
4. College of Environmental Science and Engineering, Ocean University of China, Qingdao 266100, China
 2019, Vol. 49
2019, Vol. 49


