真核细胞必须不断适应的外部条件的变化包括:温度和紫外线等物理参数; 离子浓度、pH值、氧化还原电位和代谢物浓度等化学因素; 接触依赖信号、激素、细胞因子和神经递质等胞外信号分子和微生物病原体[1]。当外部条件的变化超过一定的阈值,这种变动就被认为是“压力”。细胞对压力的反应决定了它是否能发挥正常功能和生存。在进化过程中,真核细胞已经获得了应对不同类型压力的策略,细胞可以针对特定的威胁选择性地做出对应的策略,这些威胁包括病原体的入侵,缺乏营养物质或细胞内错误折叠的蛋白质积累等[2]。细胞适应环境变化,保护自身免受潜在的伤害的反应包括各种应激反应途径,自噬就是其中之一。
自噬是一种受到严格调控的重要的细胞过程,可通过溶酶体降解和回收受损的细胞器和细胞质成分,能起到保护细胞和维持细胞内部稳态的作用[3]。现有研究表明:细胞在正常状态下,细胞自噬处于较低水平,用来维持蛋白质和细胞器的周转; 在各种生理和病理的应激条件下,如生长因子消耗、饥饿、缺氧以及化学诱导的刺激,细胞自噬水平会提高[4]。Kroemer等[5]研究发现,自噬能够通过消除潜在的毒性物质和增加细胞的适应能力来减少细胞损伤。真核细胞中至少存在3种不同类型的自噬:微自噬(Microautophagy)[6]、分子伴侣介导的自噬(Chaperonemediated autophagy, CMA)[7]和宏观自噬(Macroautophagy)[8]等。在宏观自噬过程中细胞形成双膜泡,即自噬体(Autophgosome)。自噬体吞噬细胞器、蛋白质或部分细胞质,并将其输送到溶酶体,被吞噬的成分在溶酶体中被降解。细胞通过分解代谢和循环来消除损坏或有害的成分,维持营养和能量稳态。宏观自噬是真核细胞转化、利用和降解受损蛋白质和细胞器的主要机制,有助于细胞在多种应激条件中生存,保护生物体抵御退行性、炎症性、感染性和肿瘤性疾病[9-10],后文中自噬皆指宏观自噬。
除自噬外,细胞对压力的反应还包括许多其他途径,如调节营养吸收、控制中间代谢、细胞周期和生长控制,以及细胞凋亡等程序。调控这些细胞过程的信号和调控自噬的信号之间存在紧密的整合。细胞自噬和凋亡是2种程序性细胞死亡的形式,自噬是有选择地降解受损的细胞器和蛋白质聚合体,而细胞凋亡则是清除受损或老化的细胞。已发现许多经典的细胞凋亡信号通路与自噬的调控之间存在复杂错综的关联。自噬对细胞凋亡的调控具有双重性:轻度的自噬在一定程度上保护细胞免受有害条件的影响,促进细胞的存活; 重度或快速的自噬诱导细胞程序性死亡,称为自噬性细胞凋亡。本文就近年来细胞自噬与应激反应的关系、不同细胞应激信号和应激刺激如何调控自噬的研究进展进行综述,为进一步了解调控自噬的多种机制提供参考。
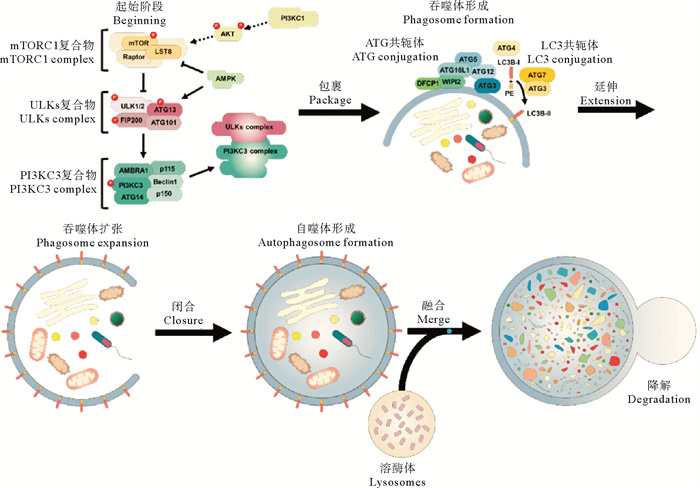
1 自噬 1.1 自噬的分子机制和调控通路自噬是包括起始、延伸、融合和溶酶体降解的动态过程。自噬途径开始于双膜囊泡的形成,即自噬体。自噬体由来自内质网膜、高尔基体晚期内膜联合形成; 在延伸步骤中,自噬体的双膜被延伸并包围细胞质成分形成一个自噬体; 最后,自噬体和溶酶体融合,溶酶体酶降解被包围的细胞质成分。该过程中的降解产物返回细胞质,被重新用于构建新的分子或在新陈代谢中发挥作用[11]。
自噬的几个核心蛋白中最著名的是微管相关蛋白1轻链-3蛋白(LC3)、p62和Beclin-1[12]。LC3是自噬体与溶酶体融合的关键分子。在自噬过程中,LC3被蛋白酶ATG4裂解产生LC3-Ⅰ,与磷脂酰乙醇胺结合形成LC3-Ⅱ,并锚定在自噬小体膜上。LC3-Ⅱ的功能是作为一个衔接蛋白,与p62蛋白相互作用,允许自噬小体吞噬底物。p62蛋白,也被称为SQSTM1(Sequestosome 1),是自噬过程的另一个常见标记物。它作为一种受体蛋白与泛素化的蛋白结合,通过膜结合的LC3-Ⅱ蛋白将其送至自噬小体进行降解[13]。p62蛋白本身能被自噬降解,因此p62蛋白在细胞质中的积累被用作自噬通量减少的标志。Beclin-1是自噬的常见标志蛋白之一,是PI3K复合物的组成部分。因Beclin-1与自噬和凋亡的相互调控相关[14],所以该蛋白被用作自噬诱导的标志物。
自噬途径的各个步骤由各种自噬相关基因编码的蛋白调节,自噬相关蛋白包括:(1)Atg1/unc-51样激酶(ULK)复合物; (2)Beclin-1/Ⅲ类磷脂酰肌醇3-激酶(PI3KⅢ)复合物; (3)2种跨膜蛋白Atg9和液泡膜蛋白1;(4)2个泛素样蛋白(Atg12和Atg8/LC3)结合系统; (5)介导自噬体和溶酶体融合的蛋白质[15],表 1为参与自噬通路不同阶段的主要蛋白。自噬通路的这些核心成分直接受到细胞应激信号的控制。
|
|
表 1 参与自噬通路不同阶段的主要蛋白 Table 1 Main proteins involved in the distinct stages of autophagy pathway |
Atg1/ULK1复合物(Atg1, Atg13等)是自噬体形成过程中重要的正向调控因子。雷帕霉素(mTOR)复合物1(mTOCR1)是一种包含mTOR、Raptor、mLST8/GβL, Deptor和PRAS40的多蛋白复合物[31]。在正常状况下mTOCR1作为激酶与Atg1/ULK1复合物结合,饥饿等应激条件下Atg1/ULK1复合物与mTOCR1分离,导致ULK1(或ULK2)和Atg13特定残基去磷酸化,催化激活ULK1(或ULK2)蛋白,同时促进Atg13和FIP200中其他残基的磷酸化。因此,mTORC1的抑制可能与ULK1(或ULK2)活性的激活关联,这一过程涉及到一个大型蛋白复合物的解离、磷酸化和去磷酸化事件。
1.1.2 Beclin-1/Ⅲ类磷脂酰肌醇3-激酶(PI3K)复合物“Beclin-1/Ⅲ类磷脂酰肌醇3-激酶(PI3K)复合物”包括Beclin-1、Vps15、Vps34以及Ambra1[19]。这类复合物可以通过激活PI3K Ⅲ蛋白,产生磷脂酰肌醇-3-磷酸(PI3P),从而招募效应因子,如含双FYVE域的蛋白1(DFCP1)和与磷脂酰肌醇相互作用的WD重复蛋白(WIPI),这类复合物的形成对于介导囊泡成核/自噬体形成的初始阶段至关重要[32]。许多与Beclin-1相互作用的蛋白诱导或抑制自噬,如:Atg14是代表Beclin-1相关的自噬关键调节器,对PI3K活性和自噬诱导至关重要; UVRAG是抗紫外线辐射相关蛋白,其与Atg14竞争与Beclin-1的结合,促进PI3K的活性,并通过与C类Vps/HOPS复合物的相互作用,促进自噬体与晚期内体/溶质体的融合[29]; Toll样受体(TLR)适配器分子MyD88和TRIF也可能与Beclin-1相互作用,通过减少Beclin-1与Bcl-2的结合,促进自噬[33]; 在TLR4诱导的自噬过程中,肿瘤坏死因子受体(TNFR)相关因子6,TRAF6与Beclin-1相互作用,并介导Beclin-1的K63连接泛素化,从而增强PI3KⅢ活性,促进自噬[34]。
1.1.3 Atg9和液泡膜蛋白1(VMP1)Atg9可以通过在不同的亚细胞间隔间循环,为隔离膜提供脂质。Atg9的循环不但需要Atg1/ULK1和Vps34的激酶活性,还涉及到含有UVRAG/Bif-1的Beclin-1复合物,因为Bif-1在饥饿后会暂时与Atg9结合[21]。VMP1作为一种跨膜蛋白与Beclin-1相互作用,将Beclin-1(以及Beclin-1复合体的其他成分)招募到自噬体处[35]。
1.1.4 2个泛素样蛋白(Atg12和Atg8/LC3)结合系统有2种泛素样(UBL)蛋白结合系统参与自噬体延伸过程:第一个途径是在E1泛素激活酶Atg7和E2连接酶Atg10的帮助下,Atg12和Atg5的共价键结合形成偶联物,该偶联物以非共价方式与Atg16结合形成多聚体Atg12-Atg5-Atg16复合物,这种复合物可以识别LC3蛋白并将其泛素化[22]; 第二种途径是首先蛋白Atg4激活Atg7(E1泛素激活酶),进而使E2连接酶Atg3发挥作用,将磷脂酰乙醇胺(PE)结合到Atg8/LC3的甘氨酸(Gly)残基上。
以上自噬相关蛋白在溶酶体的发生、自噬体的形成、自噬体-溶酶体的融合以及溶酶体中物质降解过程中发挥至关重要的作用。此外,细胞质中存在刺激和调节自噬的信号蛋白,如5′AMP激活蛋白激酶(AMPK)和丝氨酸/苏氨酸激酶(mTOR)等。除了AMPK和mTORC1外,一些应激信号在细胞水平上也可调节自噬(如TFEB、TP53/p53、FOXO1、E2F1、STAT3、NF-κB)。microRNAs(miRNAs)和组蛋白修饰,后者与自噬的调控有关[36]。其中,p53蛋白在各种应激反应中被激活,Vousden等[37]研究表明p53蛋白在遗传毒性应激反应中起到维持基因组稳定性的作用。许多p53调控的基因产物可以通过AMPK,mTORC1途径刺激自噬,或直接编码核心自噬和溶酶体蛋白(如ULK1、ULK2和DRAM)[38]。p53在非应激条件下也能抑制自噬。细胞核中的p53可以介导刺激自噬的发生,而细胞质中的p53则会抑制细胞自噬的发生。除此以外,Harrison等[23]发现死亡相关蛋白激酶(DAPK),通过与LC3相互作用的细胞骨架分子MAP1B结合,正向调节自噬。调控的自噬机制详见图 1。
1.2 鉴定细胞自噬的方法目前检测海洋无脊椎动物自噬的方法主要分为以下几种:
(1) 透射电镜技术(Transmission electron microscopy),自20世纪50年代Bhutia[40]首次使用透射电镜观察到细胞自噬到现在,透射电镜作为唯一一种可以在纳米范围内揭示自噬结构和形态的工具,已经成为了观察细胞自噬的“标准”。例如Picot等[8]在使用NH4Cl和卡马西平(Carbamazepine)处理太平洋牡蛎血淋巴细胞后使用透射电镜观察到了细胞中自噬体的存在。Luo等[41]将七鳃鳗(Lampetra japonicum)进行饥饿处理后,使用透射电镜观察到了细胞中自噬小体膜的形成、延伸过程,细胞中自噬体和溶酶体的融合以及自噬体内容物溶解过程。
(2) LC3B蛋白的检测和定量:在细胞自噬过程中,蛋白酶将胞浆中的LC3B(microtubule-associated protein light chain 3B)前体蛋白切割,暴露出羧基末端的甘氨酸残基,形成可溶性的LC3B,即LC3B-Ⅰ; 随后,LC3B-Ⅰ蛋白被E1泛素激活酶Atg7活化后传递给E2泛素结合酶Atg3,在Atg3的作用下通过泛素化修饰与磷脂酰乙醇胺(PE)结合形成自噬体膜,即脂溶性的LC3B,即LC3B-Ⅱ,LC3B蛋白在自噬体膜形成过程中发挥重要作用。由于LC3B-Ⅱ的含量与自噬程度呈正比,因此,LC3-Ⅰ和LC3-Ⅱ比例的变化已经被用来作为检测细胞自噬程度的分子标志[24]。LC3B蛋白具有高度的保守性,随着组学技术的快速发展,在越来越多的海洋无脊椎动物中发现了LC3B蛋白。余振兴等[42]采用纯化后的MgLC3B-His融合蛋白对新西兰兔进行多次免疫,采集分离兔抗血清并利用protein A纯化,成功制备出紫贻贝的LC3B多克隆抗体。Han等[43]使用干扰素诱导剂[poly (I∶C)]刺激太平洋牡蛎后,牡蛎外套膜细胞中LC3-Ⅰ向LC3-Ⅱ的转化(LC3-Ⅱ/LC3-Ⅰ)显著增加。Yue等[44]使用RNA干扰降低仿刺海参(Apostichopus japonicus)Ajlst8蛋白(一种新型的免疫调节剂)的表达后,其细胞中LC3-Ⅱ/LC3-Ⅰ的比例为对照组的0.71倍。目前海洋无脊椎生物LC3蛋白抗体的制备还存在许多问题,例如许多生物中并未有相应的LC3蛋白抗体,这阻碍了海洋无脊椎动物自噬研究的发展,因此希望未来有更多的研究人员能够参与到LC3蛋白抗体制备的工作中。
除了使用Western Blot来检测LC3-Ⅱ/LC3-Ⅰ比例变化外,还可以利用mRFP-GFP-LC3双荧光自噬指示体系,GFP是一种对酸敏感的蛋白,而mRFP是一种不受外界影响表达稳定的荧光表达基团。当自噬小体进入第二阶段后,与溶酶体融合形成自噬溶酶体。自噬溶酶体由于溶酶体内的酸性环境,可导致pH值下降,使GFP发生淬灭。因此,GFP荧光减弱可以指示自噬溶酶体的数量,GFP越少,从自噬小体进入自噬溶酶体阶段越顺利。反之,自噬泡与溶酶体融合受到抑制,自噬溶酶体形成阶段受阻,但mRFP是一直稳定表达的,因此mRFP红色荧光可以全程标记和跟踪LC3,而GFP绿色荧光的减弱可以说明自噬体和溶酶体的融合增多。当自噬被诱导时,黄色亮点(同时发出GFP绿色荧光和mRFP红色荧光,主要是自噬体)和红色亮点(自噬溶酶体)都会增加; 当自噬被抑制导致自噬体产生减少时,黄色和红色亮点都会减少; 当自噬被抑制导致自噬体降解受损时,黄色亮点增加,红色亮点减少或保持不变[45]。Martino等[46]使用这一方法观察到海胆(Paracentrotus lividus)胚胎细胞自噬的变化。目前使用mRFP-GFP-LC3双荧光自噬指示体系主要用于观察癌细胞系中的自噬行为,在观察海洋无脊椎动物自噬的研究还较少,相信随着技术的发展,该技术可以有更广的应用范围。
(3) SQSTM1的定量检测:SQSTMI1(Sequestosome1,也称p62)是参与细胞自噬的重要受体分子,SQSTMI1识别泛素化修饰的错误折叠蛋白、功能异常细胞器或入侵的病原体[47]。SQSTMI1通过与自噬体膜上的Atg8(Autophagy-related protein 8)、LC3家族蛋白相互作用,被整合到自噬体中,在溶酶体中被降解,因而其蛋白表达量的降低可以作为细胞自噬完成的指标。Balbi等[48]观测到感染弧菌(Vibrio tapetis)后的贻贝(Mytilus galloprovincialis)血淋巴中SQSTMI1蛋白表达水平下降,同时使用TEM观察到自噬体和溶酶体的快速形成,证明了贻贝血淋巴细胞在应对细菌感染时的自噬变化。
(4) 自噬相关基因的转录和调控:在某些情况下,自噬的发生伴随着某些自噬相关基因mRNA水平的增加或降低。Wu等[49]使用雷帕霉素(Rapamycin)诱导南美白对虾(Litopenaeus vannamei)细胞发生自噬后,转录组结果显示在3 050个差异基因中,有34个差异基因与PI3K-Akt信号通路有关,50个差异基因与自噬体形成相关,为更全面完整的检测南美白对虾细胞自噬行为提供了数据支持。
目前还没有能够适用于任何生物或实验背景的绝对标准来确定自噬状态,所以在观察自噬时应采取多层次、多角度的检测方法,能够在细胞、蛋白和分子水平利用不同的技术和方法检测自噬。
2 自噬与应激反应自噬是由多种刺激诱导的,包括营养和能量应激、内质网应激、病原体相关分子模式(PAMPs)、损伤相关分子模式(DAMPs)、缺氧、氧化还原应激和线粒体损伤等。这些刺激对自噬的刺激涉及多种信号通路,这些信号在调控自噬和其他应激反应中发挥重叠的功能。
2.1 自噬与氧化应激 2.1.1 自噬与ROS活性氧(Reactive oxygen species, ROS)是一种体积小、寿命短、活性高的分子,通常是线粒体电子传递链中氧代谢的副产物(mETC)[50]。在氧化磷酸化的过程中,mETC的复合物Ⅰ和Ⅲ的电子泄漏会导致O2分子的部分还原(如负氧离子)和高活性代谢物的形成[51]。在饥饿状态下,能量胁迫会增加线粒体产生ATP的需求,导致电子泄漏的增加,从而产生相对过剩的ROS。ROS的积累会导致细胞成分如蛋白质、DNA和脂质的氧化损伤,从而激发自噬[52]。Filomeni等[53]研究发现ROS与剥夺营养物质(如葡萄糖、氨基酸或血清)时的自噬诱导有关。自噬在氧化应激条件下被激活,保护细胞避免凋亡。此外,已有研究表明抗氧化分子在一定程度可以抑制自噬的发生。
线粒体中的过氧化氢在细胞信号传递中发挥重要作用,它比其他ROS分子稳定,很容易扩散到细胞中[54]。已有大量研究表明过氧化氢会参与到自噬信号通路中,主流观点认为过氧化氢可以通过硫醇修饰Atg4第81位半胱氨酸使LC3-Ⅰ转化为LC3-Ⅱ,从而增加自噬体的数量[55]。外源性过氧化氢也可导致氧化应激和线粒体功能障碍,从而诱导自噬发生。利用过氧化氢与自噬之间的关联,Zhang等[56]使用过氧化氢治疗可刺激恶性胶质瘤细胞的自噬和凋亡。Amelotti使用TNFα处理肿瘤细胞增加了细胞中ROS的水平,从而诱导肿瘤细胞的自噬和死亡。同时,脂多糖(LPS)也可诱导过氧化氢的产生,从而导致自噬[57]。
ROS可以通过激活丝裂原活化蛋白激酶(MAPKs)诱导自噬,过氧化氢通过氧化半胱氨酸残基的α和β亚单位或氧化ATM蛋白激酶直接激活AMPK[58]。氧化应激激活的ATM诱导其下游信号AMPK -TSC2抑制mTORC1,从而诱导自噬发生[59]。Alexander等[60]发现在过氧化氢刺激下通过肝激酶B1 (LKB1)磷酸化AMPK第172位苏氨酸(T172),抑制mTORC1,从而诱导自噬。
在病理学方面,多项研究也揭示了ROS和自噬在多种疾病进展过程中的相互作用。过量的ROS产生导致氧化应激,这与神经退行性疾病、心血管病等慢性病及癌症有关。大多数细胞自噬起到减少氧化应激的作用。诱导自噬可以作为一种治疗与氧化应激相关疾病的策略。
2.1.2 自噬与NONO是在氧化应激过程中由NO合成酶(NOS)产生。在自噬信号中,NO通过降低c-Jun-N末端激酶1 (JNK1)和核因子κB激酶亚基β (IKKβ)抑制剂的活性来抑制自噬体的形成[61]。B淋巴瘤2 (Bcl-2)可以与Beclin-1相互结合抑制自噬的发生,而JNK1可以通过磷酸化B淋巴瘤2 (Bcl-2)来破坏其与Beclin-1的相互作用,从而诱导自噬[62]。IKKβ可以通过增强AMPK磷酸化抑制mTOR和JNK1介导的Bcl-2磷酸化诱导自噬[63]。Janjetovic等[64]通过NO供体物质(硝普钠SNP和S-亚硝基谷胱甘肽GSNO)来增加胶质瘤细胞内NO含量,从而使细胞的自噬过程受到抑制作用。
2.2 自噬与免疫信号感染(或细胞暴露于微生物产物)会产生一种特殊形式的细胞应激,从而导致自噬。在感染过程中细胞因子如IFNg和下游免疫相关的GTP酶以及病原体识别受体(PRRs)调节自噬的发生[65]。PRRs包括TLRs家族、NOD样受体(NLRs)及RIG-Ⅰ样受体(RLRs)。TLRs家族通过NF-κB、MAPK和干扰素调节途径激活促炎细胞因子和Ⅰ型干扰素的产生; NOD样受体(NLR)主要通过NF-κB和MAPK以及炎症体的成分激活信号传导; RIG-Ⅰ样受体(RLRs)负责识别病毒RNA[66]。由于这些PRRs不仅能识别病原体相关分子模式(PAMPs)还能识别损伤相关分子模式(DAMPs,包括坏死细胞、缺氧、细胞内离子梯度异常、活性氧物种和错误折叠的蛋白质积累的产物),因此在细胞应对不同形式的压力时,可以利用这个免疫信号家族引起自噬。
转录因子NF-κB及其一些上游调节因子的功能是将包括免疫信号在内的各种应激信号与自噬途径进行整合。当NF-κB的抑制剂(IκB)被IκB激酶(IKK)磷酸化时,NF-κB被激活。IKK复合体由1个调节亚基(NEMO)和2个催化亚基(IKKα、IKKβ)组成。该复合物在活性氧、DNA损伤、死亡受体和PRRs等多个不同的压力刺激时被另一个上游激酶TAK1激活。IKK亚单位通过依赖AMPK和JNK1激活的NF-κB独立通路刺激自噬[67]。Criollo等[63]在小鼠和人类细胞中,敲除或敲降TAK1或IKK亚单位可阻止饥饿、雷帕霉素、p53抑制或内质网应激等刺激反应对自噬的诱导。同时Copetti等[68]研究表明NF-κB家族成员p65/RelA可以在T细胞激活过程中通过上调Beclin-1的表达诱导自噬,而缺少NF-κB的细胞在热休克诱导的自噬方面存在缺陷。因此,自噬诱导可能同时存在IKK依赖性和NF-κB依赖性机制。此外,NF-κB的p105亚基可以与某些自噬蛋白(包括Beclin-1)相互作用,但这些相互作用的意义还有待探讨[69]。
除了NF-κB对自噬的调节外,自噬反过来可以调节NF-κB的信号传导[70]。例如,通过自噬降解IKK和NF-κB诱导激酶NIK,会抑制NF-κB的表达。当自噬被抑制时,会导致p62的积累并改变NF-κB信号通路[71]。
早期研究认为双链RNA依赖蛋白质激酶PKR的主要功能是应对病毒感染。后来,Bonnet等[72]发现PKR可以激活IKKβ-NF-κB通路。Nakamura等[73]研究发现PKR参与到内质网应激反应和脂肪酸暴露反应,并通过磷酸化eIF2a、激活“炎症激酶”IKKβ和JNK1或磷酸化IRS1诱导自噬,这项研究解释了JNK1、IKK和eIF2a通路在诱导自噬过程中功能重叠且相互作用。
然而,目前炎症通路促进自噬活性的机制,还不能解释炎症因子带来的炎症反应和不良代谢效应,因为一些研究中发现自噬基因的遗传缺失反而会激活炎症信号,例如Yang等[74]发现抑制肝脏细胞的Atg7基因的会导致ER应激和胰岛素抵抗的增加。因此炎症分子激活自噬的机制及其作用需要进一步的研究,以解释炎性因子在自噬激活中的作用与自噬的细胞保护作用之间的关联。图 2为细胞内膜损伤后自噬与促炎症信号的传递过程[75]。
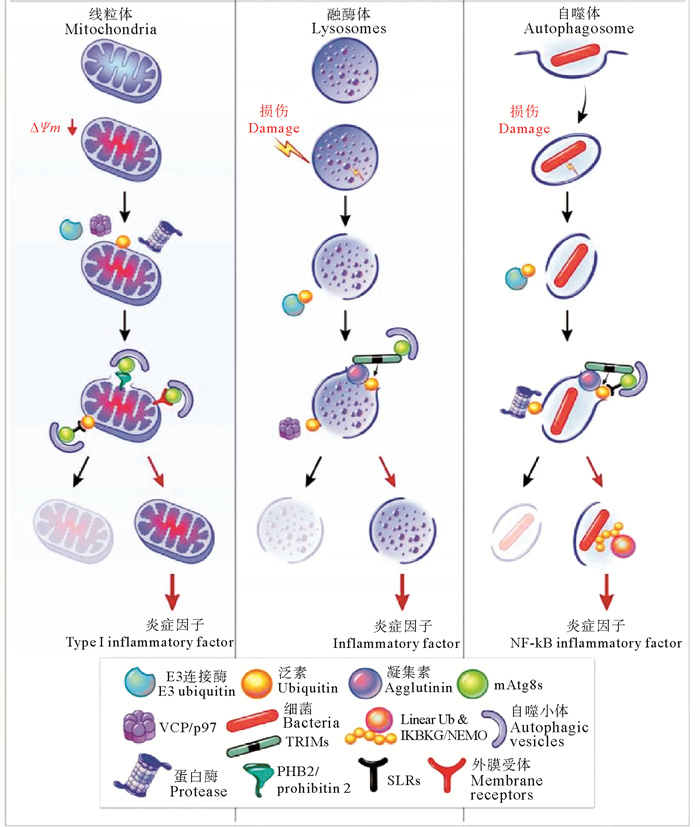
|
( 参考Deretic等[75]。Reference from Deretic, et al[75]. ) 图 2 细胞内膜损伤后自噬与促炎症信号的传递过程 Fig. 2 Common themes in autophagic homeostasis of endomembrane damage and proinflammatory signaling |
低氧和缺氧(氧浓度分别 < 3%和 < 0.1%)都可以通过各种不同的机制引起自噬。低氧诱导的自噬依赖于低氧诱导因子HIF,而缺氧诱导的自噬不依赖于HIF[76]。HIF是一个由β亚基和一个由氧调节的α亚基组成的二聚体,只有当氧浓度下降到5%以下时,它才会被激活表达。当氧浓度在1%~3%时,HIF会激活BNIP3和BNIP3L(NIX)的转录,这2种蛋白可以破坏Beclin-1和Beclin-2之间的相互抑制作用[77]。同时BNIP3L常存在于线粒体的外表面,因其拥有一个WXXL基团,故其可与LC3及其同源物GABARAP结合,从而诱导线粒体自噬。低氧诱导的自噬则涉及其他途径,在低氧条件下细胞中转录因子ATF4和CHOP通过增加自噬必需基因LC3和Atg5的转录诱导自噬[26]。
2.4 自噬与饥饿饥饿是目前已知最有效的自噬生理诱导因子。在大多数培养的哺乳动物细胞中,饥饿会在几分钟内引起细胞自噬,当细胞同时缺乏营养(如氨基酸和葡萄糖)和生长因子(如血清中所含的生长因子)时,细胞自噬达到最高水平。mTOR和AMPK在饥饿诱导的自噬中发挥关键的调节作用,最近的研究表明Sirtuins家族也负责调控饥饿的自噬[78]。
Sirtuins是一个能够感知环境压力并依赖于NAD的脱乙酰酶家族,Sirt1可以去乙酰化Atg5、Atg7和LC3诱导自噬发生。Lee等[79]在敲除了Sirt1的小鼠中发现了与自噬受损一致的表型,包括自噬底物p62水平的增加、受损细胞器的积累、能量稳态的破坏和围产期早期的死亡。Sirt1还可以使转录因子forkhead box O3a(FOXO3a)脱乙酰化。FOXO3a能激活自噬因子Bnip3,是自噬调控的另一个重要分子。FOXO3去磷酸化后能进入细胞核,上调多个自噬相关基因的表达,如ULK2,Beclin-1, VPS34, BNIP3和BNIP3L, ATG12, ATG4B, LC3, GABARAPL1等[25]。Sirt2在饥饿时与FOXO1解离,导致FOXO1的乙酰化,有利于FOXO1与细胞质中的Atg7相互作用,从而刺激自噬的发生[80]。
AMPK能够增加自噬水平,而mTORC1会抑制自噬水平的增加。AMPK通过感知饥饿刺激诱导自噬的方式有2种:①通过磷酸化ULK复合物间接诱导自噬; ②通过抑制mTOR复合物1(mTORC1)直接诱导自噬。AMPK还可以磷酸化Atg13的Ser224位点,从而抑制自噬[17]。Lan等[81]发现AMPK与Sirt1之间存在相互关联。Sirt1介导的LKB1去乙酰化,促进其从细胞核转运到细胞质,随后激活AMPK。
除AMPK外,其他一些激酶也在饥饿诱导的自噬上调中发挥作用。如JNK1,它既能通过磷酸化Bcl2降低其对Beclin-1的BH3域的亲和力,也可以使Sirt1磷酸化[82]。此外,eIF2a的磷酸化和IKK的激活也是饥饿诱导自噬的必要条件[83]。其他激酶如p38α丝裂原活化蛋白激酶(MAPK14)能通过作用于饥饿诱导的mAtg9转运和自噬小体形成所必需的p38IP抑制自噬[84]。因此,饥饿状态下需要几种激酶的协同作用才能激活自噬,然而,目前对这种协调的确切机制及其关联仍然知之甚少。
2.5 自噬与凋亡虽然自噬通常有助于细胞在不利条件下的存活,但已有证据表明,高强度和持续的自噬也与细胞死亡有关[85]。自噬依赖性细胞死亡的经典观点,即Ⅱ型程序性细胞死亡(PCD),涉及细胞死亡之前的细胞质空泡化,自噬体结构的大量积累[86]。自噬和凋亡之间存在显著的交叉作用,细胞凋亡通常被称为Ⅰ型PCD,通常涉及启动和执行凋亡的半胱氨酸蛋白酶家族的Caspases。因为一些ATG蛋白具有Caspase识别域,所以Caspases可以切割自噬相关分子进而调节PCD过程中的自噬活性。在大多数情况下,Caspase介导的ATG蛋白的裂解会导致自噬的下调或抑制,从而增加细胞死亡,如之前报道的Caspase介导的Beclin-1、ATG16L或AMBRA1的裂解[87]。自噬也能通过Caspases来抑制细胞凋亡信号,使其在特定环境下降解[88]。
然而某些Caspase裂解的ATG产物能够增强自噬活性。例如,Caspase-3裂解半胱氨酸蛋白酶ATG4D后的产物具有更高的活性,起到促进自噬的作用。同样,Caspase9在与自噬核心蛋白ATG7相互作用时催化活性降低,与单独的ATG7相比,ATG7/Caspase9复合物对LC3B表现出更高的亲和力和更强的自噬诱导作用[89]。
Calpains是钙离子依赖性的半胱氨酸蛋白酶,能够将细胞内钙离子含量与自噬和凋亡信号传递联系起来。类似于Caspase介导的ATGs的切割,Calpains可以切割ATG5,并作为自噬和凋亡之间的额外开关[90]。Calpains裂解的ATG5产物可以从胞体迁移到线粒体,有助于细胞色素c的释放,从而增加细胞凋亡。除ATG5外,Calpain也被证明可以切割其他不同的ATG蛋白,从而增加自噬和细胞凋亡之间的联系[91]。
上文介绍的p53蛋白在自噬与凋亡中也发挥作用。在正常条件下p53位于细胞质中,对FIP200发挥抑制作用,从而限制自噬的启动[92]。轻微的细胞应激会使p53向细胞核移动,增加促自噬因子AMPK、损伤调节自噬调节剂1 (DRAM1)和Sestrins1和Sestrins2的表达,发挥诱导细胞自噬的作用。然而,持续的高应激水平导致p53线粒体膜定位触发凋亡[93]。
2.6 自噬与环境胁迫许多被认为对生物有毒的环境污染物能够通过直接或间接影响自噬和溶酶体系统发挥毒性作用。一些污染物会抑制或阻断自噬的最后阶段,降低溶酶体降解的效率,从而导致有害物质的积累[94]。同时,暴露于污染物/外来生物也可能影响自噬的初始阶段或自噬相关信号通路,阻碍自噬过程,进而影响细胞对环境的适应。自噬作为一种细胞自我保护机制,当细胞暴露于污染物或外来生物时细胞自噬水平增加,而当损伤严重且持续时,会导致自噬的持续诱导,这在某些情况下可能会导致细胞死亡。为了更好地理解外源性物质、自噬和细胞凋亡之间复杂的关系,本文作者总结了一些重要的工业污染物对自噬通路的作用(见表 2)。
|
|
表 2 重要工业污染物对自噬通路的作用 Table 2 Effects of major industrial pollutants on autophagy pathways |
除了环境污染物外,感染细菌或病毒也会诱导自噬。目前多数研究认为细胞自噬是动物应对病菌感染的一种保护机制:比如Xu等[109]发现感染对虾白斑综合症病毒(WSSV)36 h后的日本沼虾(Marsupenaeus japonicas)体内中LC3-Ⅱ和LC3-Ⅰ比例相对于未感染组显著提高,表明病毒感染会诱导日本沼虾体内细胞发生自噬。Pierrick等[110]发现牡蛎疱疹病毒1型(Ostreid herpesirus-1, OsHV-1)和弧菌(Vibrio aestuarianus)能诱导太平洋牡蛎细胞自噬,且使用自噬抑制剂处理后,牡蛎感染OsHV-1病毒后的死亡率远高于未加自噬抑制剂处理组,证明自噬在保护太平洋牡蛎免受病毒细菌的感染中起着重要作用。除此之外,Balbi等[48]也通过TEM观测、Western Blot实验等方法证明了贻贝血淋巴细胞在应对弧菌感染时的自噬变化。
目前病菌感染后对细胞自噬影响的研究多集中于观测生物感染病菌后体内自噬的变化,未来可通过药物等方法促进机体细胞自噬来应对病菌感染,或使用促进自噬的药物降低动物感染病菌后的死亡率。
在过去的几十年里,针对暴露于环境污染物对健康影响的研究表明,细胞毒性通常会影响线粒体的稳定性以及ROS的产生和积累,而这些刺激都可以调节自噬活性。因此,自噬相关基因和蛋白可作为污染物危害初期快速可靠的生物标志物。
对暴露于环境污染物后的自噬调节、自噬依赖的细胞死亡和凋亡机制的研究,有利于进一步理解细胞和生物体对污染物的反应,为开发自噬相关基因和蛋白作为生物标志物提供实验支撑。此外,依据相关研究设计新的自噬相关治疗方法,以解决污染物对人体健康的有害影响并开发相关的生物传感器,有助于检测环境污染物的毒性效应。
2.7 自噬与细胞其他应激反应自噬和细胞其他应激反应的整合可以分3个方面。第一,单一类型的压力刺激会触发细胞内多种应激反应的信号通路,引发不同的细胞反应,其中之一是自噬。过氧化导致的应激反应,是通过低氧诱导因子(HIF)、NF-κB和p53的激活诱导转录编程,从而引起未折叠蛋白反应(UPR),并刺激一般和选择性的自噬[71]。缺氧引发的应激反应在刺激自噬的同时也激活了血管生成和细胞保护相关的细胞因子[77]。第二,不同类型的应激刺激通过单一的分子事件刺激多个应激反应发生。TAK1和IKK的激活通过NF-κB信号通路调节自噬[67]。第三,不同的细胞应激反应途径能够相互调控,因此自噬与其他应激反应途径能够相互调控。例如自噬缺陷导致p62积累,从而改变NF-κB信号。p62在自噬缺失的情况下积累可激活应激应答转录因子Nrf2。环境污染物诱导自噬的应激途径由图 3所示。
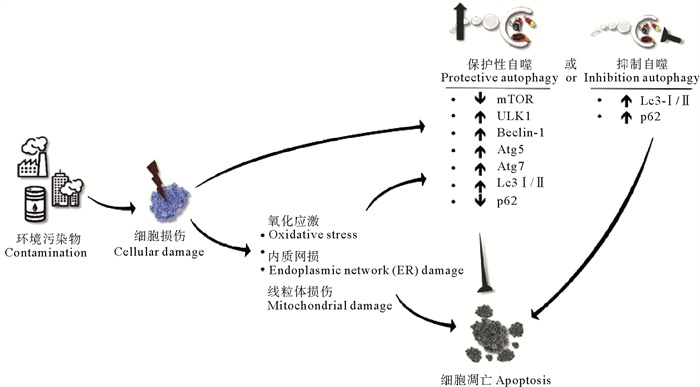
|
图 3 环境污染物诱导自噬的应激途径 Fig. 3 Effects and signaling modulation on autophagy by environmental pollutants |
该综述描述了常见的自噬调控分子机制及目前鉴定自噬的主要方法,重点关注了自噬和不同类型的应激反应之间的密切联系,这些联系由多种复杂的机制组成,这些机制在复杂的信号通路中交织在一起,构成分子开关和稳态装置,对这些复杂机制间的分子联系进一步研究,以及利用系统生物学方法对其进行整合,可能为更精细的自噬实验和治疗操作提供可测试的模型。目前,对自噬调控的研究仍处在初始阶段,为了充分了解细胞和生物体如何应对分子损伤及环境胁迫带来的压力,需要更深入研究自噬调节,自噬依赖性细胞死亡和细胞凋亡的调控机制。希望将来有更多应激反应与自噬调控通路的研究出现。
自噬与免疫反应和化学应激诱发的其他细胞功能之间仍有许多联系,尚待进一步阐明。需要更多的基础研究来确定暴露于环境化学物质后自噬调控途径的分子机制。这些研究将有助于进一步了解自噬在化学毒性和组织发病机制中的作用。
| [1] |
Kroemer G, Mario G, Levine B. Autophagy and the integrated stress response[J]. Molecular Cell, 2010, 40(2): 280-293. DOI:10.1016/j.molcel.2010.09.023 (  0) 0) |
| [2] |
Dikic I, Elazar Z. Mechanism and medical implications of mammalian autophagy[J]. Nature Reviews Molecular Cell Biology, 2018, 19(6): 349-364. DOI:10.1038/s41580-018-0003-4 (  0) 0) |
| [3] |
He C, Klionsky D J. Regulation mechanisms and signaling pathways of autophagy[J]. Annual Review of Genetics, 2009, 43(1): 67-93. DOI:10.1146/annurev-genet-102808-114910 (  0) 0) |
| [4] |
Singh R, Cuervo A. Autophagy in the cellular energetic balance[J]. Cell Metabolism, 2011, 13(5): 495-504. DOI:10.1016/j.cmet.2011.04.004 (  0) 0) |
| [5] |
Kroemer G, Reed J C. Mitochondrial control of cell death[J]. Nature Medicine, 2001, 1(5): 513-519. (  0) 0) |
| [6] |
Chora S, Starita-Geribaldi M, Guigonis J M, et al. Effect of cadmium in the clam Ruditapes decussatus assessed by proteomic analysis[J]. Aquatic Toxicology, 2009, 94(4): 300-308. DOI:10.1016/j.aquatox.2009.07.014 (  0) 0) |
| [7] |
Herpin A, Lescat L, Bobe J, et al. Lighting chaperone-mediated autophagy (CMA) evolution with an ancient LAMP: The existence of a functional CMA activity in fish[J]. Autophagy, 2020, 16(10): 1-3. (  0) 0) |
| [8] |
Picot S, Morga B, Faury N, et al. A study of autophagy in hemocytes of the Pacific oyster, Crassostrea gigas[J]. Autophagy, 2019, 15(10): 1-9. (  0) 0) |
| [9] |
Mizushima N, Yoshimori T, Levine B. Methods in mammalian autophagy research[J]. Cell, 2010, 140(3): 313-326. DOI:10.1016/j.cell.2010.01.028 (  0) 0) |
| [10] |
Chakraborty D, Felzen V, Hiebel C, et al. Enhanced autophagic-lysosomal activity and increased BAG3-mediated selective macroautophagy as adaptive response of neuronal cells to chronic oxidative stress[J]. Redox Biology, 2019, 24: 101181. DOI:10.1016/j.redox.2019.101181 (  0) 0) |
| [11] |
Hamasaki M, Furuta N, Matsuda A, et al. Autophagosomes form at ER-mitochondria contact sites[J]. Nature, 2013, 495(7441): 389-393. DOI:10.1038/nature11910 (  0) 0) |
| [12] |
Komatsu M. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice[J]. The Journal of Cell Biology, 2005, 169(3): 425-434. DOI:10.1083/jcb.200412022 (  0) 0) |
| [13] |
Kang R, Zeh H J, Lotze M T, et al. The Beclin1 network regulates autophagy and apoptosis[J]. Cell Death & Differentiation, 2011, 18: 571-580. (  0) 0) |
| [14] |
Klionsky D J, Boyer-Guittaut M, Marie-Agnès B, et al. Guidelines for the use and interpretation of assays for monitoring autophagy (3rd edition)[J]. Autophagy, 2016, 12(1): 1-9. DOI:10.1080/15548627.2015.1100356 (  0) 0) |
| [15] |
Cheng Y, Ren X, Hait W N, et al. Therapeutic targeting of autophagy in disease: Biology and pharmacology[J]. Pharmacological Reviews, 2013, 65(4): 1162. DOI:10.1124/pr.112.007120 (  0) 0) |
| [16] |
Hashemi-Sadraei N, Müller-Greven G M, Abdul-Karim F W, et al. Expression of LC3B and FIP200/Atg17 in brain metastases of breast cancer[J]. Journal of Neuro-Oncology, 2018, 140(2): 237-248. DOI:10.1007/s11060-018-2959-5 (  0) 0) |
| [17] |
Ruderman N B, Xu X J, Nelson L, et al. AMPK and SIRT1: A long-standing partnership?[J]. American Journal of Physiology Endocrinology & Metabolism, 2010, 298(4): 751. (  0) 0) |
| [18] |
Funderburk S F, Wang Q J, Yue Z. The Beclin 1-VPS34 complex--at the crossroads of autophagy and beyond[J]. Trends in Cell Biology, 2010, 20: 355-362. DOI:10.1016/j.tcb.2010.03.002 (  0) 0) |
| [19] |
Axe E L, Simon A, Manifava M, et al. Autophagosome formation from membrane compartments enriched in phosphatidylinositol 3-phosphate and dynamically connected to the endoplasmic reticulum[J]. The Journal of Cell Biology, 2008, 182(4): 685-701. DOI:10.1083/jcb.200803137 (  0) 0) |
| [20] |
Tang Z, Takahashi Y, Chen C, et al. Atg2A/B deficiency switches cytoprotective autophagy to non-canonical caspase-8 activation and apoptosis[J]. Cell Death and Differentiation, 2017, 24(12): 2127-2138. DOI:10.1038/cdd.2017.133 (  0) 0) |
| [21] |
Orsi A, Polson H E, Tooze S A. Membrane trafficking events that partake in autophagy[J]. Current Opinion in Cell Biology, 2010, 22(2): 150-156. DOI:10.1016/j.ceb.2009.11.013 (  0) 0) |
| [22] |
Klionsky D J, Emr S D. Autophagy as a regulated pathway of cellular degradation[J]. Science, 2000, 290: 1717-1721. DOI:10.1126/science.290.5497.1717 (  0) 0) |
| [23] |
Harrison B, Kraus M, Burch L, et al. DAPK-1 binding to a linear peptide motif in MAP1B stimulates autophagy and membrane lebbing[J]. Journal of Biological Chemistry, 2008, 283(15): 9999-10014. DOI:10.1074/jbc.M706040200 (  0) 0) |
| [24] |
Mcleland C B, Rodriguez J, Stern S T. Autophagy monitoring assay: Qualitative analysis of MAP LC3-Ⅰ to Ⅱ conversion by immunoblot[J]. Methods in Molecular Biology, 2011, 697: 199-206. (  0) 0) |
| [25] |
Mammucari C, Milan G, Romanello V, et al. FoxO3 controls autophagy in skeletal muscle in vivo[J]. Cell Metabolism, 2007, 6(6): 458-471. DOI:10.1016/j.cmet.2007.11.001 (  0) 0) |
| [26] |
Novak I, Kirkin V, McEwan D G, et al. Nix is a selective autophagy receptor for mitochondrial clearance[J]. EMBO Reports, 2010, 11(1): 45-51. DOI:10.1038/embor.2009.256 (  0) 0) |
| [27] |
Tian X, Teng J, Chen J. New insights regarding SNARE proteins in autophagosome-lysosome fusion[J]. Autophagy, 2020, 24: 1-9. (  0) 0) |
| [28] |
Takáts S, Nagy P, Varga Á, et al. Autophagosomal syntaxin17-dependent lysosomal degradation maintains neuronal function in Drosophila[J]. The Journal of Cell Biology, 2013, 201(4): 531-539. DOI:10.1083/jcb.201211160 (  0) 0) |
| [29] |
Wesselborg S, Stork B. Autophagy signal transduction by ATG proteins: From hierarchies to networks[J]. Cellular and Molecular Life Sciences CMLS, 2015, 72(24): 4721-4757. DOI:10.1007/s00018-015-2034-8 (  0) 0) |
| [30] |
Sarafian V S, Koev I, Mehterov N, et al. LAMP-1 gene is overexpressed in high grade glioma[J]. Acta Pathologica, Microbiologica, et Immunologica Scandinavica, 2018, 126(8): 657-662. DOI:10.1111/apm.12856 (  0) 0) |
| [31] |
He C C. The Beclin-1 interactome[J]. Current Opinion in Cell Biology, 2010, 22(2): 140-149. DOI:10.1016/j.ceb.2010.01.001 (  0) 0) |
| [32] |
Polson H E J, Lartigue J D, Rigden D J, et al. Mammalian Atg18 (WIPI2) localizes to omegasome-anchored phagophores and positively regulates LC3 lipidation[J]. Autophagy, 2010, 6(4): 506-522. DOI:10.4161/auto.6.4.11863 (  0) 0) |
| [33] |
Shi C S, Kehrl J H. Myd88 and Trif Target Beclin1 to trigger autophagy in macrophages[J]. Journal of Biological Chemistry, 2008, 283(48): 33175. DOI:10.1074/jbc.M804478200 (  0) 0) |
| [34] |
Shi C S, Kehrl J H. TRAF6 and A20 regulate lysine63-linked ubiquitination of Beclin-1 to control TLR4-induced autophagy[J]. Science Signaling, 2010, 3(123): ra42. (  0) 0) |
| [35] |
Yang Z, Klionsky D J. Mammalian autophagy: Core molecular machinery and signaling regulation[J]. Current Opinion in Cell Biology, 2010, 22(2): 124-131. DOI:10.1016/j.ceb.2009.11.014 (  0) 0) |
| [36] |
Rabanal-Ruiz Y, Byron A, Wirth A, et al. mTORC1 activity is supported by spatial association with focal adhesions[J]. The Journal of Cell Biology, 2021, 220(5): e202004010. DOI:10.1083/jcb.202004010 (  0) 0) |
| [37] |
Vousden K H, Lane D P. P53 in health and disease[J]. Nature Reviews Molecular Cell Biology, 2007, 8(4): 275-283. DOI:10.1038/nrm2147 (  0) 0) |
| [38] |
Feng Z, Zhang H, Levine A J, et al. The coordinate regulation of the p53 and mTOR pathways in cells[J]. Proceedings of the National Academy of Sciences, 2005, 102(23): 8204-8209. DOI:10.1073/pnas.0502857102 (  0) 0) |
| [39] |
Yun H R, Yong H J, Kim J, et al. Roles of autophagy in oxidative stress[J]. International Journal of Molecular Sciences, 2020, 21(9): 3289. DOI:10.3390/ijms21093289 (  0) 0) |
| [40] |
Bhutia S K, Praharaj P P, Bhol C S, et al. Monitoring and measuring mammalian autophagy[M]// Turksen K. Methods in Molecular Biology. Heidelberg: Springer Nature, 2018: 209-222.
(  0) 0) |
| [41] |
Luo L, Lu J, Qin W, et al. Autophagy participates in innate immune defense in lamprey[J]. Fish & Shellfish Immunology, 2018, 83: 416-424. (  0) 0) |
| [42] |
余振兴, 朱倩, 姚翠鸾. 紫贻贝LC3B蛋白的原核表达及抗血清制备[J]. 水产学报, 2017, 41(4): 498-505. Yu Z X, Zhu Q, Yao C L. Prokaryotic expression and antiserum preparation of LC3B protein in purple mussel (Mytilus edulis)[J]. Journal of Aquaculture, 2017, 41(4): 498-505. (  0) 0) |
| [43] |
Han Z R, Wang W L, Lv X J, et al. ATG10 regulates the formation of autophagosome in the anti-virus immune response of pacific oyster (Crassostrea gigas)[J]. Fish & Shellfish Immunology, 2019, 91: 325-332. (  0) 0) |
| [44] |
Yue Z X, Lv Z M, Shao Y, et al. Cloning and characterization of the target protein subunit LST8 of rapamycin in Apostichopus japonicus[J]. Fish & Shellfish Immunology, 2019, 92: 460-468. (  0) 0) |
| [45] |
黄桢钧, 刘彬, 刘本荣, 等. 双荧光mRFP-eGFP-LC3体系在细胞自噬中的作用[J]. 贵阳医学院学报, 2015, 40(10): 1029-1032. Huang Z J, Liu B, Liu B R, et al. Role of dual fluorescent mRFP-eGFP-LC3 system in cellular autophagy[J]. Journal of Guiyang Medical College, 2015, 40(10): 1029-1032. (  0) 0) |
| [46] |
Martino C, Chiarelli R, Bosco L, et al. Induction of skeletal abnormalities and autophagy in Paracentrotus lividus sea urchin embryos exposed to gadolinium[J]. Marine Environmental Research, 2017, 130: 12-20. DOI:10.1016/j.marenvres.2017.07.007 (  0) 0) |
| [47] |
Bjorkoy G, Lamark T, Brech A, et al. P62/SQSTM1 forms protein aggregates degraded by autophagy and has a protective effect on huntingtin-induced cell death[J]. Journal of Cell Biology, 2005, 171(4): 603-614. DOI:10.1083/jcb.200507002 (  0) 0) |
| [48] |
Balbi T, Cortese K, Ciacci C. Autophagic processes in Mytilus galloprovincialis hemocytes: Effects of Vibrio tapetis[J]. Fish & Shellfish Immunology, 2018, 73: 66-74. (  0) 0) |
| [49] |
Wu W, Lin X, Wang C, et al. Transcriptome of white shrimp Litopenaeus vannamei induced with rapamycin reveals the role of autophagy in shrimp immunity[J]. Fish & Shellfish Immunology, 2019, 86: 1009-1018. (  0) 0) |
| [50] |
Brand M D. Mitochondrial generation of superoxide and hydrogen peroxide as the source of mitochondrial redox signaling[J]. Free Radic Biol Med, 2016, 100: 14-31. DOI:10.1016/j.freeradbiomed.2016.04.001 (  0) 0) |
| [51] |
Thannickal V J, Fanburg B L. Reactive oxygen species in cell signaling[J]. American Journal of Physiology Lung Cellular & Molecular Physiology, 2018, 122(6): 877-902. (  0) 0) |
| [52] |
Bhatti J S, Bhatti G K, Reddy P H. Mitochondrial dysfunction and oxidative stress in metabolic disorders—A step towards mitochondria based therapeutic strategies[J]. Biochimica et Biophysica Acta (BBA)—Molecular Basis of Disease, 2016, 1863(5): 1066. (  0) 0) |
| [53] |
Filomeni G, Desideri E, Cardaci S, et al. Under the ROS: Thiol network is the principal suspect for autophagy commitment[J]. Autophagy, 2014, 6(7): 999-1005. (  0) 0) |
| [54] |
Lee J, Giordano S, Zhang J. Autophagy, mitochondria and oxidative stress: Cross-talk and redox signalling[J]. Biochemical Journal, 2012, 441(2): 523-540. DOI:10.1042/BJ20111451 (  0) 0) |
| [55] |
Scherz-Shouval R, Shvets E, Elazar Z. Oxidation as a post-translational modification that regulates autophagy[J]. Autophagy, 2007, 3(4): 371-373. DOI:10.4161/auto.4214 (  0) 0) |
| [56] |
Zhang X, Cheng X, Yu L, et al. MCOLN1 is a ROS sensor in lysosomes that regulates autophagy[J]. Nature Communications, 2016, 7: 12109. DOI:10.1038/ncomms12109 (  0) 0) |
| [57] |
Djavaheri-Mergny M, Amelotti M, Mathieu J, et al. NF-kappaB activation represses tumor necrosis factor-alpha-induced autophagy[J]. The Journal of Biological Chemistry, 2006, 281(41): 30373-30382. DOI:10.1074/jbc.M602097200 (  0) 0) |
| [58] |
Trudel L J, Wogan G N, Tripathi D N, et al. Reactive nitrogen species regulate autophagy through ATM-AMPK-TSC2-mediated suppression of mTORC1[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(32): 2950-2957. (  0) 0) |
| [59] |
Zhi G, Kozlov S, Lavin M F, et al. ATM activation by oxidative stress[J]. Science, 2010, 330(6003): 517-521. DOI:10.1126/science.1192912 (  0) 0) |
| [60] |
Alexander A, Cai S L, Kim J, et al. ATM signals to TSC2 in the cytoplasm to regulate mTORC1 in response to ROS[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(9): 4153-4158. DOI:10.1073/pnas.0913860107 (  0) 0) |
| [61] |
Knowles R G, Moncada S. Nitric oxide synthases in mammals[J]. Biochemical Journal, 1994, 298(2): 249-258. DOI:10.1042/bj2980249 (  0) 0) |
| [62] |
Pattingre S, Tassa A, Qu X, et al. Bcl-2 antiapoptotic proteins inhibit beclin1-dependent autophagy[J]. Cell, 2005, 28(10): 677-685. (  0) 0) |
| [63] |
Criollo A, Senovilla L, Authier H. The IKK complex contributes to the induction of autophagy[J]. The EMBO Journal, 2010, 3(4): 371-373. (  0) 0) |
| [64] |
Janjetovic K, Misirkic M, Vucicevic L, et al. Synergistic antiglioma action of hyperthermia and nitric oxide[J]. European Journal of Pharmacology, 2008, 583(1): 1-10. DOI:10.1016/j.ejphar.2007.12.028 (  0) 0) |
| [65] |
Rhea S, Beth L. Autophagy and innate immunity: Triggering, targeting and tuning[J]. Seminars in Cell and Developmental Biology, 2010, 21(7): 699-711. DOI:10.1016/j.semcdb.2010.04.003 (  0) 0) |
| [66] |
Baud V, Karin M. Is NF-kappaB a good target for cancer therapy? Hopes and pitfalls[J]. Nature Reviews Drug Discovery, 2009, 8(1): 33-40. DOI:10.1038/nrd2781 (  0) 0) |
| [67] |
Herrero-Martín G, Høyer-Hansen M, García-García C, et al. TAK1 activates AMPK-dependent cytoprotective autophagy in TRAIL-treated epithelial cells[J]. The EMBO Journal, 2009, 28(10): 677-685. (  0) 0) |
| [68] |
Copetti T, Bertoli C, Dalla E, et al. p65/RelA modulates BECN1 transcription and autophagy[J]. Molecular & Cellular Biology, 2009, 29(6): 2594-608. (  0) 0) |
| [69] |
Mathew R, Karp C M, Beaudoin B, et al. Autophagy suppresses tumorigenesis through elimination of p62[J]. Cell, 2009, 137(6): 1062-1075. DOI:10.1016/j.cell.2009.03.048 (  0) 0) |
| [70] |
Hu N, Zhang Y. TLR4 knockout attenuated high fat diet-induced cardiac dysfunction via NF-κB/JNK-dependent activation of autophagy[J]. Biochimica et Biophysica Acta, 2017, 63(8): 2001-2011. (  0) 0) |
| [71] |
Nivon M, Richet E, Codogno P, et al. Autophagy activation by NFkappaB is essential for cell survival after heat shock[J]. Autophagy, 2009, 5(6): 766-783. DOI:10.4161/auto.8788 (  0) 0) |
| [72] |
Bonnet M C, Weil R, Dam E, et al. PKR stimulates NF-kappaB irrespective of its kinase function by interacting with the IkappaB kinase complex[J]. Molecular & Cellular Biology, 2000, 20(13): 4532-4542. (  0) 0) |
| [73] |
Nakamura T, Furuhashi M, Ping L, et al. Double-stranded RNA-dependent protein kinase links pathogen sensing with stress and metabolic homeostasis[J]. Cell, 2010, 140(3): 338-348. DOI:10.1016/j.cell.2010.01.001 (  0) 0) |
| [74] |
Ling Y, Ping L, Fu S, et al. Defective hepatic autophagy in obesity promotes ER stress and causes insulin resistance[J]. Cell Metabolism, 2010, 11(6): 467-478. DOI:10.1016/j.cmet.2010.04.005 (  0) 0) |
| [75] |
Deretic V, Levine B. Autophagy balances inflammation in innate immunity[J]. Autophagy, 2018, 14(2): 243-251. DOI:10.1080/15548627.2017.1402992 (  0) 0) |
| [76] |
Majmundar A J, Wong W J, Simon M C. Hypoxia-inducible factors and the response to hypoxic stress[J]. Molecular Cell, 2010, 40(2): 294-309. DOI:10.1016/j.molcel.2010.09.022 (  0) 0) |
| [77] |
Bellot G, Garcia-Medina R, Gounon P, et al. Hypoxia-induced autophagy is mediated through Hypoxia-inducible factor induction of BNIP3 and BNIP3L via their BH3 Domains[J]. Molecular & Cellular Biology, 2009, 29(10): 2570-2581. (  0) 0) |
| [78] |
Haigis M C, Sinclair D A. Mammalian sirtuins: Biological insights and disease relevance[J]. Annual Review of Pathology Mechanisms of Disease, 2010, 5(1): 253-295. DOI:10.1146/annurev.pathol.4.110807.092250 (  0) 0) |
| [79] |
Lee I H, Cao L, Mostoslavsky R, et al. A Role for the NAD-Dependent deacetylase Sirt1 in the regulation of autophagy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(9): 3374-3379. DOI:10.1073/pnas.0712145105 (  0) 0) |
| [80] |
Zhao Y, Yang J, Liao W, et al. Cytosolic FoxO1 is essential for the induction of autophagy and tumour suppressor activity[J]. Nature Cell Biology, 2010, 12(7): 665-675. DOI:10.1038/ncb2069 (  0) 0) |
| [81] |
Lan F, Cacicedo J M, Ruderman N, et al. SIRT1 modulation of the acetylation status, cytosolic localization, and activity of LKB1[J]. Journal of Biological Chemistry, 2008, 283(41): 27628-27635. DOI:10.1074/jbc.M805711200 (  0) 0) |
| [82] |
Behrends C, Sowa M E, Gygi S P, et al. Network organization of the human autophagy system[J]. Nature, 2010, 466(7302): 68-76. DOI:10.1038/nature09204 (  0) 0) |
| [83] |
Talloczy Z, Jiang W, Virgin H W, et al. Regulation of starvation- and virus-induced autophagy by the elF2alpha kinase signaling pathway[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(1): 190-195. DOI:10.1073/pnas.012485299 (  0) 0) |
| [84] |
Webber J L, Tooze S A. Coordinated regulation of autophagy by p38α MAPK through mAtg9 and p38IP[J]. The EMBO Journal, 2009, 29(1): 27-40. (  0) 0) |
| [85] |
Yang L, Shoji-Kawata S, Sumpter R M, et al. Autosis is a Na+, K+-ATPase-regulated form of cell death triggered by autophagy-inducing peptides, starvation, and hypoxia-ischemia[J]. Proceedings of the National Academy of Sciences, 2013, 110(51): 20364-20371. DOI:10.1073/pnas.1319661110 (  0) 0) |
| [86] |
Liu Y, Levine B. Autosis and autophagic cell death: The dark side of autophagy[J]. Cell Death and Differentiation, 2015, 22(3): 367-376. DOI:10.1038/cdd.2014.143 (  0) 0) |
| [87] |
Luo S, Rubinsztein D C. Apoptosis blocks Beclin 1-dependent autophagosome synthesis-an effect rescued by Bcl-xL[J]. Cell Death and Differentiation, 2010, 17(2): 268-277. DOI:10.1038/cdd.2009.121 (  0) 0) |
| [88] |
MarinO G, Niso-Santano M, Baehrecke E H, et al. Self-consumption: The interplay of autophagy and apoptosis[J]. Nature Reviews. Molecular Cell Biology, 2014, 15(2): 81-94. DOI:10.1038/nrm3735 (  0) 0) |
| [89] |
Jeong H S, Choi H Y, Lee E R, et al. Involvement of caspase-9 in autophagy-mediated cell survival pathway[J]. Molecular Cell Research, 2011, 1813(1): 80-90. (  0) 0) |
| [90] |
Xia H G, Zhang L, Chen G, et al. Control of basal autophagy by calpain1 mediated cleavage of ATG5[J]. Autophagy, 2010, 6(1): 61-66. DOI:10.4161/auto.6.1.10326 (  0) 0) |
| [91] |
Yousefi S, Perozzo R, et al. Calpain-mediated cleavage of Atg5 switches autophagy to apoptosis[J]. Nature Cell Biology, 2006, 8(10): 1124-1132. DOI:10.1038/ncb1482 (  0) 0) |
| [92] |
Park A, Koh H C. NF-κB/mTOR-mediated autophagy can regulate diquat-induced apoptosis[J]. Archives of Toxicology, 2019, 93(5): 1239-1253. DOI:10.1007/s00204-019-02424-7 (  0) 0) |
| [93] |
Morselli E, Shen S, Ruckenstuhl C, et al. P53 inhibits autophagy by interacting with the human ortholog of yeast Atg17, RB1CC1/FIP200[J]. Cell Cycle, 2011, 10(16): 2763-2769. DOI:10.4161/cc.10.16.16868 (  0) 0) |
| [94] |
Tsai M S, Lin C Y, Lin C C, et al. Association between perfluoroalkyl substances and reproductive hormones in adolescents and young adults[J]. International Journal of Hygiene & Environmental Health, 2015, 218(5): 437-443. (  0) 0) |
| [95] |
Liu L, Liao J Z, He X X, et al. The role of autophagy in hepatocellular carcinoma: Friend or foe[J]. Oncotarget, 2017, 8(34): 57707-57722. DOI:10.18632/oncotarget.17202 (  0) 0) |
| [96] |
Huang H, Zhu J, Li Y, et al. Upregulation of SQSTM1/p62 contributes to nickel-induced malignant transformation of human bronchial epithelial cells[J]. Autophagy, 2016, 12(10): 130-145. (  0) 0) |
| [97] |
Messner B, Türkcan A, Ploner C, et al. Cadmium overkill: Autophagy, apoptosis and necrosis signalling in endothelial cells exposed to cadmium[J]. Cellular and Molecular Life Sciences, 2016, 73(8): 1699-1713. DOI:10.1007/s00018-015-2094-9 (  0) 0) |
| [98] |
Duarte F V, Simões A M, Teodoro J S, et al. Exposure to dibenzofuran triggers autophagy in lung cells[J]. Toxicology Letters, 2012, 209(1): 35-42. DOI:10.1016/j.toxlet.2011.11.029 (  0) 0) |
| [99] |
Park J H, Lee J E, Shin I C, et al. Autophagy regulates chlorpyrifos-induced apoptosis in SH-SY5Y cells[J]. Toxicol Appl Pharmacol, 2013, 268(1): 55-67. DOI:10.1016/j.taap.2013.01.013 (  0) 0) |
| [100] |
Mireia N S, Bravo-San P, Rubén G S, et al. ASK1 overexpression accelerates Paraquat-induced autophagy via endoplasmic reticulum stress[J]. Toxicological Sciences, 2011(1): 156-168. (  0) 0) |
| [101] |
Venkatesan R, Park Y U, Ji E, et al. Malathion increases apoptotic cell death by inducing lysosomal membrane permeabilization in N2a neuroblastoma cells: A model for neurodegeneration in Alzheimer's disease[J]. Cell Death Discovery, 2017, 3: 17007. (  0) 0) |
| [102] |
Zhao J M. Autophagy potentially protects against 2, 3, 7, 8-tetrachlorodibenzo-p-Dioxin induced apoptosis in SH-SY5Y cells[J]. Environmental Toxicology, 2016, 31(9): 1068-1079. DOI:10.1002/tox.22116 (  0) 0) |
| [103] |
Bodas M, Van Westphal C, Carpenter-Thompson R, et al. Nicotine exposure induces bronchial epithelial cell apoptosis and senescence via ROS mediated autophagy-impairment[J]. Free Radic Biol Med, 2016, 97: 441-453. DOI:10.1016/j.freeradbiomed.2016.06.017 (  0) 0) |
| [104] |
Yuan L, Liu J, Deng H, et al. Benzo[a]pyrene induces pyroptotic and autophagic death through inhibiting PI3K/Akt signaling pathway in HL-7702 human normal liver cells[J]. The Journal of Toxicological Sciences, 2019, 44(2): 121-131. DOI:10.2131/jts.44.121 (  0) 0) |
| [105] |
Chao Q, Wang C, Peng D, et al. Bisphenol a induces autophagy and apoptosis concurrently involving the Akt/mTOR pathway in testes of pubertal SD rats[J]. Environmental Toxicology, 2017, 32(7-8): 1977. (  0) 0) |
| [106] |
Park J H, Lee J E, Lee S J, et al. Potential autophagy enhancers protect against fipronil-induced apoptosis in SH-SY5Y cells[J]. Toxicology Letters, 2013, 223(1): 25-34. DOI:10.1016/j.toxlet.2013.08.015 (  0) 0) |
| [107] |
Gui Y X, Fan X N, Wang H M, et al. Glyphosate induced cell death through apoptotic and autophagic mechanisms[J]. Neurotoxicology & Teratology, 2012, 34(3): 344-349. (  0) 0) |
| [108] |
Duan P, Hu C, Quan C, et al. 4-Nonylphenol induces apoptosis, autophagy and necrosis in sertoli cells: Involvement of ROS-mediated AMPK/AKT-mTOR and JNK pathways[J]. Toxicology, 2016, 341-343: 28-40. DOI:10.1016/j.tox.2016.01.004 (  0) 0) |
| [109] |
Xu Y, Hu Y, Zhou Y, et al. Rab9 defense against white spot syndrome virus by participation in autophagy in Marsupenaeus japonicas[J]. Fish & Shellfish Immunology, 2020, 104: 245-251. (  0) 0) |
| [110] |
Moreau P, Moreau K, Segarra A, et al. Autophagy plays an important role in protecting Pacific oysters from OsHV-1 and Vibrio aestuarianus infections[J]. Autophagy, 2015, 11(3): 516-526. DOI:10.1080/15548627.2015.1017188 (  0) 0) |
 2022, Vol. 52
2022, Vol. 52