分解代谢是维持正常生命活动的必要保证,细胞内物质分解代谢途径主要有三个:①溶酶体依赖途径; ②泛素-蛋白酶体途径; ③钙依赖蛋白酶系统。自噬作为细胞内分解代谢的机制之一,属于溶酶体依赖途径,主要降解长效蛋白和受损细胞器。正常水平的自噬对维持细胞代谢很重要,但也有潜在的风险,因为胞质内不受调节的降解对细胞是有害的[1]。自噬水平应当被控制在合适的范围内,因此揭示自噬的启动和调控机制具有重要意义。
1 自噬的基本介绍自噬是细胞降解自身蛋白质、细胞器或其他细胞内成分,实现降解产物再利用和细胞器更新的过程。基础水平的自噬对细胞内环境的稳态、受损细胞器的清除、异常蛋白质的聚集和抵抗病原体的入侵起着至关重要的作用。
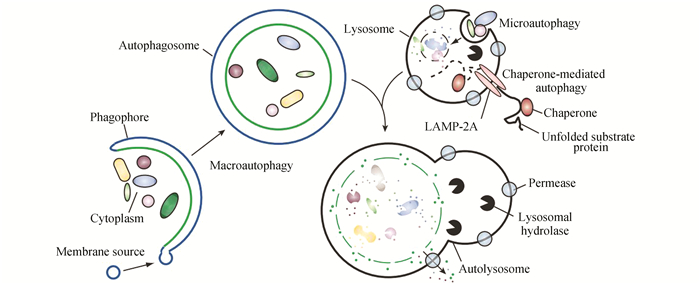
自噬的类型有三种:微自噬(microautophagy),巨自噬(macroautophagy)和分子伴侣依赖的自噬(chaperone-mediated autophagy),他们的机制和功能各有不同。微自噬通过膜内陷直接包绕胞质内成分,酵母菌中的微自噬能选择性降解特定的细胞器; 巨自噬通过被称为自噬小体的独特双层膜结构包绕细胞内成分,包括选择性地包绕特定细胞器或病原体和非选择性地吞噬细胞质内成分。自噬小体与含有水解酶类的溶酶体融合形成自噬溶酶体,自噬小体及其内含物在溶酶体内被酶裂解并释放出大分子物质; 分子伴侣依赖的自噬通过胞质和溶酶体的分子伴侣hsc70将非折叠蛋白底物直接定位在溶酶体膜上[2]。微自噬和巨自噬能特异或非特异地吞噬较大的细胞内结构,而分子伴侣依赖的自噬只能特异降解可溶性蛋白。
|
图 1 三种不同类型的自噬[2] |
自噬的启动与Atg1激酶复合体有关。当外界环境发生变化时,自噬作为细胞保护机制的自噬会被启动,而mTOR是最初感受营养条件变化的信号通路之一。饥饿能通过抑制TOR复合体1(TORC1)启动细胞内的级联反应,使Atg1激酶复合体激活。Atg1激酶复合体处于TORC1下游,由Atg1和Atg17-Atg31-Atg29构成[3],它能招募其他Atg蛋白至吞噬泡的招募位点(phagophore assembly site,PAS)或称前自噬体结构(pre-autophagosomal structure,PAS)并通过磷酸化激活下游位点[4]。
2.2 延伸自噬启动后,会形成不完整的双层膜结构,称吞噬泡(phagophore)。吞噬泡会逐渐延伸形成完整的囊泡,变为吞噬泡的成熟形式,即自噬体(autophagosome)。自噬体的形成是一个动态过程,期间空泡不断与吞噬泡融合并最终形成自噬体。但相对于自噬体来说,吞噬泡只是一个短暂的阶段,所以目前的研究主要集中在自噬体。
吞噬泡的延伸依赖于成核(nucleation),成核是指招募一些特定分子定位到吞噬泡的过程,成核在一定程度上具有放大作用,能进一步招募自噬体延伸所需的蛋白。在成核的过程中,Ⅲ型磷脂酰肌醇3激酶(calss Ⅲ PI3K)复合体Ⅰ也被招募到吞噬泡上,该复合体由5种不同蛋白组成:脂激酶Vps34和调节激酶Vps15,Vps30/Atg6,Atg14和Atg38[5]。Class Ⅲ PI3K的主要作用是催化磷脂酰肌醇产生磷脂酰肌醇-3-磷酸盐(PtdIns3P)[6],而PtdIns3P对于Atg18,Atg2,Atg8,Atg9和Atg12等自噬蛋白的正确定位起着重要作用[6]。
吞噬泡的膜的延伸涉及到两种泛素样反应:①ATG12通过ATG7和ATG10结合到ATG5上,形成ATG5-ATG12复合体(ATG7和ATG10的作用分别类似于泛素化酶E1和E2),ATG5-ATG12复合体通过非共价键与ATG16结合,形成Atg12-Atg5-Atg16的二聚体复合物。②微管相关蛋白轻链3(MAP-LC3/ATG8/LC3)通过ATG7、Atg4(两者类似于E1的作用)、ATG3(类似于E2的作用)和Atg12-Atg5-Atg16复合体(类似于E3的作用)以共价键结合到磷脂酰乙醇胺脂质体上形成LC3-Ⅱ。其中,两种Ubl蛋白(Atg12蛋白和Atg8蛋白)[7]的结构与泛素类似,但并非泛素的同系物; 目前没有充足证据表明第一种泛素样反应中Atg12与Atg5的结合需要E3酶,而第二种泛素样反应中的E3样酶并不是自噬过程中所必需的[8]。在两种泛素样反应作用下,吞噬泡的膜逐渐延伸,最终形成完整的自噬小体。多个LC3阳性的自噬小体在胞质内的任意位置随机生成,然后被微管以动力蛋白依赖的方式结合到溶酶体上[9],并由SNARE蛋白VAMP8和Vti1B介导,融合为自噬溶酶体。哪些分子参与了吞噬泡的延伸是当前的研究热点,现有的研究已经证明了Atg2和Atg18招募两种Ubl蛋白到PAS上,但Atg2或Atg18能否直接招募Atg8和Atg12仍有待进一步研究。
Ath9被认为是吞噬泡形成过程中膜的运输分子,因为:①Atg9是唯一的跨膜蛋白,并对吞噬泡的延伸至关重要[10];②经mTOR抑制剂雷帕霉素处理后,Atg9在胞质中具有高度的流动性[11];③这种蛋白能与自身结合,并转运到PAS。但目前还没有直接证据证明Atg9在膜穿梭中的作用[12]。
2.3 融合自噬体成熟后会定位到空泡(vacuole)并与之融合。融合后的自噬体会将其运输的底物释放到空泡中。但哺乳动物中,自噬体会与溶酶体融合成自噬溶酶体(Autolysosome),而溶酶体直径普遍小于自噬体,所以与空泡融合并不是真核生物的一般特点。目前控制融合时机的机制还不清楚,但有一种防止未成熟的自噬体与囊泡融合的机制,这种机制能阻止自噬体内运输的底物释放到囊泡内。例如,Atg8-PE复合体要经过由Atg4介导的再次降解(Atg4同时也是Atg8早期结合阶段所需的分子),这种降解是自噬体与空泡融合所必需的过程[13]。其原因可能是裂解会触发其他自噬蛋白从成熟自噬体中解离下来,而解离后的自噬体才能与空泡的融合。其他将内含物运往空泡的细胞内过程也需要类似的促进融合的机制[14]。当底物被运到空泡内,自噬体的膜会被一种假定存在的脂肪酶Atg15所降解[15],自噬体内的底物随之被空泡内在的水解酶降解。降解的产物通过各种通透酶(如Atg22)被释放回胞质[16]。
3 自噬的调节 3.1 mTOR途径哺乳动物雷帕霉素靶蛋白(mTOR)是氮依赖的负向调控真核生物自噬的靶点,具有丝氨酸/苏氨酸激酶活性,可以感受环境的变化,并调节细胞代谢和生长。在富营养的环境下,TORC1能直接将Atg13、Atg1和Atg14磷酸化,阻止Atg1-Atg13-Atg17-Atg31-Atg29复合物的形成和激活,抑制自噬特异的Ptdlns3K,阻止自噬的激活。在氨基酸剥夺条件下mTOR会被抑制,进而激活ATG13、ULK1和ULK2,产生饥饿诱导的自噬[9]。胰岛素样生长因子与其受体(IGFIR)的结合能调节mTOR通路的活性,该受体具有酪氨酸激酶活性,能使底物(如IRS1和IRS2)磷酸化,被磷酸化的底物又能激活Akt,而Akt能抑制TSC1/TCS2复合体的活性并负向调控mTOR。如此一来,IGF1R信号通路能激活mTOR并抑制自噬,而在饥饿环境下则相反。在极度饥饿的环境下,mTOR的活性也受GTPases的调控,这种调控主要影响溶酶体的定位,并让mTOR复合体1从溶酶体上解离下来[17]。而在温和的饥饿环境下,虽然mTOR也会被抑制,但这时的mTOR复合体仍结合在溶酶体上。当重新恢复营养状态后,溶酶体与其结合的mTOR移动到质膜,这个过程对于mTOR抑制自噬的形成至关重要[18]。转录调节在氮剥夺的条件下也发挥着作用,如Gcn2是一个能感受氨基酸水平的激酶,激活后的Gcn2能特异地激活ATG基因转录[19]。另外,失活的MTORC1能让去磷酸化的转录因子EB定位到细胞核,并激活自噬基因的转录[20]。在急性早幼粒细胞白血病中,三氧化二砷可通过mTOR通路调节自噬缓解病情[21]。
3.2 AMPK途径腺苷酸活化蛋白激酶(AMPK)是细胞内主要的能量感受器,可以感受葡萄糖水平的变化。在葡萄糖含量较低时,AMP/ATP之比升高将激活AMPK,激活的AMPK会激活Ulk1[22],但这种激活也能被MTORC1依赖的磷酸化所抑制。AMPK也能通过使MTORC1复合体的RPTOC/rapor亚基去磷酸化,或磷酸化并激活MTORC1的负向调控因子TSC1/2复合体,来激活自噬。芍药醇能通过AMPK通路上调自噬抑制平滑肌细胞增殖,从而起到抗动脉粥样硬化作用[23]。
3.3 PKA途径蛋白激酶A(PKA)也能感受葡萄糖的变化,但起到负调控的作用。当葡萄糖含量丰富时,PKA能与cAMP结合并使Atg1和Atg13磷酸化,阻止Atg13定位到PAS。另外,PKA也能磷酸化并激活TORC1或通过抑制AMPK间接激活MTORC1从而抑制自噬。
3.4 Jnk-1途径另一个与饥饿有关的自噬调控通路是通过阻碍Beclin 1(ATG6)和凋亡相关蛋白Bcl-2或Bcl-XL间的相互作用而介导的。饥饿能激活Jnk-1激酶,激活的Jnk-1能使Bcl-2磷酸化从而阻碍Beclin 1与Bcl-2间的相互作用并启动自噬。这种机制也与内质网应激导致的自噬相关[9]。在人膀胱癌细胞中,actein可通过增强Jnk-1通路激活自噬来抑制膀胱癌细胞的增殖[24]。
3.5 P53途径P53能通过mTOR双向调控自噬。在细胞核内,激活的P53四聚物能结合到多种能编码自噬上游分子的启动子基因上启动该自噬,如AMPK的β1和β2亚基,DAPK-1,DRAM,TSC2等。而细胞质内的P53能通过某种机制抑制自噬[25]。致癌基因或基因毒性应激能激活P53,激活的P53能通过AMPK途径或上调磷酸酶与张力蛋白同源物基因(PTEN)或上调TSC1来激活自噬; 而利用P53的化学抑制剂也能激活自噬[26]。另外,溴氯哌喹酮可通过抑制P53激活自噬,发挥抗乳腺癌作用[27]。
3.6 游离脂肪酸某些脂质代谢与自噬的调控密切相关。游离脂肪酸能通过EIF2AK2/PKR依赖途径使EIF2S1/elF2α和MAPK8激活,或使MTORC1受到抑制,从而增强自噬水平。以脂滴形式存在于细胞内的膳食脂肪能激活自噬,并被自噬体包裹进而被溶酶体降解。游离脂肪酸等降解产物又能调节自噬,从而避免脂毒性的产生[28]。
3.7 IP3途径IMPase能减少游离的肌醇和1, 4, 5-三磷酸肌醇(IP3),IP3能通过与内质网表面IP3受体结合,促进内质网内的钙离子释放,激活钙依赖的蛋白酶家族,通过裂解并激活Gsα产生大量cAMP抑制自噬[29]。其他机制也可能参与了这个过程,如下降的IP3水平减少内质网的钙离子流入线粒体,并损伤线粒体的呼吸链,从而通过AMPK途径上调自噬[30]。另外,IP3受体能与Beclin 1结合从而负向调控自噬[31]。丙戊酸钠、卡马西平等神经精神类药物能通过该途径激活自噬[28]。
4 总结自噬在生物进化过程中高度保守,在细胞代谢平衡、生存、生长中发挥着重要作用,自噬水平的紊乱会导致各种疾病的发生。随着越来越多的学者在自噬领域的探索,人们对自噬的理解不断深入,但目前仍有许多尚未解决的问题:①转录后修饰多种多样,但最终会怎样影响自噬仍然未知; ②有学者发现了几种只需一个ATG蛋白亚单位的非经典自噬,而这种自噬的运作机制仍是个谜; ③自噬体膜的来源仍然有争议; ④参与自噬过程的相关蛋白仍未研究透彻; ⑤对底物具有特异性的选择性自噬的机制需要进一步研究。解决以上问题能帮助我们深入地理解自噬,更好地探究自噬与疾病的关系。
| [1] | Gozuacik D, Kimchi A. Autophagy and cell death[J]. Curr Top Dev Biol, 2007, 78(4): 217-245. |
| [2] | Massey AC, Zhang C, Cuervo AM. Chaperone-mediated autophagy in aging and disease[J]. Curr Top Dev Biol, 2006, 73: 205-235. DOI: 10.1016/S0070-2153(05)73007-6. |
| [3] | Kamada Y, Funakoshi T, Shintani T, et al. Tor-mediated induction of autophagy via an Apg1 protein kinase complex[J]. Journal of Cell Biology, 2000, 150(6): 1 507-1 513. DOI: 10.1083/jcb.150.6.1507. |
| [4] | Papinski D, Schuschnig M, Reiter W, et al. Early steps in autophagy depend on direct phosphorylation of Atg9 by the Atg1 kinase[J]. Mol Cell, 2014, 53(3): 471-483. DOI: 10.1016/j.molcel.2013.12.011. |
| [5] | Kihara A, Noda T, Ishihara N, et al. Two distinct Vps34 phosphatidylinositol 3-kinase complexes function in autophagy and carboxypeptidase Y sorting in Saccharomyces cerevisiae[J]. J Cell Biol, 2001, 152(3): 519-530. DOI: 10.1083/jcb.152.3.519. |
| [6] | Burman C, Ktistakis NT. Regulation of autophagy by phosphatidylinositol 3-phosphate[J]. FEBS Lett, 2010, 584(7): 1 302-1 312. DOI: 10.1016/j.febslet.2010.01.011. |
| [7] | Ohsumi Y. Molecular dissection of autophagy: two ubiquitin-like systems[J]. Nat Rev Mol Cell Biol, 2001, 2(3): 211-216. DOI: 10.1038/35056522. |
| [8] | Cao Y, Cheong H, Song H, et al. In vivo reconstitution of autophagy in Saccharomyces cerevisiae[J]. J Cell Biol, 2008, 182(4): 703-713. DOI: 10.1083/jcb.200801035. |
| [9] | Ravikumar B, Sarkar S, Davies JE, et al. Regulation of mammalian autophagy in physiology and pathophysiology[J]. Physiol Rev, 2010, 90(4): 1 383-1 435. DOI: 10.1152/physrev.00030.2009. |
| [10] | Noda T, Kim J, Huang WP, et al. Apg9p/Cvt7p is an integral membrane protein required for transport vesicle formation in the Cvt and autophagy pathways[J]. J Cell Biol, 2000, 148(3): 465-480. DOI: 10.1083/jcb.148.3.465. |
| [11] | Yamamoto H, Kakuta S, Watanabe TM, et al. Atg9 vesicles are an important membrane source during early steps of autophagosome formation[J]. J Cell Biol, 2012, 198(2): 219-233. DOI: 10.1083/jcb.201202061. |
| [12] | Reggiori F, Shintani T, Nair U, et al. Atg9 cycles between mitochondria and the pre-autophagosomal structure, in yeasts[J]. Autophagy, 2005, 1(2): 101-109. DOI: 10.4161/auto.1.2.1840. |
| [13] | Nair U, Yen WL, Mari M, et al. A role for Atg8-PE deconjugation in autophagosome biogenesis[J]. Autophagy, 2012, 8(5): 780-793. DOI: 10.4161/auto.19385. |
| [14] | Jiang P, Nishimura T, Sakamaki Y, et al. The HOPS complex mediates autophagosomelysosome fusion through interaction with syntaxin 17[J]. Mol Biol Cell, 2014, 25(8): 1 327-1 337. DOI: 10.1091/mbc.e13-08-0447. |
| [15] | Epple U, Suriapranata I, Eskelinen E, et al. Aut5/Cvt17p, a putative lipase essential for disintegration of autophagic bodies inside the vacuole[J]. Journal of Bacteriology, 2001, 183(20): 5 942-5 955. DOI: 10.1128/JB.183.20.5942-5955.2001. |
| [16] | Yang Z, Huang J, Geng J, et al. Atg22 recycles amino acids to link the degradative and recycling functions of autophagy[J]. Molecular Biology of the Cell, 2006, 17(12): 5 094-5 104. DOI: 10.1091/mbc.e06-06-0479. |
| [17] | Sancak Y, Barpeled L, Zoncu R, et al. Ragulator-Rag complex targets mTORC1 to the lysosomal surface and is necessary for its activation by amino acids[J]. Cell, 2010, 141(2): 290-303. DOI: 10.1016/j.cell.2010.02.024. |
| [18] | Korolchuk VI, Shinji S, Maike L, et al. Lysosomal positioning coordinates cellular nutrient responses[J]. Nature Cell Biology, 2011, 13(4): 453-460. DOI: 10.1038/ncb2204. |
| [19] | B'Chir W, Maurin AC, Carraro V, et al. The eIF2α/ATF4 pathway is essential for stress-induced autophagy gene expression[J]. Nucleic Acids Research, 2013, 41(16): 7 683-7 699. DOI: 10.1093/nar/gkt563. |
| [20] | Settembre C, Di MC, Polito VA, et al. TFEB links autophagy to lysosomal biogenesis[J]. Science, 2011, 332(6 036): 1 429-1 433. |
| [21] | Li T, Ma RS, Zhang Y, et al. Arsenic trioxide promoting ETosis in acute promyelocytic leukemia through mTOR-regulated autophagy[J]. Cell Death Dis, 2018, 9(2): 75. DOI: 10.1038/s41419-017-0018-3. |
| [22] | Egan DF, Shackelford DB, Mihaylova MM, et al. Phosphorylation of ULK1 (hATG1) by AMP-activated protein kinase connects energy sensing to mitophagy[J]. Science, 2011, 331(6 016): 456-461. |
| [23] | Wu HF, Song AW, Hu WJ, et al. The anti-atherosclerotic effect of paeonol against vascular smooth muscle cell proliferation by up-regulation of autophagy via the AMPK/mTOR signaling pathway[J]. Front Pharmacol, 2017, 8: 948. |
| [24] | Ji L, Zhong B, Jiang X, et al. Actein induces autophagy and apoptosis in human bladder cancer by potentiating ROS/JNK and inhibiting AKT pathways[J]. Oncotarget, 2017, 8(68): 112. |
| [25] | Maiuri MC, Galluzzi L, Morselli E, et al. Autophagy regulation by p53[J]. Current Opinion in Cell Biology, 2010, 22(2): 181-185. DOI: 10.1016/j.ceb.2009.12.001. |
| [26] | Fleming A, Noda T, Yoshimori T, et al. Chemical modulators of autophagy as biological probes and potential therapeutics[J]. Nature Chemical Biology, 2011, 7(1): 9-17. DOI: 10.1038/nchembio.500. |
| [27] | Xia XJ, Wang L, Zhang XJ, et al. Halofuginone-induced autophagy suppresses the migration and invasion of MCF-7 cells via regulation of STMN1 and p53[J]. J Cell Biochem, 2018, 119(5): 4 009-4 020. DOI: 10.1002/jcb.v119.5. |
| [28] | Kim KH, Lee MS. Autophagy—a key player in cellular and body metabolism[J]. Nature Reviews Endocrinology, 2014, 10(6): 322-337. DOI: 10.1038/nrendo.2014.35. |
| [29] | Williams A, Sarkar S, Cuddon P, et al. Novel targets for Huntington's disease in an mTOR-independent autophagy pathway[J]. Nature Chemical Biology, 2008, 4(5): 295-305. DOI: 10.1038/nchembio.79. |
| [30] | Cárdenas C, Miller RA, Smith I, et al. Essential regulation of cell bioenergetics by constitutive InsP 3 receptor Ca2+ transfer to mitochondria[J]. Cell, 2010, 142(2): 270-283. DOI: 10.1016/j.cell.2010.06.007. |
| [31] | Vicencio JM, Ortiz C, Criollo A, et al. The inositol 1, 4, 5-trisphosphate receptor regulates autophagy through its interaction with Beclin 1[J]. Cell Death & Differentiation, 2009, 16(7): 1 006-1 017. |
 2018, Vol. 39
2018, Vol. 39


