| 碳水化合物结合结构域的研究进展 |
随着工业的日益发达, 能源需求日渐紧张, 生物质资源因其来源广泛、绿色低耗逐步被众多研究者注意, 其中生物酶降解生物质资源可以有效缓解能源问题, 但在具体实践中存在诸多问题, 例如酶的利用效率低下、生物质资源的结构复杂性与商品化酶种类少的矛盾等。生物质原料中碳水化合物含量巨大, 以纤维素、淀粉、几丁质等形式存在[1], 通常情况下分子量大、降解困难, 为应用生产带来巨大挑战。
针对碳水化合物建立的碳水化合物活性酶数据库(http://www.cazy.org), 是涉及能够降解、修饰或者产生糖苷键酶类的相关结构域的数据库资源[2-3], 基于酶蛋白序列相似性可归类为不同的蛋白质家族, 其中包括糖苷水解酶(GHs)、糖基转移酶(GTs)、多糖裂解酶(PLs)、碳水化合物酯酶(CEs)、辅助活性酶类(AAs)以及碳水化合物结合结构域(CBMs)。
碳水化合物结合结构域是一类非催化活性的具有折叠结构的蛋白质结构, 能够与多糖(特别是不溶性多糖)进行特异性结合[4]。根据最初发现的几个结合纤维素的模块, CBM曾经被归类为纤维素结合域(Cellulose binding domain, 简称CBD)。随着不断发现碳水化合物活性酶中的其他模块结合纤维素以外的碳水化合物, 开始出现“碳水化合物结合结构域”这一术语[5]。截至2021年10月, 基于氨基酸序列的相似性, CBM被分为88个家族, 大部分来自于细菌中的酶类, 并且与细胞壁聚合物结合的家族居多。其中第50家族占比最高, 包含123 000多种CBM, 占总CBM的44%左右, 另外48、5、13和32家族也占比较高, 分别占比15.6%、4.8%、4.3%、4.1%。大部分CBM的氨基酸残基数在30~250个之间, 可以位于肽链的N端、C端或中间[6]。
近年来, 关于CBM的结构、作用机制、重组融合以及与底物结合的研究逐渐增多, 这对促进碳水化合物的研究以及推动碳水化合物结合结构域的广泛应用具有重大作用。
1 CBM的命名、分类以及结构 1.1 CBM的命名与分类最早发现的几种CBM根据氨基酸序列相似性依次命名为Type I至Type XIII, 随着越来越多CBM被发现, CBM改用家族进行命名, 其中第1家族至第13家族等同于最早命名的Type I至Type XIII, 基于蛋白质序列的一致性, 目前CBM分为88个家族。为了提高准确度, CBM命名也可以加入物种来源及其来源的酶, 例如来源于日本纤维弧菌(Cellvibrio japonicus Ueda107)木聚糖酶XynA的第10家族CBM, 可以命名为CjCBM10或CjXynACBM10。如果酶含有相同家族的串联CBM, 则加入CBM在酶中相对于N-末端的位置相匹配的数字, 例如CBM10-1、CBM10-2。
1.2 CBM的结构与分类过去几十年通过用X射线晶体学和核磁共振谱测定了不同家族CBM的三维结构。在CBM分类中, 根据蛋白质折叠和三维结构分类为7个“折叠族”[7], 分别为β-三明治、β-三叶折叠、半胱氨酸结、独特型、OB折叠、hevein折叠和独特型含hevein-折叠, 其中以β折叠最为常见。
β-三明治“折叠族”CBM通常包括两个β折叠, 每个β折叠由3~6条反平行β链组成。大部分此类折叠能够结合金属离子[8], 这一特性利于结构稳定性。另外2006年Hashimotoz[9]在Boraston[7]研究的基础上提出可以将β-三明治结构细分为β-果冻卷和免疫球蛋白两类结构。β-三叶折叠是一个连续的氨基酸序列, 具有四个β链以及两个发夹结构。Valerie等[10]在研究β-三叶折叠结构的CBM时发现Streptomyces lividans的CBM13因其独特的结构特征, 大大增加了对底物的高度亲和性。Hevein折叠是一个仅具有大约40个氨基酸的结构域, 具有两个小β片和一个小的螺旋区域, 但是可以有效容纳延伸的结合位点, 剩余的折叠族是仅含β-折叠和卷曲的30~60个AA的小多肽, 其配体特异性的多样性较低。
除此之外, 还可以根据CBM底物特性将其再次分为三种类型[7, 11], A型(CBMs TypeA), 也称为表面结合型; B型(CBMs TypeB), 也称为链式结合型; C型(CBMs TypeC), 也称为寡糖结合型(见表 1)。
| 表 1 CBM类型 |
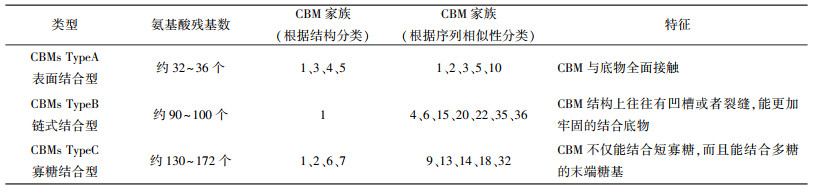 |
A型CBM表面具有芳香族氨基酸, 因此有疏水性, 同时能够形成相对平坦的结合位点, 故而对于不可溶的底物有较强的亲和性, 可以与结晶多糖结合, 但是对于可溶性的多糖却难以结合[12-13]; B型CBM采用结合单聚糖链的碳水化合物结合凹槽, 底物的聚合程度能够影响结合能力, 通常能够结合聚合度较高的底物(如木聚糖、甘露聚糖、半乳聚糖、混合键的葡聚糖和非结晶纤维素)[14-15], 另外除了保守芳香族氨基酸影响, 氢键在B型CBM底物特异性的结合过程中具有关键作用; C型CBM可以有效结合短寡糖(一糖、二糖、三糖), 由于其不具有类似B型的延伸结合位点凹槽, 所以底物结合较为集中[16], 同时结合位点表现的口袋拓扑结构, 能够有效识别聚糖的非还原末端[17]。
由图 1可见, CBM的结合位点与周围残基的独特互补能够显示不同的表面拓扑结构, YeCBM32结合位点的不同拓扑结构可能是与多糖链内部结合有关系, 例如, 聚半乳糖醛酸裂解酶能够利用碱性氨基酸侧链和糖的酸性糖醛酸基稳定相互作用的盐桥。另外, 通过定点诱变可以验证碳水化合物活性位点的位置, YeCBM32中色氨酸用丙氨酸取代, 发现色氨酸丢失会影响他们的环境[18]。肠道共生菌Roseburia中CBM86中不变赖氨酸(K95)的侧链对于底物结合至关重要, 因为它为堆叠在Y62上的木糖基环提供了唯一的带电氢键, 实验表明, 突变体能够保留整体蛋白质结构, 但是亲和力大大下降[19]。
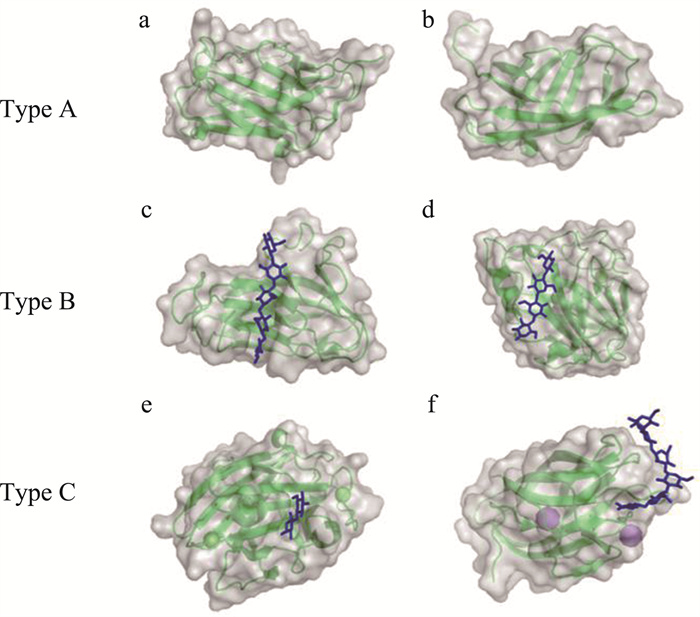 |
| 注: PDB ID a—4B9C;b—1EXG; c—1GU3;d—1J84;e—1I82;f—1W9W。 图 1 根据底物特异性结合的三类CBM分类 |
2 作用机制 2.1 邻近作用
CBM可以增加底物附近的酶浓度[20], 导致多糖水解增加。Cécile Hervé[21]研究发现, 在植物细胞壁完整的情况下, 拥有纤维素导向的CBM可能会为多糖降解酶带来选择性优势。主要是通过酶与细胞壁物质保持紧密接触来完成。酶通过其纤维素特异性CBM结合到细胞壁, 而催化模块能够接近目标底物, 该底物能够达到与纤维素微纤维紧密结合的效果。
2.2 叠加作用通常碳水化合物活性酶中含有不止一个CBM[22], 多CBM会产生一定的叠加效应。产气荚膜梭菌分泌一种多模块唾液酸酶, 其在N末端具有CBM32和CBM40。CBM32结合N-乙酰半乳糖胺和半乳糖, 而CBM40识别唾液酸, 它们的串联排列可能起到增强对含有各自配体的糖缀合物的亲和作用[23]。具有3个CBM的双功能酶CelA, 其中具备纤维素结合能力的有2个, 剩余1个起辅助作用。酶作用底物时由表面向深层降解, 从而底物产生空腔结构[24], 刘亮等[25]研究发现, 甘露聚糖酶-乙酰酯酶双功能酶44884具有2个CBM65, 对底物甘露聚糖以及结晶纤维素有良好的结合活性, 并且对催化域的水解均有明显促进作用。
2.3 底物靶向作用CBM可以通过将酶引导到富含木葡聚糖的细胞壁区域来增强活性[26]。唐存多[27]通过构建C端带有CBM的reAuMan5A-CBM融合蛋白, 经过在P.pastoris GS115中进行蛋白表达及纯化后, 发现重组融合蛋白能够与微晶纤维素具有92.3%的结合, 但是底物与reAuMan5A并无结合能力, 这表现出重组融合蛋白reAuMan5A-CBM具有良好的纤维素结合能力。GHs的CBM靶向不溶性多糖, 即附加在木聚糖降解酶上的CBM35能识别GlcA。酶被转移到半纤维素多糖上, 从而使酶能够到达目标底物[17]。
2.4 微晶破坏作用研究发现, 碳水化合物活性酶中的某些CBM能够破坏结晶型多糖, 从而有利于酶的催化结构结合底物, 提高酶的催化效率, 但是这种破坏作用的研究尚不透彻[28]。目前, 研究发现可以分为两种破坏方式, 直接氧化催化以及通过底物氢键断裂实现。CBM2a和CBM44在单独存在时具有破坏纤维素纤维结构的作用, 同时可以释放小碎片, 表现出疏解结晶纤维素的能力[29]。后来研究中发现对淀粉也有此作用, CBM与淀粉特异性结合的同时破坏淀粉表面, 从而提高底物分解率。
3 CBM融合表达与应用 3.1 CBM融合及重组表达基因工程的快速发展, 使得可以通过CBM的融合来提高酶的热稳定性、活性和底物结合能力[30-31]。来源于某些生物体的CBM稳定性较高[32-33], 属于热稳定性结构域。有研究显示, 热稳定性的结构域在和不稳定的蛋白结构域发生融合后, 通常能够达到提高后者热稳定性的效果。因此在酶分子理性改造过程中, 借助稳定CBM可以改善酶学性质, 例如稳定性、最适温度以及pH等。另外借助CBM能够特异性结合底物的特性, 通过融合和重组表达的生物方法, 使不同CBM与同一种催化结构域融合, 可以达到降解不同类型的碳水化合物的效果。CBM融合与重组表达见表 2。
| 表 2 CBM融合与重组表达 |
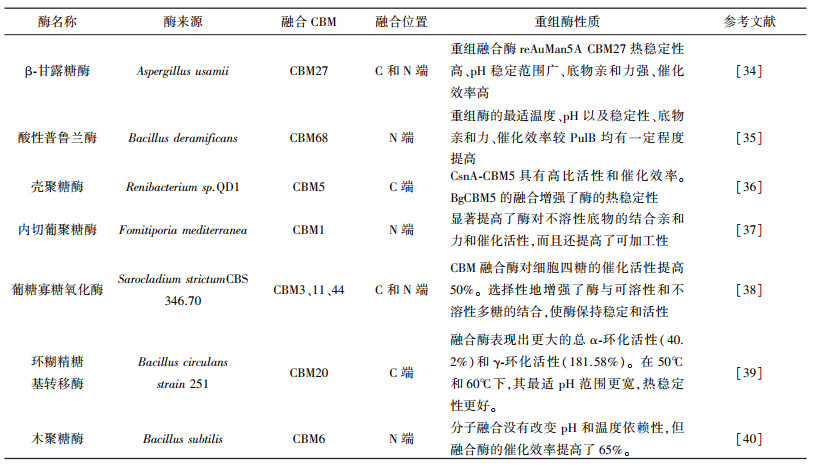 |
但是并非所有CBM融合都会产生积极作用, 有些结构域能够相互融合, 但与此同时也影响了功能。CBM融合位置不同产生的作用也存在区别, 在之前的研究中表明, CBM可以融合在C端或N端, 少数还会位于中间[6]。β-甘露聚糖酶AuMan5A与来源于海栖热孢菌Thermotoga maritima CBM27分别结合在C端和N端, 对酶学性质产生了不同影响[41]。
除此之外, CBM与催化活性结构域融合时使用的连接肽(Linker)对于融合效果也会有一定程度的影响[42]。连接肽通常具有较高的脯氨酸、丝氨酸、苏氨酸。壳聚糖酶与CBM5的重组融合中将FNIII作为连接催化模块和CBM的接头[36], 能够有效提高融合效率和酶稳定性。接头的选择对多模块酶的组装和功能具有重要意义, 连接体的长度和刚性调整可以影响催化结构域和CBM之间的分子内相互作用。
3.2 碳水化合物结合结构域的应用 3.2.1 纤维素改性CBM能够识别并特异性结合到晶体表面的特性, 为纤维素的修饰改性提供了可能性。Kevin Aïssa等[43]研究发现CBM2a对结晶纤维素有很强的亲和能力, 将CBM2a引入到纤维素表面进行改性, 能够为纤维素表面提供强有力的非共价修饰, 可以增加干燥后功能化纤维素纳米晶体的再分散性, 并基于空间相互作用提高了悬浮稳定性。Zhang等[44]通过CBM与角质酶对醋酸纤维素纤维进行表面改性, 另外造纸过程中加入CBM也使成纸机械性得到改善[45]。
3.2.2 生物材料近年来, 借助大肠杆菌以及毕赤酵母等重组表达系统, CBM还被研究为生物医学工具, 即通过生物材料的功能化, 促进了不同领域的应用。Barbosa等[46]研究发现, CBM3能精确修饰纤维素水凝胶, 在温和的生物条件下能够促进纤维素水凝胶的功能化, 而不需要复杂的化学接枝过程, 能够实现纤维素基水凝胶在生物医学领域的应用。
3.2.3 分子探针重组CBM可用于分子探针, 用于表征和监测天然、工程多糖的变化以及固定和改善酶性质。分子探针的相对荧光强度可以用来量化在酶促分解期间发生的结构变化, 为阐明纤维素的超分子亚结构及其对酶可及性提供了一种定量方法[47]。Gourlay等[48]通过监测纤维尺寸和评估纤维表面荧光标记的CBM的结合特征, 证明膨胀素靶向纤维素纤维位错内的无定形区域, 促进这些位错处的纤维断裂。
CBM重组有助于阐明分子进化, 以及许多生物过程中涉及的碳水化合物-蛋白质相互作用, 除了以上应用, CBM重组融合也可以应用于改善酶性质、细胞固定化、新型金属吸附剂等生物医学、复合材料和电子相关领域, 也可以借助CBM作高通量筛选碳水化合物活性酶的新型多功能微阵列方法, CBM的广泛应用展示出其作为生物技术工具的广阔前景。
4 展望CBM作为结构功能域, 在近年来吸引众多研究人员关注。碳水化合物与蛋白质相互作用对于许多生物过程是必不可少的, 而CBM在碳水化合物的识别中起着关键作用。随着新家族CBM的探索以及与多糖结合机制的研究, 对将来改造碳水化合物活性酶和高效利用碳水化合物提供理论基础。另外基因工程的发展大大促进CBM融合技术, 未来CBM在功能材料、生物、医学等领域将具有更为广泛的应用。
| [1] |
SIDAR A, ALBUQUERQUE E D, VOSHOL G P, et al. Carbohydrate binding modules: diversity of domain architecture in amylases and cellulases from filamentous microorganisms[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 871. DOI:10.3389/fbioe.2020.00871 |
| [2] |
CANTARELB L, COUTINHO P M, RANCUREEL C, et al. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for glycogenomics[J]. Nucleic Acids Research, 2009, 37: 233-238. DOI:10.1093/nar/gkn663 |
| [3] |
王帅, 陈冠军, 张怀强, 等. 碳水化合物活性酶数据库(CAZy)及其研究趋势[J]. 生物加工过程, 2014, 12(1): 102-108. |
| [4] |
李恒, 唐双焱. 碳水化合物结合结构域研究进展[J]. 微生物学报, 2017, 57(8): 1160-1167. |
| [5] |
杨昂. 碳水化合物结合模块CBM9-2引入Thumb获得木聚糖酶活性[D]. 郑州: 河南农业大学, 2018.
|
| [6] |
CARVALHO C C, PHAN N N, CHEN Y, et al. Carbohydrate-binding module tribes[J]. Biopolymers, 2015, 103(4): 203-214. DOI:10.1002/bip.22584 |
| [7] |
BORASTON A B, BOLAM D N, GILBERT H J, et al. Carbohydrate-binding modules: fine-tuning polysaccharide recognition[J]. Biochemical Journal, 2004, 382(3): 769-781. DOI:10.1042/BJ20040892 |
| [8] |
LIU T, YI Z W, ZENG R Y, et al. The first characterization of a Ca2+-dependent carbohydrate-binding module of β-1, 3-xylanase from Flammeovirga pacifica[J]. Enzyme and Microbial Technology, 2019, 131: 109418. DOI:10.1016/j.enzmictec.2019.109418 |
| [9] |
HASHIMOTO H. Recent structural studies of carbohydrate-binding modules[J]. Cellular and Molecular Life Sciences CMLS, 2006, 63(24): 2954-2967. DOI:10.1007/s00018-006-6195-3 |
| [10] |
NOTENBOOM V, BORASTON A B, WILLIAMS S J, et al. High-resolution crystal structures of the lectin-like xylan binding domain from Streptomyces lividans xylanase 10A with bound substrates reveal a novel mode of xylan binding[J]. Biochemistry, 2002, 41(13): 4246-4254. DOI:10.1021/bi015865j |
| [11] |
GILBERT H J, KNOX J P, BORASTON A B. Advances in understanding the molecular basis of plant cell wall polysaccharide recognition by carbohydrate-binding modules[J]. Current Opinion in Structural Biology, 2013, 23(5): 669-677. DOI:10.1016/j.sbi.2013.05.005 |
| [12] |
SHOSEYOV O, SHANI Z, LEVY I. Carbohydrate binding modules: biochemical properties and novel applications[J]. Microbiology and Molecular Biology Reviews, 2006, 70(2): 283-295. DOI:10.1128/MMBR.00028-05 |
| [13] |
PIRES V M R, PEREIRA P M M, BRáS J L A, et al. Stability and ligand promiscuity of type A carbohydrate-binding modules are illustrated by the structure of Spirochaeta thermophila StCBM64C[J]. Journal of Biological Chemistry, 2017, 292(12): 4847-4860. DOI:10.1074/jbc.M116.767541 |
| [14] |
OLIVEIRA C, CARVALHO V, DOMINGUES L, et al. Recombinant CBM-fusion technology: applications overview[J]. Biotechnology Advances, 2015, 33(3/4): 358-369. |
| [15] |
KOGNOLE A A, PAYNE C M. Cellulose-specific Type B carbohydrate binding modules: understanding oligomeric and non-crystalline substrate recognition mechanisms[J]. Biotechnology for Biofuels, 2018, 11(1): 1-16. DOI:10.1186/s13068-017-1003-x |
| [16] |
NOTENBOOM V, BORASTON A B, KILBURN D G, et al. Crystal structures of the family 9 carbohydrate-binding module from Thermotoga maritima xylanase 10A in native and ligand-bound forms[J]. Biochemistry, 2001, 40(21): 6248-6256. DOI:10.1021/bi0101704 |
| [17] |
JIA X, WANG C, DU X, et al. Specific hydrolysis of curdlan with a novel glycoside hydrolase family 128 β-1, 3-endoglucanase containing a carbohydrate-binding module[J]. Carbohydrate Polymers, 2021, 253: 117276. DOI:10.1016/j.carbpol.2020.117276 |
| [18] |
ABBOTT D W, HRYNUIK S, BORASTON A B. Identification and characterization of a novel periplasmic polygalacturonic acid binding protein from Yersinia enterolitica[J]. Journal of Molecular Biology, 2007, 367(4): 1023-1033. DOI:10.1016/j.jmb.2007.01.030 |
| [19] |
LETH M L, EJBY M, MADLAND E, et al. Molecular insight into a new low-affinity xylan binding module from the xylanolytic gut symbiont roseburia intestinalis[J]. The FEBS Journal, 2020, 287(10): 2105-2117. DOI:10.1111/febs.15117 |
| [20] |
Guillén D, Sánchez S, Rodríguez-Sanoja R. Carbohydrate-binding domains: multiplicity of biological roles[J]. Applied Microbiology and Biotechnology, 2010, 85(5): 1241-1249. DOI:10.1007/s00253-009-2331-y |
| [21] |
Hervé C, ROGOWSKI A, BLAKE A W, et al. Carbohydrate-binding modules promote the enzymatic deconstruction of intact plant cell walls by targeting and proximity effects[J]. Proceedings of the National Academy of Sciences, 2010, 107(34): 15293-15298. |
| [22] |
MOLLER M S, El BOUABALLATI S, HENRISSAT B, et al. Functional diversity of three tandem C-terminal carbohydrate-binding modules of a β-mannanase[J]. Journal of Biological Chemistry, 2021, 296. |
| [23] |
BORASTON A B, FICKO-BLEAN E, HEALEY M. Carbohydrate recognition by a large sialidase toxin from Clostridium perfringens[J]. Biochemistry, 2007, 46(40): 11352-11360. DOI:10.1021/bi701317g |
| [24] |
BRUNECKY R, ALAHUHTA M, XU Q, et al. Revealing nature's cellulase diversity: the digestion mechanism of Caldicellulosiruptor bescii CelA[J]. Science, 2013, 342(6165): 1513-1516. DOI:10.1126/science.1244273 |
| [25] |
刘亮, 刘佳文, 王若楠, 等. 新型甘露聚糖酶-乙酰酯酶双功能酶的功能和结构域协同研究[J]. 微生物学报, 2020, 60(12): 2690-2704. |
| [26] |
VENDITTO I, NAJMUDIN S, LUIS A S, et al. Family 46 Carbohydrate-binding modules contribute to the enzymatic hydrolysis of xyloglucan and β-1, 3-1, 4-Glucans through distinct mechanisms[J]. Journal of Biological Chemistry, 2015, 290(17): 10572-10586. DOI:10.1074/jbc.M115.637827 |
| [27] |
唐存多. β-甘露聚糖酶的基因克隆、分子改造及低聚甘露糖的酶法制备[D]. 无锡: 江南大学, 2013.
|
| [28] |
周顺华. 碳水化合物结合结构域及其应用[J]. 现代食品, 2016(5): 113-115. |
| [29] |
GOURLAY K, ARANTEA V, SADDLER J N. Use of substructure-specific carbohydrate binding modules to track changes in cellulose accessibility and surface morphology during the amorphogenesis step of enzymatic hydrolysis[J]. Biotechnology for Biofuels, 2012, 5(1): 1-14. DOI:10.1186/1754-6834-5-1 |
| [30] |
李娜, 夏欢, 江燕斌. 融合糖苷水解酶在生物质转化中的研究进展[J]. 中国科学: 化学, 2021, 51(7): 831-843. |
| [31] |
刘亮伟. 底物结合模块C1/C2双端融合提高黑曲霉木聚糖酶稳定性和活性[D]. 郑州: 河南农业大学, 2016.
|
| [32] |
LEE J P, SHIN E S, CHO M Y, et al. Roles of Carbohydrate-Binding Module (CBM) of an Endo-β-1, 4-Glucanase (Cel5L)from Bacillus sp.KD1014 in Thermostability and Small-Substrate Hydrolyzing Activity[J]. Journal of Microbiology and Biotechnology, 2018, 28(12): 2036-2045. DOI:10.4014/jmb.1802.10001 |
| [33] |
李相前, 邵蔚蓝. 海栖热袍菌内切葡聚糖酶Cel12B与木聚糖酶XynA CBD结构域融合基因的构建, 表达及融合酶性质分析[J]. 微生物学报, 2006, 46(5): 726-729. DOI:10.3321/j.issn:0001-6209.2006.05.008 |
| [34] |
LI J, WANG C, HU D, et al. Engineering a family 27 carbohydrate-binding module into an Aspergillus usamii β-mannanase to perfect its enzymatic properties[J]. Journal of Bioscience and Bioengineering, 2017, 123(3): 294-299. |
| [35] |
曾艳, 郑宏臣, 付晓平, 等. 普鲁兰酶N端结构域CBM68对酶学性质的影响[J]. 南开大学报, 2018, 51(4): 94-99. |
| [36] |
HAN Y, GAO P, YU W, et al. Thermostability enhancement of chitosanase CsnA by fusion a family 5 carbohydrate-binding module[J]. Biotechnology Letters, 2017, 39(12): 1895-1901. |
| [37] |
PAN R, HU Y, LONG L, et al. Extra carbohydrate binding module contributes to the processivity and catalytic activity of a non-modular hydrolase family 5 endoglucanase from Fomitiporia mediterranea MF3/22[J]. Enzyme and Microbial Technology, 2016, 91: 42-51. |
| [38] |
FOUMANI M, VUONG T V, MACCORMICK B, et al. Enhanced polysaccharide binding and activity on linear β-glucans through addition of carbohydrate-binding modules to either terminus of a glucooligosaccharide oxidase[J]. PLoS One, 2015, 10(5): e0125398. |
| [39] |
JIA X, GUO Y, LIN X, et al. Fusion of a family 20 carbohydrate‐binding module (CBM20)with cyclodextrin glycosyltransferase of Geobacillus sp.CHB1 improves catalytic efficiency[J]. Journal of Basic Microbiology, 2017, 57(6): 471-480. |
| [40] |
HOFFMAM Z B, ZANPHORLIN L M, COTA J, et al. Xylan-specific carbohydrate-binding module belonging to family 6 enhances the catalytic performance of a GH11 endo-xylanase[J]. New Biotechnology, 2016, 33(4): 467-472. |
| [41] |
刘亮伟, 程洁, 陈红歌. 木聚糖酶碳水化合物结合结构域研究进展[J]. 生物工程学报, 2010, 26(3): 29-296. |
| [42] |
袁风娇, 王春娟, 李雪晴, 等. CBM融合位置对AuMan5A酶学性质的影响[J]. 食品与生物技术学报, 2019, 38(4): 1-7. |
| [43] |
AïSSA K, KARAASLAN M A, RENNECKAR S, et al. Functionalizing cellulose nanocrystals with click modifiable carbohydrate-binding modules[J]. Biomacromolecules, 2019, 20(8): 3087-3093. |
| [44] |
ZHANG Y, CHEN S, WU J, et al. Enzymatic surface modification of cellulose acetate fibre by cutinase-CBM (carbohydrate-binding module) fusion proteins[J]. Biocatalysis and Biotransformation, 2012, 30(2): 184-189. |
| [45] |
张雨, 王晓燕, 张鑫宇, 等. CBM的结构、应用及与多糖的相互作用研究进展[J]. 纤维素科学与技术, 2019, 27(2): 66-73. |
| [46] |
BARBOSA M, SIMõES H, PRAZERES D M F. Functionalization of cellulose-based hydrogels with bi-functional fusion proteins containing carbohydrate-binding modules[J]. Materials, 2021, 14(12): 3175. |
| [47] |
NOVY V, AïSSA K, NIELSEN F, et al. Quantifying cellulose accessibility during enzyme-mediated deconstruction using 2 fluorescence-tagged carbohydrate-binding modules[J]. Proceedings of the National Academy of Sciences, 2019, 116(45): 22545-22551. |
| [48] |
GOURLAY K, HU J, ARANTES V, et al. The use of carbohydrate binding modules (CBMs) to monitor changes in fragmentation and cellulose fiber surface morphology during cellulase-and swollenin-induced deconstruction of lignocellulosic substrates[J]. Journal of Biological Chemistry, 2015, 290(5): 2938-2945. |
 2022, Vol. 36
2022, Vol. 36


