| 壳聚糖酶的分子结构与作用机制的研究进展 |
2. 烟台大学 生命科学学院, 烟台 264005;
3. 山东大学 公共卫生学院微生物检验学系, 济南 250012
2. School of Life Sciences, Yantai University, Yantai 264005, China;
3. Department of Microbiological Laboratory Technology, School of Public Health, Shandong University, Jinan 250012, China
壳聚糖是由N-乙酰-α-氨基-D-葡萄糖胺以β(1→4)糖苷键连结而成的线性含氮多糖, N—乙酰度在50%以下[1](壳聚糖结构如图 1所示[2])。壳聚糖具有独特的物理化学性质和多样的生物学活性, 广泛应用于农业、医学、化妆品和废水处理等领域[3-4]。除壳聚糖外, 壳聚糖的水溶性降解产物也具有广泛的生物活性和极高的实用价值[5-6]。
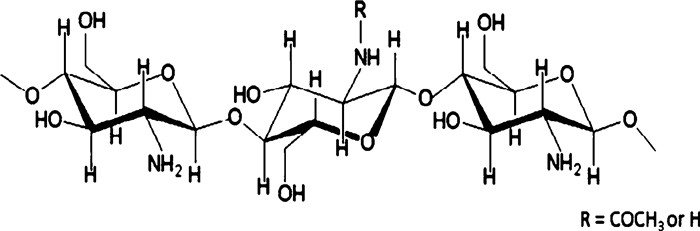 |
| 图 1 壳聚糖结构[2] |
壳聚糖降解获得水溶性降解产物, 降解方法有物理法、化学法和酶法[5]。因为酶法的降解过程温度较低, 且处于pH值近中性的环境中, 故具有更大的优势[7]。1990年国际酶学委员会首次命名壳聚糖酶为壳聚糖N-乙酰氨基壳聚糖水解酶, 2004年补充为壳聚糖酶是以内切方式催化水解部分乙酰化壳聚糖中的β-1, 4氨基葡萄糖苷键的酶;作用于壳聚糖分子, 最终产生寡聚糖终产物。
依据酶的结构特征, 即氨基酸序列分类, 壳聚糖酶分为GH3、GH5、GH7、GH8、GH46、GH75和GH80七种糖苷水解酶家族[8]。目前, 7种家族中的GH46、GH75、G8和GH80的结构已经解析出来。本文将对这四种三维结构已经解析出的壳聚糖酶结构和壳聚体酶作用机制进行阐述, 揭示各种壳聚糖酶结构与作用机制的相互关系及酶解过程进行综述, 揭示了不同壳聚糖酶如何作用于壳聚糖生产活性寡聚糖的微观世界, 为筛选和改造生产新型壳聚糖酶奠定基础, 对于提高酶法生产效率具有重要的意义。
1 GH46壳聚糖酶家族 1.1 GH46壳聚糖家族酶种类与作用GH46家族一般来源于细菌, 主要是杆菌和链霉菌属, 属于II型壳聚糖酶亚类[9-10]。依照蛋白序列的系统发育树分析, GH46家族成员本质上分为A、B、C、D和E五个集群[11]。
GH46家族是典型的壳聚糖内切酶, 在壳聚糖内部剪切β-1, 4连接产生一系列的低聚糖[8]。GH46的ScCsn46A酶降解壳聚糖在酸性环境下得到高收率的低聚物, 包括单体(D), 二聚体(DD或DA), 酶能够切割所有4种糖苷连(D-D, D-A, A-D, A-A);ScCsn46A酶对富含A的壳聚糖降解不广泛, 对富含D的壳聚糖广泛降解;ScCsn46A酶降解壳聚糖程度随着壳聚糖脱乙酰化的降低而降低;ScCsn46A酶对完全脱乙酰化的壳聚糖充分降解后主要得二聚体(DD或DA)和单体(D)[12]。
1.2 GH46壳聚糖家族酶的晶体结构部分GH46家族壳聚糖酶的结构被解析出来, 目前来源于Bacillus circulans MH-K1的Csn(Ch01)[13][PDB编码: 1QGI]、来源于Microbacterium sp.OU01的OU01[14][PDB编码: 4OLT](图 2a)), 来源于Streptomyces sp.N174的Csn174[PDB编码: 1CHK][15]和来源于Streptomyces sp.SirexAA-E的SACTE_5457 [PDB编码: 4ILY]四种酶的晶体结构已经测定出来[16]。
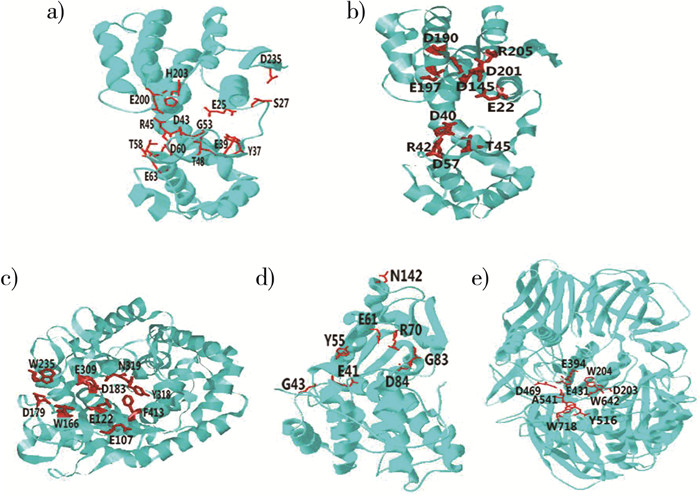 |
| 注: a)GH46家族CsnOU01与(GlcN)6复合体的晶体结构(PDB编码, 4OLT)及关键氨基酸;b)GH46家族来源于Streptomyces sp.N174的Csn174晶体结构(PDB编码: 1CHK)及关键氨基酸;c)GH8家族CsnK17的晶体结构(PDB编码: 1V5D)及关键氨基酸;d)GH80家族ChoK的晶体结构(PDB编码: 5B4S)及关键氨基酸;e)外切-β-氨基葡糖苷酶CsxA与(GlcN)2的复合物结构(PDB编码: 2VZS)及关键氨基酸 图 2 壳聚糖酶晶体结构和关键氨基酸图 |
GH46家族的壳聚糖酶由两个裂片组成, 一个较大的裂片和一个较小的裂片(图 2a)和2b)), 都富含α-螺旋, 两个裂片之间的结合槽可能用于结合壳聚糖链[17]。
1.3 GH46家族酶解作用机制壳聚糖酶两个裂片之间是结合槽, 结合槽打开时, 酶能夹住长链的壳聚糖多糖[17], 两个裂片之间的结合槽从变窄到变宽促成了酶产物的释放[18]。
在GH46壳聚糖酶CsnN174中, 结合槽中的一些带电氨基酸和疏水氨基酸在酶解过程中起到关键作用。其中, 结合槽中心位置的Glu22为糖苷氧原子提供一个质子来剪切β-1, 4连接, 然后一个水分子被Asp40的羧酸根激活后攻击了α侧-1位点上的GlcN残基上的C1位碳原子, 从而完成水解[19]。Glu22, Arg205, Asp145和Arg190的带电侧链组成的相互作用网络维持Glu22的催化效能[20]。CsnN174酶与(GlcN)6残基结合过程中, Arg45与-2位的GlcN的C6的羟基形成了2个氢键, Asp60与-2位的GlcN上的C3的羟基和C2的氨基相作用[9];Asp57, Glu197和Asp201位于酶与底物结合槽的表面, 可能是与糖基相结合的氨基酸[9, 21];Glu197和Asp201与GlcN以高亲和力和低亲和力进行结合[22];Arg42与壳聚糖GlcN残基产生静电相互作用并具有保持催化残基Asp40功能的作用[23];而Glu197突变可能扰动Arg42侧链的正确定向位置从而影响到酶的壳聚糖结合能力[10]。疏水氨基酸Thr45的羟基侧链的氢键可能用于固定催化水分子[24]。
另外, GH46家族某些壳聚糖酶的-2, -1和+1位处与壳聚糖多糖形成的氢键, 对酶与底物结合和酶催化过程起主要作用, 对于酶的催化活性是必须的[8]。
GH46家族其他壳聚糖酶的催化机制和酶-底物相互作用研究也不断深入。GH46家族CsnA壳聚糖酶的螺旋铰链上的E160附近存在一个氢键网络, 由T114, E160和K163之间形成氢键来保持酶的结构稳定性[16]。GH46家族GsCsn46A壳聚糖酶, 有6个底物结合糖苷单元, Glu42(催化酸)和Asp58(催化碱)是两个催化氨基酸, Asn44、Tyr52、Arg60、Thr73、Asp75和Asp259是酶与底物相互作用中的关键氨基酸[8, 17]。GsCsn46A酶的柔性环区域(216RHPANLETEDEW227)位于+3和+2亚位点处, 具有不同的底物结合位点。GsCsn46A酶的芳香氨基酸残基Tyr233与+2位的糖苷基团通过堆积作用形成一个疏水糖结合平面[8]。
2 GH8壳聚糖酶家族 2.1 GH8壳聚糖酶家族的种类与作用GH8壳聚糖酶家族中多个酶的结构被解析出来, 如, 来源于芽孢杆菌菌种Bacillus spec.MN的GH8家族的壳聚糖酶(CSN-MN)、来源于芽孢杆菌菌种Bacillus sp.K17的ChoK酶和来源于热纤梭菌Clostridium thermocellum的CelA酶, 它们属于II型壳聚糖酶, 水解-1到+1位之间的β-1, 4-链接(图 3a))。Liu Ya-Li等克隆表达了革兰氏阳性菌株的GH8家族的壳聚糖酶, 此酶6 h能将约400个残基的壳聚糖链酶解到平均5.1个残基的寡聚糖, 产物范围从2聚糖到9聚糖[25]。
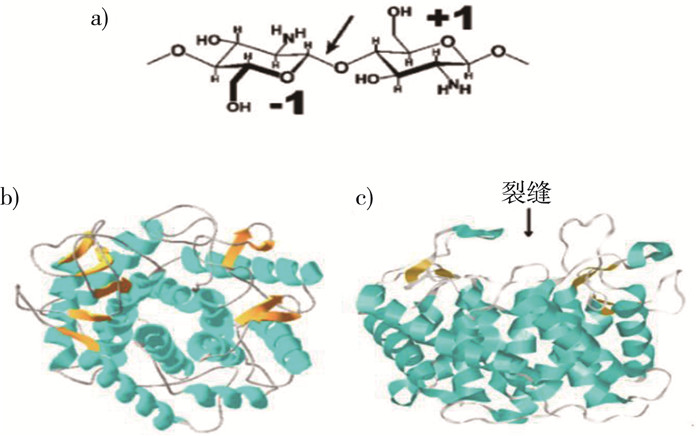 |
| 注: a)GH8壳聚糖酶水解示意图;b)ChoK壳聚糖酶活性状态晶体结构(pH6.4, PDB编码, 1V5D);c)ChoK壳聚糖酶非活性状态晶体结构(pH3.7, PDB编码: 1V5C) 图 3 GH8壳聚糖酶水解示意图和GH8家族ChoK壳聚糖酶的晶体结构图 |
2.2 GH8壳聚糖酶家族的晶体结构
GH8壳聚糖酶具有一个(α/α)6折叠, 由6个重复的螺旋-环-螺旋模式组成, 共12个α螺旋, 类似“双桶”(图 3b))。具有310螺旋和短β-股的长环伸出螺旋外部, 并向螺旋内部折叠, 在“双桶”顶部形成一个“裂缝”, 形成底物结合的口袋(图 3c))[26]。
2.3 GH8壳聚糖酶家族作用机制与结构的关系GH8壳聚糖酶家族与糖基特异性结合机制得到广泛研究。有研究发现CSN-MN酶与糖基之间存在特异性结合作用。CSN-MN酶结合糖基的位点在-3位至+3位, 切割发生-1位和+1位[26]。CSN-MN酶的催化槽是高度电负性的, GlcN具有正电氨基酸基团, 因此酶与GlcN易于结合, 体现了酶的绝对专一性[18]。
研究者进一步研究结合机制发现, CSN-MN酶的空间结构对酶与糖基的特异性结合是必须的, 其作用机制如下: CSN-MN酶的环L1和环L2在-2位点建立了封闭面样的表面[27]。依据两环的距离, 可以将酶分为完全打开、打开和关闭三种状态, 分别可能表示与底物结合处于低亲和状态, 酶与底物结合及产物释放的高亲和状态, 酶的催化构象。L2环上的N270与L1环上的T61、G62和D63在底物和酶相结合时接近, 在-2亚位点L1和L2环接近达到最近距离。-2位的曲面封闭是底物驱动过程, 相互作用发生在L1的D63、L2的N260与质子化的GlcN氨基酸基团之间[27]。
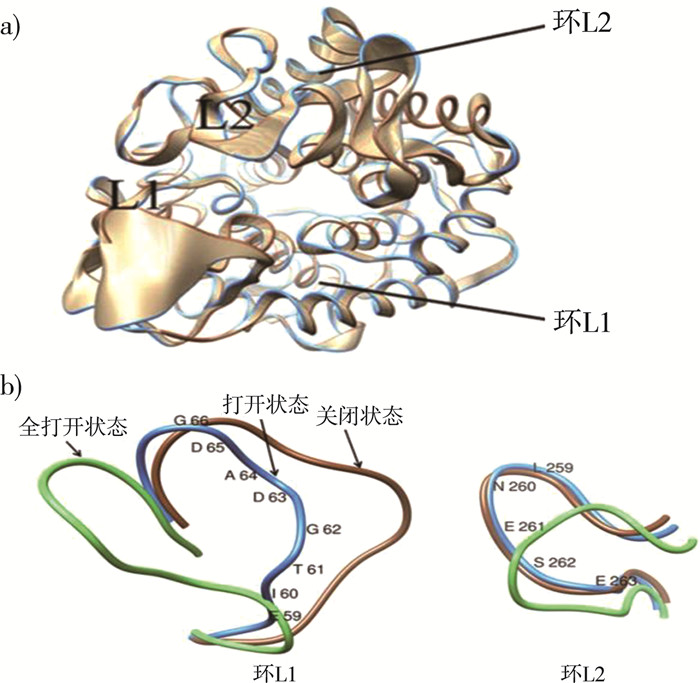 |
| 注: a)CSN-MN酶的环L1和环L2;b)酶与底物结合的三种状态(全打开状态、打开状态和关闭状态)中环L1和环L2的位置 图 4 CSN-MN酶与糖基的特异性结合机制示意图[27] |
又有研究认为GH8壳聚糖酶家族酶解壳聚糖的作用机制是酸碱催化。如, ChoK酶的Glu122位于-1和+1位糖基之间的氧化糖基旁边, 是催化酸, 而Glu309是催化碱。催化水分子位于-1位糖基的C1、Glu309的羧酸根氧原子和Asn319的氨基氧原子形成的三角的中间位置, 活化水分子攻击α侧的-1位糖基的C1位碳原子。ChoK酶的Asn319侧链氨基酸可能用于固定催化水分子[26]。
另, GH8壳聚糖酶家族活性区域中的带电氨基酸和疏水氨基酸对壳聚糖酶的酶解有重要作用。
CSN-MN酶与糖基结合过程中, 在-2位上, 酶的E309与GlcN形成H键相互作用;在-1位和+1位, D183和E107形成H键。ChoK酶的四个酸性氨基酸Asp179, Glu309, Asp183和Glu107, 位于(α/α)6核心结构之上突出的环状结构形成的结合槽中, 分别与-3、-2、-1和+1位的GlcN残基通过静电作用结合[26]。
CSN-MN酶的酸性氨基酸-3位的D131、-2位的D63、-1位的D135、+1位的E59、+2位的E263、+3位的D315和E362与底物通过直接或间接溶解水分子和酶通过氢键相互作用。此外, -1位的S198、-2位的N260、+2位的Y270、-1位的N271和+3位的S363也与底物通过H键相互作用。进一步研究发现, -3位的D131、-1位的D135、-1位的S198、-2位的N260与-1位的N271在酶与质子化的GlcN亚基相互作用中形成最重要的稳定H键。-3至-1位是底物结合的主要位置, 此酶易于结合GlcN[27]。
CSN-MN酶的疏水氨基酸W187、W118、F365和Y270分别呈现在-3、-2、+1和+2位上, 与壳聚糖的糖环存在芳香堆积相互作用, 在壳聚糖分子定位和固定中起到决定性作用[27]。
ChoK酶的疏水氨基酸Trp235、Trp166、Phe413和Tyr318的疏水侧链也存在于结合槽中, 分别与-3、-2、+1和+2位的吡喃环形成面对面的堆积相互作用[26]。这些疏水残基识别Glc和GlcN残基, 可能与GH8壳聚糖酶的广泛特异性有关。Guerin等揭示了与ChoK酶结构同源的来源于Clostridium thermocellum(CelA)的GH8木质纤维素酶的催化机理[28], 与ChoK酶存在类似之处。
3 GH80壳聚糖酶家族 3.1 GH80壳聚糖酶家族的种类目前, GH80壳聚糖酶家族中的ChoA蛋白的重组蛋白McChoA结构已经破解出来, 是破解的GH80壳聚糖酶家族中的唯一一个蛋白, 来源于解壳聚糖松江菌(Mitsuaria chitosanitabida)。
3.2 GH80壳聚糖酶家族的晶体结构McChoA蛋白由311个氨基酸组成, 结构(图 3d)中包含12个α螺旋和5个β股, 蛋白折叠成大小类似的2个部分;McChoA蛋白中包含6个半胱氨酸, 组成了3个二硫键, 分别是Cys56-Cys151、Cys227-Cys235、Cys260-Cys297, 这6个半胱氨酸对酶的催化活性是必需的, 可能对于酶处于合适的稳定的折叠结构状态非常重要。
3.3 GH80壳聚糖酶家族作用机制与结构的关系McChoA蛋白活性区的带电氨基酸、疏水氨基酸等为酶催化功能关键氨基酸, 如, Glu41、Glu61、Gly43、Tyr55、Arg70、Gly83、Asp84和Asn142是与酶的催化功能相关的关键氨基酸(图 2d))。其中, 带电氨基酸Glu41和Glu61是催化残基, 分别位于催化中心口袋的上下两端;Gly43、Tyr55、Arg70、Gly83和Asp84是酶的-2、-1和+1位上进行底物结合和催化的关键氨基酸;位于-2位上的带电氨基酸Arg70和Asp84突变导致酶催化活性丧失;Asn142Ser突变导致酶活性的增强。
4 外切-β-氨基葡糖苷酶 4.1 外切-β-氨基葡糖苷酶的种类与作用外切-β-氨基葡糖苷酶只产生于青霉菌属的菌株中, 与其它的壳聚糖酶不同, 它通过外切机制酶解壳聚糖。外切-β-氨基葡糖苷酶酶解高纯度壳聚糖只产生壳聚糖单体(GlcN)。
4.2 外切-β-氨基葡糖苷酶的晶体结构Van Bueren等报道了来源于Amycolatopsis orientalis的外切-β-氨基葡糖苷酶(CsxA)的晶体结构(图 2e))[29]。CsxA具有GH2催化区域, 这个局域具有一个不完整的(αβ)8桶状区域, 连接了4个具有免疫球蛋白样拓扑结构的β三明治区域。
4.3 外切-β-氨基葡糖苷酶作用机制与结构的关系有研究认为外切-β-氨基葡糖苷酶酶解壳聚糖的作用机制是酸碱催化。如, 研究发现CsxA酶从壳聚糖非还原端外切的方式水解β氨基葡萄糖苷, 催化活性由Asp469(酸/碱)和Glu541(亲核体)完成, Asp469提供质子给糖苷的氧原子从而切割β-1, 4-连接。Asp469的共轭碱从β侧攻击糖基的C1碳激活水分子, 形成的中间体与Glu541侧链形成共价键(制动机制)。如图 2e)显示, CsxA-(GlcN)2复合物的晶体结构表明CsxA的-1位的Asp469(催化酸/碱)周围是由Glu394和Glu541的羧酸基形成的负电子环境, Asp469通过不断地增加它的pKa持续地质子化。-1位GlcN残基的氨基可能促进了pKa循环, 保持了酶高度的催化效力。+1位上, Glu431的侧链羧基与GlcN的氨基基团形成了一个氢键, Tyr516的羟基氧与C6的羟基形成氢键, Trp781与GlcN的吡喃环形成了面对面的堆积相互作用(图 2e))。因为CsxA酶与底物作用时, GlcN上的C2有充分的空间进行其它替代(如GlcNAc), 所以酶的底物特异性较低。
5 总结与展望通过壳聚糖相关酶结构与功能的研究发现, 壳聚糖酶与壳聚糖结合机制与空间结构、氨基酸的带电性和疏水性有关, 大多数壳聚糖相关酶被发现通过酶蛋白的酸性氨基酸(如天冬氨酸和谷氨酸)的负电荷和壳聚糖多糖的氨基酸基团的正电荷之间的静电相互作用进行配体识别[9]。
壳聚糖酶解机制大多以酸碱催化为基础, 酶的活性中心能结合底物, 形成一个“口袋”样空间结构(如图 3), 酶活性中心的带电氨基酸和疏水氨基酸是酶解作用的关键氨基酸。如, 在外切-β-氨基葡萄糖苷酶(CsxA)的底物识别中, 静电相互作用非常重要, 酸性氨基酸E394、E431、D203和D469形成了负电荷区域(图 2e)), GlcN残基的正电荷被发现控制了催化中心的静电环境[29]。GH8壳聚糖酶除了静电作用, 还有部分芳香氨基酸残基(如图 3c的W166和W235)参与了与吡喃酮环的面对面堆积相互作用, 诱导了β-1, 4-葡聚糖的水解活性, 因此, GH8壳聚糖酶除了水解壳聚糖还水解β-1, 4-葡聚糖, 其作用底物范围较广。
壳聚糖酶酶解作用的高效机制也被研究出来, 通常, 壳聚糖酶的壳聚糖结合结构域的结合位点只容纳2个GlcN残基, 主要识别壳聚糖GlcN残基的非还原端。值得注意的是, 2个壳聚糖结合结构域串联, 沿多糖链方向协同进行, 促进了壳聚糖的有效降解。
另外, 有学者研究了壳聚糖酶酶解作用产物的聚糖长短的因素, 发现GH46家族的ScCsn46A酶的酶切程度与壳聚糖的乙酰化相关[9]。
这些重要发现对于我们生产不同寡聚糖应选择哪种壳聚糖酶提供了依据, 为有效利用不同种壳聚糖酶、改造壳聚糖酶和提高寡聚糖生产效率提供了重要的理论基础, 为今后壳聚糖生产行业研发的方向提供了依据。
| [1] |
KUMAR MNVR, MUZZARELLI RAA, MUZZARELLI C, et al. Chitosan chemistry and pharmaceutical perspectives[J]. Chemical Reviews, 2004, 104(12): 6017-6084. DOI:10.1021/cr030441b |
| [2] |
THADATHI N, VELAPPAN SP. Recent developments in chitosanase research and its biotechnological applications: a review[J]. Food Chemistry, 2014, 150: 392-399. DOI:10.1016/j.foodchem.2013.10.083 |
| [3] |
ZHANG J, XIA W, LIU P, et al. Chitosan modigfications and pharmaceutical/biomedical applications[J]. Marine Drugs, 2010, 8(7): 1962-1987. DOI:10.3390/md8071962 |
| [4] |
BRZEZINSKI R. Uncoupling chitosanase production from chitin[J]. Bioeng Bugs, 2011, 2(4): 226-229. DOI:10.4161/bbug.2.4.16111 |
| [5] |
AAM B B, HEGGSET E B, NORBERG A L, et al. Production of chitooligosaccharides and their potential applications in medicine[J]. Marine Drugs, 2010, 8(5): 1482-1517. DOI:10.3390/md8051482 |
| [6] |
JUNG W J, PARK R D. Bioproduction of chitooligosaccharides: present and perspectives[J]. Marine Drugs, 2014, 12(11): 5328-5356. DOI:10.3390/md12115328 |
| [7] |
AKTUGANOV G E, MELENT'EV A I. Specific features of chitosan depolymerization by chitinases, chitosanases, and nonspecific enzymes in the production of bioactive chitooligosaccharides (review)[J]. Applied Biochemistry and Microbiology, 2017, 53(6): 611-627. DOI:10.1134/S0003683817060023 |
| [8] |
QIN Z, CHEN Q, LIN S, et al. Expression and characterization of a novel cold-adapted chitosanase suitable for chitooligosaccharides controllable preparation[J]. Food Chemistry, 2018, 253: 139-147. DOI:10.1016/j.foodchem.2018.01.137 |
| [9] |
SHINYA S, FUKAMIZO T. Interaction between chitosan and its related enzymes: a review[J]. International Journal of Biological Macromolecules, 2017, 104: 1422-1435. DOI:10.1016/j.ijbiomac.2017.02.040 |
| [10] |
EIJSINK V, HOELL I, VAAJE-KOLSTADA G. Structure and function of enzymes acting on chitin and chitosan[J]. Biotechnology & Genetic Engineering Reviews, 2010, 27: 331-366. |
| [11] |
VIENS P, LACOMBE-HARVEY M, BRZEZINSKI R, et al. Chitosanases from family 46 of gly-coside hydrolases: from proteins to phenotypes[J]. Marine Drugs, 2015, 13(11): 6566-6587. DOI:10.3390/md13116566 |
| [12] |
HEGGSET EB, DYBVIK AI, HOELL IA, et al. Degradation of chitosans with a family 46 chitosanase from Streptomyces coelicolor A3(2)[J]. Biomacromolecules, 2010, 11(9): 2487-2497. DOI:10.1021/bm1006745 |
| [13] |
SAITO J, KITA A, HIGUCHHI Y, et al. Crystal structure of chitosanase from Bacillus circulans MH-K1 at 1.6-A resolution and its substrate recognition mechanism[J]. Journal of Biological Chemistry, 1999, 274(43): 30818-30825. DOI:10.1074/jbc.274.43.30818 |
| [14] |
LYU Q, WANG S, XU W, et al. Structural insights into the substrate-binding mechanism for a novel chitosanase[J]. Biochemical Journal, 2014, 461(2): 335-345. DOI:10.1042/BJ20140159 |
| [15] |
FUKAMIZO T, HONDA Y, GOTO S, et al. Reaction mechanism of chitosanase from Streptomyces sp.N174[J]. Biochemical Journal, 1995, 311(Pt 2): 377-383. |
| [16] |
HAN Y J, YU R L, GAO P X, et al. The hydrogen-bond network around Glu160 contributes to the structural stability of chitosanase CsnA from Renibacterium sp QD1[J]. International Journal of Biological Macromolecules, 2018, 109: 880-887. DOI:10.1016/j.ijbiomac.2017.11.071 |
| [17] |
SHINYA S, GHINET MG, BRZEZINSKI R, et al. NMR line shape analysis of a multi-state ligand binding mechanism in chit-osanase[J]. Journal of Biomolecular NMR, 2017, 67(4): 309-319. DOI:10.1007/s10858-017-0109-6 |
| [18] |
LYU Q, SHI Y, WANG S, et al. Structural and biochemical insights into the degradation mechanism of chitosan by chitosanase OU01[J]. Biochimica et Biophysica Acta, 2015, 1850(9): 1953-1961. DOI:10.1016/j.bbagen.2015.06.011 |
| [19] |
MARCOTTE EM, MONZINGO AF, ERNST SR, et al. X-ray structure of an anti-fungal chitosanase from Streptomyces N174[J]. Nature Structural Biology, 1996, 3(2): 155-162. DOI:10.1038/nsb0296-155 |
| [20] |
FUKAMIZO T, JUFFER A-H, VOGEL H-J, et al. Theoretical calculation of pKa reveals an important role of Arg205 in the activity and stability of Streptomyces sp.N174 chitosanase[J]. Journal of Biological Chemistry, 2000, 275(33): 25633-25640. DOI:10.1074/jbc.M002574200 |
| [21] |
TREMBLAY H T, YAMAGUCHI T, FUKAMIZO R, et al. Mechanism of chitosanase-oligosaccharide interaction: subsite structure of Streptomyces sp.N174 chitosanase and the role of Asp57 carboxylate[J]. Journal of Biochemistry, 2001, 130(5): 679-686. DOI:10.1093/oxfordjournals.jbchem.a003034 |
| [22] |
KATSUMI T, LACOMBE-HARVEY ME, TREMBLAY H, et al. Role of acidic amino acid residues in chitooligosaccharide-binding to Streptomyces sp.N174 chitosanase[J]. Biochemical and Biophysical Research Communications, 2005, 338(4): 1839-1844. DOI:10.1016/j.bbrc.2005.10.157 |
| [23] |
LACOMBE-HARVEY M-ō, FUKAMIZO T, GAGNON J, et al. Accessory active site residues of Streptomyces sp.N174 chitosanase: variations on a common theme in the lysozyme superfamily[J]. FEBS Journal, 2009, 276(3): 857-869. DOI:10.1111/j.1742-4658.2008.06830.x |
| [24] |
LACOMBE-HARVEY M ō, FORTIN M, OHNUMA T, et al. A highly conserved arginine residue of the chitosanase from Streptomyces sp.N174 is involved both in catalysis and substrate binding[J]. BMC Biochemistry, 2013, 14: 23. DOI:10.1186/1471-2091-14-23 |
| [25] |
LIU Y L, JIANG S, KE Z M, et al. Recombinant expression of a chitosanase and its application in chitosan oligosaccharide production[J]. Carbohydrate Research, 2009, 344(6): 815-819. DOI:10.1016/j.carres.2009.01.027 |
| [26] |
ADACHI W, SAKIHAMA Y, SHIMIZU S, et al. Crystal structure of family GH-8 chitosanase with subclass II specificity from Bacillus sp.K17[J]. Journal of Molecular Biology, 2000, 343(3): 785-795. |
| [27] |
SINGH R, WEIKERT T, BASA S, et al. Structural and biochemical insight into mode of action and subsite specificity of a chitosan degrading enzyme from Bacillus spec.MN[J]. Scientific Report, 2019, 9(1): 1132. DOI:10.1038/s41598-018-36213-6 |
| [28] |
GUERIN D M, LASCOMBE M B, COSTABEL M, et al. Atomic resolution structure of an inverting glycosidase in complex with substrate[J]. Journal of Molecular Biology, 2002, 316(5): 1061-1069. DOI:10.1006/jmbi.2001.5404 |
| [29] |
VAN BUEREN A L, GHINET M G, GREGG K, et al. The structural basis of substrate recognition in an exo-D-glucosaminidase involved in chitosan hydrolysis[J]. Journal of Molecular Biology, 2009, 385(1): 131-139. DOI:10.1016/j.jmb.2008.10.031 |
 2021, Vol. 35
2021, Vol. 35


