| 沙门氏菌跨物种感染基因的挖掘及鉴定 |
沙门氏菌是一种重要的食源性致病菌, 其致病性研究对维护公共卫生安全具有重要意义[1]。我国约有70起食源性病原微生物中毒事故受沙门氏菌感染, 每年感染200多万人[2], 造成不可估量的经济损失。
沙门氏菌属于肠杆菌科, 为革兰氏阴性短杆菌。目前全世界已分离出2 543个血清型, 其中中国发现了287个血清型[3]。沙门氏菌有两个种, 即肠道沙门氏菌和邦戈尔沙门氏菌。现在已知的沙门氏菌有人类致病菌、动物致病菌, 也有对人和动物都致病的细菌(人兽共患沙门氏菌)。吃沙门氏菌感染的食物会导致食物中毒。沙门氏菌在自然界中分布广泛, 是引起食物中毒的重要致病菌。它很容易在没有中间宿主参与的情况下通过直接或间接途径传播给人类或动物。感染后, 沙门氏菌可引起三种疾病, 即肠热、败血症和肠胃炎[4]。
传统沙门氏菌检测方法存在操作复杂、检测时间长、结果误差大等缺点[5]。因此, 迫切需要建立一种快速、简便、准确的检测沙门氏菌中致人兽共患病基因的方法, 为食品生产的各个环节加强食源性微生物的检测提供方法依据, 确保食品质量安全[6]。
1 材料与方法 1.1 实验菌株肠炎沙门氏菌菌株30株。
1.2 实验试剂LB营养肉汤培养基, 沙门氏菌显色培养, 缓冲蛋白胨水(BPW), 亚硒酸盐胱氨酸(SC)增菌液, 细菌基因组提取试剂盒(TIA Namp bacteria DNA Kit离心柱型), PCR试剂, TAE缓冲液。
1.3 研究方法 1.3.1 全基因组测序及序列比对分析使用离心柱法对30株不同来源的沙门氏菌进行基因组DNA提取, 将提取的30株沙门氏菌的全基因组使用Roche 454焦磷酸测序进行全基因组DNA的测定, 并将测序片段使用SOAP、Spades、Abyss软件进行组装, 并使用CISA软件进行整合, 最终得到30株菌的全基因组DNA序列信息。
使用Ridom SeqspHere+软件(生物信息学软件)进行cgMLST分析, 肠炎沙门氏菌菌株P125109(GenBank登录号NC 011294)的基因组用作参照基因组[7]。然后使用肠炎沙门氏菌菌株EC20121175(GenBank登录号CP-007269.2)的基因组与参考基因组进行比较以建立核心和辅助基因组基因的列表。鉴定试验菌株之间的所有具有等位基因差异的基因[8]。然后通过在参考基因组中搜索特定序列并验证每个密码子中的等位基因变化来对每个等位基因表征多态性。最后, 使用了中国医学科学院病原生物学研究所、北京协和医学院(北京, 中国)的致病菌毒力因子数据库, 以确定预测的人兽共患病致病基因。
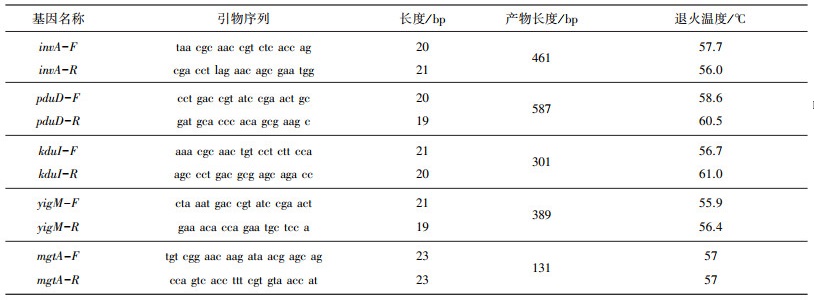
1.3.2 PCR引物设计根据五个预测的人兽共患病致病性基因(invA、pduD、kduI、yigM、mgtA), 在GeneBank数据库中搜索相同物种的五个预测致病性基的DNA序列。把结果进行对比找到同源性高的保守区域的致病基因的扩增靶序列, 并将其从NCBI网站下载下来, 运用Primer Premier 5.0进行引物设计, 用Primer Premier 5.0软件根据引物设计原则, 设计5对引物, 根据多重PCR引物设计原则, 设计的引物产物长度之间相差都不低于50 bp, 引物序列见表 2。设计相关引物并进行验证。
| 表 2 引物的详细信息s |
 |
1.3.3 PCR扩增条件优化
反应采用25 μL体系, 在0.2 mL离心管中进行。以沙门氏菌菌株G1为4个基因阳性菌株, 参考表 2引物的标准退火温度依次对每个基因的退火温度进行梯度设置。
1.3.4 沙门氏菌的培养鉴定针对沙门氏菌扩增培养、分离过程, 以国家卫生学检验标准(GB/T 4789.4—2008)为参考。先在离心管内吸取5 mL灭菌的生理盐水, 然后加入沙门氏菌样品, 室温静置30 min;涡旋振荡使样品均匀混合后吸取出1 mL混合的生理盐水转入50 mL BPW中, 36 ℃水浴10 h。再取出1 mL的BPW培养物, 转入盛有10 mL的SC的锥形瓶中, 42 ℃培养18~24 h[9]。用已灭菌的接种环蘸取菌液, 通过三区划线法来把细菌接种于沙门氏菌显色培养基上, 于36 ℃培养42 h后观察结果。
1.3.5 多重PCR扩增PCR扩增体系(25 μL): 根据多次单重PCR摸索成功的扩增体系, 在各个基因单重PCR的条件范围内, 筛选出多重PCR的扩增体系, 通过调节各引物的浓度比、PCR Buffer浓度、dNTP浓度建立检测这5种致病基因的多重PCR。PCR产物进行凝胶电泳检测。
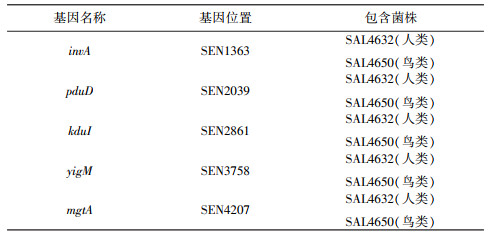
2 结果与分析 2.1 人兽共患基因的预测结果通过对鸡蛋和病人中分离的30种肠炎沙门氏菌菌株进行全基因组测序然后用软件Ridom SeqspHere +进行cgMLST基因组分析, 预测invA、pduD、kduI、yigM、mgtA这5个致病基因为沙门氏菌的人兽共患病基因(表 1)。
| 表 1 基因信息 |
 |
2.2 PCR引物设计结果
通过表 2看出, 设计的引物是为了后续做五重PCR的引物, 因此设计的产物长度之间相差不低于50 bp, 相差最小的为88 bp, 退火温度在55.9~60.0 ℃之间, BLAST比对同物种同源性较高, 有很好的特异性。
2.3 沙门氏菌的培养鉴定结果图 1是沙门氏菌菌株G1显色培养基三区划线法培养24 h后的结果, 平板上出现肉眼可见的紫红色菌落, 扁平透明, 表面光滑, 直径约2 mm, 初步判定培养出的菌落是沙门氏菌菌落, 其他菌株的鉴定结果也均是肉眼可见的品红色菌落。
 |
| 图 1 G1菌株培养24 h结果 |
2.4 PCR扩增条件优化结果
根据各引物单重PCR所建立的扩增条件和参数范围, 最后优化出一个适合五个引物的五重PCR方法的条件, 建立的五重PCR方法为: 25 μL的扩增体系在0.2 mL离心管中加入, 依次加入以下组分: 15 μL ddH2O(双蒸水);2.5 μL 10× PCR Buffer(含Mg2+);dNTP 2 μL;invA-F/R各0.25 μL;kduI-F/R各0.4 μL;pduD-F/R各0.25 μL;yigM-F/R各0.5 μL;mgtA-F/R各0.25 μL;Taq酶0.5 μL;DNA模板2 μL。短暂离心后立即PCR。
PCR扩增条件为: 94 ℃预变性5 min; 94 ℃变性30 s, 62 ℃退火30 s; 72 ℃延伸1 min, 30个循环, 最后72 ℃延伸5 min。PCR扩增完毕, 取5 μL PCR产物进行1.5%琼脂糖凝胶电泳25 min, 然后在凝胶成像系统下拍照记录结果(见图 2)。
 |
| 图 2 invA、pduD、kduI、yigM、mgtA电泳图 |
利用上面建立的五重PCR扩增体系对同时含有五种致病基因的沙门氏菌G1阳性菌进行PCR检测, PCR扩增产物的电泳图如图 2所示。没有出现杂条带, 扩增的目的条带大小跟引物长度大小一致, 从下到上依次是mgtA 131 bp、kduI 301 bp、yigM 389 bp、invA 461 bp、pduD 587 bp。说明建立的多重PCR特异性良好。
3 结论沙门氏菌被认为是一种新兴的食源性人兽共患病病原体, 有证据表明商业家禽产品与人类疾病之间存在明显的流行病学联系[8]。现在沙门氏菌已经成为公共卫生影响广泛的病原体, 家禽业和公共卫生服务部门都对这种病原体进行了积极的监测。本实验在查阅国内外文献的基础上, 通过实验进一步验证沙门氏菌的人兽共患病基因, 建立了检测沙门氏菌人兽共患病基因的五重PCR方法, 与常规PCR方法的符合率达100%, 本实验建立的五重PCR方法操作不仅简单快速省时省力, 在敏感性和特异性方面与标准PCR几乎没有差异, 可以同时对多个菌株的不同致病基因进行检测和鉴定, 该套方法能对沙门氏菌的致病性从遗传基因的角度进行检测, 为以后的沙门氏菌人兽共患病靶基因的检测和溯源提供依据和参考资料, 可用于沙门氏菌致病基因的快速检测和不同人兽共患病基因流行病学调查, 为指导沙门氏菌人兽共患病的研究提供辅助技术, 在科研方面具有实用性和研究价值[10]。
| [1] |
肖茜文, 王艳蕊, 刘桐, 等. 沙门氏菌和金黄色葡萄球菌DNA提取方法的比较[J]. 农产品加工, 2017(3): 48-50. |
| [2] |
TEERARAT P, CHOKESAJJAWATEE N, SANTIYANONT P, et al. Quantification and rep-PCR characterization of Salmonella spp.in retail meats and hospital patients in Northern Thailand[J]. Zoonoses and public health, 2019, 66(3): 301-309. DOI:10.1111/zph.12565 |
| [3] |
KHALTABADI R F, SHAHROKHI N, MINA E R, et al. Salmonella TypHimurium in Iran: Contribution of molecular and IS200 PCR methods in variants detection[J]. Plos one, 2019, 14(3). |
| [4] |
郭龙宗. 种鸡场沙门氏菌感染状况的调查与防控[D]. 泰安: 山东农业大学, 2017.
|
| [5] |
张萍, 冯芳. 沙门氏菌的检测技术和方法的研究进展[J]. 食品安全质量检测学报, 2015, 6(5): 1834-1841. |
| [6] |
ZHAO J X, SONG Q F, LI C H, et al. Characterization of ciprofloxacin-resi-stant and ESBL-producing Salmonell-enteric serotype Derby in Eastern China[J]. BMC Microbiology, 2019, 19(61): 2-6. |
| [7] |
刘斌. 沙门氏菌血清分型分子靶点的发掘及鉴定体系的建立[D]. 上海: 上海交通大学, 2012.
|
| [8] |
翟立公. 沙门氏菌及其致病性血清型分子检测技术研究及在食品中的应用[D]. 南京: 南京农业大学, 2015.
|
| [9] |
ITACIR F, MEDEIROS L, CARVALHO Y, et al. The urban and rural capybaras (Hydrochoerus hydrochaeris) as reserveoir of Salmonella in the western Amazon, Brazil[J]. Pesquisa veterinarya brasileira, 2019, 39(1): 66-69. DOI:10.1590/1678-5150-pvb-5761 |
| [10] |
申志勇. PCR引物特异性评估体系及多重PCR引物设计系统的构建与应用[D]. 北京: 中国人民解放军军事医学科学院, 2009.
|
 2022, Vol. 36
2022, Vol. 36


