| 柠檬酸杆菌基因组多位点序列分型(in silico MLST)研究 |
2. 济南市历下区明德中学, 山东 济南 250000
2. Jinan Mingde Middle School, Jinan 250000, China
柠檬酸杆菌(Citrobacter)为革兰氏阴性杆菌, 肠杆菌科柠檬酸属, 兼性厌氧, 生长能力强, 在自然界中分布广泛, 如土壤、污水及食物中, 此外动物肠道中也发现有它们的存在。柠檬酸杆菌属目前共含有18个种, 其中弗氏柠檬酸杆菌是柠檬酸杆菌属最早发现的一个种[1]。近年来, 已经有许多关于动物感染柠檬酸杆菌的报道, 如布氏柠檬酸杆菌可引起菌血症[2]; 人和动物可因弗氏柠檬酸杆菌感染而引发腹泻、败血症、食物中毒。1982年, Sato等[3]从发病的翻车鲀上首次分离到弗氏柠檬酸杆菌, 随后相继在发病的鲑鱼、虹鳟、斑马鱼、鲟鱼、中华鲟、狗和巴西龟等动物中分离发现弗氏柠檬酸杆菌[4]。柠檬酸杆菌作为一种条件致病菌严重威胁到人类的生命健康且容易造成水产、畜禽养殖业病疫的发生。
全基因组测序技术是以研究核苷酸序列为基础的新兴技术方法, 该技术可与生物信息学工具结合后, 提供了更全面的关于病原体基因型特征的信息与多种检测的可能性, 例如抗生素耐药性和毒力的表征以及系统发育分析等[5-8]。多位点序列分型(Multilocus Sequence Typing, MLST)是一种通过测定多个管家基因的序列来进行分型的方法[9]。MLST通常选取7~10个管家基因来进行测序。被广泛应用于细菌的分型研究中。该方法具有分辨率高、数据可靠、重复性好、可比性好等特点, 对细菌溯源具有重要意义[10]。目前, 已经广泛应用于细菌的分型研究中。
通过全基因组测序技术对4株疑似柠檬酸杆菌进行了测序获得全基因组序列, 在此基础上通过生信分析手段对4株菌进行16S rRNA分析和多位点序列分型, 从而进一步确认其物种信息以及与数据库中现存的柠檬酸杆菌菌株的分子进化关系, 为柠檬酸杆菌防控及流行病学调查提供技术支持。
1 材料与方法 1.1 菌株与试剂试验用的4株柠檬酸菌株为齐鲁工业大学微生物与免疫研究所自行分离、鉴定及保藏的菌株。菌株的背景信息详见表 1。试验在齐鲁工业大学微生物与免疫实验室, 于2019年9月至2022年9月份进行。
| 表 1 本研究使用的柠檬酸杆菌信息 |
 |
1.1.1 主要试剂
Typtone、Yeast Extract, 英国OXOID公司; 氯化钠、磷酸氢二钠、磷酸二氢钠, 国药集团化学试剂有限公司; Agar Powder, Solarbio公司; 细菌基因组提取试剂盒, 天根生化科技(北京)有限公司; 营养肉汤, 北京陆桥技术责任有限公司。
1.1.2 主要仪器恒温培养振荡器ZWY-2102C, 上海智城分析仪器制造有限公司生产; 超净工作台SW-CJ-2F, 苏州安泰空气技术有限公司生产; 恒温生物培养箱HH-B11·420-BS, 上海跃进医疗器械有限公司; 超微量紫外分光光度计MD2000H, Biofuture公司; 台式高速离心机TGL-16B, 上海安亭科学仪器厂。
1.2 细菌培养首先取出保藏在超低温冰箱中的试验菌株冻存管, 于室温下解冻, 然后在无菌环境下, 先用缓冲蛋白胨水(BPW)进行预增菌, 吸取200 μL菌液转接到提前灭菌好的BPW中, 37 ℃培养12~18 h。对预增菌后的菌株在LB固体培养基上进行划线培养, 挑取生长良好的菌落到加到有1 mL无菌营养肉汤的离心管中, 并放置于培养箱中过夜培养, 温度设定为37 ℃。
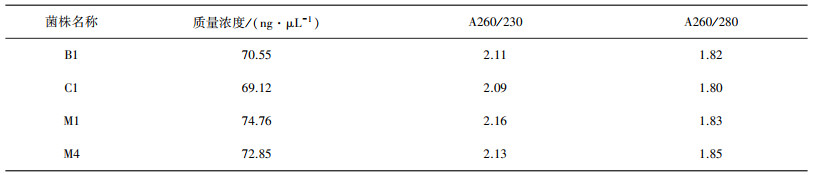
1.3 柠檬酸杆菌基因组DNA的提取使用细菌基因组提取试剂盒(TIANGEN, DP302-02)分别提取基因组DNA。使用MD2000超微量核酸分析仪检测提取DNA的浓度、A260/280以及A260/230, 保证提取的基因组质量浓度≥60 ng/μL、A260/280≥1.8和A260/230≥2.0。
1.4 细菌基因组测序提取的基因组样本使用北京诺禾致源公司的Illumina Novaseq PE250平台进行测序, 进行2×250个循环, 覆盖深度为100~260倍, 使用readfq软件进行预处理后获得Clean Data, 使用SOAP denovo(Version 2.04)[11]、SPAdes[12]、ABySS[13]等组装软件对获得的数据进行组装, 使用CISA[14]软件对上述组装结果进行整合; 然后采用gapclose(Version 1.12)[15]软件处理组装结果中的gap, 从而得到最终的组装结果; 最后对基因组序列进行评估和统计分析, 得到4株柠檬酸杆菌的全基因组序列信息。
1.5 基于16S rRNA的物种鉴定NT核酸序列数据库(Nucleotide Sequence Database)是目前最全的物种基因信息数据库, 数据库内存放已知物种的DNA序列信息和全部的人类DNA信息。对组装的沙门氏菌基因组DNA使用rRNAmmer软件(Version 1.2)[16]进行rRNA预测, 将预测得到的16S rRNA基因序列使用BLASTn在NT数据库进行比对, 以序列相似度97%为最低标准, 获取每个菌株最佳匹配物种信息。
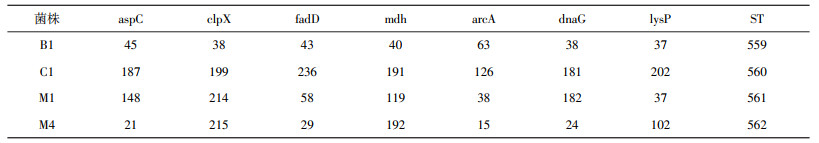
1.6 柠檬酸杆菌基因组MLST(in silico MLST)分型研究对于实验菌的MLST分析, 参照柠檬酸杆菌多位点序列分型数据库的分型方案(http://pubmlst.org/cfreundii/), 选用7个等位基因(aspC, clpX, fadD, mdh, arcA, dnaG, lysP)进行分析, 在PubMLST数据库中提供了基因组上传进行MLST分析的途径, 将4株柠檬酸杆菌的基因组上传至PubMLST数据库中进行7个管家基因等位基因的比对, 最终得到菌株的序列型。
1.7 基于基因组MLST的柠檬酸杆菌的遗传进化分析通过软件Ridom Seqsphere+ v7.2.3(Ridom GmbH, Germany)对菌株的MLST分型数据进行建树分析[17]。在4株柠檬酸杆菌的基础上, 添加了两株人源的柠檬酸杆菌: Cifr 10955(PubMLST ID: 427)和IR5464(PubMLST ID: 463)。选用Ridom Seqsphere+提供的邻居加入树(neighbor joining tree, NJT), NJT建树方法参照Saitou建立的算法[18]。在进行NJT建树分析时候, 只保留7个等位基因皆成功分型的菌株信息。全部4株菌的7个管家基因皆分型成功。
2 结果与分析 2.1 柠檬酸杆菌基因组提取如表 2所示, 为4株柠檬酸杆菌基因组DNA MD 2000检测结果。结果显示, 4株柠檬酸杆菌的基因组质量浓度均在70 ng/μL左右, A260/A230均在2.1左右, A260/280均在1.8左右, 符合基因组质量浓度大于60 ng/μL、A260/A230的比值大于2.0、A260/A280大于1.8的DNA样品标准, 可以进行测序。
| 表 2 4株柠檬酸杆菌基因组纯度检测结果 |
 |
2.2 柠檬酸杆菌基因组分析
获得基因组组装结果后, 统计其基因组大小、基因数量、基因总长度、平均长度等信息, 具体如表 3所示。4株柠檬酸杆菌的基因组大小在4.7 ~5.5 Mb之间, 平均基因组大小为5.0 Mb。4株柠檬酸杆菌基因组中包含的平均基因数量为4 871, 每株菌所包含的基因数量在4 591~5 334之间。基因的平均长度均在900 bp左右。此外, 基因长度占基因组的比例均在87%左右。
| 表 3 4株柠檬酸杆菌基因组组装结果 |
 |
2.3 柠檬酸杆菌16S rRNA分析结果
使用rRNAmmer软件对组装的菌株序列进行预测、调取16S rRNA的基因序列, 在NCBI网站中进行BLASTx比对, 结果表明, 4株菌在16S rRNA水平鉴定为柠檬酸杆菌。其中B1、C1、M1与布雷氏柠檬酸杆菌同源性香近, M4与沃克曼氏柠檬酸杆菌同源性相近。
2.4 柠檬酸杆菌基因组MLST分型分析结果将4株菌的全基因组序列上传至PubMLST数据库进行分型比对, 4株柠檬酸杆菌的基因组MLST分型结果见表 4。分析结果表明, 4株菌的序列型(ST)分别为B1(ST-559)、C1(ST-560)、M1(ST-561)、M4(ST-562), 4株菌中, B1、C1和M1虽同属于布雷氏柠檬酸杆菌, 但它们的7个管家基因的等位基因赋值完全不同, 同时M4与另外3株菌的7个管家基因等位基因赋值也不相同。此外, 通过与PubMLST数据库中的数据进行比较, 发现这4个ST均为新ST, 此前从未被报道。
| 表 4 4株柠檬酸杆菌多位点序列分型结果 |
 |
2.5 基于基因组MLST的柠檬酸杆菌的遗传进化分析
基于基因组MLST分型结果, 对柠檬酸杆菌构建Neighbor-joining系统进化树, 结果见图 1。分析结果表明, 菌株M1与Cifr10955分离株(ST-568)亲缘关系最近, 它们在arcA位点的等位基因数值均为38, 其余的菌株未发现有亲缘关系较近的分离株。
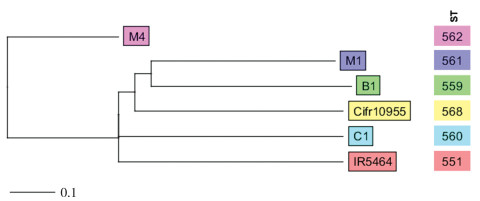 |
| 图 1 4株菌与PubMLST中国上传的柠檬酸杆菌MLST分型结果邻居加入树(NJT) |
3 结论与讨论
柠檬酸杆菌作为一种常见的细菌, 国内外对分型做过很多研究。Mansour Rezaei等对从医院采集得到的50株柠檬酸杆菌分离株通过药敏试验和超广谱-β内酰胺酶(ESBL)筛选后, 用脉冲场凝胶电泳(PFGE)进行基因分型, 结果显示, 12株(24%)ESBL阳性菌株和38株(76%)ESBL阳性菌株的PFGE结果分别产生了39个不同基因型类型的聚类群, 其中有些不同病房的分离株在同一个聚类群中, 可能反映出菌株在医院病房内的传播[19]。叶明亮等[20]首次利用116株弗氏柠檬酸杆菌分离株建立了弗氏柠檬酸杆菌的随机扩增多态性DNA法基因分型图谱, 结果显示, 116株菌被分成78种RAPD型, 分型结果明显。
柠檬酸杆菌是一种常见的人与动物共患的疾病, 通常会引起腹泻等肠道疾病。其在感染婴儿、老年人等免疫力低下人群后易引起菌血症、脑膜炎等严重疾病甚至死亡[21]。此外, 柠檬酸杆菌的感染也会对养殖业产生巨大的影响[22]。因此, 提高对柠檬酸杆菌的溯源与分型水平, 有利于柠檬酸杆菌的防控, 减少因柠檬酸杆菌的感染而造成的经济损失。
由于16S rRNA序列具有高度保守、不易变异等优点, 被广泛应用于细菌分类学研究中[23]。目前NCBI和EBI数据库中储存了大量物种的16S rRNA序列, 通过BLST比对可快速准确的找到未知细菌的16S rRNA同源序列, 并确定其在细菌分类学中的地位。通过16S rRNA分析, 本研究中的4株菌均被确定为柠檬酸杆菌, 其中B1、C1、M1被确定为布雷氏柠檬酸杆菌, M4被确定为沃克曼氏柠檬酸杆菌。
细菌分型是细菌溯源、细菌鉴定、流行病学调查以及食品安全监管的重要手段, 对食品安全、公共卫生具有重要意义。随着基因测序技术的发展, 具有准确、快速并且重复性好、分辨力高等特点的分子分型技术已被广泛应用于致病菌的鉴定、溯源、系统演化分析等多个方面[24]。MLST是一种常用的分子分型方法, 根据不同的菌株中管家基因序列的不同, 对菌株赋予序列型(ST)。目前, 已有Enterobase、PubMLST等公共数据库用于分析和储存细菌的分型信息。柠檬酸杆菌常用的MLST的方案通常为7个位点, 包括aspC, clpX, fadD, mdh, arcA, dnaG和lysP。本研究中的4株柠檬酸杆菌被分为了4个不同的ST(ST559、ST560、ST561、ST562)。通过与PubMLST数据库中的数据进行比对, 发现这4个ST型此前未被报道过, 均为新的ST型。MLST由于具有操作简单、结果可靠、具有较为成熟的分析数据库、可进行标准化等优点, 使细菌分型信息得以在不同的实验室之间交流传播[25-26]。此外, 与脉冲场凝胶电泳(PFGE)等传统的分子分型方法相比, MLST分型的结果具有准确性高、可重复等优点。本研究对4株柠檬酸杆菌进行了全基因组测序, 并采用PubMLST数据库中现有的柠檬酸杆菌属的多位点序列分型方案进行了in silico MLST分析。虽然, 全基因组测序的方法与传统的PCR扩增7个管家基因的方法相比所花费的时间和成本更高, 但全基因组测序可以获得大量的基因序列, 并为后续对菌株进行致病性与耐药性等分析打下基础。
综上所述, 通过16S rRNA分析将从鸡种分离得到的4株菌鉴定为柠檬酸杆菌, 其中B1、C1、M1被确定为布雷氏柠檬酸杆菌, M4被确定为沃克曼氏柠檬酸杆菌。通过in silico MLST分析, 获得了4个新的ST型。有关柠檬酸杆菌感染的报道多集中于水生生物, 在禽类中的报道相对较少。本研究所获得的数据丰富了我国柠檬酸杆菌的遗传信息的多样性, 有利于柠檬酸杆菌的检测、流行病学调查和防控。由于选取的菌株数量有限, 无法更好的探究柠檬酸杆菌的遗传进化规律以及跨物种间的致病机理。因此, 希望后续的研究可以通过对大量的柠檬酸杆菌进行分析, 以探究柠檬酸杆菌的耐药机制和致病机制等。
| [1] |
何晓青. 柠檬酸杆菌属(Citrobacter)的分类与鉴定[J]. 中国卫生检验杂志, 2005, 15(12): 1535-1536. |
| [2] |
HIRAI J, UECHI K, HAGIHARA M, et al. Bacteremia due to Citrobacter braakii: a case report and literature review[J]. Journal of Infection and Chemotherapy, 2016, 22(12): 819-821. DOI:10.1016/j.jiac.2016.07.003 |
| [3] |
SATO N, YAMANE N, KAWAMURA T. Systemic Citrobacter freundii Infection among Sunfish Mola mola in Matsushima Aquarium[J]. Nihon Suisan Gakkaishi, 1982, 48(11): 1551-1557. DOI:10.2331/suisan.48.1551 |
| [4] |
张明洋, 胡安东, 程振涛, 等. 虹鳟弗氏柠檬酸杆菌的分离鉴定与多位点序列分型[J]. 基因组学与应用生物学, 2020(6): 2521-2528. |
| [5] |
ALLARD M W, BELL R, FERREIRA C M, et al. Genomics of foodborne pathogens for microbial food safety[J]. Current Opinion in Biotechnology, 2018, 49: 224-229. DOI:10.1016/j.copbio.2017.11.002 |
| [6] |
PETTENGILL J B, TATE H, GENSHEIMER K, et al. Distribution of antimicrobial resistance genes across salmonella enterica isolates from animal and nonanimal foods[J]. Journal of Food Protection, 2020, 83(2): 295-304. DOI:10.4315/0362-028X.JFP-19-310 |
| [7] |
MCDERMOTT P F, TYSON G H, KABERA C, et al. Whole-genome sequencing for detecting antimicrobial resistance in nontyphoidal Salmonella[J]. Antimicrobial Agents and Chemotherapy, 2016, 60(9): 5515-5520. DOI:10.1128/AAC.01030-16 |
| [8] |
TYSON G H, MCDERMOTT P F, LI C, et al. WGS accurately predicts antimicrobial resistance in Escherichia coli[J]. Journal of Antimicrobial Chemotherapy, 2015, 70(10): 2763-2769. DOI:10.1093/jac/dkv186 |
| [9] |
JAUREGUY F, LANDRAUD L, PASSET V, et al. Phylogenetic and genomic diversity of human bacteremic Escherichia coli strains[J]. BMCGenomics, 2008, 9(1): 1-14. |
| [10] |
TURNER K M E, FEIL E J. The secret life of the multilocus sequence type[J]. InternationalJournal of Antimicrobial Agents, 2007, 29(2): 129-135. DOI:10.1016/j.ijantimicag.2006.11.002 |
| [11] |
LI R, LI Y, KRISTIANSEN K, et al. SOAP: short oligonucleotide alignment program[J]. Bioinformatics, 2008, 24(5): 713-4. DOI:10.1093/bioinformatics/btn025 |
| [12] |
BANKEVICH A, NURK S, ANTIPOV D, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing[J]. J Comput Biol, 2012, 19(5): 455-77. DOI:10.1089/cmb.2012.0021 |
| [13] |
SIMPSON J T, WONG K, JACKMAN S D, et al. ABySS: a parallel assembler for short read sequence data[J]. Genome Res, 2009, 19(6): 17-23. |
| [14] |
LIN SH, LIAO YC. CISA: contig integrator for sequence assembly of bacterial genomes[J]. PLoS One, 2013, 8(3): 843. |
| [15] |
LUO R, LIU B, XIE Y, et al. SOAP denovo2:an empirically improved memory-efficient short-read de novo assembler[J]. Gigascience, 2012, 1(1): 18. DOI:10.1186/2047-217X-1-18 |
| [16] |
LAGESEN K, HALLIN P, RøDLAND E A, et al. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [17] |
JÜNEMANN S, SEDLAZECK F J, PRIOR K, et al. Updating benchtop sequencing performance comparison[J]. NatureBiotechnology, Nature Publishing Group, 2013, 31(4): 294-296. |
| [18] |
SAITOU N, NEI M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. MolecularBiology and Evolution, 1987, 4(4): 406-425. |
| [19] |
REZAEI M, AKYA A, ELAHI A, et al. The clonal relationship among the Citrobacter freundii isolated from the main hospital in Kermanshah, west of Iran[J]. Iranian Journal of Microbiology, 2016, 8(3): 175-180. |
| [20] |
叶明亮, 黄象艳, 吕波, 等. 弗氏柠檬酸杆菌随机扩增多态性DNA法基因分型[J]. 中华医院感染学杂志, 2005(10): 32-34. |
| [21] |
闫革彬, 隋吉林, 芦丹, 等. 一起弗氏柠檬酸杆菌所致食源性疾病调查[J]. 疾病监测, 2013, 28(3): 247-248. |
| [22] |
吕爱军, 胡秀彩, 朱静榕, 等. 弗氏柠檬酸杆菌感染诱导斑马鱼皮肤免疫相关基因的差异表达[J]. 水产学报, 2012, 36(3): 359-366. |
| [23] |
努尔夏提·努尔买买提, 李焕宇, 杨成德. 辣椒根腐病菌拮抗链霉菌的筛选及16S rRNA基因系统发育分析[J]. 中国农学通报, 2022, 38(33): 116-123. |
| [24] |
SANKARASUBRAMANIAN J, VISHNU U S, GUNASEKARAN P, et al. Development and evaluation of a core genome multilocus sequence typing (cgMLST) scheme for Brucella spp[J]. Infect Genet Evol, 2019, 67: 38-43. |
| [25] |
刘佳妍, 金莉莉, 王秋雨. 细菌基因组重复序列PCR技术及其应用[J]. 微生物学杂志, 2006(3): 94-97. |
| [26] |
刘金华, 贺丹, 杨艳秋, 等. 多位点测序分型技术在病原微生物分型鉴定中的应用[J]. 微生物学通报, 2007, 34(6): 1188-1191. |
 2023, Vol. 37
2023, Vol. 37


