| 基因编辑技术及其在生物医学领域应用的研究进展 |
基因编辑技术可以完成目标基因的基因敲除和替换,能够对基因序列进行修饰。它是近年来发展出来的一种编辑工具。随着基因组测序的实现,基因功能的研究已成为基因时代关注的焦点。基因编辑技术能够修改基因及调控不同类型的细胞基因以研究基因功能,能够产生不同的CAG重复序列等的基因细胞系,治疗突变引起的疾病。与传统的基因靶向技术比较,基因编辑技术拥有不会限制ES细胞,而且能够适用于更多的物种等优点。在生物医学领域,基因编辑技术将有助于进一步探讨疾病的发病机制,更准确的定义基因功能,以达到精确医学的目的。基因编辑技术有着非常广阔的发展前景和应用价值。
1 基因编辑技术基因编辑技术已经得到了广泛应用,促进了靶位点的基因敲除及敲入,据此进一步用网络和调控基因序列的功能,创建遗传性疾病的模型,制定相关基因治疗方法。介绍基因编辑工具的组成部分以及优缺点。
1.1 ZFN技术ZFN(锌指核酶)作为嵌合核酸酶由两部分组成[1],第一部分是锌指结构域的DNA结合盒,每个锌指蛋白结合3个核苷酸,每个亚基提供足够的特异性位点特异性结合DNA。研究员专门对靶基因组构建了八至十个锌指结构域[2]。第二部分是FokⅠ核酸内切酶结构域,其可以使紧密位于ZFN亚基二聚体的双链断裂[3],ZFNs技术靶向切割DNA如图 1所示。kim等人[4]于1996年构建出了首个锌指核酸内切酶,从这项研究中发现人工构建的ZFN具备特定识别切割位点的特点。
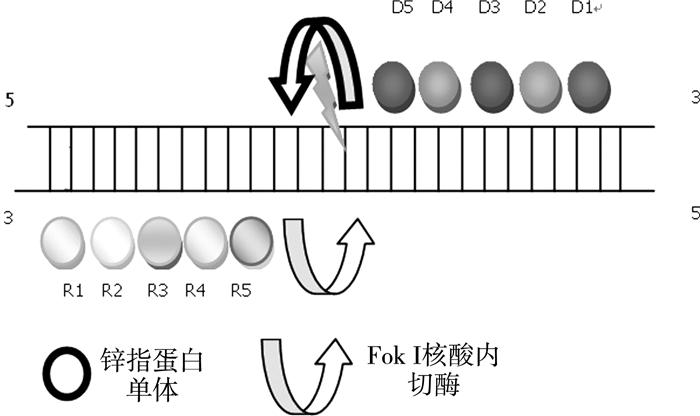 |
| 图 1 ZFNs靶向切割DNA示意图 |
这个工具成为对人类细胞进行基因编辑的基础,包括多能干细胞[5],动物和植物细胞[6-7]。当前ZFN体系技术尚存问题有:需要改善锌指结构域与靶分子的结合特异性以及亲和性;要找到高效导入、基因调控性、基因的瞬时表达等问题的解决办法;亟需提高ZFN的FokⅠ域对细胞的潜在效果和潜在的免疫反应的综合评价。
1.2 TALENs技术TALEN(转录激活效应因子核酸酶)具备序列特异性结合的能力,通过连接FokⅠ与经改造的TALE,研究者将TALE形成了一类特殊的基因组编辑功能强大的工具。
TALEN组成包括N端序列(含核定位信号)、中央结构域(含可特异性识别基因序列的TALE重复序列)以及C端序列(具备FokⅠ核酸内切酶)[8]。不同的特定的基因序列的长度判别是由类型差异的TALE因子确定。TALENs技术靶向切割DNA如图 2所示,该技术的转录激活因子样效应核酸酶与通过DNA特定识别的基因结合,而后FokⅠ核酸酶执行剪切特定位点功能,并通过细胞内在的同源性指导修复或非同源末端连接修复过程,以实现特定序列的插入、缺失及基因融合。
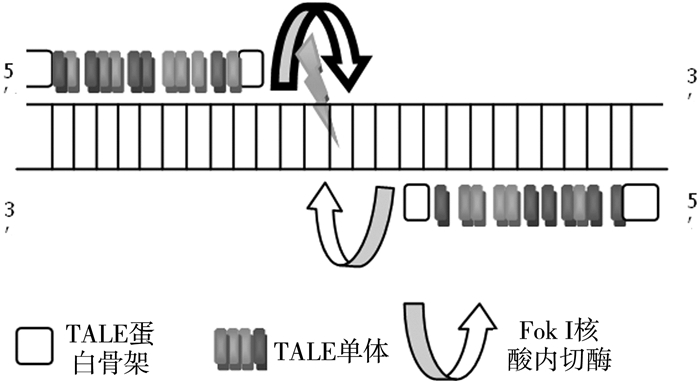 |
| 图 2 TALENs靶向切割DNA示意图 |
同ZFN相比较,TALEN具有明显的优点。首先,设计高效率的TALEN所需的分子克隆条件极其简易,TALEN明显简化了ZFN复杂的筛选过程;其次,该体系技术结合DNA序列比较严格且靶向效率高于ZFN;第三,TALEN可以减少脱靶的发生率,对受体细胞引起的毒性也会减少。该体系技术在基因敲除方面的应用较多,例如它适用于进行较大片段基因的插入。
但该技术仍然存在以下问题:TALEN蛋白可能会引发相应的机体免疫反应,降低其在细胞中的靶向效率[9];TALEN技术工具对靶向序列有一定的局限性,它需要靶向序列5’末端为碱基T。
1.3 CRISPR-Cas技术CRISPR-Cas9[10]是源于(古)细菌的降解入侵噬菌体和质粒DNA片段的获得性免疫机制。该技术体系于2012年首次被报道[11]。文献[12]中提到其新颖性在于它的RNA引导核酸内切酶活性提高了其效率、可扩展性和易用性。
CRISPR-Cas9系统靶向切割DNA如图 3所示,该技术的作用原理是将crRNA与tracrRNA进行改造,然后相互连接组成复合物[13],该复合物特异性能够识别靶基因序列。通过RNA的两种不同的人工改造设计组成sgRNA (singleguide RNA),sgRNA完全能够引导Cas9定点切割DNA。
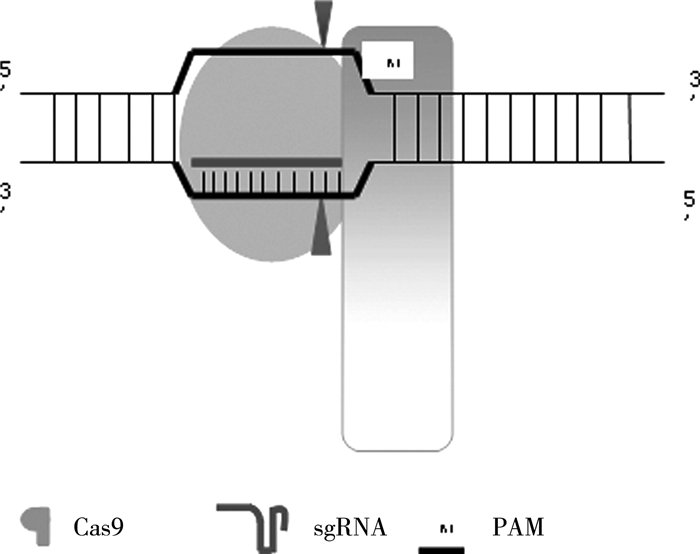 |
| 图 3 CRISPR-Cas9系统靶向切割DNA示意图 |
该技术体系具有有效性、特异性及易用性等显著优势[14],摆脱了与特定DNA识别的蛋白模块组件装配操作过程的复杂性,其毒性远低于ZFN技术。该技术设计与合成gRNA的效率明显高于前两项技术的DNA识别模块的构造过程。CRISPR-Cas9基因组工程工具箱以前所未有的精度和灵敏度帮助研究人员了解基因的功能[15]。这项技术可以用来研究目标基因的功能或通过基因编辑校正细胞中的基因突变,为未来的基因治疗方法铺平道路。CRISPR修复效率的改进,特别是增加基因矫正率和减少脱靶效应以及更有效的传递方法将发展出更广泛的治疗应用[16]。CRISPR系统也被用于nongene编辑,如激活基因表达的抑制作用,以及对染色体区域和个人基因的追踪定位[17]。总之,该系统的应用率以惊人的速度增长,将带来更好的治疗人类疾病的方法。
该技术的局限性,是其同样对上下文产生依赖,当前仅能应用于PAM序列上游。脱靶效应可能导致基因组不稳定和破坏正常基因的功能,而这是制约CRISPR/Cas9在生物医学和临床得到更为广泛应用的原因[18]。此外,该技术的特异性有待改善[19]。由此,Bolukbasi等[20]为Cas9融集编程DNA结合结构域,结果表明它能显著提高CRISPR-Cas9的准确性,使其特异性和亲和力得到进一步改善。
2 基因编辑技术在生物医学方面的应用 2.1 药物发现模型ZFNs、TALENs和CRISPR/Cas9系统使用这些机制来进行有针对性的基因组修饰,已成功地用于人类细胞的基因操作。Pellagatti等人[21]提出这些基因组编辑工具可用于研究基因功能,发现新的治疗靶点,并建立了药物模型。
动物病理和生理模型是药物筛选和药物靶点研究的重要手段,但是有些疾病的动物模型无法真实反映人类的病理、生理特点,如冠状动脉病、变胆固醇代谢和丙型肝炎感染等。随着基因编辑技术和人类多功能干细胞技术的结合,可以得到符合人体特征的多种细胞模型,为新药的开发、选择和评价奠定基础。经过Liu等人[22]的一系列实验,得出的结论是,TALEN介导的基因突变可作为人类疾病遗传建模的有效工具。例如:已经被建立的丙型肝炎病毒侵染模型是通过TALEN技术敲除人的肝癌细胞系特定基因形成的,与传统的细胞模型相比,它具备更加完整的载脂蛋白B表达缺失,分选SORT1基因编码蛋白,apoB-100蛋白分泌的调控,都会对低密度脂蛋白以及胆固醇水平造成一定程度的影响。
Pablo等人[23]提出利用小鼠作为研究人类疾病的首选动物模型,这是由于较低的成本和易于使用。TALENs技术实现人类胚胎干细胞的细胞hues1 sort1基因敲除,冠状动脉疾病模型的建立(包括排除物种差异的影响);另外也有相关文献报道了利用TALENs敲除编码RAC-b丝氨酸苏氨酸蛋白激酶的AKT2,以此完成胰岛素抵抗和脂肪代谢相关模型的建立;针对处于外周蛋白的特定基因(PLIN1) 进行编辑以创建存储、释放TG(甘油三酯)和FFA(游离脂肪酸)的相关模型[24]。基因编辑技术在该领域的应用具有高效、灵活及基因完整敲除等多方面的优势。
通过CRISPR/Cas9技术体系能够创建点突变,缺失和复杂染色体重排的生殖细胞和体细胞动物模型。在胚胎干细胞中,通过基因定位与同源重组,进行基因敲除产生了小鼠模型[25],研究在体内的与疼痛相关基因的功能。应用该系统可进一步阐明潜在的可作为治疗慢性疼痛疾病药物的互补策略机制。运用Cas9表达稳定的人胃癌细胞株,这些细胞株可用于大规模药物筛选、新靶点筛选和基因功能研究[26]。
这些基因编辑工具中与规律成簇间隔短回文重复相关的核酸酶已经成为药物筛选和构建动物模型的新型工具,也为编辑特异性基因序列提供了支持。
2.2 基因工程药物糖基化目前,美国已经批准了13个单克隆抗体以治疗癌症,IgG1是主要结构类型[27]。靶向肿瘤细胞的单抗药物主要作用机理是通过效应细胞的Fcγ受体与IgG1抗体Fc结构域结合介导抗体依赖细胞毒作用(ADCC)和抗体依赖吞噬作用[28]。通过ZFNs技术敲除FUT8基因,得到Alginate缺失的细胞系以进行相关抗体表达[29],研究结果得知,对于抗体介导ADCC效应的能力,通过缺失盐藻酸糖基化修饰的抗体进行介导是正常盐藻酸糖基化修饰抗体的100倍[30]。该课题人员已通过特异性CRISPR/Cas体系成功完成敲除CHO细胞FUT8基因。
SAFC最近介绍了通过ZFNs建造的N-乙酰氨基葡糖转移酶敲除细胞,这种细胞能够完成高甘露糖的糖基化蛋白分子表达[31],该策略可提高所表达的细胞与抗原的结合能力。另外,由抗体介导的诊疗方法,普通药物会随着血液循环产生一定的毒性,而利用高甘露糖基化的抗体可降低这种风险。
2.3 工程细胞改造与优化ZFN技术现在已经成功地应用于人类干细胞基因的相关编辑或校正与疾病相关的基因。Rahman等人[32]利用该技术敲除来自HIV患者的T细胞中分离的CCR5受体,旨在启动保护这些细胞免遭病毒感染。此外,通过ZFNs技术靶向野生型DHFR和GS CHO-K1细胞基因用来建立双基因敲除的细胞系,细胞能够同时用DHFR、GS两种筛选系统进行筛选,使工程细胞的构建效率得到极大改善[33]。
在工程细胞构造领域的另一个研究热点是如何在生物反应器的连续培养中提高工程细胞的存活率,Bax、Bak在细胞凋亡的机制中促凋亡因子相对明确,通过ZFN体系使Bax,Bak基因敲除,能够显著改善CHO工程细胞抗凋亡能力,参考文献[34]中的试验在连续培养12~17 d时,其产物表达量是野生型细胞株的2~5倍。
2.4 基因治疗基因编辑技术的出现,使人类基因组序列可以被精准修饰,以达到治疗效果。这包括校正突变基因、将治疗基因的插入到基因组中的特定位置、以及敲除有害基因或基因组序列。
基因编辑技术能够控制人类基因的表达,通过基因工程在他们的原生基因组上下文合成的转录因子,可以有针对性的准确调控任何DNA序列[35]。Maeder等人[36]指出一些治疗性基因太大而无法通过载体有效的转移。而且,插入外源基因不能总是解决显性突变或删除不需要的遗传物质,如病毒基因组或受体。为了解决这些传统的基因加成方法限制,基因编辑领域已经出现有针对性的精确修改基因组序列。现在新的基因组编辑技术基于工程核酸酶,允许更精确的遗传操作。基因组编辑包括基因中断的可能结果,尤其是应用于CCR5 coreceptor基因,或引入小突变或更大的整体基因盒插入靶位点。Wang等人[37]利用基因组编辑试剂输送和高效的同源性示范进展引导T细胞和HSPCs编辑,发展这一技术用于HIV治疗等应用。
CRISPR/Cas9基因编辑工具的成功之处是由RNA以极高的特异性及高效率的指引Cas9蛋白至目标DNA位点,使DNA链断裂。参考文献[38]中的最近几项研究使用CRISPR/Cas9成功调节了在动物模型和体外细胞内引起疾病的等位基因以及体外诱导多能干细胞,提高了基因编辑对于基因治疗的希望。基因编辑技术在生物医学领域的应用如图 4所示。
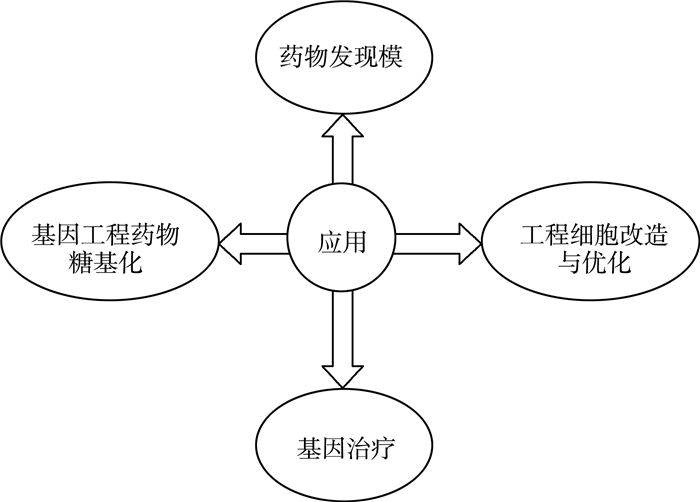 |
| 图 4 基因编辑技术在生物医学领域的应用 |
3 结语与展望
经过几年的时间,从ZFNs技术、TALENs技术到CRISPR/Cas技术,基因编辑技术飞速发展,ZFNs技术和TALENs技术在基因编辑平台仍被广泛应用,CRISPR/Cas技术也相继问世,目前,最新工具Ngago尚未被证实,或许在未来将能够与CRISPR系统相竞争。
1) ZFNs技术作为第一代基因编辑技术为基因编辑的发展发挥了重要作用,但该技术仍存在提高结合特异性和亲和性、改善潜在的免疫反应等问题。
2) TALENs基因编辑技术的脱靶率明显降低,对受体细胞引起的毒性也比ZFNs技术显著降低,应用也更加广泛。但是TALENs技术对靶向序列有一定的局限性,它需要靶向序列5’末端为碱基T。
3) CRISPR/Cas技术是目前应用最广泛的基因编辑技术,该技术的有效性、特异性及易用性有着显著的改善。然而该技术的局限性是其同样对上下文产生依赖,当前仅能应用于PAM序列上游。
基因编辑技术对分子机制研究和基因治疗产生了重大的影响。目前自体细胞治疗和干细胞治疗正迅速发展,细胞治疗中许多靶基因都可以使用基因组编辑技术进行操作,将基因组编辑技术与细胞治疗相结合已经成为一种趋势。另外,基因编辑技术在生物制药方面也起了关键作用,特别是在蛋白质药物分子糖基化修饰领域的贡献是巨大的。在分子水平的体外糖基化修饰相比传统技术更有效,基因编辑技术对生物医学的发展起到了巨大的推动作用。
| [1] |
XIAO A, HU Y Y, WANG W Y, et al. Artificial zinc finger nucleic acid enzyme mediated genome site modification technology[J]. Hereditas, 2011, 33(7): 665-683. |
| [2] |
KLUQ A. The discovery of zinc fingers and their development for practical applications in gene regulation and genome manipulation[J]. Quat Rev Biophys, 2010, 43(01): 1-21. DOI:10.1017/S0033583510000089 |
| [3] |
OSBOM M J, DEFEO A P, BLAZAR B R, et al. Synthetic zinc finger nuclease design and rapid assembly[J]. Hum Gene Ther, 2011, 22(9): 1155-1165. DOI:10.1089/hum.2011.072 |
| [4] |
KIM C A, BERQ J M. Aresolution crystal structure of a designed zinc finger protein bound to DNA[J]. Nat Struct Biol, 1996, 3(11): 940-945. DOI:10.1038/nsb1196-940 |
| [5] |
YAO Y, NASHUN B, ZHOU T, et al. Generation of CD34+ cells from CCR5-disrupted human embryonic and induced pluripotent stem cells[J]. Hum Gene Ther, 2012, 5(23): 238-242. |
| [6] |
CANTOS C, FRANCISCO P, TRIJATMIKO K R, et al. Identification of "safe harbor" loci in indica rice genome by harnessing the property of zinc-finger nucleases to induce DNA damage and repair[J]. Front Plant Sci., 2014, 5(26): 302. |
| [7] |
ZHANG W, WANG D, LIU S, et al. Multiple copies of a linear donor fragment released in situ from a vector improve the efficiency of zinc-finger nuclease-mediated genome editing[J]. Gene Ther, 2014, 21(3): 282-288. DOI:10.1038/gt.2013.83 |
| [8] |
BOCH J, SCHOLZE H, SCHORNACK S, et al. Breaking the code of DNA binding specificity of TAL type Ⅲ effectors[J]. Science, 2009, 326(5959): 1509-1512. DOI:10.1126/science.1178811 |
| [9] |
张金脉, 任兆瑞. TALENs:一种新的基因定点修饰技术[J]. 生命科学, 2013, 25(1): 126-132. |
| [10] |
KIM D, KIM J, HUR J K, et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells[J]. Nat Biotechnol, 2016, 34(6): 863-868. |
| [11] |
JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [12] |
GO D E, STOTTMANN R W. The impact of CRISPR/Cas9-based genomic engineering on biomedical research and medicine[J]. Curr Mol Med, 2016, 16(4): 343-352. DOI:10.2174/1566524016666160316150847 |
| [13] |
JINEK M, CHYLINSKI K, Fonfara I, et al. A programmable dual RNAguided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [14] |
SEEGER T, PORTEUS M, WU J C. Genome editing in cardiovascular biology[J]. Circ Res, 2017, 120(5): 778-780. DOI:10.1161/CIRCRESAHA.116.310197 |
| [15] |
TYCKO J, MYER V E, HSU P D. Methods for optimizing CRISPR-Cas9 genome editing specificity[J]. Mol Cell, 2016, 63(3): 355-370. DOI:10.1016/j.molcel.2016.07.004 |
| [16] |
PELLAGATTI A, DOLATSHAD H, VALLETTA S, et al. Application of CRISPR/Cas9 genome editing to the study and treatment of disease[J]. Archives of Toxicology, 2015, 89(7): 1023-1034. DOI:10.1007/s00204-015-1504-y |
| [17] |
DOETSCHMAN T, GEORGIEVA T. Gene Editing With CRISPR/Cas 9 RNA-Directed Nuclease[J]. Circ Res, 2017, 120(5): 876-894. DOI:10.1161/CIRCRESAHA.116.309727 |
| [18] |
ZHANG X H, TEE L Y, WANG X G, et al. Off-target effects in CRISPR/Cas9-mediated genome engineering[J]. Mol Ther Acids, 2015, 4(264): 1038-1046. |
| [19] |
YUAN W M, LIAN F Z, XING X H. Genome modification by CRISPR/Cas9[J]. FEBS Journal, 2014, 281(23): 5186-5193. DOI:10.1111/febs.2014.281.issue-23 |
| [20] |
BOLUKBASI M F, GUPTA A, OIKEMUS S, et al. DNA-binding-domain fusions enhance the targeting range and precision of Cas 9[J]. Nat Methods, 2015, 12(12): 1150-1156. DOI:10.1038/nmeth.3624 |
| [21] |
PELLAGATTI A, DOLATSHAD H, YIP B H, et al. Application of genome editing technologies to the study and treatment of hematological disease[J]. Advances in Biological Regulation, 2016, 60(9): 122-134. |
| [22] |
LIU H, CHEN Y, NIU Y, et al. TALEN-Mediated Gene Mutagenesis in Rhesus and Cynomolgus Monkeys[J]. Cell Stem Cell, 2014, 14(3): 323-328. DOI:10.1016/j.stem.2014.01.018 |
| [23] |
PABLO P P, ZHENG Y C. Biomedical applications of gene editing[J]. Hum Genet, 2016, 135(9): 967-969. DOI:10.1007/s00439-016-1723-1 |
| [24] |
DING Q, LEE Y K, SCHAEFER E A, et al. A TALEN genome editing system for generating human stem cell-based disease models[J]. Cell Stem Cell, 2013, 13(2): 238-251. |
| [25] |
SUN L, LUTZ B M, TAO Y X. The CRISPR/Cas9 system for gene editing and its potential application in pain research[J]. HHS Public Access, 2016, 1(3): 22-33. |
| [26] |
BIAN X C, YANG Z L, FENG H L. Establishment and validation of human cancer cell lines with stable Cas9 expression[J]. Zhonghua Bing Li Xue, 2017, 46(1): 43-48. |
| [27] |
SLIWKOWSKI M X, MELLMAN I. Antibody therapeutics in cancer[J]. Science, 2013, 341(6151): 1192-1198. DOI:10.1126/science.1241145 |
| [28] |
STAGG J, LOI S, DISEKERA U, et al. Anti-ErbB-2 mAb therapy requires type Ⅰand Ⅱinterferons and synergizes with anti-PD-1 or anti-CD137 mAb therapy[J]. Proc Natl Acad Sci USA, 2011, 108(17): 7142-7147. DOI:10.1073/pnas.1016569108 |
| [29] |
MALPHETTES L, FREYVERT Y, CHANG J, et al. Highly efficient deletion of FUT8 in CHO cell lines using zinc-finger nucleases yields cells that produce compieteiy nonfucosylated antibodies[J]. Biotechnol Bioeng, 2010, 106(05): 774-783. DOI:10.1002/bit.v106:5 |
| [30] |
YAMANE O N, KINOSHITA S, INOUE U M, et al. Establishment of FUT8 knockout chinese hamster ovary cells:an ideal host cell line for producing completely defucosylated antibodies with enhanced antibody-dependent cellular cytotoxicity[J]. Biotechnol Bioeng, 2004, 87(5): 614-622. DOI:10.1002/(ISSN)1097-0290 |
| [31] |
SEALOVER N R, DAS A M, BROOKS J K, et al. Engineering chinese hamster ovary(CHO) cells for producing recombinant proteins with simple glycoforms by zinc-finger nuclease(ZFN)-mediated geneknockout of mannosyⅠ(alpha-1, 3-)-glycoprotein beta-1, 2-N-acetylglucosaminyltransferase(Mgatl)[J]. Biotechnol, 2013, 167(1): 24-32. |
| [32] |
RAHMAN S H, MAEDE R M L, JOUNG J K, et al. Zinc-finger nucleases for somatic gene therapy:the next frontier[J]. Human Gene Therapy, 2011, 22(10): 1089-2011. |
| [33] |
LIU P Q, CHAN E M, COST G J, et al. Generation of a triple-gene knockout mammalian cell line using engineered zinc-finger nucleases[J]. Biotechnol Bioeng, 2010, 106(1): 97-105. |
| [34] |
COST G J, FREYVERT Y, VAFIADIS A, et al. BAK and BAX deletion using zinc-finger nucleases yields apoptosis-resistant CHO cells[J]. Biotechnol Bioeng, 2010, 105(2): 330-340. DOI:10.1002/bit.v105:2 |
| [35] |
CHARLES A G, PABLO P P. Activating human genes with zinc finger proteins, transcription activator-like effectors and CRISPR/Cas9 for genetherapy and regenerative medicine[J]. Expert Opinion on Therapeutic Targets, 2014, 18(8): 835-839. DOI:10.1517/14728222.2014.913572 |
| [36] |
MAEDER M L, GERSBACH C A. Genome editing technologies for Gene and Cell Therapy[J]. Molecular Therapy, 2016, 24(3): 430-446. DOI:10.1038/mt.2016.10 |
| [37] |
WANG C X, CANNON P M. The clinical applications of genome editing in HIV[J]. Blood, 2016, 127(21): 46-52. |
| [38] |
SAVIC N, SCHWANK G. Advances in therapeutic CRISPR/Cas9 genome editing[J]. Transl Res, 2016, 168(10): 15-21. |
 2017, Vol. 31
2017, Vol. 31


