2. 新余学院 基础医学教研室, 江西 新余 338004
2. Department of Foundamental Medicine, Xinyu University, Xinyu 338004, China
手性醇是一类重要手性模块化合物,在医药、化工和农业等领域有极高的应用价值[1-3],如(S)-(4-氯苯基)(吡啶-2-基)甲醇、(S)-3-氯-1-苯基-1-丙醇和(R)-2-羟基-4-苯基丁酸乙酯可分别合成抗过敏药物倍他司汀[4]、抗抑郁药氟西汀[5]和降压药物卡托普利[6]等。利用还原酶或全细胞为催化剂不对称生物还原潜手性羰基化合物是制备手性醇的有效途径。还原酶是一类以NAD(P)H为辅酶、在酶学分类上属于短链脱氢酶/还原酶(short-chain dehydrogenases/reductases,SDR)类,因具有立体选择性强、反应条件温和、安全性和环境相容性好等特点,日益受到研究者关注,且投入大量研究工作[7-9]。随着生物技术和手性技术的快速发展,还原酶的需求越来越大,开发更多高效、新型还原酶催化剂已成为当前研究热点之一。
(R)-1-[3, 5-二(三氟甲基)苯基]乙醇((R)-1-[3, 5-bis(trifluoromethyl)phenyl]ethanol,(R)-BTPE)是合成阿瑞吡坦、福沙吡坦和罗拉吡坦等药物的关键手性模块[10-11]。(R)-BTPE的制备方法有化学法合成[12-13]、酶法拆分[14-15]和生物催化不对称还原[11, 16-25]等,基于还原酶或全细胞为催化剂的不对称生物还原法具有高立体选择性、副产物少和理论产率可达100% 等特征,越来越多的还原酶或微生物细胞被报道用于不对称还原制备(R)-BTPE,如Leifsoniaxyli[17]、Lactobacillus kefir[11, 18]、Penicilliumexpansum[19]、Microbacteriumoxydans[20]、Trichodermaasperellum ZJPH0801[21]、Chryseobacterium sp.CA49[22]、Burkholderiacenocepacia[23]、Leifsonia sp.S749[24]和固定化的酮还原酶[25]等。然而,大多数报道的还原酶在生物催化制备(R)-BTPE时存在催化活性低、底物浓度限制和反应条件敏感等问题,不利于工业化应用。本研究基于基因挖掘技术和还原酶保守序列分析,经筛选和设计获得不对称生物催化还原[3, 5-二(三氟甲基)苯基]乙酮(3, 5-bis(trifluoromethyl)acetophenone,BTAP)制备(R)-BTPE的新型还原酶SDR05;以表达还原酶SDR05的重组大肠杆菌为催化剂,采用单因素实验和Box-Behnken法构建重组菌不对称还原BTAP制备(R)-BTPE的反应体系,提高还原酶SDR05的催化效率,克服底物浓度限制,实现底物在高质量浓度下转化。
2 材料和方法 2.1 试剂[3, 5-二(三氟甲基)苯基]乙酮、(R)-1-[3, 5-二(三氟甲基)苯基]乙醇、(S)-1-[3, 5-二(三氟甲基)苯基]乙醇、十二烷、异丙基β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)和卡那霉素、酵母抽提物、胰蛋白胨、氧化型烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+)、氧化型烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate,NADP+)和乙二胺四乙酸二钠(EDTA-Na2)等。
2.2 菌体培养和收集方法(1) 种子培养:从解冻的甘油管中取0.5 mL菌液接种于装有50 mL Luria-Bertani(LB)液体培养基的250 mL三角瓶中,然后置于37 ℃、150 r⋅min−1条件下振荡培养12 h。
(2) 重组菌静息细胞的获得:取1.0 mL培养好的种子液,接种于装有100 mL LB液体培养基的500 mL三角瓶中,置于37 ℃、150 r⋅min−1条件下振荡培养90 min(菌体的光密度值OD600nm为0.25~0.30),无菌条件下加入终浓度为0.6 mmol⋅L−1 IPTG,再置于29 ℃、150 r⋅min−1条件下继续培养14 h,然后在4 ℃、10 000 r⋅min−1条件下离心5 min,弃上清液后用质量分数为0.9% 的NaCl溶液洗涤2次,即得重组湿菌体。
2.3 还原酶基因挖掘、设计及含还原酶基因的重组菌构建以还原酶氨基酸序列(GenBank:AGW01025)为探针,利用基因挖掘技术从GenBank数据库(http://www.ncbi.nlm.nih.gov/genbank/)中通过序列比对获得与探针序列相似度为70%~80% 的还原酶基因,文献检索筛选出还原酶功能未被报道的基因为目的基因;再基于还原酶高度保守氨基酸的序列分析,对筛选获得的目的基因进行设计,比对探针序列和已报道的相关还原酶序列,改变目的基因氨基酸残基,使其保守氨基酸序列得以保留,得到新型还原酶基因,并在其5’和3’端添加Nco I和Hind III酶切位点,然后送于苏州金唯智生物科技有限公司进行基因全合成和构建含还原酶基因的重组菌。
2.4 重组菌静息细胞不对称还原制备(R)-BTPE的条件研究(1) 单因素实验构建反应体系:以2.2节部分获得的湿菌体为催化剂,基于单因素实验,考察反应温度、缓冲液pH值、添加剂种类和浓度、底物浓度和菌体浓度等对产物(R)-BTPE产率的影响,构建还原反应体系。单因素实验初始反应体系:异丙醇体积分数为25%,重组菌湿菌体质量浓度为40 g⋅L−1,BTAP浓度为300 mmol⋅L−1和一定体积的0.1 mmol⋅L−1、pH 7.4磷酸缓冲液组成5 mL反应体系;反应条件为30 ℃、150 r⋅min−1和12 h。
(2) Box-Behnken法确定重要因素的最优水平:基于Box-Behnken法,考察影响重组菌催化还原反应显著因素(菌体浓度、异丙醇体积分数和NAD+浓度)的交互作用,以反应12 h的产物产率为响应值,通过对实验结果二次方模型的回归分析和方差分析,确定它们的最优水平,验证最优水平下产物产率。
(3) 反应时间和较大规模对重组菌静息细胞制备(R)-BTPE的影响:在以构建获得的重组菌静息细胞不对称还原BTAP的反应体系和反应条件下,考察不同反应时间和较大规模反应体系对生物还原制备(R)-BTPE的影响。
2.5 酶活力、催化常数kcat和米氏常数Km测定(1) 酶活力测定:实验在构建获得的反应体系(异丙醇体积分数为69%,重组菌质量浓度为300 g⋅L−1,NAD+浓度为3.9 mmol⋅L−1,MgCl2浓度为1 mmol⋅L−1、BTAP浓度为3 000 mmol⋅L−1和一定体积的0.1 mol⋅L−1、pH 7.4磷酸缓冲液)中,32 ℃和150 r⋅min−1条件下,反应30 min后萃取,用气相色谱法分析和计算产物和剩余底物浓度。定义每分钟催化BTAP产生1 μmol(R)-BTPE所需酶量(细胞质量,g)为一个酶活力单位(U)。
(2) 催化常数kcat和米氏常数Km测定:以构建获得的反应体系为基础,分别配制底物质量浓度为256、384、512、640和768 g⋅L−1的反应体系,在32 ℃、150 r⋅min−1的条件下,反应30 min后,乙酸乙酯萃取、离心收集乙酸乙酯相,气相色谱法分析、计算产物和剩余底物浓度。应用双倒数作图法,以产物(R)-BTPE的生成速度倒数与底物浓度倒数作图,求出kcat和Km。
2.6 产物的分离纯化及鉴定还原反应结束后,反应液用乙酸乙酯萃取,离心收集乙酸乙酯相,再经旋转蒸发器真空蒸馏获得产物浓缩液。产物浓缩液上样硅胶层析柱,用洗脱剂(体积比V(石油醚): V(乙酸乙酯)=8:1)进行洗脱分离,用硅胶薄板层析检测所含产物的收集液,合并含产物的收集液,经旋转蒸发器真空蒸馏,即得产物。用气相色谱法和旋光法测定产物纯度和旋光度,用CDCl3溶解后进行氢谱1H-NMR和碳谱13C-NMR分析。
2.7 气相色谱法测定产物产率和对映体过量值还原反应结束后,反应液用一定体积的乙酸乙酯萃取,离心收集乙酸乙酯相(即气相分析样品),参照文献[26],采用气相色谱法测定产物和剩余底物浓度,计算产物产率(yield)和对映体过量值(enantiomeric excess,ee),方法分别为yield = (cP/c0)100%和ee = [(cR – cS)/(cS + cR)] × 100%,其中,c0为底物的起始浓度,cP为产物浓度,cR、cS分别为产物(R)-BTPE和(S)-BTPE的浓度。
3 结果与讨论 3.1 还原酶基因的挖掘、设计及其重组菌构建以Leifsoniaxyli HS0904源还原酶(GenBank:AGW01025)为探针,利用基因挖掘技术从GenBank数据库中通过序列比对获得15个来源于不同微生物种属的还原酶基因,它们与探针序列相似度为70%~80%;经文献检索筛选出5个还原酶功能未见有相关文献报道的基因为目的基因,它们均属于SDR家族的氧化还原酶基因,GenBank号分别为WP_134172672、BAP47552、WP_066039821、WP_147782688和WP_180934612;基于还原酶保守氨基酸的序列分析,比对目的基因与探针高度保守氨基酸残基(如T14G15-(X)3-G19-X-G21,D65,N90-N91-A92-G93,催化四联体N116-S144-Y157-K161,N179,P187-G188和T192。)和其他已报道具有催化功能的还原酶序列,对选定的5个目的基因进行设计,改变其氨基酸残基,使目的基因保留高度保守氨基酸残基,或同时对其他位置的氨基酸进行适当改变,获得5个新型还原酶(命名为SDR01、SDR02、SDR03、SDR04和SDR05)基因,并分别进行全基因合成和构建含相应还原酶基因的重组菌。对构建获得的重组菌进行诱导培养,以培养获得的重组菌静息细胞为催化剂,进行不对称还原BTAP制备相应手性醇的功能验证。结果表明,表达还原酶SDR05的重组菌具有较高的不对称还原BTAP制备(R)-BTPE的能力(产物产率为35.1%,ee值为99.9%),而表达还原酶SDR01(产率为0)、SDR02(产率为0)、SDR03(产率为0)和SDR04(产率为1.2%)的重组菌没有或仅有较低的催化能力。比对原有目的基因序列,还原酶SDR01、SDR02和SDR03分别改变0、1(K179N)和1(K179N)个氨基酸残基,仅使高度保守氨基酸序列保留;还原酶SDR04和SDR05分别改变8(E60R/K61A/A148S/K179N/ T182V/N183V/H191R/V249T)和18(L27T/R42E/G43E/E46N/T49V/V73E/E80V/A81E/P129D/V145I/S154Y/ G163A/H175Y/Q178K/Q179N/A197S/A202D/A249T)个氨基酸残基,它们不仅使高度保守氨基酸序列保留,且通过比对探针和其他已报道具有催化功能的还原酶序列,对其他位置氨基酸残基进行改变。功能验证和序列分析结果表明,还原酶只保留高度保守氨基酸残基序列,不能确保其具有催化活性,其他位置的保守氨基酸对其催化活性也有较大的影响。
如图 1所示为用ESPript 3.0[27]在线绘制的还原酶SDR05氨基酸序列的BLAST(Basic Local alignment search tool)比对分析图,发现还原酶SDR05氨基酸序列与其原有目的基因[来源于Nocardioidesungokensis的还原酶(GenBank:WP_180934612)]有最高的序列相似性(92.83%),与探针序列有81.27%的序列相似性;与文献报道的催化制备(R)-BTPE的Leifsoniasp. S749源还原酶(GenBank:BAD99642)有75.70%的序列相似性。还原酶SDR05与unclassifiedLeifsonia(GenBank:WP_018191686)、Cryocola sp. 340MFSha3.1(GenBank:WP_020077127)和Pimelobactersimplex(GenBank:WP_038681732)源的酶也有较高的序列相似性,分别为80.88%、80.48% 和80.48%。因此,SDR05是一种新型的还原酶,确定表达还原酶SDR05的重组菌为本研究的后续菌株。

|
图 1 还原酶SDR05氨基酸序列的BLAST比对分析(还原酶的保守序列用“*”标记) Fig.1 Blast analysis of amino acid sequences of reductase SDR05 and other proteins (The conserved sequence of reductases were marked with "*") |
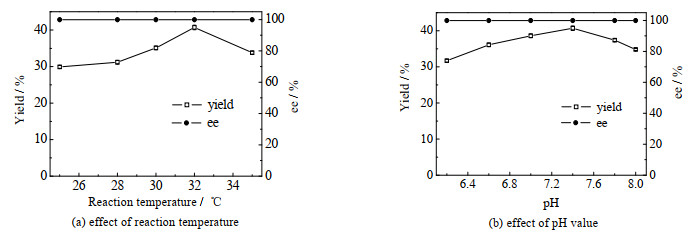
反应温度和缓冲液pH值是不对称生物还原反应的关键影响因素。图 2为不同反应温度和缓冲液不同pH值对还原产率和ee值的影响,从图 2中可看出,最佳反应温度和缓冲液pH值分别为32 ℃和7.4。
|
图 2 反应温度和pH值对生物催化还原的影响 Fig.2 Effects of reaction temperature and pH on biocatalytic reduction |
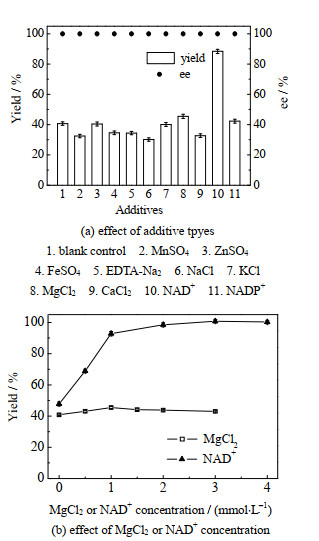
金属离子[28]、NAD+[24]和NADP+等添加剂可作为酶蛋白结构的一部分或作为辅助因子对酶蛋白的稳定性和活性有一定促进作用。实验考察了反应体系中添加金属离子、NAD+和NADP+等对产物产率和ee值的影响(见图 3(a))。结果表明,反应体系中添加MgCl2和NAD+对还原反应有促进作用,尤其是NAD+可显著提高产物产率,可能是因为还原酶SDR05是NADH依赖型还原酶(辅酶依赖型与探针还原酶一致[28]),NAD+的添加能强化辅酶的再生。考察反应体系中不同浓度的MgCl2和NAD+对还原反应的影响(见图 3(b)),结果表明,MgCl2的最佳添加浓度为1 mmol⋅L−1,与不添加MgCl2相比,产物产率提高了11.5%;NAD+最佳添加浓度为3 mmol⋅L−1,产物产率为96.1%,与不添加NAD+相比,产率提高了111.2%。
|
图 3 添加剂对生物催化还原的影响 Fig.3 Effects of additives on biocatalytic reduction |
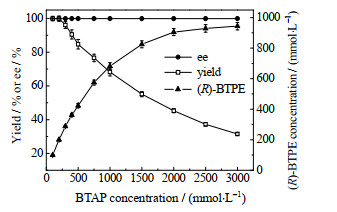
制备手性醇的底物是有机物质,在不对称还原反应中,它们存在底物水溶性差、对细胞有毒性、底物抑制和产物抑制等问题,导致底物浓度受到限制,影响反应效率。目前已报道的大多数制备(R)-BTPE用催化剂存在底物浓度限制,不适于工业化生产。考察了底物BTAP不同浓度对生物催化还原的影响,结果见图 4。随着BTAP浓度增大,产物浓度也明显增多,但产物产率下降,BTAP浓度为3 000 mmol⋅L−1时,产物浓度最大(943.7 mmol⋅L−1),产物产率为31.5%。为了实现底物在高浓度下的转化,确定反应体系中底物添加浓度为3 000 mmol⋅L−1。
|
图 4 BTAP浓度对生物催化还原的影响 Fig.4 Effects of BTAP concentration on biocatalytic reduction |
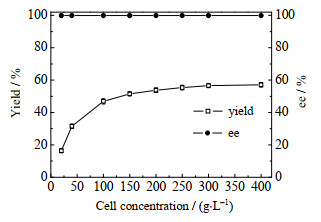
因反应体系底物浓度(3 000 mmol⋅L−1)增加,生物催化剂的用量也要提高。因此,实验考察菌体质量浓度对生物催化还原的影响(见图 5)。结果表明,菌体质量浓度对产物产率有显著影响,当菌体质量浓度由20 g⋅L−1增大到150 g⋅L−1时,产物产率由16.4% 提高到51.5%;继续提高菌体质量浓度,产物产率也继续增大,但提高幅度不明显。当菌体质量浓度为300 g⋅L−1时,产物产率为56.6%,菌体质量浓度增大到400 g⋅L−1时,产物产率仅增加了0.5%,因此,确定最佳菌体质量浓度为300 g⋅L−1。
|
图 5 静息细胞质量浓度对生物催化还原的影响 Fig.5 Effects of resting cells concentration on biocatalytic reduction |
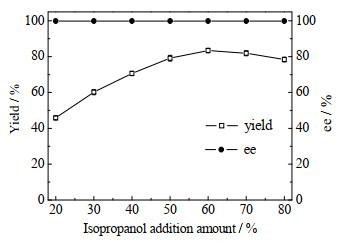
辅酶的持续再生是生物催化不对称还原反应关键影响因素。异丙醇作为辅酶再生的偶联基质,其添加量对还原反应的辅酶再生至关重要。实验考察异丙醇体积分数对还原产率和ee值的影响(见图 6)。结果表明,随着异丙醇体积分数的增加,产物产率大幅度提高,在异丙醇体积分数为60% 时,产物产率最大(83.4%),比优化前(异丙醇体积分数为25% 时的产物产率为56.6%)提高了47.3%,可能是因为异丙醇作为辅酶再生的偶联基质,能强化辅酶再生,且异丙醇作为有机溶剂,可以增加细胞膜的通透性和底物溶解性,有利于底物进入胞内和产物流出胞外,从而提高还原反应的效率;继续提高异丙醇体积分数,产物产率下降,可能是因为过量的异丙醇,会对菌体有毒性,使酶失活,降低反应效率。因此,异丙醇的最佳体积分数为60%。
|
图 6 异丙醇添加量对生物催化还原的影响 Fig.6 Effects of isopropanol amounts on biocatalytic reduction |
由单因素实验结果可知,菌体质量浓度(A)、异丙醇体积分数(B)和NAD+浓度(C)是影响不对称还原制备(R)-BTPE的显著因素。研究采用Box-Behnken法进行实验设计,考察3个显著因素间的交互作用以及对还原反应的影响,并确定它们最优水平,实验设计及实验结果如表 1所示。表 1中A=−1、0、1分别为菌体质量浓度100、200、300 g⋅L−1对应的水平值,B=−1、0、1分别为异丙醇体积分数25%、50%、75%对应的水平值,C=−1、0、1分别为NAD+浓度1、3、5 mmol⋅L−1对应的水平值。
|
|
表 1 Box-Behnken法实验设计及结果 Table 1 Experimental design and results of Box-Behnken method |
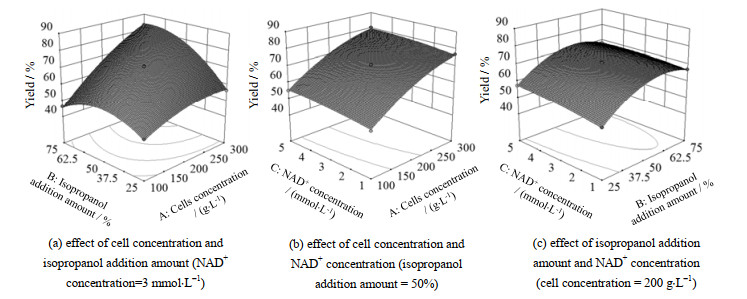
利用Design-Expert 10.0.7软件,对表 1响应值(产率)进行模型二次方回归分析和方差分析(见表 2),建立二次回归方程Y = 70.58 + 12.24A + 6.50B + 1.84C + 6.80AB −0.22AC − 0.95BC −3.93A2 − 8.40B2 − 0.93C2。模型方程决定系数R2、校正决定系数Radj2和预测决定系数Rpred2分别为0.998、0.996和0.978,它们的数值均接近于1,且相互间的差值较小,表明模型的拟合程度良好,模型可较准确反映实验值,可较好地预测结果和优化试验参数。由表 2可知,模型一次项A和B、二次项A2和B2、交叉项AB具有高度显著性(概率p < 0.000 1),模型一次项C,二次项C2,交叉项BC具有显著性(p < 0.050 0)。二次回归方程的三维响应面曲线图(见图 7)也表明,因素A(菌体质量浓度)和B(异丙醇体积分数)为显著变量,对产物产率影响较大,而C(NAD+浓度)在选定浓度范围内仅有轻微影响,结果与文献[24]报道一致。此外,回归模型的高F检验统计量值(F值=445.13)和低概率(p < 0.000 1)表明,回归模型方程具有高度显著性,可以预测最佳反应条件。预测结果为菌体质量浓度300 g⋅L−1、异丙醇体积分数69% 和NAD+浓度3.9 mmol⋅L−1,模型预期产物产率为84.4%。由于预测的最佳反应条件未包括在实验设计的17组实验中,需验证和确认模型预测的准确性。验证试验结果表明,产物产率为88.1%,稍高于预期的产率,基本与预期产率相符。
|
|
表 2 Box-Behnken法二次方模型的回归分析和方差分析 Table 2 Regressive analysis and ANOVA results of the Box-Behnken quadratic model |
|
图 7 Box-Behnken实验二次回归方程的三维响应面曲线图 Fig.7 Analysis of the experimental results by Box-Behnken method |
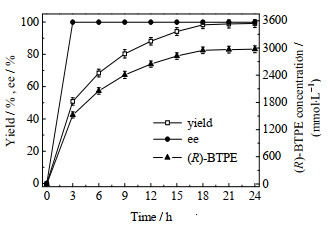
在50 mL反应瓶中加入质量浓度为300 g⋅L−1的重组菌、体积分数为69%的异丙醇、浓度为3.9 mmol⋅L−1的NAD+、1 mmol⋅L−1的MgCl2、3 000 mmol⋅L−1的BTAP和一定体积的pH 7.4的磷酸缓冲液组成5 mL反应体系,在32 ℃和150 r⋅min−1条件下分别反应不同时间。结果表明(见图 8),随着反应时间的延长,产物产率增大,当时间为18 h,产物产率为98.3%,继续延长时间,产物产率增加不大(98.8%/21h,99.1%/24h),因此,确定反应时间为18 h。整个反应过程中,产物ee值变化不大,都保持在99.9% 以上。研究结果与优化前相比,BTAP浓度由300 mmol⋅L−1提高到3 000 mmol⋅L−1,实现底物在高浓度下转化;与文献报道的不对称生物还原制备(R)-BTPE用还原酶相比,如Chryseobacteriumsp.CA49源还原酶ChKRED20[22](底物浓度为586 mmol⋅L−1,产率为92%)、Codexis源还原酶P1B2[25] (底物浓度为586 mmol⋅L−1,产率为97%)、LeifsoniaxyliHS0904源突变还原酶LXCAR-S154Y[29] (底物浓度为1 000 mmol⋅L−1,产率为98.7%)、Lactobacillus kefir源还原酶LkCR[18] (底物浓度为1 191 mmol⋅L−1,产率为96.7%)和Leifsonia sp.S749源还原酶KR01[24] (底物浓度为2 344 mmol⋅L−1,产率为98.3%)等,本研究筛选和设计获得的新型还原酶SDR05具有最高的底物浓度(底物浓度为3 000 mmol⋅L−1,产率为98.3%)。
|
图 8 反应时间对重组菌制备(R)-BTPE的影响 Fig.8 Effects of reaction time on (R)-BTPE preparation by recombinant E. coli cells |
进一步考察了较大规模反应体系(2.0 L反应瓶中加0.4 L反应液)对制备(R)-BTPE的影响。结果表明,在32 ℃、150 r⋅min−1的条件下反应18 h,产物产率和ee值分别为97.8% 和99.9%。单位质量(每克)细胞产生产物量为9.8 mmol⋅L−1,与文献报道的最高值7.7 mmol⋅L−1相比[24],提高了27.2%。
3.5 酶活力、催化常数kcat和米氏常数Km测定在构建获得的反应体系和反应条件下,测得还原酶SDR05的酶活力为0.023 U,单位质量每克菌体细胞酶活力为43.5 U。以产物(R)-BTPE的生成速度倒数与底物浓度的倒数作图获得双倒数方程y=62.665x+0.207(R2=0.992),得出Vmax (最大反应速率)、Km和kcat分别为4.8 g⋅L−1⋅min−1、300.8 g⋅L−1和0.016 min−1。
3.6 产物的分离纯化与鉴定上述较大规模制备(R)-BTPE的反应液经乙酸乙酯萃取、旋转蒸发器真空蒸馏和硅胶柱层析等方法分离,获得274 g的分离产物,分离收率为90%,经气相色谱检测,产物纯度为99% 以上。分离产物经CDCl3溶解后进行1H-NMR和13C-NMR鉴定[1H-NMR (600 MHz, CDCl3): 7.83 (单峰s,2H),7.79 (s,1H),5.04 (四重峰q,1H,耦合常数J=6.78 Hz),2.46 (s, 1H),1.56 (双峰d,3H,J=6.78 Hz)],碳谱13C-NMR(100 Hz,CDCl3),化学位移δ = 147.9,131.7,131.8,125.6,124.9,121.8,121.1,119.4,69.4,25.5],确定分离产物为BTPE,再经过旋光度测定([α]D25 = +21.5)及与标准品(R)-BTPE的气相结果比对分析,确证产物为(R)-BTPE。
4 结论基于基因挖掘技术和还原酶保守序列分析,经筛选和设计获得新型还原酶SDR05;以表达还原酶SDR05的重组菌为催化剂,通过向反应体系中添加异丙醇和NAD+,强化辅酶再生,获得了不对称还原BTAP制备(R)-BTPE的反应体系(质量浓度为300 g⋅L−1的重组菌,浓度为3.9 mmol⋅L−1的NAD+,浓度为1 mmol⋅L−1的MgCl2,体积分数为69% 的异丙醇,浓度为3 000 mmol⋅L−1的BTAP和一定体积的0.1 mol⋅L−1、pH 7.4的磷酸盐缓冲液)和反应条件(32 ℃、150 r⋅min−1和18 h),提高了还原反应效率(产物产率98.3% 和ee值99.9%),克服了底物浓度限制,实现底物在高质量浓度(768 g⋅L−1)下的转化,为工业化制备(R)-1-[3, 5-二(三氟甲基)苯基]乙醇等手性化合物奠定了基础。
| [1] |
LI Y H, ZHANG R Z, XU Y. Structure-based mechanisms: On the way to apply alcohol dehydrogenases/reductases to organic-aqueous systems[J]. International Journal of Biological Macromolecules, 2021, 168(31): 412-427. |
| [2] |
JIANG W, FANG B. Synthesizing chiral drug intermediates by biocatalysis[J]. Applied Microbiology and Biotechnology, 2020, 192(1): 146-179. |
| [3] |
ZHENG Y G, YIN H H, YU D F, et al. Recent advances in biotechnological applications of alcohol dehydrogenases[J]. Applied Microbiology and Biotechnology, 2017, 101(3): 1-15. |
| [4] |
NI Y, ZHOU J Y, SUN Z H. Production of a key chiral intermediate of Betahistine with a newly isolated Kluyveromyces sp. in an aqueous two-phase system[J]. Process Biochemistry, 2012, 47(7): 1042-1048. DOI:10.1016/j.procbio.2012.03.022 |
| [5] |
TANG Y P, ZHANG G M, WANG Z, et al. Efficient synthesis of a (S)-fluoxetine intermediate using carbonyl reductase coupled with glucose dehydrogenase[J]. Bioresource Technology, 2018, 250: 457-463. DOI:10.1016/j.biortech.2017.10.097 |
| [6] |
XU G C, NI Y. Bioreductive preparation of ACE inhibitors precursor (R)-2-hydroxy-4-phenylbutanoate esters: Recent advances and future perspectives[J]. Bioresources and Bioprocessing, 2015, 2(1): 1-11. DOI:10.1186/s40643-014-0030-8 |
| [7] |
QIN Y L, RUAN T, HOU H S, et al. A novel thermal stable carbonyl reductase from bacillus cereus by gene mining as biocatalyst for β-carbonyl ester asymmetric reduction reaction[J]. Catalysis Letters, 2019, 149(2): 610-618. DOI:10.1007/s10562-018-2645-4 |
| [8] |
WANG J, CHENG P, WU Y, et al. Discovery of a new NADPH-dependent aldo-keto reductase from Candida orthopsilosis catalyzing the stereospecific synthesis of (R)-pantolactone by genome mining[J]. Journal of Biotechnology, 2019, 291: 26-34. DOI:10.1016/j.jbiotec.2018.12.011 |
| [9] |
王亚军, 吴配配, 罗希, 等. E. coli BL21(DE3)/pET28a(+)-cr羰基还原酶分离纯化及酶学性质研究[J]. 高校化学工程学报, 2015, 29(3): 607-615. WANG Y J, WU P P, LUO X, et al. Purification and enzymatic characterization of E. coli BL21 (DE3)/pET28a(+)-cr carbonyl reductase[J]. Journal of Chemical Engineering of Chinese Universities, 2015, 29(3): 607-615. DOI:10.3969/j.issn.1003-9015.2015.03.016 |
| [10] |
WU C E, LIAW C C. Using aprepitant as secondary antiemetic prophylaxis for cancer patients with cisplatin-induced emesis[J]. Support Care Cancer, 2012, 20(10): 2357-2361. |
| [11] |
GELO-PUJIC M, GUYADER F L, SCHLAMA T. Microbial and homogenous asymmetric catalysis in the reduction of 1-[3, 5-bis(trifluoromethyl)phenyl] ethanone[J]. Tetrahedron: Asymmetry, 2006, 17(13): 2000-2005. DOI:10.1016/j.tetasy.2006.06.033 |
| [12] |
LI W, SUN X, ZHOU L, et al. Highly efficient and highly enantioselective asymmetric hydrogenation of ketones with TunesPhos/1, 2-Diamine-Ruthenium (II) complexes[J]. The Journal of Organic Chemistry, 2008, 74(3): 1397-1399. |
| [13] |
NAUD F, SPINDLER F, RUEGGEBERG C J, et al. Enantioselective ketone hydrogenation: From R & D to pilot scale with industrially viable Ru/phosphine-oxazoline complexes[J]. Organic Process Research & Development, 2007, 11(3): 519-523. |
| [14] |
VANKAWALA P J, KOLLA N, ELATI C R, et al. Efficient synthesis of (1R)-[3, 5-bis(trifluoromethyl)phenyl] ethanol, a key intermediate for aprepitant, an NK-1 receptor antagonist[J]. Synthetic Communications, 2007, 37(19): 3439-3446. DOI:10.1080/00397910701487473 |
| [15] |
王亮, 徐刚, 杨立荣. 动力学拆分制备(R)-3, 5-双三氟甲基苯乙醇[J]. 化工进展, 2013, 32(11): 2691-2694. WANG L, XU G, YANG L R. Preparation of (R)-3, 5-bis(trifluoromethyl)phenyl ethanol by kinetic resolution[J]. Chemical Industry and Engineering Progress, 2013, 32(11): 2691-2694. |
| [16] |
HOMANN M J, VAIL R B, PREVITE E, et al. Rapid identification of enantioselective ketone reductions using targeted microbial libraries[J]. Tetrahedron, 2004, 60(3): 789-797. |
| [17] |
WANG N Q, SUN J, HUANG J, et al. Cloning, expression, and directed evolution of carbonyl reductase from Leifsoniaxyli HS0904 with enhanced catalytic efficiency[J]. Applied Microbiology and Biotechnology, 2014, 98(20): 8591-601. |
| [18] |
CHEN K, LI K, DENG J, et al. Carbonyl reductase identification and development of whole-cell biotransformation for highly efficient synthesis of (R)-[3, 5-bis(trifluoromethyl)phenyl] ethanol[J]. Microbial Cell Factories, 2016, 15(1): 191. DOI:10.1186/s12934-016-0585-5 |
| [19] |
KURBANOGLU E B, ZILBEYAZ K, TASKIN M, et al. Total production of (R)-3, 5-bistrifluoromethylphenyl ethanol by a symmetric reduction of 3, 5-bis(trifluoromethyl)-acetophenone in the submerged culture of Penicilliumexpansum isolate[J]. Tetrahedron: Asymmetry, 2009, 20(23): 2759-2763. DOI:10.1016/j.tetasy.2009.11.006 |
| [20] |
盖萍, 汤传根, 刘静媛, 等. 氧化微杆菌C3催化3, 5-双三氟甲基苯乙酮的不对称反-Prelog还原[J]. 应用与环境生物学报, 2013, 19(1): 37-42. GAI P, TANG C G, LIU J Y, et al. Asymmetric anti-Prelog reduction of 3, 5-bis(trifluoromethyl)acetophenone by Microbacteriumoxydans C3[J]. Chinese Journal of Applied and Environmental Biology, 2013, 19(1): 37-42. |
| [21] |
LI J, WANG P, HE J Y, et al. Efficient biocatalytic synthesis of (R)-[3, 5-bis(trifluoromethyl) phenyl] ethanol by a newly isolated Trichodermaasperellum ZJPH0810 using dual cosubstrate: Ethanol and glycerol[J]. Applied Microbiology and Biotechnology, 2013, 97(15): 6685-6692. DOI:10.1007/s00253-013-4973-z |
| [22] |
LIU Y, TANG T X, PEI X Q, et al. Identification of ketone reductase ChKRED20 from the genome of Chryseobacterium sp. CA49 for highly efficient anti-Prelog reduction of 3, 5-bis(trifluoromethyl)acetophenone[J]. Journal of Molecular Catalysis B: Enzymatic, 2014, 102: 1-8. DOI:10.1016/j.molcatb.2014.01.009 |
| [23] |
YU S, LI H, LU Y, et al. A catalyst from Burkholderiacenocepacia for efficient anti-Prelog's bioreduction of 3, 5-bis(trifluoromethyl) acetophenone[J]. Applied Biochemistry and Biotechnology, 2018, 184(4): 1319-1331. DOI:10.1007/s12010-017-2628-8 |
| [24] |
TANG J, WEI T, NI G, et al. Development of an efficient and cost-effective enzymatic process for production of (R)-[3, 5-bis(trifluoromethyl)phenyl] ethanol using carbonyl reductase derived from Leifsonia sp. S749[J]. Applied Biochemistry and Biotechnology, 2019, 188(1): 87-100. |
| [25] |
LI H, MONCECCHI J, TRUPPO M D. Development of an immobilized ketoreductase for enzymatic (R)-1-(3, 5- bis(trifluoromethyl)phenyl)ethanol production[J]. Organic Process Research & Development, 2015, 19(7): 695-700. |
| [26] |
WANG N, XU Y, PENG C, et al. Identification of a newly isolated Rhodotorulamucilaginosa NQ1 and its development for the synthesis of bulkycarbonyl compounds by whole-cell bioreduction[J]. Letters in Applied Microbiology, 2021, 72(4): 399-407. |
| [27] |
ROBERT X, GOUET P. Deciphering key features in protein structures with the new ENDscript server[J]. Nucleic Acids Research, 2014, 42(1): 320-324. |
| [28] |
WANG N Q, HUANG J, LUO H D, et al. Purification and characterization of a new carbonyl reductase from Leifsoniaxyli HS0904 involved in stereoselective reduction of 3, 5-bis(trifluoromethyl) acetophenone[J]. Journal of Molecular Catalysis B: Enzymatic, 2013, 92: 1-6. DOI:10.1016/j.molcatb.2013.03.007 |
| [29] |
WANG N Q, LI J, SUN J, et al. Bioreduction of 3, 5-bis(trifluoromethyl)acetophenone using ionic liquid as a co-solvent catalyzed by recombinant Escherichia coli cells[J]. Biochemical Engineering Journal, 2015, 101: 119-125. |




