2. 沈阳双鼎制药有限公司, 辽宁 沈阳 110179
2. Shenyang Shuangding Pharmaceutical Co. Ltd., Shenyang 110179, China
转录激活因子样效应物(TALE)是黄单胞菌分泌的针对特定DNA序列的蛋白质[1-2]。TALE家族成员包含一个高度保守和重复的区域,其与DNA的结合能力取决于每个模块重复的第12和第13个氨基酸残基(即重复可变双氨基酸残基(RVD))。每个RVD中都有一个简单的代码,用来指定对特定碱基的识别,这决定了TALE核苷酸结合的特异性。例如,NI(Asn/Ile)对应A,NG(Asn/Gly)对应T,HD(His/Asp)对应C,NH(Asn/His)对应G[3-5]。人工构建的带有功能域的DNA结合域(DBD)补充的TALE可应用于基因工程的许多方面,包括用于转录调控的转录因子(TALE-TF) [6-9]和用于基因组编辑的转录激活因子样效应核酸酶(TALEN)[10-12]。因此,TALE在基因工程领域发挥了巨大的作用。
TALE的脱靶率比目前热门的CRISPR/Cas9(Clustered Regularly Interspaced Short Palindromic Repeats/Cas9)[13-15]要低,识别DNA长度也较长;基因调控和编辑的位置不受前间区序列邻近基序(PAM)的限制;具有更低的非靶点结合能力,更低的细胞毒性,基于TALE具有高靶向能力[16],所以,已由法国Cellectis公司用于生产通用嵌合抗原受体T细胞[17],并已被批准用于癌症免疫疗法。接受该基因编辑疗法的患者已显示出显著的治疗效果[18]。最重要的是,在设计和构建TALE时,理论上可以靶向任何DNA序列。但是构建TALE的方法比较繁琐复杂,而且构建的TALE长度有限,容易脱靶。并且,大多数研究人员仍然使用软件来预测TALE靶向的目标区域,然后构建一个有针对性的TALE。在这种情况下,需要构建更多的TALE,构建周期太长,效率偏低[19-21]。
本研究发明了一种新的简单的TALE文库的构建方法,设计了一种可以轻松实现靶向较长DNA序列的TALE文库的方法。该组装方法是快速且标准化的,能够精确控制产生的RVD和整体DNA结合域的组成。而且研究人员可以直接将其作为实验材料,为TALE研究人员节省了大量的时间。
2 材料与方法 2.1 材料金门组装试剂盒来自Addgene;限制酶BsaI、牛血清白蛋白(BSA)、T4 DNA连接酶、腺嘌呤核苷三磷酸(ATP)来自NEB公司;质粒核酸安全酶来自Epicentre公司;BsmBI来自Thermo Scientific公司;DH5α感受态细胞、壮观霉素、卡那霉素、X-gal、IPTG(Isopropyl β-D-Thiogalactoside,异丙基硫代半乳糖苷)来自南京源恩浩生物试剂;PrimeSTAR® GXL DNA Polymerase高保真聚合酶混合液来自Takara公司。
2.2 方法采用基于金门组装试剂盒优化改进的新方法构建了18 bp-TALE-VP64文库(bp表示碱基对)。
第1部分:构建含10个串联重复序列(RVDs:NH1-NH10)组合的PFUS-A+10×NH质粒。
步骤1(第1天,3 h):第1次金门酶切连接反应。将10个RVD构建模块质粒(NH1到NH10)各加入150 ng于0.5 mL离心管中。然后,加入150 ng pFUS-A质粒、1 µL BsaI、1 µL BSA、1 µL T4 DNA连接酶、2 µL T4 DNA连接酶反应缓冲液。然后加入蒸馏水,使总反应体积达到10 µL。
步骤2(第1天,10 min):上述反应体系混合并短暂旋转,涡旋混匀30 s。
步骤3(第1天,3 h):将反应体系在PCR仪上孵育。孵育条件为37 ℃、5 min和16 ℃、10 min共计10个循环,然后50 ℃、5 min;80 ℃、5 min。
步骤4(第1天,1 h):反应体系(10 µL)加入1 µL质粒核酸安全酶和1 µL 10 mmol⋅L−1 ATP在37 ℃下孵育1 h。
步骤5(第1天,4 h):将步骤4的反应体系转化到100 µL DH5α中,加入溶菌肉汤(LB)液体培养基得到1 mL该细胞悬液,涂布于LB琼脂细菌培养皿(含有壮观霉素50 μg⋅mL−1和20 mg⋅mL−1 X-gal) (X-gal是β-半乳糖苷酶的显色底物)以及0.1 mol⋅L−1 IPTG,由于PFUS-A+10×NH,PFUS-B7+7×NH和pLR-NH骨架质粒都具有壮观霉素抗性,所以,培养微生物大肠杆菌细胞DH5α,有抗性的说明质粒构建转染成功,没有转染成功的没有抗性,不会生长出菌落。
步骤6(第2天,6 h):从培养皿上挑选几个白色菌落,并使用引物pCR8-F1和pCR8-R1(表 1)进行菌落PCR检验。PCR程序:96 ℃预变性3 min;96 ℃变性20 s,55 ℃退火20 s,72 ℃延伸2 min,进行30个循环;72 ℃总延伸3 min,4 ℃保存。PCR检测正确的菌落经过培养、提取,得到PFUS-A+10×NH质粒。
|
|
表 1 质粒突变体和菌落PCR的引物 Table 1 Primers for plasmid mutation and colony PCR |
第2部分:构建含有7个串联重复序列(RVDs:NH1-NH7)组合的PFUS-B7+7×NH质粒。
步骤1(第1天,3 h):取7个RVD构建质粒模块(NH1到NH7)各加入150 ng,加入pFUS-B7质粒150 ng、1 µL BsaI、1 µL BSA、1 µL T4 DNA连接酶、2 µL T4 DNA连接酶反应缓冲液。然后加入蒸馏水,使总反应体积达到10 µL。
步骤2(第1天,10 min):反应体系短暂旋转混合,涡旋混匀30 s。
步骤3(第1天,3 h):将反应体系在PCR仪上孵育,孵育条件为37 ℃、5 min和16 ℃、10 min共计10个循环;然后50 ℃、5 min;80 ℃、5 min。
步骤4(第1天,1 h):反应体系(10 µL)加入1 µL质粒核酸安全酶和1 µL 10 mmol⋅L−1 ATP在37 ℃下孵育1 h。
步骤5(第1天,4 h):将步骤4的反应体系转化到100 µL DH5α感受态细胞。然后加入LB培养基得到1 mL细胞悬液涂布于LB琼脂细菌培养皿(含有壮观霉素50 µg⋅mL−1和20 mg⋅mL−1 X-gal以及0.1 mol⋅L−1 IPTG)。
步骤6(第2天,6 h):从培养皿中挑选几个白色菌落,使用引物pCR8-F1和pCR8-R1(见表 1)进行菌落PCR检测。PCR程序:96 ℃预变性3 min;96 ℃变性20 s,55 ℃退火20 s,72 ℃延伸1 min,进行30个循环;72 ℃总延伸3 min,4 ℃保存。PCR检测正确的菌落经过培养、提取,得到PFUS-B7+7×NH质粒。
第3部分:PFUS-A+10×NH质粒突变体、PFUS-B7+7×NH质粒突变体及pLR-NH质粒突变体的构建。
步骤1(第3天,3 h):PFUS-A+10×NH质粒突变体、PFUS-B7+7×NH质粒突变体、pLR-NH质粒突变体均采用PCR扩增得到,引物如表 1所示。利用HD-C、NH-G、NG-T和NI-A这4种特殊引物(表 1)对PFUS-A+10×NH质粒、PFUS-B7+7×NH质粒、pLR-NH质粒分别进行PCR扩增。3个PCR反应(240 μL)均包含1 ng质粒、10 μmol⋅L−1 HD-C、10 μmol⋅L−1 NH-G、10 μmol⋅L−1 NG-T、10 μmol⋅L−1 NI-A和PrimeSTAR® GXL DNA Polymerase高保真聚合酶混合液。PCR程序:96 ℃预变性3 min;96 ℃变性20 s,58 ℃退火20 s,72 ℃延伸3 min,进行28个循环;72 ℃总延伸3 min,4 ℃保存。PCR产物用1% 琼脂糖凝胶电泳检测。PCR产物经苯酚/氯仿萃取法纯化,用无水乙醇沉淀。PCR产物在水中重悬,3种PCR产物即PFUS-A+10×NH质粒突变体、PFUS-B7+7×NH质粒突变体、pLR-NH质粒突变体。
第4部分:构建TALE-VP64文库(RVDs:NH1-NH17和pLR-NH)。
步骤1(第3天,3 h):在20 µL反应体系中,包括150 ng PFUS-A+10×NH质粒突变体,150 ng PFUS-B7+7×NH质粒突变体,150 ng pLR-NH质粒突变体和75 ng TALE-backbone-VP64。加入1 µL BsmBI,1 µL T4 DNA连接酶、2 µL T4 DNA连接酶反应缓冲液。最后加入蒸馏水使总反应体积为20 µL。
步骤2(第3天,10 min):该反应体系短暂旋转混合,涡旋混匀30 s。
步骤3(第3天,3 h):将反应体系在热循环PCR仪上孵育。热循环条件为37 ℃、5 min和16 ℃、10 min,共计10个循环;然后37 ℃、15 min;80 ℃、5 min。
步骤4(第3天,4 h):将20 µL的反应体系转化到200 µL DH5α感受态细胞中。加入LB培养基得到1 mL细胞悬液涂布于LB琼脂细菌培养皿(含有卡那霉素50 µg⋅mL−1和20 mg⋅mL−1 X-gal以及0.1 mol⋅L−1 IPTG)。
步骤5(第4天,4 h):利用引物TAL-F和TAL-R(表 1)进行菌落PCR实验,根据电泳条带鉴定阳性菌落。最后PCR检测正确的菌落,过夜培养。
步骤6(第5天,2 h):提取培养的菌液中的质粒为18 bp-TALE-VP64文库质粒表达载体。
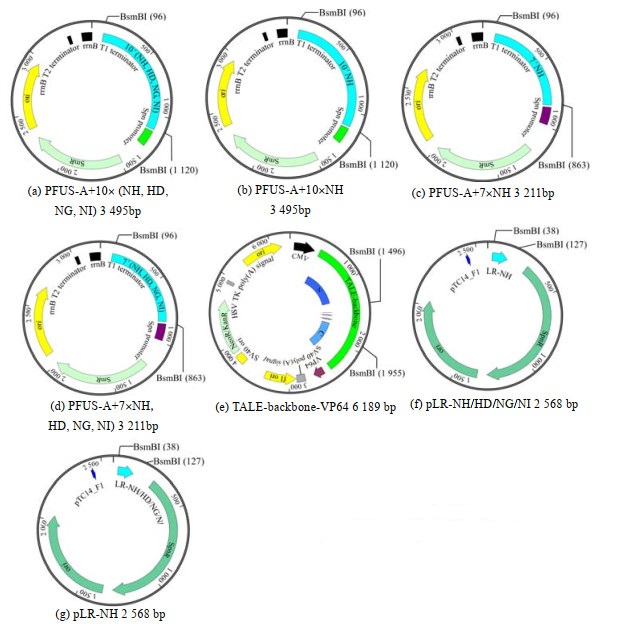
3 实验结果与分析 3.1 TALE文库构建流程的建立经过对金门法的优化构建了TALE文库,详细流程见图 1,此流程显示了构建TALE-VP64文库的示例,该质粒文库可以结合18 bp DNA长度的目标靶向位点。其中PFUS-A+10×NH是一种由10个单体质粒和一个骨架质粒PFUS-A在第1次酶切连接反应中制备的质粒;PFUS-B7+7×NH是一种由7个单体质粒和1个骨架质粒PFUS-B7在第1次酶切连接反应中制备的质粒。因此,PFUS-A +10×NH是表达与N1-N10核苷酸结合的氨基酸的质粒载体,PFUS-B7+7×NH是表达与N11-N17核苷酸结合的氨基酸的质粒载体。编码与最后一个核苷酸(N18)结合的氨基酸的序列包含在另一个单体质粒(pLR-NH)中。第2次酶切连接反应包含4个质粒,包括PFUS-A+10×NH质粒突变体(PFUS-A+10×(NH,HD,NG,NI))、PFUS-B7 +7×NH质粒突变体(PFUS-B7+7× (NH,HD,NG,NI))、pLR-NH质粒突变体(pLR-NH/HD/NG /NI)和TALE骨架载体(TALE-backbone-VP64)(见图 2,图中bp为碱基对)。

|
图 1 利用金门法基于4种特异性引物构建TALE-VP64文库的流程 Fig.1 Flowchart of the construction of TALE-VP64 libraries by the Golden-Gate method based on new specific primers pipeline |
|
图 2 质粒图谱及其组成 Fig.2 Plasmids maps and their elements |
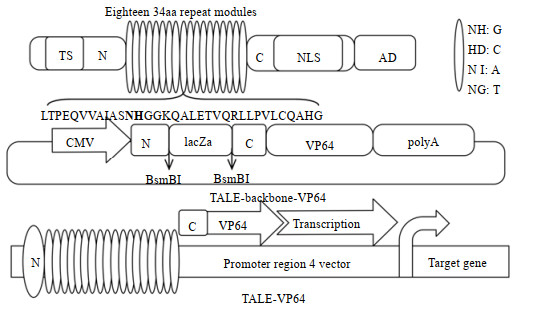
另外,在构建过程中所使作的各种骨架质粒图谱(即突变前质粒)见图 2中PFUS-A+10×NH、PFUS-B7×NH、pLR-NH质粒。最后,预计构建成功的TALE-VP64质粒文库具有转染各种细胞的能力,并可以激活下游基因的表达(见图 3),Zhang等[21]单独构建的TALE-VP64质料转染293T细胞,确实激活了绿色荧光基因ZsGreen,使绿色荧光蛋白高表达。TALE-VPR质料转染HepG2细胞和PANCl细胞,分别使得内源基因HNF4a和E47基因上调,从而证实了TALE-VP64/VPR强大的靶向和激活功能。TALE-VP64文库中每个DNA结合模块由34个氨基酸组成,其中每个模块的第12和13位氨基酸变化较大,其他氨基酸高度保守,这2个不保守的氨基酸被命名为重复可变残基(RVD),RVD氨基酸对应DNA碱基之间的分子密码是NI对应A、HD对应C、NG对应T、NH对应G,所有的TALE由N末端的转移结构域(AD)组成(图 3)。TALE的末端转移结构域以及C末端的核定位信号和转录激活结构域具有高度的保守性,不同成员之间互换这些功能区域并不影响其正常发挥作用。TALE-DNA结合域与合成的VP64转录激活因子融合,该激活因子募集RNA聚合酶和启动转录所需的其他因子,激活下游基因的表达(见图 3)。
|
图 3 用于基因组工程的TALE-VP64文库 Fig.3 The TALE -VP64 libraries for genome engineering |
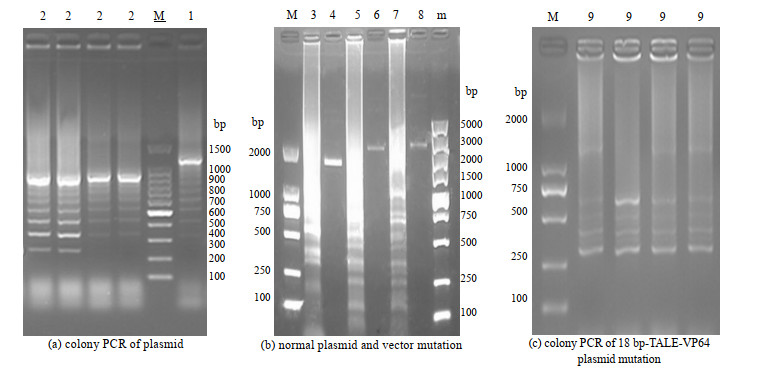
第1部分和第2部分中从每个平板上选取白色菌落,使用引物pCR8-F1和pCR8-R1进行菌落PCR检验。条带应达到预期大小(PFUS-A+10×NH质粒约12 kb、PFUS-B7+7×NH载体约0.8 kb (kb表示1 000碱基对)),但一般不可能得到单一的条带,一般条带“阶梯”从约200 bp开始,这是正确克隆的标志,是克隆中重复的结果[22](见图 4中1和2泳道)。
|
图 4 质粒和菌落PCR产物的琼脂糖凝胶电泳结果
Fig.4 Results of agarose gel electrophoresis of plasmid and colony PCR products
M: DL2 000 DNA marker; m: DL5 000 DNA marker; M: 100-bp DNA ladder. 1. Colony PCR of PFUS-A+10×NH plasmid 2. Colony PCR of PFUS-B7+7×NH plasmid 3: DNA from pLR-NH plasmid vector mutation 4. pLR-NH plasmid 5. DNA from PFUS-B7+7×NH plasmid vector mutation 6. PFUS-B7+7×NH plasmid 7. DNA from PFUS-A+10×NH plasmid vector mutation 8. PFUS-A+10×NH plasmid 9. Final colony PCR of 18 bp-TALE-VP64 |
第3部分中用4种特异引物和长链高保真DNA聚合酶分别扩增PFUS-A+10×NH质粒、PFUS-B7+7×NH和pLR-NH质粒(见图 4中4、6、8泳道),获得各个质粒载体突变体(见图 4中3、5、7泳道),由于质粒突变体实际是PCR产物,并且主要是各种质粒的混合物,但是也不排除有短链的一些PCR产物,所以,从电泳图观察,质粒突变体主要条带的分子量与对应的质粒基本一致。
第4部分中挑取4个白色菌落用菌落PCR检测最终18 bp-TALE-VP64文库。全长PCR产物通常不太突出,大约是2 051 bp,从结果观察,主条带确实在2 051 bp左右,但是不明显。而“阶梯效应”是成功组装的有力指标[22](见图 4中9泳道)。
PFUS-A+10×NH、PFUS-B7+7×NH、pLR-NH骨架质粒分别与各自的质粒突变体不同,3种质粒突变体是各自以3种质粒(PFUS-A+10×NH、PFUS-B7+7×NH和pLR-NH骨架质粒)为模板,加入4种特异性引物经过PCR反应后得到3种PCR产物,PFUS-A+10×NH质粒突变体就是PFUS-A+10× (NH,HD,NG,NI不同排列组合)的混合物,即410种质粒的混合物;PFUS-B7+7×NH质粒突变体就是PFUS-B7+7× (NH,HD,NG,NI不同排列组合)的混合物,即47种质粒的混合物;pLR-NH质粒突变体就是pLR-NH、pLR-HD、pLR-NG、pLR-NI的混合物,即4种质粒的混合物。
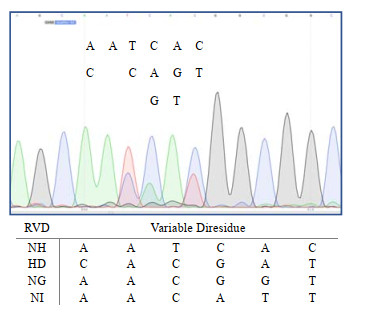
3.3 TALE文库质粒的基因测序将最后提取的18bp-TALE-VP64质粒文库送基因公司测序,最后得到的测序结果见图 5,图中有6个核苷酸的重复序列(RVDs),显示“噪声”信号。RVD结构域预期核苷酸组成与构建的18 bp-TALE-VP64文库一致(图 5)。通过基因测序验证了该文库的所有序列是正确的,并且包含所有可能的DNA靶点的序列组合,该TALE-VP64质粒文库对18个碱基(ATCG)的排列组合都有靶向性,所以就是418,共计68 719 476 736个组合。所以涵盖了可能的核苷酸DNA靶点。因为也有很多研究者构建针对11个碱基或者12个碱基的TALE文库质粒,由于靶向的碱基越多,脱靶的可能性越小,此处主要是体现该质粒文库的高靶向性。另外从测序结果观察,虽然NH对应的序列较多,但是也存在HD、NG、NI对应的序列,由于测序的质粒文库实际是多种质粒的混合物,里面相当于418的各种排列组合的质粒的混合物。注意观察峰图,RVD对应有几个是双峰、三峰,所以从测序结果得出,构建的文库涵盖了预期的RVD组成。
|
图 5 18-bp TALE-VP64文库的DNA测序图谱 Fig.5 DNA sequencing profiles of the 18 bp-TALE-VP64 libraries |
先前文献报道中的构建TALE的方法都是构建独立个体的方法,无论用什么方法来单独构建若干TALE质粒,都不可能形成一个巨大的文库,这些方法是低效和耗时的。在本研究中,建立了一种基于独特引物组装完整、高效的TALE文库构建的方法,与其他技术相比[23-24],TALE靶向DNA目标的长度可以控制。增加DNA靶标的长度将获得更高的特异性,然而,构建TALE文库的成本和时间也将大大增加;另一种增加文库靶向DNA目标长度的方法,不增加额外的实验负担或增加复杂性,包括利用RVD特异性。例如,已知RVD-NN可以同时靶向A和G,因此,只有4个模块(即NH、NI、NG和HD)可用于组装完整的TALE文库。所以,研究构建的这4个模块的18 bp-TALE文库将产生最多68 719 476 736种组合。靶向DNA长度更大的TALE文库的组装在实验上可能很麻烦,在某些情况下,实际上是无法实现的。另一个问题在于,在破译给定的RVD序列的TALE-DNA结合方面存在一定程度的不确定性,这可能也会使全基因组分析中潜在目标序列的识别复杂化,这个问题可以通过染色质免疫共沉淀-测序技术(ChIP-seq)[25]进行进一步研究。总之,本研究成功建立了一种基于TALE的多功能文库的组装方法,构建成功的TALE文库可以利用酵母单杂交试验探索其功能完整性[26]。进一步说明,TALE-DBD可以完全定制,以靶向任何核苷酸含量(如GC含量丰富的DNA区域)、核苷酸限制(如转录因子基序)或其他修饰[27-28]。最后,TALE蛋白的编码序列易于分离和测序,便于快速识别其靶基因。总之,考虑到该组装方法的高度模块化的特性,控制其靶向DNA长度大小从而调整其文库特异性的能力,以及构建的文库的多功能性和方便性,预计未来将有更广泛的应用。
5 结论在本研究中,使用TALE试剂盒和独特设计的引物,建立了一种快速构建TALE文库的新方法。利用该方法构建了可靶向18bp-DNA序列的TALE文库,并对其进行了DNA测序验证,满足预期结果。为基因组调控和基因组编辑技术提供了简单高效的方法和实验工具。
| [1] |
BONAS U, STALL R E, STASKAWICZ B. Genetic and structural characterization of the avirulence gene Avrbs3 from Xanthomonas-Campestris Pv vesicatoria[J]. Molecular and General Genetics MGG, 1989, 218(1): 127-136. DOI:10.1007/BF00330575 |
| [2] |
BOCH J, BONAS U. Xanthomonas AvrBs3 family-type Ⅲ effectors: Discovery and function[J]. Annual Review of Phytopathology, 2010, 48(1): 419-436. DOI:10.1146/annurev-phyto-080508-081936 |
| [3] |
WOOD A J, LO T W, ZEITLER B, et al. Targeted genome editing across species using ZFNs and TALENs[J]. Science, 2011, 333(6040): 307-307. DOI:10.1126/science.1207773 |
| [4] |
MOSCOU M J, BOGDANOVE A J. A simple cipher governs DNA recognition by TAL effectors[J]. Science, 2009, 326(5959): 1501-1501. DOI:10.1126/science.1178817 |
| [5] |
BOCH J, SCHOLZE H, SCHORNACK S, et al. Breaking the code of DNA binding specificity of TAL-Type Ⅲ effectors[J]. Science, 2009, 326(5959): 1509-1512. DOI:10.1126/science.1178811 |
| [6] |
MORBITZER R, RÖMER P, BOCH J, et al. Regulation of selected genome loci using de novo-engineered transcription activator-like effector (TALE)-type transcription factors[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(50): 21617-21622. |
| [7] |
LI T, HUANG S, JIANG W Z, et al. TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain[J]. Nucleic Acids Research, 2011, 39(1): 359-372. DOI:10.1093/nar/gkq704 |
| [8] |
ZHANG F, CONG L, LODATO S, et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription[J]. Nature Biotechnology, 2011, 29(2): 149-153. DOI:10.1038/nbt.1775 |
| [9] |
GEIβLER R, SCHOLZE H, HAHN S, et al. Transcriptional activators of human genes with programmable DNA-Specificity[J]. Plos One, 2011, 6(5): e19509. DOI:10.1371/journal.pone.0019509 |
| [10] |
CERMAK T, DOYLE E L, CHRISTIAN M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Research, 2011, 39(17): 7879-7879. DOI:10.1093/nar/gkr739 |
| [11] |
TESSON L, USAL C, MÉNORET S, et al. Knockout rats generated by embryo microinjection of TALENs[J]. Nature Biotechnology, 2011, 29(8): 695-696. DOI:10.1038/nbt.1940 |
| [12] |
SUN N, LIANG J, ABIL Z, et al. Optimized TAL effector nucleases (TALENs) for use in treatment of sickle cell disease[J]. Molecular Biosystems, 2012, 8(4): 1255-1263. DOI:10.1039/c2mb05461b |
| [13] |
GASIUNAS G, BARRANGOU R, HORVATH P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): E2579-E2586. |
| [14] |
CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [15] |
KOIKE-YUSA H, LI Y L, TAN E P, et al. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library[J]. Nature Biotechnology, 2014, 32(3): 267-273. DOI:10.1038/nbt.2800 |
| [16] |
PARK C Y, KIM J, KWEON J, et al. Targeted inversion and reversion of the blood coagulation factor 8 gene in human iPS cells using TALENs[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25): 9253-9258. |
| [17] |
QASIM W, AMROLIA P J, SAMARASINGHE S, et al. First clinical application of TALEN engineered universal CAR19 T cells in B-ALL[J]. Blood, 2015, 126(23): 2046. DOI:10.1182/blood.V126.23.2046.2046 |
| [18] |
QASIM W, ZHAN H, SAMARASINGHE S, et al. Molecular remission of infant B-ALL after infusion of universal TALEN gene-edited CAR T cells[J]. Science Translational Medicine, 2017, 9(374): eaaj2013. DOI:10.1126/scitranslmed.aaj2013 |
| [19] |
SANDER J D, CADE L, KHAYTER C, et al. Targeted gene disruption in somatic zebrafish cells using engineered TALENs[J]. Nature Biotechnology, 2011, 29(8): 697-698. DOI:10.1038/nbt.1934 |
| [20] |
MUSSOLINO C, MORBITZER R, LÜTGE F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity[J]. Nucleic Acids Research, 2011, 39(21): 9283-9293. DOI:10.1093/nar/gkr597 |
| [21] |
ZHANG S Y, CHEN H T, WANG J K. Generate TALE/TALEN as easily and rapidly as generating CRISPR[J]. Molecular Therapy-Methods & Clinical Development, 2019, 13(1): 310-320. |
| [22] |
HOMMELSHEIM C M, FRANTZESKAKIS L, HUANG M M, et al. PCR amplification of repetitive DNA: A limitation to genome editing technologies and many other applications[J]. Scientific Reports, 2014, 4(1): 5052. DOI:10.1038/srep05052 |
| [23] |
FU Y F, FODEN J A, KHAYTER C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nature Biotechnology, 2013, 31(9): 822-826. DOI:10.1038/nbt.2623 |
| [24] |
MALI P, YANG L, ESVELT K M, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [25] |
FUREY T S. ChIP-seq and beyond: New and improved methodologies to detect and characterize protein-DNA interactions[J]. Nature Reviews Genetics, 2012, 13(12): 840-852. DOI:10.1038/nrg3306 |
| [26] |
LI Y, EHRHARDT K, ZHANG M Q, et al. Assembly and validation of versatile transcription Activator-Like effector libraries[J]. Scientific Reports, 2014, 4(1): 4857. DOI:10.1038/srep04857 |
| [27] |
VALTON J, DUPUY A, DABOUSSI F, et al. Overcoming transcription activator-like effector (TALE) DNA binding domain sensitivity to cytosine methylation[J]. Journal of Biological Chemistry, 2012, 287(46): 38427-38432. DOI:10.1074/jbc.C112.408864 |
| [28] |
DENG D, YIN P, YAN C Y, et al. Recognition of methylated DNA by TAL effectors[J]. Cell Research, 2012, 22(10): 1502-1504. DOI:10.1038/cr.2012.127 |




