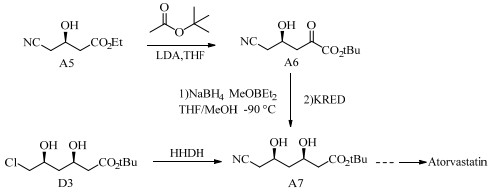
他汀类药物具有良好的降血脂疗效[1],其中的代表性品种阿托伐他汀钙是唯一一个全球年销售额连续多年过百亿美元的药物[2-3]。近年来,文献报道了多种阿托伐他汀钙的合成方法[1, 4],具有光学活性的侧链(3R, 5R)-6-氰基-3, 5-二羟基己酸叔丁酯(A7)是其合成的关键手性砌块(图 1)。
|
图 1 阿托伐他汀关键中间体A7的合成方法 Fig.1 Synthetic route of atorvastatin intermediate (A7) |
传统化学合成路线先由(R)-4-氰基-3-羟基丁酸乙酯(A5)经二异丙基氨基锂(LDA)介导的克莱森缩合制备(5R)-6-氰基-5-羟基-3-羰基己酸叔丁酯(A6),再经NaBH4选择性还原获得A7,反应涉及超低温,工业化应用难度大,且能耗很高[1-2]。现工艺采用羰基还原酶(KRED)催化A6到A7的不对称还原反应,但仍涉及A5到A6的化学反应步骤[2-3],而利用卤醇脱卤酶催化(3R, 5S)-6-氯-3, 5-二羟基己酸叔丁酯(D3)的脱卤氰化反应制备A7反应条件温和,副反应少,极具应用潜力。2013年,陈少云[5]首次利用卤醇脱卤酶HHDH[6]催化D3制备A7,之后王金玉[7]对此HHDH进行分子改造得到突变酶Mut-HHDH(W86I),比酶活约为0.65 U·mg-1(37℃、pH 7.0)。2017年,Luo Yu等[8]对HheC2360 [9]进行分子改造得到突变酶Mut-HheC2360(V84G/W86F),其对D3的催化效率提高了15倍,比酶活约为0.02 U·mg-1(45℃,pH 7.0)。目前卤醇脱卤酶筛选和改造研究中关于催化(S)-4-氯-3-羟基丁酸乙酯(A4)制备A5的比较多[5, 9-10],但是由于底物差异比较大,针对A4筛选和改造得到的卤醇脱卤酶并不适合催化D3制备A7,改造策略基本没有可以借鉴的地方。因此通过筛选和分子改造提高卤醇脱卤酶催化D3制备A7的酶活具有重要的研究意义。
本研究使用基因挖掘技术来筛选对D3有较高催化活性的卤醇脱卤酶,发现来源于Agromyces mediolanus sp.的卤醇脱卤酶HheAAM酶活较高,然后运用定向进化和半理性设计方法确定影响酶活的关键位点,并使用迭代饱和突变(ISM)策略对这些位点进行组合突变以进一步提高酶活,希望获得优势突变酶,为后续研究奠定良好的基础。
2 材料与方法 2.1 菌株和质粒大肠杆菌E. coli BL21(DE3)和表达质粒pET-28a(+)均为本实验室保藏。
2.2 在线工具与软件用BLAST (http://www.ncbi.nlm.nih.gov)进行基因挖掘,用Clustal X进行多序列比对,用Swiss-Model (http://swissmodel.expasy.org)进行蛋白结构模拟,用Discovery Studio 4.0软件进行分子对接。
2.3 卤醇脱卤酶基因的合成、克隆与表达基因均由生工生物工程(上海)股份有限公司合成(针对E. Coli内表达密码子优化),上下游引物均由擎科梓熙生物技术(杭州)有限公司合成。目的基因构建至pET-28a(+)载体的BamH Ⅰ和Hind Ⅲ酶切位点之间。将成功转化的单克隆接种于5 mL LB液体培养基(50 μg·mL-1 Kan)中,37℃、200 r·min-1条件下培养8 h,然后转接到50 mL LB液体培养基(50 μg·mL-1 Kan)中,培养至OD600为0.8~1.0,添加IPTG至终浓度0.5 mmol·L-1,18℃下诱导过夜。
2.4 突变文库的构建 2.4.1 易错PCR文库构建以pET-28a(+)-HheAAM为模板,利用Trans EasyTaq DNA Polymerase试剂盒在HheAAM全长基因上引入随机突变,PCR体系和PCR程序见说明书,以2.3的方式构建重组突变质粒,转化大肠杆菌感受态细胞得到突变体库。
2.4.2 ISM文库构建以pET-28a(+)-HheAAM为模板,利用全质粒PCR方法在HheAAM基因上引入特定位点的突变,Dpn Ⅰ消化后转化大肠杆菌感受态细胞得到突变体库,筛选获得文库中最优突变体,以其质粒作为下一轮PCR的模板,引入新位点的突变。
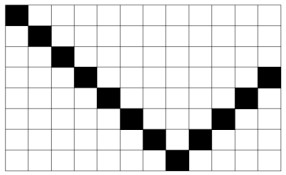
2.5 高通量筛选方法[5, 7]在96浅孔板(母板)的每孔中加200 μL LB液体培养基(50 μg·mL-1 Kan),用灭菌牙签从野生型HheAAM (Wt-HheAAM)平板上挑取单菌落接种到图 2中黑色位置,将突变菌株接种到图 2中空白位置,37℃、200 r·min-1条件下培养8 h。在96深孔板(子板)的每孔中加400 μL LB液体培养基(50 μg·mL-1 Kan),再将母板中的种子液按对应位置转接到子板中(接种量为50 μL),37℃、200 r·min-1培养条件下3 h左右,添加IPTG至终浓度0.5 mmol·L-1,18℃下诱导过夜。转接后母板的每个孔中加入50 μL的50%甘油,-80℃下保藏。
|
图 2 96 孔板中野生型 HheAAM 的位置 Fig.2 Position of Wt-HheAAM in a 96-well plate |
将上述经过诱导表达的子板发酵液离心(4℃、4000 r·min-1、10 min),弃上清,用HEPES缓冲液(100 mmol·L-1、pH 7.5)洗两遍后重悬细胞。在新的96浅孔板(反应板)的每个孔中加入250 μL HEPES缓冲液,从子板中取50 μL细胞重悬液按照对应位置加入反应板中,加底物D3至终浓度10 mmol·L-1,反应30 min (37℃、200 r·min-1)。反应结束后离心(4℃、4000 r·min-1、10 min),取40 μL反应上清液,加60 μL饱和Hg(SCN)2-乙醇溶液和150 μL浓度为60 g·L-1 FeNH4(SO4)2-1 mol·L-1硝酸溶液进行显色反应,用酶标仪测定460 nm处吸光值,挑取吸光值相对于Wt-HheAAM的吸光值增大的突变株进行复筛检测。复筛时将保藏的菌株划线得到单菌落,按2.3节方法诱导表达,按2.7节方法测定酶活。
2.6 目的蛋白的纯化与浓度测定将成功诱导表达的菌体超声破碎,离心取上清,过滤后缓慢上柱,先用缓冲液A (20 mmol·L-1 Na2HPO4-NaH2PO4、500 mmol·L-1 NaCl、50 mmol·L-1咪唑、pH 7.5)洗涤,再用缓冲液B (20 mmol·L-1 Na2HPO4-NaH2PO4、500 mmol·L-1 NaCl、含50 mmol·L-1、100 mmol·L-1、250 mmol·L-1咪唑、pH 7.5)梯度洗脱,收集250 mmol·L-1咪唑的洗脱峰。用Amicon Ultra-15 mL超滤管(截留分子量为30 kDa) 3000 r·min-1下离心浓缩,加15mL缓冲液C (100 mmol·L-1 HEPES、pH 7.5),离心稀释高浓度咪唑,重复一次,加入终浓度10%的甘油,-80℃下保存。蛋白浓度用考马斯亮蓝法测定[11]。
2.7 酶活测定500 μL反应体系(100 mmol·L-1 HEPES、pH 7.5)中,含10 mmol·L-1底物(D3),30 mmol·L-1 NaCN以及适量酶,37℃反应30 min,加入500 μL乙腈终止反应,过滤后进行液相检测,色谱柱采用PntulipsTM QS-C18(5 μm×4.6 mm×250 mm),流动相采用乙腈与10 mmol·L-1乙酸钠溶液(pH 5.5),它们的体积之比为3:7,流速为1.0 mL·min-1,进样量为5 μL,检测波长为210 nm,柱温为37℃。底物D3保留时间为14.3 min,产物A7保留时间为8.6 min。
酶活单位(U)定义:在所述条件下,每分钟转化生成1 μmol产物(A7)所需的酶量。酶活和比酶活计算公式如下:
| $ EA = \frac{{{c_{\rm{p}}} \times V \times n}}{{t \times {V_{\rm{E}}}}} $ |
| $ SA = \frac{{{c_{\rm{p}}} \times V \times n}}{{t \times {m_{\rm{E}}}}} $ |
式中,EA表示酶活(U·L-1),SA表示比酶活(U·g-1),cp表示产物浓度(μmol·L-1),V表示反应体系体积(L),n表示反应液稀释倍数,t表示反应时间(min),VE表示所加的酶体积(L),mE表示所加的酶量(g)。
2.8 动力学参数测定测定纯酶在不同浓度底物(1~10 mmol·L-1 D3)下的酶活,采用双倒数作图法[11]计算得到目标蛋白的动力学参数,酶活测定方法如2.7节。
3 结果与分析 3.1 卤醇脱卤酶的基因挖掘卤醇脱卤酶可分为HheA、HheB、HheC 3类,不同类型的卤醇脱卤酶底物特异性差别较大。HheA和HheB对长链(C5和C6)以及含大取代基的底物表现出较高催化活性,而HheC则偏好催化短链(C2和C3)底物的转化[12],但是目前催化D3制备A7的研究报道中所用均为HheC类型。本研究克隆表达了多个卤醇脱卤酶(HheA[13],HheAAD2[12],HheB[13],HheBGP1以及HheC[12, 14])并测定了酶活,发现HheA相对于HheB、HheC对底物D3表现出更好的催化活性(表 1)。
|
|
表 1 卤醇脱卤酶的酶活测定 Table 1 Enzyme activities of halohydrin dehalogenases |
因此本研究以HheA为筛选模板,在NCBI上筛选与HheA、HheAAD2序列同源性超过30%的基因序列。将筛选得到的基因序列进行多序列比对,挑选出符合卤醇脱卤酶序列模式(T-X4-(F/Y)-X-G- X93-131-S-X12-Y-X3-R)[15]的基因序列(HheAAM、HheA4~HheA12),人工合成这些序列,克隆表达后测定酶活。由表 1可见,除HheA7和HheA10~12外,所筛选出的卤醇脱卤酶均对底物D3表现出一定的催化活性,其中HheAAM酶活最高(约为13 U·L-1),高于现有报道的HheC类型的HHDH[5-6]和Mut-HheC2360 (V84G/W86F)[8],因此选择HheAAM为对象进行后续研究。总的来说,卤醇脱卤酶对D3的酶活不高,这可能与D3本身较大的侧链结构有关,导致底物难以进入酶催化中心附近,因此需要进一步对酶进行改造。
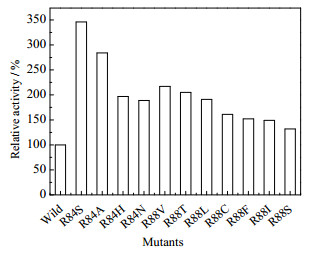
3.2 突变位点的设计与选择 3.2.1 定向进化由于目前鲜有通过酶改造提高卤醇脱卤酶HheA对D3的酶活的报道,缺乏可借鉴的突变热点。为提高HheAAM的催化活性,采用易错PCR方法构建随机突变文库,筛选约5000个突变株后得到两个阳性突变株R84A和R88L,酶活分别是Wt-HheAAM的2.84和1.91倍,说明84位和88位是影响HheAAM酶活的关键位点。对84位和88位分别进行饱和突变(NNK/NNS),得到了其他9个阳性突变株,酶活分别是Wt-HheAAM的1.32~3.46倍(图 3)。
|
图 3 定向进化方法得到的阳性突变株及其相对酶活 Fig.3 Relative activity of mutants obtained by directed evolution |
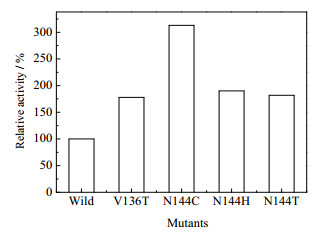
由于易错PCR方法筛选工作量大,获得阳性突变株概率低,故继而采用半理性设计方法继续寻找其他关键位点。经分子对接发现,以底物为中心2.5 Å范围内一共有9个氨基酸(F12、N87、S134、V136、L141、N144、Y147、P177、N178)。其中,S134和Y147是卤醇脱卤酶的催化活性中心,故对其他7个氨基酸所在位点进行饱和突变,筛选约700个突变子后得到4个阳性突变株(136T、144C、144H、144T),如图 4所示,酶活分别提高1.78~3.13倍,说明136位和144位也是影响HheAAM酶活的关键位点。
|
图 4 半理性设计方法得到的阳性突变株及其相对酶活 Fig.4 Relative activity of mutants obtained by semi-rational design |
由于上述单点突变株酶活提高不明显,所以尝试对这4个影响酶活的关键位点(84、88、136、144位)进行组合突变。若简单地将每个位点的最优突变株直接组合则不能考虑到各位点间的相互影响;若对四个位点进行随机组合突变,即使选用NDT密码子,也需要筛选超过60000个转化子[16]。因此,本研究使用迭代饱和突变策略(ISM)[17]对之前找到的关键位点行组合突变,按一定迭代顺序在一个位点的最优突变株上引入另一个位点的饱和突变,避免了直接组合可能带来的消极作用,也大大降低了筛选工作量[18]。
由于对136位进行饱和突变只筛得一个阳性突变株M1(V136T),故以其为第一轮最优突变株,逐级往上叠加突变位点(图 5)。而88位处在一个与136位相似的空间位置,靠近底物叔丁基的另一个甲基,所以首先在M1上引入88位的饱和突变,得到第二轮最优突变株M2(88L-136T),然后在M2上分别引入144位和84位饱和突变,得到最优突变株M3-1(88L-136T-144C)和M3-2(84S-88L-136T)。最后在M3-1和M3-2上分别引入84位和144位饱和突变,得到的是同一个最优突变株M4(84S-88L-136T-144C),其比酶活为887.80 U·g-1,是Wt-HheAAM的13倍(表 2)。
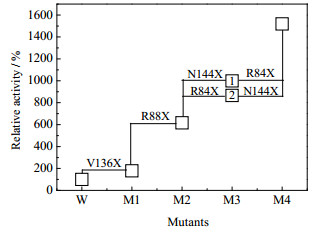
|
图 5 ISM策略组合突变得到的阳性突变株及其相对酶活 Fig.5 Relative activity of positive mutants obtained by ISM strategy |
|
|
表 2 Wt-HheAAM和Mut-HheAAM的酶活与比酶活测定 Table 2 Enzyme activities and specific activities of Wt-HheAAM and Mut-HheAAM |
Wt-HheAAM与Mut-HheAAM的动力学参数如表 3所示。分别比较M2与M3-1、M3-2可见,144C和84S突变均会导致Km(米氏常数)的降低和kcat(反应常数)的升高,144C突变则更倾向于提高酶与底物亲和力(Km↓),而84S突变更倾向于加快酶转化底物的速率(kcat↑)。由M2可知,88L-136T突变使kcat升高的同时导致了Km的升高,说明酶转化底物的速率变快而酶与底物的亲和力变差了。但是88L-136T突变对底物结合的负性影响可以通过84S和144C突变相抵消,所以突变体M4的Km值降低,其催化效率(kcat·Km-1)是Wt-HheAAM的17倍。
|
|
表 3 Wt-HheAAM和Mut-HheAAM的动力学参数测定 Table 3 Kinetic parameters of Wt-HheAAM and Mut-HheAAM |
以与HheAAM序列同源性为97.54%的HheA酶晶体结构模型(PDB code: 4z9f.1.A)为模板,用Swiss- Model对HheAAM进行同源建模。用Discovery Studio 4.0中的Ramachandran评价蛋白模型的合理性,再将蛋白与底物D3用CDOCKER进行分子对接,选择合适的模型进行后续分析(图 6)。
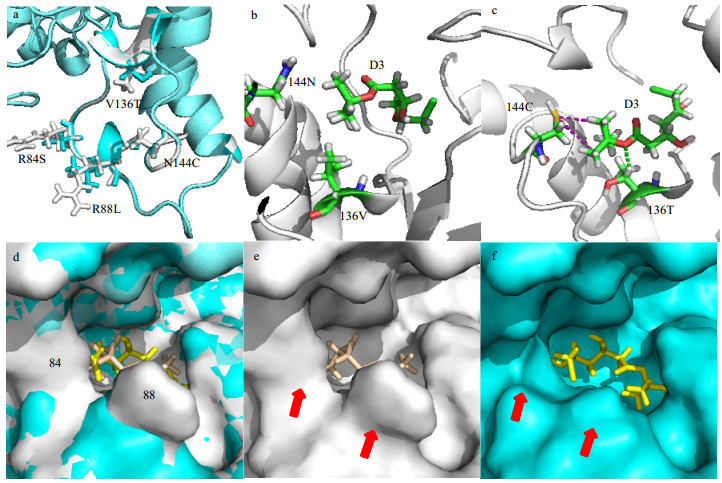
|
图 6 Wt-HheAAM与M4-HheAAM的结构模拟 Fig.6 Structural simulation results of Wt-HheAAM and M4-HheAAM a. an overview of mutation sites. Wt-HheAAM and M4-HheAAM were colored with white and cyan, respectively. b.136V and 144N in Wt-HheAAM c.136T and 144C in M4-HheAAM d-f. substrate binding pockets. Wt-HheAAM and M4-HheAAM were colored with white and cyan, and their substrate were colored with yellow and peachpuff, respectively |
HheAAM酶分子在底物与活性中心结合口袋附近有3条loop结构(图 6(a)),84位与88位处于loop1上,136位和144位处于loop2上,而针对loop3上的位点进行改造并没有得到阳性突变株。136位和144位虽然处于活性中心附近,但是136V和144N与底物之间并无明显的相互作用力,当136位的Val变为Thr后能与底物D3形成一个氢键,144位的Asn变为Cys后能与底物D3的叔丁基产生两个疏水作用力,可以更好地结合底物,从而提高酶与底物的亲和力(图 6(b)、(c))。84位和88位在空间结构中同活性位点并无直接作用,但是当84位和88位由体积较大的Arg变为体积较小的Ser和Leu时,会导致loop1构象发生轻微变化,底物进入酶活性中心的通道变大,空间位阻减小,从而使得酶转化底物的速率加快,所以84S和88 L突变后kcat增大明显(图 6(d)~(f))。而根据3.4.1节测得的动力学参数可知88L-136T突变实际上在加快底物转化速率的同时导致酶与底物亲和力变差,而136T突变会导致酶与底物结合增强,推测88 L突变可能导致底物结合口袋变大,从而导致酶催化中心与底物之间相对距离变远,影响酶与底物的结合。
4 结论本研究利用基因挖掘手段筛选获得一系列卤醇脱卤酶,其中HheAAM(来源于Agromyces mediolanus sp.)对底物D3表现出相对较好的催化活性。为提高HheAAM对D3的酶活对其进行改造,结合定向进化和半理性设计方法找到4个影响酶活的关键位点,使用ISM策略对这些位点进行组合突变,突变株M4-HheAAM(84S-88L-136T-144C)的比酶活为Wt-HheAAM的13倍。通过动力学参数测定和分子模拟发现,84S和88L突变会导致底物通道变大,使得酶转化底物的速率加快,而136T和144C突变则会导致酶与底物亲和力提高。
卤醇脱卤酶催化的脱卤氰化反应包括卤代醇脱卤反应和环氧化物开环反应,本研究所采用高通量筛选方法是基于Cl-的释放建立的,后续研究中可以尝试建立针对环氧化物开环的高通量筛选方法,提高环氧中间态开环上氰基的速度,从而提高产物A7的生产速率。
| [1] | WANG Zheng-lin(王正林), YING Jun(应俊), LIN Sheng-chao(林圣超), et al. Progress in synthesis of atorvastatin calcium(阿托伐他汀钙的合成研究进展)[J]. Chinese Journal of Modern Applied Pharmacy(中国现代应用学), 2011, 28(5): 423-428. |
| [2] | Bornscheuer U T, Huisma G W, Kazlauskas R J, et al. Engineering the third wave of biocatalysis[J]. Nature, 2012, 485(7397): 185. DOI:10.1038/nature11117. |
| [3] | Bommarius A S. Biocatalysis:a status report[J]. Annual Review of Chemical & Biomolecular Engineering, 2015, 6(1): 319. |
| [4] | Mothana B, Boyd R J. A density functional theory study of the mechanism of the Paa-Knorr pyrrole synthesis[J]. Journal of Molecular Structure Theochem, 2007, 811(1): 97-107. |
| [5] | CHEN Shao-yun (陈少云). Multi-enzymatic synthesis of chiral β-hydroxynitriles with oxidoreductase and halohydrin dehalogenase (氧化还原酶-卤代醇脱卤酶多酶法合成手性β-羟基腈)[D]. Hangzhou (杭州): Zhejiang University (浙江大学), 2013. |
| [6] | Davis C S, Fox R J, Huisman G W, et al. Halohydrin dehalogenases and related polynucleotides: US, 7588928[P]. 2009. |
| [7] | WANG Jin-yu (王金玉). Improving the catalytic activity of halohydrin dehalogenase HHDH by directed evolution (定向进化提高卤代醇脱卤酶HHDH的催化活性)[D]. Hangzhou (杭州): Zhejiang University (浙江大学), 2016. |
| [8] | Luo Y, Chen Y Z, Ma H M, et al. Enhancing the biocatalytic manufacture of the key intermediate of atorvastatin by focused directed evolution of halohydrin dehalogenase[J]. Scientific Reports, 2017. |
| [9] | Schallmey M, Floor R J, Hauer B, et al. Biocatalytic and structural properties of a highly engineered halohydrin dehalogenase[J]. ChemBioChem, 2013, 14(7): 870-881. DOI:10.1002/cbic.201300005. |
| [10] | Chen S Y, He X J, Wu J P, et al. Identification of halohydrin dehalogenase mutants that resist COBE inhibition[J]. Biotechnology & Bioprocess Engineering, 2014, 19(1): 26-32. |
| [11] | Xue F, Liu Z Q, Wang Y J, et al. Biochemical characterization and biosynthetic application of a halohydrin dehalogenase from Tistrella mobilis ZJB1405[J]. Journal of Molecular Catalysis B Enzymatic, 2015, 115: 105-112. DOI:10.1016/j.molcatb.2015.02.008. |
| [12] | van Hylckama Vlieg J E, Tang L, Lutje Spelberg J H, et al. Halohydrin dehalogenases are structurally and mechanistically related to short-chain dehydrogenases/reductases[J]. Journal of bacteriology, 2001, 183(17): 5058-5066. DOI:10.1128/JB.183.17.5058-5066.2001. |
| [13] | Yu F, Nakamura T, Mizunashi W, et al. Cloning of two halohydrin hydrogen-halide-lyase genes of Corynebacterium sp. strain N-1074 and structural comparison of the genes and gene products[J]. Biosci Biotechnol Biochem, 1994, 58(8): 1451-1457. DOI:10.1271/bbb.58.1451. |
| [14] | Higgins T P, Hope S J, Effendi A J, et al. Biochemical and molecular characterisation of the 2, 3-dichloro-1-propanol dehalogenase and stereospecific haloalkanoic dehalogenases from a versatile Agrobacterium sp.[J]. Biodegradation, 2005, 16(5): 485-492. DOI:10.1007/s10532-004-5670-5. |
| [15] | Schallmey M, Koopmeiners J, Wells E, et al. Expanding the halohydrin dehalogenase enzyme family:identification of novel enzymes by database mining[J]. Applied & Environmental Microbiology, 2014, 80(23): 7303. |
| [16] | Sun Z T, Lonsdale R, Kong X D, et al. Reshaping an enzyme binding pocket for enhanced and inverted stereoselectivity:use of smallest amino acid alphabets in directed evolution[J]. Angewandte Chemie, 2015, 54(42): 12410-12415. DOI:10.1002/anie.201501809. |
| [17] | Reetz M T, Kahakeaw D, Sanchis J. Shedding light on the efficacy of laboratory evolution based on iterative saturation mutagenesis[J]. Molecular Biosystems, 2009, 5(2): 115-122. DOI:10.1039/B814862G. |
| [18] | Valetti F, Gilardi G. Improvement of biocatalysts for industrial and environmental purposes by saturation mutagenesis[J]. Biomolecules, 2013, 3(4): 778. DOI:10.3390/biom3040778. |




