近年来,随着生物技术的快速发展,基因治疗在生物医药领域越来越受到关注,目前已有多个基因治疗产品获得美国食品和药物管理局(FDA)或欧洲药品管理局(EMA)批准[1]。
质粒DNA(pDNA)是一种小型环状DNA,在基因治疗中可以作为DNA疫苗直接参与疾病防控,也可以作为病毒载体或mRNA等药物的生产原料[2]。pDNA存在三种不同构象:超螺旋(supercoiled,sc),开环(open circular,oc)和线性(linear,ln)。作为基因治疗药物,pDNA存在转染效率低的缺点,大约1 000个分子中只有1个能进入细胞并被表达[3]。研究表明,超螺旋质粒DNA (supercoiled plasmid DNA,sc pDNA)具有更高的细胞转染效率[4],因此,如何制备高同质性即高超螺旋比例的pDNA是关键。FDA对pDNA产品中杂质含量做了详细规定[5],其中RNA因电荷和疏水性存在较大差异而较容易分离,oc pDNA和sc pDNA相对分子质量及总带电量相近,差别仅在于局部电荷密度以及sc pDNA过旋引起的碱基暴露[6],因此如何有效分离oc pDNA和sc pDNA成为pDNA纯化过程的关键。
pDNA分离方法有沉淀[7]、膜分离[8]、双水相萃取[9]、固相萃取[10]、磁性纳米颗粒吸附[11]等,但普遍存在分离效果有限、收率低等问题,而层析具有更精细的分离效果。Valente等[12]使用精氨酸大孔亲和层析,从澄清的细胞裂解液中分离出pcDNA3-FLAG-p53(碱基对数量为6 070 bp),采用NaCl浓度cNaCl=120 mmol·L−1的洗脱液使oc pDNA和RNA洗脱,再升高cNaCl至160 mmol·L−1使sc pDNA洗脱,得到的sc pDNA纯度为92%,但收率仅为43%;作者进一步通过试验设计法(DoE)优化制备了O-磷-L-酪氨酸介质,sc pDNA纯度提高到98.2%,但收率仍只有49.7% [13]。Santos等[14]采用L-酪氨酸作为亲和配基分离pPH600质粒(碱基对数量为3 562 bp),纯度为98.2%、收率为56.3%,但介质的动态载量较低。Bicho等[15]采用乙二胺作为离子交换配基制备整体柱用于pDNA分离,sc pDNA纯度为97.1%、收率为47.0%。疏水相互作用层析也有报道[16],可达到98%的同质性和96%的收率,但需要偶联其他分离步骤。整体而言,sc pDNA分离仍有较大挑战,难以同时达到高纯度与高收率。
混合模式层析是一种兼有多种分子相互作用的新型层析分离方法[17-20]。pDNA分离涉及的3种主要核酸成分既有荷电性质的差异,也有疏水性的差异,因而利用混合模式层析纯化更具有优势。Silva-Santos等[21]使用Capto adhere介质,得到92.2%同质性的sc pDNA;又采用胍丁胺整体柱,获得纯度为98.3%的sc pDNA,收率为51.8%[22]。近些年新推出的混合模式介质Capto PlasmidSelect是以2-巯基吡啶为配基,兼有离子和疏水相互作用,对sc pDNA具有较好的选择性。本研究针对sc pDNA分离,采用Capto PlasmidSelect作为混合模式介质,澄清的细胞裂解液直接上样,考察该介质对分离过程中主要成分sc pDNA、oc pDNA和RNA的吸附和分离性能,优化冲洗、洗脱、上样量等分离条件,实现sc pDNA的高效分离。
2 材料与方法 2.1 材料混合模式介质Capto PlasmidSelect,Cytiva公司;质粒pVAX1(碱基对数量为3.0 kbp),武汉淼灵生物科技有限公司;质粒纯化中量试剂盒,上海生工生物工程有限公司;CIMac pDNA分析柱,BIA Separations公司;超滤离心管(0.5 mL,截留分子量为10 000),Millipore公司;其他试剂为市售分析纯。AKTA层析系统,GE Healthcare公司。
2.2 菌体培养取少量经pVAX1转化的DH5α大肠杆菌,在含有卡那霉素100 mg·mL−1的固体溶菌肉汤(LB)培养基上37 ℃下活化24 h,挑取单菌落经两级扩大培养,以2%的接种量接入250 mL、含100 mg·mL−1卡那霉素的液体LB培养基,在220 r·min−1、37 ℃条件下摇床培养12~16 h。
2.3 pDNA初步提取采用改良的碱裂解法进行pDNA初步分离。细菌培养液经4 000 r·min−1离心30 min,取沉淀物用10 mL溶液I(葡萄糖浓度cGlu=50 mmol·L−1、三羟甲基氨基甲烷浓度cTris= 25 mmol·L−1、乙二胺四乙酸浓度cEDTA=10 mmol·L−1,pH=8.0)重悬,加入15 mL溶液II(cNaOH = 0.2 mol·L−1、SDS的质量浓度为0.01g·mL−1)进行裂解,室温下放置5 min,立即加入12 mL溶液III(醋酸钾浓度cKAc= 3 mol·L−1,pH=5.5)中和,此时产生大量絮状沉淀。冰浴20 min,10 000 r·min−1下离心30 min,取上清液加入0.6倍体积的异丙醇,室温放置30 min使其充分沉淀,10 000 r·min−1下离心20 min,将收集到的沉淀物用少量质量分数为70%的乙醇洗涤,自然挥发掉多余的乙醇。用5 mL、cTris=10 mmol·L−1的Tris-HCl缓冲液(pH=8.0)重悬,加入(NH4)2SO4至c(NH4)2SO4= 2.5 mol·L−1,冰浴15 min使部分RNA和蛋白质析出,10 000 r·min−1下离心20min,得到上清液作为初分离料液,用于后续分离。料液中含有sc pDNA 0.82 mg·mL−1、oc pDNA 0.16 mg·mL−1和RNA 1.63 mg·mL−1。
2.4 静态吸附测定取适量Capto PlasmidSelect介质,用去离子水充分清洗,抽干后用c(NH4)2SO4= 2.5 mol·L−1、cTris=10 mmol·L−1的缓冲液(pH=7.5)平衡30 min,再抽滤15 min。称取约0.03 g介质置于2 mL离心管,将上述初分离料液pH值调至7.5,稀释成8种不同浓度,分别取0.8 mL加入装有介质的离心管,25℃、1 200 r·min−1下恒温混匀仪振荡3 h,充分吸附达到平衡;3 000 r·min−1下离心2 min,使吸附介质沉降,得到上清液,经孔径为0.22 μm的滤膜过滤,用高效液相色谱(HPLC)分析各组分的平衡质量浓度ρ(μg·mL−1)。根据物料衡算得到sc pDNA、oc pDNA和RNA这3种核酸的吸附量Q(mg·g−1),采用Langmuir吸附平衡式对吸附等温线进行拟合,得到饱和吸附量Qm(mg·g−1)和结合常数Ka(mL·μg−1),具体如下:
| $ Q=\frac{\rho Q_{\mathrm{m}}}{\rho+1 / K_{\mathrm{a}}} $ | (1) |
将Capto PlasmidSelect介质1 mL装填于Tricorn 5/50层析柱(Cytiva公司)。初分离料液pH值调至7.5,经孔径为0.45 μm的滤膜过滤后作为上样料液,上样流速为200 cm·h−1。上样前用c(NH4)2SO4=2.5 mol·L−1的平衡缓冲液(pH=7.5)充分平衡介质,上样后收集流穿组分,用HPLC分析流穿液中sc pDNA、oc pDNA和RNA的质量浓度,分别绘制穿透曲线,计算10% 穿透时的动态载量。上样完成后,用cEDTA=10 mmol·L−1、cTris=100 mmol·L−1的缓冲液(pH=7.5)洗脱,并用cNaOH =1 mol·L−1的清洗液进行清洗再生。
2.6 分离条件优化用cEDTA=10 mmol·L−1、cTris=100 mmol·L−1的缓冲液(pH=7.5)配制5种不同浓度的(NH4)2SO4与NaCl的混合盐溶液(如1.5 mol·L−1(NH4)2SO4与0.5 mol·L−1 NaCl混合溶液,其他配比为1.6+0.4、1.7+0.3、1.8+0.2、1.9+0.1)作为洗脱液。首先以 c(NH4)2SO4=2 mol·L−1、cTris=100 mmol·L−1的缓冲液(pH =7.5)作为冲洗液,考察上述混合盐洗脱液的浓度对洗脱效果的影响,即收集洗脱液,HPLC分析sc pDNA纯度、同质性和收率。确定洗脱条件后,进一步考察冲洗条件和不同上样量(2、5、10 mL)对纯化效果的影响。
2.7 凝胶电泳称取0.4 g琼脂粉溶于50 mL 1xTAE缓冲液制成凝胶,加入体积分数为1%的核酸染料4S Green Plus。样品上样量为6 μL (5 μL样品+1 μL DNA上样缓冲液),DNA Marker上样量为5 μL,110 V恒压电泳35 min,黑暗条件紫外灯下观察电泳条带并拍照,用Image J软件对图像进行分析。
2.8 HPLC分析分析柱为CIMac pDNA 0.3 mL Analytical Column,上样条件为cNaCl=0.725 mol·L−1、cTris=10 mmol·L−1、pH=8.0,洗脱梯度设置为cNaCl=0.725~0.85 mol·L−1,最后用cNaCl=1 mol·L−1的溶液进行再生。流量为1 mL·min−1,上样体积为20 μL,检测波长为260 nm,室温下进行。sc pDNA的纯度和同质性计算式如下:
| $ \text { Purity }=\frac{A_{\mathrm{sc}}}{A_{\mathrm{all}}} $ | (2) |
| $ \text { Homogenity }=\frac{A_{\mathrm{sc}}}{A_{\mathrm{sc}}+A_{\mathrm{oc}}} $ | (3) |
式中:Asc、Aoc、Aall分别为sc pDNA、oc pDNA和全部峰面积。
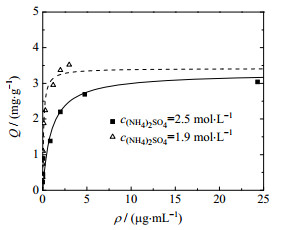
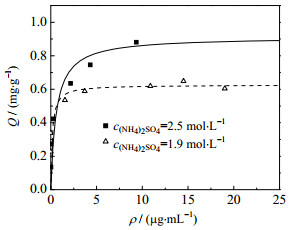
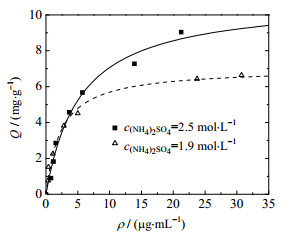
3 结果与讨论 3.1 静态吸附性能初分离料液中含有sc pDNA、oc pDNA,以及未去除完全的RNA和少量其他杂质,下面考察Capto PlasmidSelect介质对不同组分的静态吸附情况。根据预实验的结果,首先选择c(NH4)2SO4=2.5 mol·L−1、cTris=10 mmol·L−1的缓冲液(pH=7.5)。为了考察盐浓度对吸附的影响,还比较了c(NH4)2SO4=1.9 mol·L−1时的吸附,具体结果见图 1~3。由图 1可见,两种盐浓度下的Qm比较接近,分别为3.3和3.4 mg·g−1,这表明盐浓度对sc pDNA吸附影响较小。但结合常数Ka有差异,比较3个组分吸附情况,可以发现c(NH4)2SO4从2.5降到1.9 mol·L−1时,sc pDNA吸附量略有增高,而oc pDNA和RNA则有所降低,原因可能在于本研究采用的是混合模式介质,配基为2-巯基吡啶,与目标物结合存在疏水、静电和亲硫等复杂作用,sc pDNA、oc pDNA、RNA在电荷密度(sc pDNA > oc pDNA > RNA)及疏水性(RNA > sc pDNA > oc pDNA)方面存在差别,因此,随盐浓度增大,sc pDNA呈现与oc pDNA和RNA不同的变化趋势。
|
图 1 Capto PlasmidSelect介质对sc pDNA的吸附等温线(pH=7.5) Fig.1 Adsorption isotherm curves of sc pDNA on Capto PlasmidSelect resin at pH=7.5 |
|
图 2 Capto PlasmidSelect介质对oc pDNA的吸附等温线(pH=7.5) Fig.2 Adsorption isotherm curves of oc pDNA on Capto PlasmidSelect resin at pH=7.5 |
|
图 3 Capto PlasmidSelect介质对RNA的吸附等温线(pH=7.5) Fig.3 Adsorption isotherm curves of RNA on Capto PlasmidSelect resin at pH=7.5 |
根据上述结果,当初分离料液中c(NH4)2SO4=2.5 mol·L−1时,可直接作为上样料液,此时,目标物sc pDNA有较好的吸附能力;c(NH4)2SO4=1.9 mol·L−1的溶液可作冲洗液,能去除大部分oc pDNA,从而提高sc pDNA的分离效果。由于RNA在c(NH4)2SO4=2.5或1.9 mol·L−1的溶液中都具有较高的结合能力,故尚需优化洗脱,以便更好地进行核酸分离。
3.2 动态吸附性能在静态吸附结果基础上,进一步考察pH=7.5、c(NH4)2SO4=2.5 mol·L−1的含pDNA的料液动态载量,选择线性流速为200 cm·h−1,三组分的穿透曲线如图 4所示,ρ0为上样料液的质量浓度,mg·mL−1。由图可见,sc pDNA和oc pDNA达到10%穿透的时间相近,其中oc pDNA略早一些,表明二者可能不存在竞争性吸附。相对而言,sc pDNA穿透曲线更平缓,表明吸附能力强,这可能与超螺旋结构空间位阻较小有关。sc pDNA的动态载量为每毫升介质负载2.00 mg,oc pDNA为每毫升介质负载0.32 mg。图中还可以发现,当sc pDNA和oc pDNA接近饱和吸附状态时,RNA仅有少量穿透(小于5%),这与3.1节的静态吸附结果一致,表明RNA对pDNA吸附没有显著影响,推测RNA吸附有可能为非特异性吸附,并不占据pDNA的吸附位点。
|
图 4 200 cm·h−1流速下Capto PlasmidSelect介质对sc pDNA、oc pDNA和RNA的穿透曲线 Fig.4 Breakthrough curves of sc pDNA, oc pDNA and RNA on Capto PlasmidSelect resin at 200 cm·h−1 linear velocity |
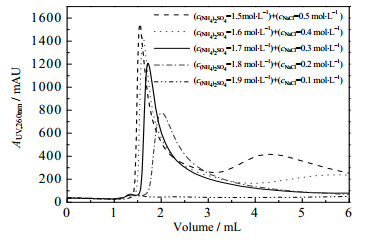
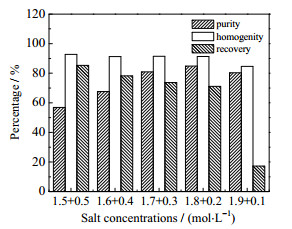
根据sc pDNA、oc pDNA和RNA在电荷密度(sc pDNA > oc pDNA > RNA)及疏水性(RNA > sc pDNA > oc pDNA)方面的差异,以及预实验结果,发现对于混合模式介质Capto PlasmidSelect,适当的c(NH4)2SO4可以促进sc pDNA结合,降低c(NH4)2SO4和添加少量NaCl可以改变sc pDNA和oc pDNA与介质的结合状态。因此,选择在c(NH4)2SO4=2 mol·L−1与(c(NH4)2SO4=1.5 mol·L−1)+(cNaCl=0.5 mol·L−1)之间设置等梯度的洗脱条件,以便考察不同盐浓度对sc pDNA纯度、同质性和收率影响。上样2 mL、c(NH4)2SO4=2 mol·L−1下冲洗,不同盐浓度的等梯度洗脱曲线见图 5,AUV, 260nm表示260 nm下的紫外响应值。由图可见,(c(NH4)2SO4=1.9 mol·L−1)+(cNaCl=0.1 mol·L−1)条件下,sc pDNA无法洗脱。随着c(NH4)2SO4降低以及cNaCl升高,洗脱条件逐渐苛刻,质粒峰后会跟随一个不可忽略的峰,如图中(c(NH4)2SO4=1.5 mol·L−1)+(cNaCl=0.5 mol·L−1)时的曲线,经电泳分析发现该峰富含RNA,从而影响sc pDNA纯度。此外,发现不同盐浓度洗脱条件对sc pDNA的同质性无显著影响。综合比较纯度、同质性和收率(见图 6),选择(c(NH4)2SO4=1.7 mol·L−1)+(cNaCl= 0.3 mol·L−1)为最佳洗脱条件,此时sc pDNA纯度为80.9%、同质性达到91.5%、收率为73.7%。
|
图 5 不同盐浓度的洗脱HPLC谱图 Fig.5 HPLC chromatogram of the elution step under different molar salt concentrations |
|
图 6 不同盐浓度的洗脱效果比较 Fig.6 Comparison of elution effect under different molar salt concentrations |
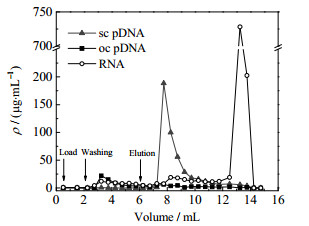
最佳洗脱条件下sc pDNA、oc pDNA和RNA三组分的层析分离结果见图 7。由图可见,在洗脱开始阶段,oc pDNA少量解吸,然后sc pDNA大量洗脱,因此收集洗脱液应以oc pDNA的小峰之后、sc pDNA峰之前为起点。RNA始终有一个小的洗脱平台,只在sc pDNA之后才大量洗脱。综合考虑收率和纯度,确定合适的收集洗脱液区间应为sc pDNA出峰之后的2~3 mL。
|
图 7 sc pDNA、oc pDNA和RNA的层析分离图谱(2 mL上样) Fig.7 Chromatogram of sc pDNA, oc pDNA and RNA separation with 2 mL loading volume |
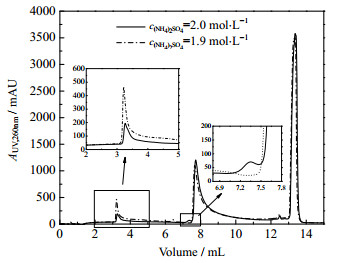
上述结果表明,c(NH4)2SO4=2.0 mol·L−1下的冲洗效果不佳。为了减少洗脱液收集组分中oc pDNA含量,需进一步对冲洗阶段盐浓度进行优化。不同c(NH4)2SO4下冲洗的层析分离谱图如图 8所示。由图可见,将冲洗液中c(NH4)2SO4由2.0 mol·L−1略微降低至1.9 mol·L−1,可以冲洗出更多的oc pDNA,同时不会影响sc pDNA。原本在洗脱阶段才能解吸的部分oc pDNA在冲洗过程中被去除,降低了洗脱峰中杂质oc pDNA的含量。优化后,洗脱峰中sc pDNA纯度从80.9%提高至87.6%、同质性从91.5% 提高至95.7%、收率则基本保持不变。
|
图 8 不同盐浓度冲洗的层析分离谱图 Fig.8 Chromatogram of washing step under different molar salt concentrations |
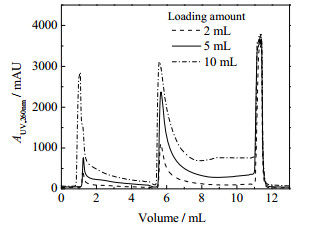
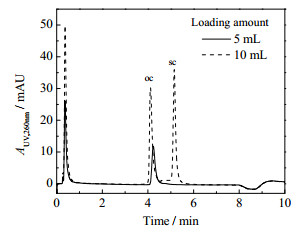
根据动态吸附实验结果,合适的上样量为5~10 mL,既达到较大处理量,又避免了sc pDNA穿透。采用优化的冲洗和洗脱条件,比较了不同上样量的sc pDNA分离结果(见图 9)。由图可见,上样量从2 mL增大到5 mL,对各组分的吸附和洗脱分离行为没有显著影响,仅仅为量级的差别。当上样量继续增大至10 mL时,冲洗峰出现异常增大,洗脱峰也较预期偏小。再通过HPLC分析(见图 10)发现,冲洗峰中含有大量sc pDNA,导致收率显著降低到59.2%。可能原因是,随上样量增大,部分sc pDNA处于弱结合状态,导致容易被冲洗掉而影响收率。
|
图 9 不同上样量的层析分离谱图 Fig.9 Chromatogram of MMC separation under different loading amounts |
|
图 10 不同上样量下冲洗峰的HPLC分析比较 Fig.10 HPLC analysis of washing peaks under different loading amounts |
最终确定最佳上样量为5 mL,此时核酸分离得到sc pDNA纯度为83.9%、同质性达到95.8%、收率为80.6%。Valente等[12]采用精氨酸大孔介质从细菌裂解液中分离pDNA,得到纯度为92%的sc pDNA,但收率仅为43%,动态载量也仅有每毫升介质负载0.256 mg。Bicho等[15]使用乙二胺整体柱,分离得到纯度为97.1% 的sc pDNA,收率仅为47.0%,且处理体积仅为20 μL,单次获得的总sc pDNA量仅为2.55 μg,而本研究单次可获得sc pDNA约为每毫升介质负载0.465 mg。与文献相比,本研究获得的sc pDNA纯度较低,主要是由于初分离料液中RNA含量较大,造成洗脱过程中RNA的部分解吸,后续可通过初分离去除RNA或后续核酸分离提高sc pDNA纯度。
4 结语采用新型混合模式层析介质Capto PlasmidSelect分离料液中的sc DNA。探讨了料液中主要组分sc pDNA、oc pDNA和RNA的吸附行为,测定了其动态载量,并优化了层析分离过程冲洗、洗脱条件及上样量。在c(NH4)2SO4=2.5 mol·L−1条件下,成分较为复杂的料液中sc pDNA的饱和吸附量达到每克介质吸咐3.3 mg,故料液可不进行额外处理,直接上样分离。200 cm·h−1流速下sc pDNA的动态载量为每毫升介质负载2.00 mg,而oc pDNA为每毫升介质负载0.32 mg。优化的分离条件为:pH=7.5、c(NH4)2SO4=2.5 mol·L−1下上样,上样量为5 mL;pH=7.5、c(NH4)2SO4=1.9 mol·L−1下冲洗,最佳洗脱盐浓度(c(NH4)2SO4=1.7 mol·L−1)+(cNaCl= 0.3 mol·L−1),可得到sc pDNA纯度为83.9%、同质性高达95.8%、收率为80.6%。结果表明,采用混合模式层析介质Capto PlasmidSelect一步分离sc pDNA效果显著、选择性好、处理量较大,具有良好的应用前景。
| [1] |
HIGH K A, RONCAROLO M G. Gene therapy[J]. The New England Journal of Medicine, 2019, 381(5): 455-464. DOI:10.1056/NEJMra1706910 |
| [2] |
KULKARNI K M, KHOT A M, LOKAPURE S G, et al. A brief review on gene therapy[J]. Indo American Journal of Pharmaceutical Sciences, 2018, 5(5): 3288-3299. |
| [3] |
FEMIRA G N M. 基因治疗和DNA疫苗质粒DNA的下游加工技术[J]. 郭利平, 章佩芬, 唐青涛, 译. 华西药学杂志, 2002, 17(2): 159-162. FEMIRA G N M. Downstream processing technologies of plasmid DNA for gene therapy and DNA vaccines[J]. GUO L P, ZHANG P F, TANG Q T, trans. West China Journal of Pharmaceutical Sciences, 2002, 17(2): 159-162. |
| [4] |
哈小琴, 吴祖泽, 张庆林, 等. 质粒超螺旋比例对其细胞转染效率及表达的影响[J]. 生物技术通讯, 2005, 16(4): 392-394. HA X Q, WU Z Z, ZHANG Q L, et al. The effect of proportion of supercoiled plasmid on transfer efficiency and expression level in target cells[J]. Letters in Biotechnology, 2005, 16(4): 392-394. |
| [5] |
US Food and Drug Administration. Guidance for industry: Considerations for plasmid DNA vaccines for infectious disease indications [R]. Washington: US Department of Health and Human Services, 2007.
|
| [6] |
SANTOS T, CARVALHO J, CORVO M C, et al. L-tryptophan and dipeptide derivatives for supercoiled plasmid DNA purification[J]. International Journal of Biological Macromolecules, 2016, 87: 385-396. DOI:10.1016/j.ijbiomac.2016.02.079 |
| [7] |
徐英黔, 吴蕾, 甘一如. CTAB法制备超螺旋质粒DNA[J]. 过程工程学报, 2007, 7(2): 390-394. XU Y Q, WU L, GAN Y R. Purification of supercoiled plasmid DNA by CTAB[J]. The Chinese Journal of Process Engineering, 2007, 7(2): 390-394. |
| [8] |
LI Y, CURRIE D, ZYDNEY A L. Enhanced purification of plasmid DNA isoforms by exploiting ionic strength effects during ultrafiltration[J]. Biotechnology and Bioengineering, 2016, 113(4): 783-789. DOI:10.1002/bit.25836 |
| [9] |
NAZER B, DEHGHANI M R, GOLIAEI B. Plasmid DNA affinity partitioning using polyethylene glycol-sodium sulfate aqueous two-phase systems[J]. Journal of Chromatography B, 2017, 1044/1045: 112-119. DOI:10.1016/j.jchromb.2017.01.002 |
| [10] |
SALEK P, FILIPOVA M, HORAK D, et al. Enhanced solid phase extraction of DNA using hydrophilic monodisperse poly (methacrylic acid-co-ethylene dimethacrylate) microparticles[J]. Molecular Biology Reports, 2019, 46(3): 3063-3072. DOI:10.1007/s11033-019-04742-6 |
| [11] |
DA SILVA R J, MACIEL B G, MEDINA-LLAMAS J C, et al. Extraction of plasmid DNA by use of a magnetic maghemite- polyaniline nanocomposite[J]. Analytical Biochemistry, 2019, 575: 27-35. DOI:10.1016/j.ab.2019.03.013 |
| [12] |
VALENTE J F A, SOUSA A, AZEVEDO G A, et al. Purification of supercoiled p53-encoding plasmid using an arginine-modified macroporous support[J]. Journal of Chromatography A, 2020, 1618: 460890. DOI:10.1016/j.chroma.2020.460890 |
| [13] |
VALENTE J F A, SOUSA A, AZEVEDO G A, et al. DoE to improve supercoiled p53-pDNA purification by O-phospho-L-tyrosine chromatography[J]. Journal of Chromatography B, 2019, 1105: 184-192. DOI:10.1016/j.jchromb.2018.12.002 |
| [14] |
SANTOS T, PEREIRA P, SOUSA F, et al. Purification of supercoiled G-quadruplex pDNA for in vitro transcription[J]. Separation and Purification Technology, 2016, 163: 59-71. DOI:10.1016/j.seppur.2016.02.036 |
| [15] |
BICHO D, SANTOS B F, CARAMELO-NUNES C, et al. Application of ethylenediamine monolith to purify a hemagglutinin influenza deoxyribonucleic acid-based vaccine[J]. Separation and Purification Technology, 2015, 154: 320-327. DOI:10.1016/j.seppur.2015.09.046 |
| [16] |
CARDOSO S, ČERNIGOJ U, LENDERO KRAJNC N, et al. Chromatographic purification of plasmid DNA on hydrophobic methacrylate monolithic supports[J]. Separation and Purification Technology, 2015, 147: 139-146. DOI:10.1016/j.seppur.2015.04.018 |
| [17] |
HALAN V, MAITY S, BHAMBURE R, et al. Multimodal chromatography for purification of biotherapeutics - A review[J]. Current Protein & Peptide Science, 2019, 20(1): 4-13. |
| [18] |
武强, 林东强, 翁叶靖, 等. 用于双水相萃取的混合模式配基制备及对IgG分配的影响[J]. 高校化学工程学报, 2013, 27(6): 925-930. WU Q, LIN D Q, WENG Y J, et al. Preparation of mixed-mode ligand for aqueous two-phase extraction and the influences of the ligand on IgG partitioning[J]. Journal of Chemical Engineering of Chinese Universities, 2013, 27(6): 925-930. DOI:10.3969/j.issn.1003-9015.2013.06.002 |
| [19] |
LUO Y D, ZHANG Q L, YAO S J, et al. Adsorption behaviors of avian immunoglobulins and purification of immunoglobulin Y from chicken serum with mixed-mode resins[J]. Chinese Journal of Chemical Engineering, 2019, 27(3): 514-518. DOI:10.1016/j.cjche.2018.05.011 |
| [20] |
WU Q C, ZHANG Q L, GAO D, et al. Mixed-mode expanded-bed adsorption for human serum albumin separation[J]. Industrial & Engineering Chemistry Research, 2018, 57(3): 1039-1047. |
| [21] |
SILVA-SANTOS A R, ALVES C P A, PRAZERES D M F, et al. Separation of plasmid DNA topoisomers by multimodal chromatography[J]. Analytical Biochemistry, 2016, 503: 68-70. DOI:10.1016/j.ab.2016.03.012 |
| [22] |
BICHO D, CARAMELO-NUNES C, SOUSA A, et al. Purification of influenza deoxyribonucleic acid-based vaccine using agmatine monolith[J]. Journal of Chromatography B, 2016, 1012/1013: 153-161. DOI:10.1016/j.jchromb.2015.12.036 |




