水凝胶作为高度亲水的三维网络结构,具有良好的亲水性、生物相容性、渗透性和机械强度等性质,由生物大分子通过物理或化学交联形成的水凝胶尤其引人关注,在生物技术、医药领域中得到广泛的应用[1]。而DNA分子由于精确的碱基识别特性、可设计的序列和可预测的二级结构,成为一种有前途的新型功能材料。此外,DNA能够响应外部微小的刺激(例如pH[2]、温度[3]、金属离子[4]等),改变DNA拓扑结构[5],并广泛用于纳米技术领域[6],DNA水凝胶也由此应运而生。
DNA水凝胶具有理想的生物学特征,利用DNA制备的水凝胶不仅保留了普通聚合物水凝胶的机械性能和高含水量,还具备以下几个特点:(1)能够实现精确的互补识别配对:基于碱基互补配对的原则,DNA链具有很强的分子识别性,能够对化学小分子或者生物大分子进行纳米级的定位,在此基础上DNA分子单链之间能够进行自组装从而快速形成水凝胶。(2)序列的可设计性:可以对DNA链的碱基组成进行编码设计,从而功能化,还可以通过对序列长短的设计,实现对凝胶中孔径大小的控制,以及特定DNA纳米结构的形成,从而实现其拓扑结构的多样性;(3)良好的环境响应性:通过形成特定的功能结构域,能够对热、pH、金属离子、限制性内切酶解等外界条件作出响应而控制凝胶的形成与崩解[7]。DNA水凝胶保留了普通水凝胶的材料特性,同时也具备了DNA的生物功能性,真正实现了水凝胶材料结构和生物功能的统一融合。
根据组成的不同,DNA水凝胶通常可分为2种,一种称为DNA化学水凝胶,由寡核苷酸-聚合物杂化体自组装而成,合成比较复杂,生物相容性也比较差[8-9];另一种称为DNA物理水凝胶,是通过DNA碱基间的互补配对作用和DNA分子链间的缠绕作用,由带黏性末端的DNA片段相互连接自组装而形成。物理水凝胶需要大量的寡核苷酸链[10],但通过DNA自组装合成足够浓度或足够长度的寡核苷酸链,需要高昂的成本,有时需利用特殊的核苷酸链合成技术来实现。滚环扩增(rolling circle amplification,RCA)是DNA单链的高效扩增方式,操作便捷,特异性较高,在生物功能材料制备方面得到了较好的应用[11]。本综述主要介绍利用滚环扩增技术制备的DNA物理水凝胶。
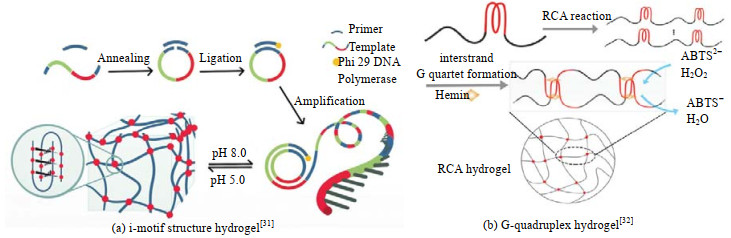
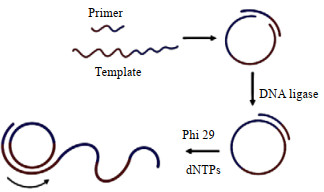
2 滚环扩增技术滚环扩增是一种酶介导的等温扩增方式,它是将一条寡核苷酸引物链退火,连接到环形DNA模板,在phi29 DNA聚合酶的作用下,将引物和dNTPs通过环形模板的滚动循环延伸而在短时间内产生大量与模板链互补配对、含数百至数千个重复单元的长链单链DNA(如图 1所示)[12-14]。滚环扩增的模板链碱基数几十到几百个均可,但必须是成环的核酸单链,相对来说引物链长度需更为固定,在16~24 nt最好。该技术借鉴了自然界中环状病原体生物DNA的分子滚环复制方式,主要应用于蛋白质[15]与核酸[16]的生物检测中。由于在检测方面的高效率与高通量,如今RCA技术也被用于扩增功能片段以生成单链核酸产物,在降低制备水凝胶成本的同时,兼具纳米结构的易于修饰与组装等性能,与此同时RCA产物也能与接枝了功能化序列的互补寡核苷酸链杂交来制备具有不同性质的多功能材料[17-18]。RCA产物能引导可编程的自组装,从而为DNA纳米结构的制备提供一种更简便、高效的策略[19],为DNA水凝胶的快捷制备注入了新动力。
|
图 1 滚环扩增示意图 Fig.1 Scheme of rolling circle amplification |
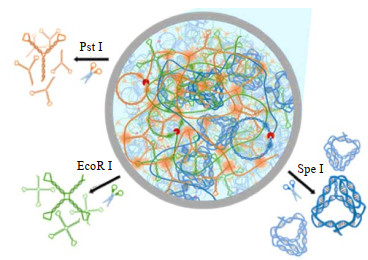
大多数DNA水凝胶是基于DNA分子碱基互补配对自组装制备而成,仅靠单链DNA,即使浓度够高、链够长也并不能形成凝胶,但不同纳米结构的单链DNA,却可以通过特定的互补配对而制备水凝胶。Ko等[20]设计出3种Y形、X形和四面体形的纳米结构DNA,滚环扩增后分别得到各自高拷贝数的重复序列的纳米结构,相互交织缠绕,堆叠,一步制得DNA水凝胶。而在设计3种结构时均包含独立的限制性内切酶酶切位点——Pst Ⅰ(TCTGCAG)、EcoR Ⅰ(GAATTC)、Spe Ⅰ(ACTAGT)。利用DNA凝胶所具有的酶响应性实现对X形、Y形和四面体形DNA纳米结构的选择性分离,从而实现对纳米结构的精确控制及对内容物的选择性控释(如图 2所示)。在单链DNA水凝胶的设计上,普通的DNA单链结构单一,而利用DNA折纸术将其设计为不同形状的纳米结构,不仅增加了内部结构的多样性,设计其他功能性位点后,也能实现对纳米结构的调控。
|
图 2 具有选择释放特性的DNA水凝胶工作机理示意图[20] Fig.2 Schematic diagram of selective release mechanism of DNA nanostructures from DNA hydrogels [20] |
单链DNA水凝胶的制备通常为一步法,即只需要一步滚环扩增即可制备。而对于双链杂合的DNA水凝胶通常需要多种扩增方式协作完成。第3节中单链DNA水凝胶中每个环状模板只结合一条引物链,称为线性滚环扩增(linear rolling circle amplification, LRCA)也被称为单引物扩增,是滚环扩增技术中最常用的扩增方式。Lee等[21]在利用引物Primer1单引物线性滚环扩增得到RCA产物ssDNA1的基础上,继续添加额外2个引物Primer2和Primer3,Primer2扩增后生成与ssDNA1互补的ssDNA2,而Primer 3用于制备ssDNA3(ssDNA3和ssDNA1具有完全相同的序列),即模板链(5’-TCGTTTGATGTTCCTAACGTACCAACGCACACGCAGTATTATGGACTG…TGGCTG-3’)中包含互补配对的Primer1(5’-TAGGAACATCAAACGACAGCCA-3’)、Primer2 (5’-ACGCAGTATTATGGACTG-3’)、Primer3(5’-TGGTACGTTAGGAACATC-3’)。ssDNA2与ssDNA1、ssDNA3互补配对,相当于消耗此前产生的长链ssDNA,Primer2也因此能够以新合成的ssDNA3为模板产生更多的ssDNA2,依次形成一个循环的链式反应体系(如图 3(a)所示),像这样的多条引物链同时进行扩增的方法,又被称为多引物链式扩增(multi-primed chain amplification,MCA),LRCA与MCA相互配合使用,是制备DNA水凝胶的有效手段。
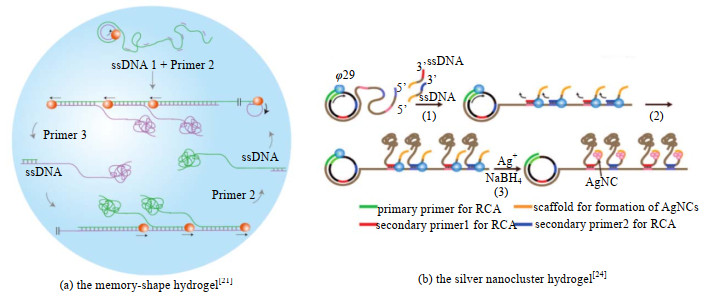
|
图 3 两种滚环扩增和多引物扩增合成DNA水凝胶示意图[21-22] Fig.3 Schematic diagram of rolling circle amplification and multi-primer amplification of DNA hydrogels [21, 24] |
在此基础上,Guo等[22-23]向ssDNA2、ssDNA3添加ATCCTCCCACCGGGCCTCCCACCTTAAC这段富含G、C碱基序列的核酸链,利用G、C碱基对银离子的较强亲和力,结合LRCA与MCA,在扩增后通过NaBH4还原Ag+,成功制备包裹有银纳米团簇的DNA水凝胶(如图 3(b)所示)[24]。将荧光物质注射进该凝胶后,由于银纳米团簇对荧光的猝灭作用,通过改变DNA与银离子的摩尔比、模板DNA的碱基序列实现对荧光强度的精准控制。同时银纳米团簇也赋予该凝胶抗菌性,使其在生物荧光标记、生物成像、组织工程等生物医学领域有广泛的应用。
值得注意的是,以上2种扩增方式协同制备的水凝胶非常柔软,具备“形状记忆”的功能,当凝胶在水中时,表现为普通“凝胶”的状态;若将其浸泡的水除去,在空气中由于表面张力与重力的作用无法保持自身的形状而塌缩,此时类似于“水”的“凝胶”的状态。
上述2种水凝胶的微观形貌均为类似于球状个体堆叠的结构,在扫描电镜下均观察到类似于“纳米花”的孔隙结构(如图 4所示),可能由于形成的“花瓣”结构柔软,密度较小而导致该类凝胶能够进行固-液转换。上述水凝胶已证明,多引物滚环扩增的DNA水凝胶能在扩增的同时赋予该水凝胶相应的功能性。此外,利用对滚环扩增与多引物链式扩增时间的调控,能够实现对该水凝胶微观结构“纳米花”密度与尺寸的控制。
Mao等[25-26]将扩增的场所转移至透明的氧化铟锡电极上(如图 5所示)。值得注意的是,该方法不用模具,就能在电极上自发地形成凸起的水凝胶。内部微观结构与固液转换的特异性与上述2种水凝胶相似。通过调节表面引物的点样面积和滚环扩增的时间来对水凝胶的形状、大小进行控制。该水凝胶能够有效地包载酶,载酶量大于其他聚合物凝胶,并且有良好的溶胀及去溶胀性,能在载酶后用于生物检测,基于该水凝胶的生物传感系统也可以在比色法与电化学分析等三维催化方面发挥应用潜力。
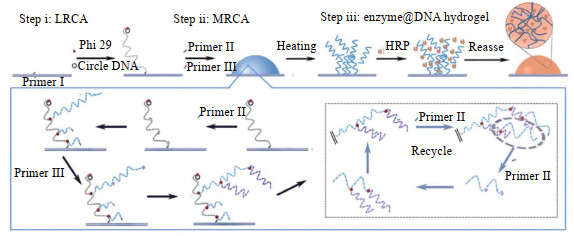
|
图 5 表面固定和自形水凝胶示意图[25] Fig.5 Schematic diagram of fabrication of surface-immobilized and self-shaped DNA hydrogels [25] |
DNA水凝胶最大的优势在于能实现功能性核酸序列或适配体的添加,利用这一优势能实现对蛋白质、细胞等原位捕获与释放。与上述LRCA与MCA协作的制备方式不同,Yao等[27]仅靠两步单引物扩增尝试了该类水凝胶的实际应用。首先设计出包含适配体Apt19S序列的DNA-chain-1(5'-AGGTCAGATGAGGAGGGGGACTTAGGACTGGGTTTATGACCTATGCGTG-3'),该序列能与骨髓间充质干细胞(BMSCs)膜上的ALPL蛋白具有高度亲和力,ALPL表位能在多能干细胞的膜上过表达,被证明是适配体Apt19S的最有效靶标,再设计出与DNA-chain-1部分互补的序列DNA-chain-2,利用2条环状模板与其对应的单引物链滚环扩增,从而构建出双链3D DNA网络,选择性地实现了BMSCs的包埋(如图 6所示),通过双LRCA过程,实现了对BMSCs的特异性捕获,并由于DNA网络的高水含量、高孔隙率和柔软性,为细胞培养提供了良好的3D微环境。
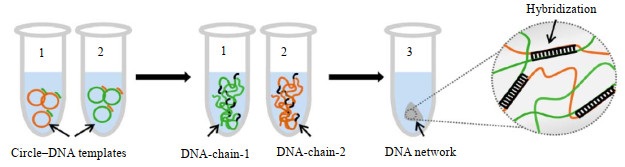
|
图 6 双滚环扩增产生用于干细胞捕捞的物理交联的DNA水凝胶[27] Fig.6 Preparation of physically cross-linked DNA network for stem cell fishing by double rolling circle amplification [27] |
双链DNA水凝胶的制备方式有2种,一类是单引物扩增与多引物链式扩增的协同作用,设计互补的引物,无论是在电极或者常规条件下反应,该方法制备出的水凝胶微观结构为“纳米花”团簇结构,同时具有固液转化特性;另一类是只利用单引物扩增,但需分开扩增,设计部分互补的序列,可以使其有更为松散的内部结构,添加合适的适配体能实现药物或细胞的特异性包埋,若设计全部互补配对的双链,内部结构会更加致密,孔径也会相对较小。
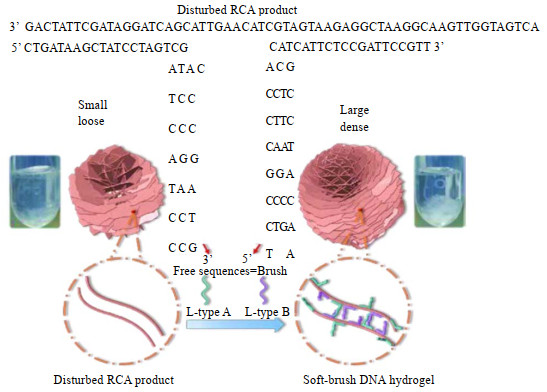
5 探针杂合DNA水凝胶从上节中双链杂合水凝胶的描述中不难看出,基于滚环扩增的DNA水凝胶本质是DNA纳米团簇的堆叠,对DNA链的碱基与长短的设计能控制其微观结构,但无法实现对其机械强度的控制。Song等[28]在滚环扩增时,引入L-型探针(如图 7所示),该序列的本质是一端与RCA产物实现碱基互补配对,而另一端为任意的自由序列,例如L-type A序列5’-CTGATAAGCTATCCTAGTCGNNNNNNNNNNNN-3’(前一部分与扩增产物互补,后一部分N为A、T、G、C中任意随机序列),该探针在改善微观形态和结构的同时强化凝胶的机械性能。在制备RCA水凝胶时,用高速离心法洗涤水凝胶,使凝胶团簇内的纳米花暴露在表面,而暴露出的纳米花“花瓣”孔隙继续吸收溶液中的水来促进溶胀,在此过程中,添加的L型探针能够干扰RCA产物,使其具有更高的凝胶化水平,并增加凝胶体积,在1 min内实现凝胶化状态的快速重建,该凝胶称之为软刷型水凝胶(soft-brush DNA hydrogel)。进一步研究发现,纳米花的大小及密度能够通过L型探针的浓度和自由序列长度来控制,不仅使水凝胶具有较高的黏弹性和致密的微观结构,也大大提高了其机械性能。
引入探针的概念,实质上是增加短链DNA的浓度,在滚环扩增基础上添加与其部分互补的短链DNA,本质上短链DNA浓度的增加,填补了微观结构中孔径的空白,因此水凝胶的整体机械强度有所提升。如果将L型探针中的解离序列更改为替代的特异性探针或功能性核酸,能够与多种靶标结合或在生物传感过程中充当信号输出方法,能够为分子检测和药物输送提供一种新技术。
6 含DNA四链体的水凝胶DNA除双螺旋结构以外,还能够形成多样的功能化二级结构。例如DNA链自组装形成的i-motif结构[29]是富含胞嘧啶(C)的DNA四链体,该结构能够被pH特异性调节,即当pH值较低时,DNA链可通过其黏性末端形成i-motif的链间互咬合结构,从而呈现凝胶状;而随着pH的升高,这种链间互咬合结构解链,DNA水凝胶崩解,故可以通过改变体系的pH使i-motif结构在形成与解离两态之间迅速转换[30]。
Wu等[31]将含C的重复序列引入环状模板链中,利用RCA技术,使其周期性地排列在RCA产物长链中,链与链之间形成i-motif结构,设计不同空白序列得到分子间(CCCCTCCCC…)、分子内(CCCAATCCCAATCCCAATCCC…)以及介于二者间的i-motif水凝胶(CCCTCCCTCCCTCCC…)(图 8(a))。研究发现,形成分子间i-motif结构的凝胶其机械强度更强,并且有自主形成i-motif结构的能力和更为显著的记忆性,而分子内i-motif水凝胶只能在pH较低的情况下形成,热稳定性较差。通过引入特定序列形成分子间i-motif水凝胶表现出更加致密的形态结构,并具有更强的机械强度与热稳定性,但pH的响应性受到了相应影响;而分子内i-motif水凝胶则表现出完全相反的状态。由此可以通过对DNA模板的精准设计,从而达到调节水凝胶的凝胶化与pH响应性的目的。
除了i-motif结构以外,还有富含鸟嘌呤(G)的DNA链自发形成的螺旋结构四链体称为G-四链体(G-quadruplex),它通过层层堆叠的方式形成一个空腔结构,该空腔能嵌入多种小分子,来增强该结构的稳定性,同时赋予该四链体不同的性质[33]。Huang等[32]制备出了G-quadruplex DNA水凝胶,需有多个重复的富含G序列的长链ssDNA,使其自主交联形成G-quadruplex,作为形成水凝胶的交联剂。与上述的i-motif四链体凝胶相同的是,分子间四链体水凝胶表现出更加致密的内部结构。G-四链体结构空腔可以结合血晶素(Hemin),形成Hemin/G-quadruplex DNA纳米酶,具有类似过氧化物酶的催化能力(如图 8(b)所示)。实验发现,该凝胶比Hemin/G-quadruplex复合物具有更高的稳定性与催化作用,并显示出更为出众的灵敏性。
DNA的2种四链体结构均具有特异功能性。设计富含C的四链体,最少需要3个相连的CCC碱基单元,较短的环路长度使得i-motif具有较高的热稳定性和pH稳定性,此外碱基序列TAA能自主折叠形成一个loop环区域,这一区域能使A、T碱基配对且堆叠在C碱基上,能有效增强该结构的稳定性,即分子间i-motif结构(CCCAAT),而随着加入C碱基与A、T碱基的数量比例不同,结构的稳定性与拓扑结构也会有所差异;另一方面,富含G-四链体的序列同样也最少需要3个G,加入A、T同样能使其链式结构更稳定。
7 各类DNA水凝胶的特点及存在问题DNA水凝胶独特的优势在于序列设计性以及智能响应性,而DNA内部的杂交或构象的变化,也为DNA水凝胶的广泛应用提供了新的思路。本文基于滚环扩增制备的DNA水凝胶的主要类型及特点归纳见表 1。不难发现,所有的DNA水凝胶在添加酶切位点后,均能具有酶响应性这一功能性;设计互补与部分互补序列,也能调节孔径大小和致密程度,从而具有不同的微观结构与机械性质。
|
|
表 1 基于滚环扩增的DNA水凝胶 Table 1 DNA hydrogels based on rolling circle amplification |
表 1所列举的几种基于滚环扩增制备的DNA水凝胶均利用DNA的序列设计,而使其具备功能性,并具有相似的微观结构与机械性能。虽然滚环扩增技术已趋于成熟,但仍然存在弊端,主要体现在:(1)无法预测及计算每一次滚环扩增产物的数量与长度,因此无法实现对该技术的精准调控;(2)在滚环扩增的整个过程中,DNA的磷酸化与环化效率无法控制,且在磷酸化、环化与扩增时存在无法预测的DNA损耗,导致DNA水凝胶制备的重复性不太理想;(3)与短链DNA自组装水凝胶相比,基于滚环扩增的DNA水凝胶的机械性能更为脆弱,更易于降解,因此在应用方面有所局限。即便如此,利用对DNA碱基的序列设计与模板链长度的控制,滚环扩增制备的水凝胶仍然可以实现对微观结构及孔径大小的调控,以此来改善DNA水凝胶的结构与机械性能,相比于传统的DNA自组装水凝胶,滚环扩增技术不仅明显降低了水凝胶的制备成本,同时也使得整个制备过程更加便捷迅速,在保留传统DNA水凝胶卓越的生物相容性、可设计性与特异性识别性的同时,赋予DNA水凝胶环境响应、记忆等功能性。如何利用DNA这一生物材料完成其特异性应用,如何实现其大规模的生产,仍然是不小的挑战。
8 展望DNA不仅充当遗传信息存储的分子,还能够成为新型生物材料基质,可以说DNA是连接生物学和材料的“理想桥梁”,为创造新的生物材料提供了关键和理想的先决条件,并日益成为构建智能水凝胶的基本单元[34-35]。DNA双螺旋独特的刚性和可设计的序列,使水凝胶从一开始就实现了从“soft matter”到“biomatter”的转变[36]。基于DNA自组装的超分子水凝胶能够快速原位形成,易于制备,具有良好的机械性能,利用DNA的高稳定性,精确的碱基配对和核酸人工进化[37]等特性,可以合理地制备具有可调稳定性和功能性的DNA水凝胶[38],这些水凝胶可用于药物输送[39]、生物传感[40]与成像[41]、3D打印[42]等领域。滚环扩增技术为DNA水凝胶制备所面临的成本问题提供了重要的解决方案,为核酸单链的扩增提供了一个有效可行的新方法,也为新型功能化的DNA纳米结构和纳米器件的设计提供了新思路。虽然利用滚环扩增技术制备DNA水凝胶仍存在很多问题,但相信随着技术上的改进,未来一定能制备出基于滚环扩增技术的“独一无二”的DNA水凝胶。
| [1] |
ZHANG Y S, KHADEMHOSSINI A. Advances in engineering hydrogels[J]. Science, 2017, 356(6337): 27-36. |
| [2] |
LU S S, WANG S. A pH-controlled bidirectionally pure DNA hydrogel: Reversible self-assembly and fluorescence monitoring[J]. Chemical Communications, 2018, 54(36): 4621-4624. DOI:10.1039/C8CC01603H |
| [3] |
UZUMCU A T, GUNEY O, OKAY O. Nanocomposite DNA hydrogels with temperature sensitivity[J]. Polymer, 2016, 100(1): 169-178. |
| [4] |
HUANG Y, FANG L, ZHU Z, et al. Design and synthesis of target-responsive hydrogel for portable visual quantitative detection of uranium with a microfluidic distance-based readout device[J]. Biosensors & Bioelectronics, 2016, 85(1): 496-502. |
| [5] |
LIU R D, MCCONNELL E M, LI J X, et al. Advances in functional nucleic acid based paper sensors[J]. Journal of Materials Chemistry B, 2020, 8(16): 3213-3230. DOI:10.1039/C9TB02584G |
| [6] |
PINHEIRO A V, HAN D R, SHIH W M, et al. Challenges and opportunities for structural DNA nanotechnology[J]. Nature Nanotechnology, 2011, 6(12): 763-772. DOI:10.1038/nnano.2011.187 |
| [7] |
YANG D Y, HARTMAN M R, DERRIEN T L, et al. DNA materials: Bridging nanotechnology and biotechnology[J]. Accounts of Chemical Research, 2014, 47(6): 1902-1911. DOI:10.1021/ar5001082 |
| [8] |
LIU L, GHAEMI A, GEKLE S, et al. One-component dual actuation: Poly(NIPAM) can actuate to stable 3D forms with reversible size change[J]. Advanced Materials, 2016, 28(44): 9792-9796. DOI:10.1002/adma.201603677 |
| [9] |
LIU J W. Oligonucleotide-functionalized hydrogels as stimuli responsive materials and biosensors[J]. Soft Matter, 2011, 7(15): 6757-6767. DOI:10.1039/c1sm05284e |
| [10] |
WANG J B, CHAO J, LIU H J, et al. Clamped hybridization chain reactions for the self-assembly of patterned DNA hydrogels[J]. Angewandte Chemie-International Edition, 2017, 56(8): 2171-2175. DOI:10.1002/anie.201610125 |
| [11] |
LI J, ZHONG W. Typing of multiple single-nucleotide polymorphisms by a microsphere-based rolling circle amplification assay[J]. Analytical Chemistry, 2008, 79(23): 9030-9038. |
| [12] |
GU L D, YAN W L, LIU L, et al. Research progress on rolling circle amplification (RCA)-based biomedical sensing[J]. Pharmaceuticals, 2018, 11(2): 35-42. DOI:10.3390/ph11020035 |
| [13] |
NILSSON M, GULLBERG M, DAHL F, et al. Real-time monitoring of rolling-circles amplification using a modified molecular beacon design[J]. Nucleic Acids Research, 2002, 30(14): 66-73. DOI:10.1093/nar/gnf065 |
| [14] |
DONG H F, WANG C, XIONG Y, et al. Highly sensitive and selective chemiluminescent imaging for DNA detection by ligation-mediated rolling circle amplified synthesis of DNAzyme[J]. Biosensors & Bioelectronics, 2013, 41(1): 348-353. |
| [15] |
CHEN C H, LUO M, et al. Sensitive colorimetric detection of protein by gold nanoparticles and rolling circle amplification[J]. The Analyst, 2015, 140(13): 4515-4520. DOI:10.1039/C5AN00485C |
| [16] |
XU H, ZHANG S X, OUYANG C HE, et al. DNA nanostructures from palindromic rolling circle amplification for the fluorescent detection of cancer-related microRNAs[J]. Talanta, 2018, 192(1): 175-181. |
| [17] |
YE J, YAN M X, ZHU L P, et al. Novel electrochemiluminescence solid-state pH sensor based on an i-motif forming sequence and rolling circle amplification[J]. Chemical Communications, 2020, 56(62): 8786-8789. DOI:10.1039/D0CC03694C |
| [18] |
ZHAO W, GAO Y, KANDADAI S A, et al. DNA polymerization on gold nanoparticles through rolling circle amplification: Towards novel scaffolds for three-dimensional periodic nanoassemblies[J]. Angewandte Chemie-International Edition, 2006, 45(15): 2609-2413. |
| [19] |
LI J, ZHENG C, CANSIZ S, et al. Self-assembly of DNA nanohydrogels with controllable size and stimuli-responsive property for targeted gene regulation therapy[J]. Journal of the American Chemical Society, 2015, 137(4): 1412-1415. DOI:10.1021/ja512293f |
| [20] |
KO O, HAN S, LEE J B. Selective release of DNA nanostructures from DNA hydrogel[J]. Journal of Industrial and Engineering Chemistry, 2020, 84(1): 46-51. |
| [21] |
LEE J, PENG S M, YANG D Y, et al. A mechanical metamaterial made from a DNA hydrogel[J]. Nature Nanotechnology, 2012, 7(12): 816-820. DOI:10.1038/nnano.2012.211 |
| [22] |
GUO W W, ORBACH R, MIRONI-HARPAZ I, et al. Fluorescent DNA hydrogels composed of nucleic acid-stabilized silver nanoclusters[J]. Small, 2013, 9(22): 3748-3752. DOI:10.1002/smll.201300055 |
| [23] |
耿金慧. 多功能荧光DNA水凝胶的构建及生物学性能研究[D]. 天津: 天津大学, 2018. GENG J H. Construction and biological properties of multifunctional fluorescent DNA hydrogel[D]. Tianjin: Tianjin University, 2018. |
| [24] |
GENG J H, YAO C, KOU X H, et al. A Fluorescent biofunctional DNA hydrogel prepared by enzymatic polymerization[J]. Advanced Healthcare Materials, 2018, 7(5): 998-1023. |
| [25] |
MAO X X, CHEN G F, WANG Z H, et al. Surface-immobilized and self-shaped DNA hydrogels and their application in biosensing[J]. Chemical Science, 2018, 9(4): 811-818. DOI:10.1039/C7SC03716C |
| [26] |
毛晓霞. 新型DNA水凝胶的制备及其在生化分析中的应用[D]. 上海: 上海大学, 2018. MAO X X. Fabrication of novel DNA hydrogel and its application in biochemical analysis[D]. Shanghai: Shanghai University, 2018. |
| [27] |
YAO C, TANG H, WU W, et al. Double rolling circle amplification generates physically cross-linked DNA network for stem cell fishing[J]. Journal of the American Chemical Society, 2020, 142(7): 3422-3429. DOI:10.1021/jacs.9b11001 |
| [28] |
SONG H, ZHANG Y Z, CHENG P, et al. A rapidly self-assembling soft-brush DNA hydrogel based on RCA products[J]. Chemical Communications, 2019, 55(37): 5375-5378. DOI:10.1039/C9CC01022J |
| [29] |
CHEN C, LI M, XING Y Z, et al. Study of pH-induced folding and unfolding kinetics of the DNA i-motif by stopped-flow circular dichroism[J]. Langmuir, 2012, 28(51): 17743-17748. DOI:10.1021/la303851a |
| [30] |
CHENG E J, XING Y Z, CHEN P, et al. A pH-triggered, fast-responding DNA hydrogel[J]. Angewandte Chemie-International Edition, 2009, 48(41): 7660-7663. DOI:10.1002/anie.200902538 |
| [31] |
XU W L, HUANG Y S, ZHAO H R, et al. DNA hydrogel with tunable pH-responsive properties produced by rolling circle amplification[J]. Chemistry-A European Journal, 2017, 23(72): 18276-18281. DOI:10.1002/chem.201704390 |
| [32] |
HUANG Y S, XU W L, LIU G Y, et al. A pure DNA hydrogel with stable catalytic ability produced by one-step rolling circle amplification[J]. Chemical Communications, 2017, 53(21): 3038-3041. DOI:10.1039/C7CC00636E |
| [33] |
MA D L, ZHANG Z H, WANG M D, et al. Recent developments in G-quadruplex probes[J]. Chemistry & Biology, 2015, 22(7): 812-828. |
| [34] |
WANG D, HU Y, LIU P F, et al. Bioresponsive DNA hydrogels: Beyond the conventional stimuli responsiveness[J]. Accounts of Chemical Research, 2017, 50(4): 733-739. DOI:10.1021/acs.accounts.6b00581 |
| [35] |
SHAO Y, JIA H Y, CAO T Y, et al. Supramolecular hydrogels based on DNA self-assembly[J]. Accounts of Chemical Research, 2017, 50(4): 659-668. DOI:10.1021/acs.accounts.6b00524 |
| [36] |
SHIBUYAMA M, Li X, SAKAI T. Gels: From soft matter to biomatter[J]. Industrial & Engineering Chemistry Research, 2018, 57(4): 1121-1128. |
| [37] |
WU J L, TIAN G X, YU W J, et al. pH-responsive hyaluronic acid-based mixed micelles for the hepatoma-targeting delivery of doxorubicin[J]. International Journal of Molecular Sciences, 2016, 17(4): 368-381. |
| [38] |
WANG Z G, DING B Q. DNA-based self-assembly for functional nanomaterials[J]. Advanced Materials, 2013, 25(28): 3905-3914. DOI:10.1002/adma.201301450 |
| [39] |
RUZICKA B, ZACCARELLI E, ZULIAN L, et al. Observation of empty liquids and equilibrium gels in a colloidal clay[J]. Nature Materials, 2011, 10(1): 56-60. DOI:10.1038/nmat2921 |
| [40] |
ZHANG L, LEI J, LIU L, et al. Self-assembled DNA hydrogel as switchable material for aptamer-based fluorescent detection of protein[J]. Analytical Chemistry, 2013, 85(22): 11077-11082. DOI:10.1021/ac4027725 |
| [41] |
XIANG A M, ZHANG K, DAI W H, et al. Multiplex micro RNA imaging in living cells using DNA-capped-Au assembled hydrogels[J]. Chemical Science, 2018, 9(37): 7419-7425. DOI:10.1039/C8SC02858C |
| [42] |
LI C, FAULKNER-JONES A, DUN A R, et al. Rapid formation of a supramolecular polypeptide-DNA hydrogel for in situ three-dimensional multilayer bioprinting[J]. Angewandte Chemie-International Edition, 2015, 54(13): 3957-3961. DOI:10.1002/anie.201411383 |