γ-谷氨酰转肽酶(γ-glutamyltranspeptidase,GGT,EC 2.3.2.2)是生物体内谷氨酰循环的关键酶,广泛存在于细菌、真菌、植物、哺乳类动物中。成熟的GGT是一种异质二聚体酶[1],包含一个大亚基(~40 KDa)和一个小亚基(~20 KDa),是由一个大小约60 KDa的没有催化活性的蛋白前体自主催化分裂而来[2]。
GGT可特异性催化谷胱甘肽的γ-谷氨酰基断裂,将其转移至受体分子(如水分子、α-氨基酸或二肽)[3]。由于该反应具有位点特异性强,且不消耗ATP等优点,在生物有机合成领域应用广泛。目前,利用GGT反应已成功实现了茶氨酸[4, 5]、γ-谷氨酰-L-多巴[6]、γ-谷氨酰-L-牛磺酸[7]、γ-谷氨酰-D-色氨酸[8, 9]、γ-谷氨酰-半胱氨酸[10]等化合物的合成。但由于GGT催化的谷氨酰基转移反应需在碱性(pH>9.0)条件下进行,而枯草芽孢杆菌B. subtilis NX-2 GGT (wt_GGT)对碱性环境的耐受性普遍较差,限制了GGT反应的工业化应用。
已经证明,大多数寡聚酶的失活往往始于四级结构的变化,即亚基解聚(subunit dissociation)[11]。本课题组在前期的研究中发现,B.subtilis NX-2 GGT在失活过程中也伴随着明显的亚基解聚,且pH值的变化对其多亚基结构的影响尤为显著[12]。
目前,寡聚酶亚基解聚的方法有提高反应压力[13]、酶的物理交联或化学交联[14]、以及酶的固定化[15]等。近年来,利用蛋白质工程手段提高寡聚酶亚基间的相互作用能,提高寡聚酶稳定性的方法逐渐成为研究热点。1987年Klibanov等首次提出了通过定点突变提高寡聚酶亚基间相互作用能的方法,并以磷酸丙糖异构酶(TIM)为对象进行了成功的尝试[16]。此后,Mishra等通过把Cu/Zn SOD二聚体表面一个保守序列上的亮氨酸突变成赖氨酸,降低了二聚体表面的疏水性,加速了其二聚体解聚[17]。Ding等在葡萄糖1-脱氢酶的亚基表面之间导入二硫键,使该酶在pH 4.5~10.5保持高度稳定的状态,在50℃下的半衰期为9900 min,是野生型酶的1868倍[18];Bjørk等将Chloroflexus aurantiacus的苹果酸脱氢酶中的Glu165替换为Gln或Lys,使突变酶在pH 7.5条件下的热稳定性显著提高[19]。Peimbert等将TIM亚基表面上的部分残基(K17,Y46,D48,Q82和D85)替换为非极性氨基酸(Ala、Val、Phe、Tyr、Ile、Leu或Pro),获得了高稳定性的突变酶DesTIMs,突变酶的Km值与野生酶相当,但催化数kcat却下降明显[20];
综上所述,采用蛋白质工程手段,在寡聚酶亚基表面引入二硫键,或增加荷电及疏水性氨基酸,提高亚基的间相互作用能,是提高其稳定性的有效途径。因此,针对野生型GGT的pH耐受性差的缺陷,本文以wt_GGT为研究对象,以B.subtilis str.168 GGT晶体结构(PDB ID: 3A75)为模板,通过分析wt_GGT大、小亚基表面残基的组成、性质及空间位置,筛选得到可能的突变位点,并将其逐一替换为缬氨酸(Valine),以增强GGT亚基间的疏水相互作用;在此基础上,考察了各种突变酶的催化特性及pH耐受性,以验证实际的突变效果。
2 材料与方法 2.1 材料与试剂用于提取基因组的菌株为B.subtilis NX-2(CGMCC 0833,wt_GGT编码基因全长为1764 bp,与B.subtilis str.168 GGT基因序列的同源性达99.21%,氨基酸序列一致性达99.15%);表达菌株E.coli BL21(DE3)和表达载体质粒pET-22b(+)均为本实验室保藏;DNA聚合酶KOD-Plus购自TOYOBO公司;限制性内切酶BanH I、Xho I及相应缓冲液购至NEB公司;DNA消化酶Dpn I购自Thermo公司;T4 DNA连接酶、标准分子量DNA、标准分子量蛋白购自TaKaRa公司;蛋白定量试剂盒Bio-Rad购自GENERAY公司;其余试剂均为国产分析纯。
2.2 主要实验仪器蛋白电泳仪、紫外分光光度计Ultrospec 7000(GE healthcare,美国);MyCyceler PCR仪(Bio-Rad,美国);pH计(METTLER TOLEDO,瑞士);蛋白纯化仪FPLC(GE Healthcare,美国)。
2.3 实验方法 2.3.1 同源建模方法和定点突变位点的选择采用Discovery studio 2.1软件(DS2.1 Accelrys Software Inc, San Diego, CA),以B.subtilis str.168 GGT晶体结构(PDB ID: 3A75)为模板经同源建模生成wt_GGT的3D结构预测模型。借助Pymol软件分析wt_GGT大、小亚基表面氨基酸残基,筛选合适的突变位点。突变的候选位点需同时满足以下条件:距离wt_GGT活性中心(T-403)距离>15 Å;保守性<100%;非疏水性氨基酸。
2.3.2 PCR方法PCR反应体系(50 μL)包括1×KOD-plus DNA聚合酶缓冲液,20 mmol·L-1 Mg2+,10 pmol·L-1正反向引物,50 ng DNA模板和1U KOD-plus DNA聚合酶。反应条件为:95℃ 5 min;95℃ 30 s;55℃30 s;68 ℃ 1~7 min,循环28次;68℃ 10 min结束程序。
2.3.3 GGT突变体质粒的获得和外源表达根据已公布的B.subtilis str. 168(Genbank: AL009126.3)的GGT基因序列设计合成引物(表 1)。在上游引物、下游引物的5′端分别引入相应的酶切位点(下划线),构建重组质粒Pet-22b(+)-GGT,并以其为模板,通过互补的正反向引物(见表 1)经PCR得到突变重组质粒;用限制性内切酶Dpn I酶切去除模板质粒;将酶切的产物转化到表达宿主E. coli BL21 (DE3)感受态细胞中,并涂布在含有氨苄青霉素(50 μg·mL-1)的LB平板,于37℃过夜培养后,挑取克隆转接LB培养基(含氨苄青霉素)培养12 h;以5 %接种量接种于新鲜的LB培养基,至OD600=0.5~0.6后加入终浓度为2.5 g·L-1的乳糖,于25℃下诱导表达12 h。为了验证所表达的突变体编码基因的正确性,每次表达过程中均取5 mL菌液,提取质粒并测序。
| 表 1 模板引物设计和定点突变设计的部分引物 Table 1 Primers design for the site-direct mutation |
将诱导表达后的菌液经12000 r·min-1离心后收集菌体;用Tris-HCl (pH 8.0,50 mmol·L-1)缓冲液洗涤菌体,再次离心收集菌体后用Tris-HCl(pH 8.0,50 mmol·L-1)缓冲液重悬菌体,冰浴超声破碎,12000 r·min-1、4℃下离心取上清液,粗酶液中GGT以Ni2+-NTA纯化,并经超滤脱盐后备用。
2.3.5 聚丙烯酰胺凝胶电泳与蛋白浓度的检测wt_GGT和突变酶的纯度由SDS-PAGE检测;蛋白浓度由Bradford蛋白定量试剂盒测定,以牛血清蛋白(BSA)为标准蛋白绘制标准曲线。
2.3.6 酶活测定[21]酶活(U)定义:以γ-谷氨酰对硝基苯胺(GpNA)为供体,以双苷二肽为受体,每分钟催化GpNA水解生成1 nmol对硝基苯胺(pNA)所需的酶量。
酶活测定方法:配制不同浓度的对硝基苯胺(pNA)标准溶液,并在410 nm下测定其吸光值,分别以pNA浓度和相应的吸光值为横、纵坐标,绘制标准曲线。取0.5 mL GpNA溶液(5 mmol·L-1)、0.5 mL双苷二肽溶液(100 mol·L-1)、0.5 mL酶液和1.5 mL Tris-HCl缓冲液(50 mmol·L-1,pH 8.0),混匀后于37 ℃水浴反应10 min。反应液于410 nm下测吸光值,根据标准曲线计算产物浓度和酶活。
2.3.7 GGT催化动力学常数的测定GGT的催化动力学方程为双底物的Michaelis-Menten方程[22],当受体(双甘二肽)浓度远大于供体(GpNA)浓度时,该方程可简化为单底物米氏方程:
| $ r = \frac{{{r_{{\rm{max}}}}{C_{s1}}}}{{{K_{\rm{m}}} + {C_{s1}}}} $ | (1) |
式中,r为反应速率[mmol·(L·min)-1];Cs1为供体GpNA浓度(mmol·L-1);Km为米氏常数(mmol·L-1);rmax为最大反应速率,可表示为式(2)
| $ {r_{{\rm{max}}}} = [E] \cdot {k_{{\rm{cat}}}} $ | (2) |
式中[E]表示酶浓度(mmol·L-1),kcat为催化数,即单位时间内一个酶分子所能催化底物发生反应的分子数,反映酶催化能力的大小(s-1)。
根据式(1)和(2),以1/r对1/Cs1作图可计算得到GGT的亲和力常数Km和催化常数kcat。
2.3.8 最适反应pH按照2.3.6节的酶活测定方法,调节缓冲液的pH值分别为6.0、7.0、8.0、9.0、10.0、11.0,分别测定突变酶及重组wt_GGT在不同pH条件下的酶活。
2.3.9 pH稳定性将重组wt_GGT和突变酶分别置于pH 6.0~11.0的缓冲液中,40℃水浴保温3 h后,分别取样,按2.3.6节的方法测定其残余酶活。设保温前各自的初始酶活为100 %。
2.3.10 失活速率常数(kd)计算将重组wt_GGT和突变酶分别置于pH 9.0~11.0的缓冲液中,并于40℃下温育,每隔一段时间取样,按2.3.6节的方法测定酶活,并计算残余相对酶活Ar(%)。假定GGT的失活为一级反应,可得失活反应方程:
| $ {r_{{\rm{max}}}} = [E] \cdot {k_{{\rm{cat}}}}\ln \frac{E}{{{E_0}}} = \ln {A_{\rm{r}}} = - {k_{\rm{d}}}t $ | (3) |
其中,kd为某一温度下的热失活速率常数。根据式(3),以Ar对时间t作图,经线性拟合可求得某一温度下的酶热失活速率常数kd。
2.3.11 重组wt_GGT和突变酶酰基化反应活化能的测定由于酶促反应速度可表示为:
| $ r = K \cdot E $ | (4) |
其中K为反应速率常数(min-1),E为酶浓度(mmol·L-1)。由于K是一个随温度变化的常数,因此根据Arrhenius方程,K可表示为:
| $ K = {K_0} \cdot {{\rm{e}}^{ - \frac{{{E_{\rm{a}}}}}{{RT}}}} $ | (5) |
式中K0为一速率常数。
根据式(4)和(5),酶促催化反应速率方程可表示为:
| $ r = E \cdot {K_0} \cdot {{\rm{e}}^{ - \frac{{{E_{\rm{a}}}}}{{RT}}}} $ | (6) |
若测定反应速率r时的反应时间很短(10 min),且温度较低( < 50℃),则可忽略酶的失活,因此有:
| $ \ln r = \ln A - \frac{{{E_{\rm{a}}}}}{{RT}} $ | (7) |
式中r表示温度为T时的反应速率(mmol·min-1),A为常数(E×K0,mmol·(L·min)-1),Ea为反应活化能(kJ·mol-1),T为反应温度(K),R为气体常数[8.314 J·(mol·K)-1]。以1/r对1/T作图,经线性拟合可计算得到GGT的酰基化反应活化能Ea。
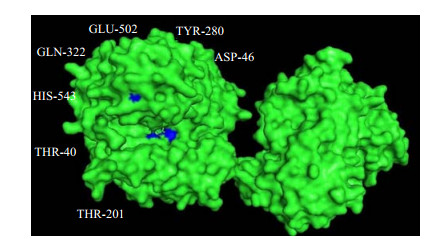
3 结果与讨论 3.1 同源建模及突变氨基酸候选位点的筛选以B.subtilis str. 168 GGT的晶体结构(PDB ID: 3A75)为模板构建了wt_GGT 3D结构的二聚体预测模型(见图 1),并运用Ramachandran模块来其进行评估。在构建的预测模型中,98.3 %的残基位于允许范围内,其中91.7 %的残基位于最优化区域,2.0 %位于待优化区域。模型的Profile-3D得分为182分,而预期最高得分为212分,表明该模型能够反映wt_GGT的真实结构,并可用于定点突变位点的选取。
|
图 1 wt_GGT二聚体的预测模型及候选突变位点的空间分布 Fig.1 Predicted 3D model for wt_GGT dimmer and the spatial distributions of the candidate mutation sites |
进一步根据2.3.1节的筛选条件,结合wt_GGT 3D结构预测模型,并选取了9条不同来源的GGT序列(Batrachochytrium dendrobatidis;Bhargavaea cecembensis;Virgibacillus halodenitrificans;Gracilibacillus lacisalsi;Thalassobacillus devorans;Desmospora sp;Salinicoccus luteus;B.acillus pumilus;Sporosarcina ureae)与模板(B. subtilis NX-2)进行同源性分析,最终选取得到了6个候选的突变位点,分别为A链上的D46、T201、Y280、Q322,和B链上的E502和H543,候选残基的空间位置、保守性,以及距活性中心(T-403)的距离见图 1和表 2。
| 表 2 候选突变位点与活性中心(T-403)的距离(Å)、保守性及位置 Table 2 Distance from the active center (T-403), conservation and positions of the candidate mutation residues |
按2.3.3节构建突变重组质粒,并转化E. coli BL21 (DE3)感受态细胞;挑选阳性克隆,经测序验证后,对所得突变菌株进行诱导表达;发酵液经离心后收集菌体,经超声破碎后,取上清液进行亲和纯化。
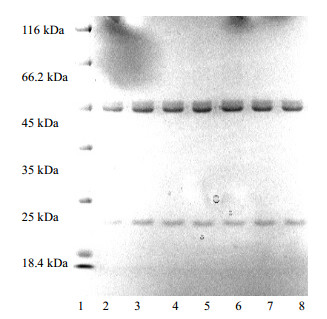
纯化后样品的SDS -PAGE电泳分析结果显示,6种突变酶在E. coli BL21 (DE3)中均可成功表达,表达产物均为二聚体蛋白,大、小亚基的分子量分别在46 kDa和20 kDa左右(图 2),与wt_BGGT一致。
|
图 2 纯化后的wt_GGT和突变酶的SDS-PAGE电泳图 Fig.2 SDS-PAGE analysis for the purified wt_GGT and mutations. Lane 1: maker; Lane 2: D46V; Lane 3:T201V; Lane 4:Y280V; Lane 5:Q322V; Lane 6: E502V; Lane 7:H543V; Lane 8:wt_ GGT |
分别配制供体(GpNA)浓度为0.5、1、2、3、4、5 mmol·L-1,受体双甘二肽浓度均为100 mmol·L-1的反应液,测定重组wt_GGT和突变酶的酰基化反应速率(rs),并按照2.3.7节的方法,计算wt_GGT和各种突变酶的催化动力学常数,结果见表 3。
| 表 3 wt_GGT和突变酶的催化动力学常数 Table 3 Kinetic parameters of wt_GGT and GGT mutations |
由表可知,大多数突变酶的催化常数与wt_GGT相近,其中D46V、Y280V、E502V、H543V的Km略小于wt_GGT,T201V和Q322V的Km较wt_GGT有所增加;此外,Y280V的kcat显著高于wt_GGT,提高幅度达43.3 %。
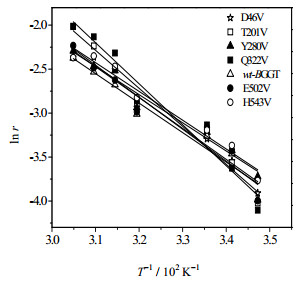
3.3.2 酰基反应活化能(Ea)的测定根据2.3.11节的方法,在不同温度下(25~50℃)分别测定wt_GGT及D46V、T201V、Y280V、Q322V、E502V、H543V的pNA的生成速率(r),以lnr对1/T作图,得到wt_GGT和突变酶的酰基化反应活化能Ea,结果如图 3和表 4所示。
反应活化能(Ea)表示GGT启动酰基转移反应所需的能量。如表所示,Y280V和E502V的反应活化能与wt_GGT相近,而D46V、T201V、Q322V,以及H543V的反应活化能较wt_GGT均有不同程度的增加。
|
图 3 wt_GGT和突变GGT酶酰基化反应活化能的拟合图 Fig.3 Activation energy fitting results of wt_GGT and GGT mutations |
| 表 4 wt_GGT和突变酶的酰基化反应活化能(Ea) Table 4 Activation energies of wt_GGT and GGT mutations |
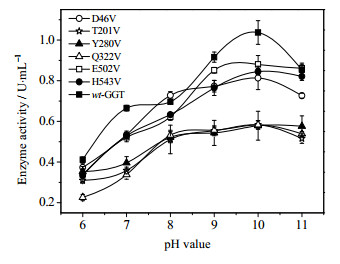
分别在pH 6.0~11.0的缓冲液中测定wt_GGT和突变酶D46V、T201V、Y280V、Q322V、E502V、H543V的活力。结果显示,无论wt_GGT还是突变酶,其最适pH范围均在pH 8.0~10.0,但突变酶酶活在这一范围内的变化均较wt_GGT更为平缓(图 4)。
|
图 4 pH对wt_GGT和突变酶活性的影响 Fig.4 Effects of pH on the activity of wt_GGT and GGT mutations |
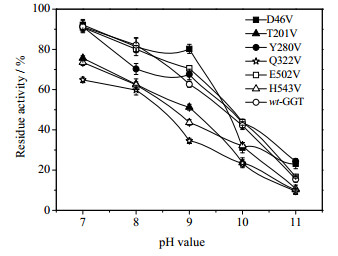
将wt_GGT和突变酶分别置于pH 6.0~11.0的缓冲液中,于40℃下保温3 h后测定酶活,以0时刻的初始酶活为100 %,计算残余酶活Ar。结果表明,随着pH值的上升,wt_GGT和突变酶的稳定性均呈下降趋势(图 5),但D46V、Y280V和E502V的pH稳定性较wt_GGT有所提高,其中Y280V在pH 11.0条件下温育3 h后,残余酶活仍可达24.32%,较wt_GGT提高了58.33%。
|
图 5 wt_GGT和突变酶的pH稳定性 Fig.5 pH stability of wt_GGT and GGT mutations |
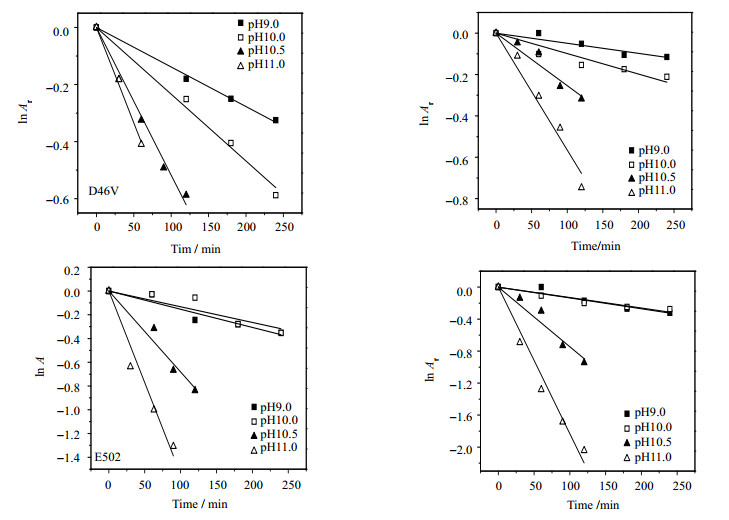
分别将wt_GGT,以及突变酶D46V、Y280V和E502V置于pH 9~11的缓冲液中,并于40℃保温,每隔一定时间取样测定酶活,以0时刻的酶活性为100%,计算相对酶活Ar。按照2.3.10节的方法计算wt_GGT和突变酶在不同温度下的失活速率常数kd,结果见表 6和图 6。
|
图 6 wt_GGT、D46V、Y280V和E502V在不同pH条件下的失活速率常数拟合 Fig.6 Fitting results of the denaturation constants (kd) of wt_GGT, D46V, Y280V and E502V under different pH values |
| 表 6 wt_GGT、D46V、Y280V和E502V在不同pH条件下的失活速率常数 Table 6 Denaturation constants (kd) of wt_GGT, D46V, Y280V and E502V under different pH values |
由表可知,wt_GGT、D46V、Y280V和E502V的失活速率均随pH值的升高呈逐渐增加的趋势,但突变酶的pH失活速率常数(kd)明显低于wt_GGT,其中Y280V在pH 11.0条件下的kd值仅为5.65×10-3。
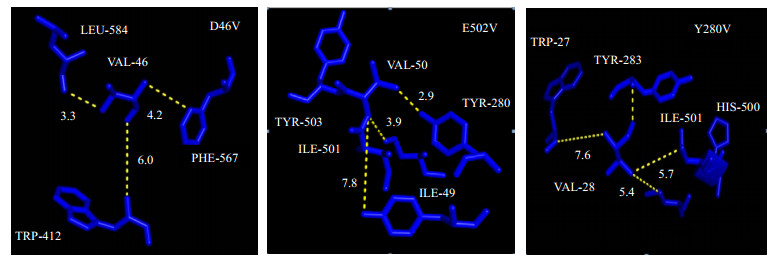
3.4 突变酶V-46、V-502和V280与附近氨基酸的相互作用分析采用Pymol软件分析了D46V、E502V和Y280V突变残基(V-46、V-502和V-280)附近的疏水性氨基酸。如图 7所示,在 < 10 Å距离内,V-46、V-502和V-280附近均存在一定数量的疏水性氨基酸,可与突变残基间发生疏水相互作用,且分别位于A链和B链(图 7),这有助于提高突变酶亚基间的相互作用能,并稳定其三、四级结构;其中,V-280可与附近4个氨基酸(Y-283、W-277、I-501和I-497)的侧链产生疏水相互作用,形成的疏水力场更强,从而使Y280V具有更强的pH稳定性。
|
图 7 D46V、E502V及Y280V与附近疏水残基的相互作用示意图 Fig.7 Illustration of the interaction between mutation residues (V-46, V-502 and V-280) and the closed hydrophobic residues |
GGT是由大、小两个亚基构成的二聚体酶,在高pH环境(pH>9.0)下易发生亚基解聚,进而引发酶的不可逆失活。为此,本文提出了采用定点突变的方法,在GGT亚基表面引入疏水性氨基酸,增强亚基间的疏水相互作用。由于疏水相互作用的强度不易受环境pH值影响,因而有助于提高GGT的pH耐受性。得到的具体结论如下:
(1) 采用同源建模的方法得到了wt_GGT的3D结构预测模型,并通过对wt_GGT表面氨基酸残基的性质、空间位置,以及保守性的分析,筛选得到了6个候选的突变位点;
(2) 采用定点突变的方法,将候选位点分别替换为缬氨酸,并成功实现了6种突变酶的异源表达;
(3) 分别测定了6种突变酶的酶学性质和催化常数。结果表明,D46V、Y280V、E502V、H543V的Km略小于wt_GGT,而T201V和Q322V的Km较wt_GGT有所增加;Y280V和D46V的kcat均较wt_GGT有所提高,其中Y280V的提高幅度高达43.3 %;
(4) 突变酶中D46V、Y280V和E502V的pH稳定性均较wt_GGT有所提高,其中Y280V在pH 11.0条件下温育3 h后,残余酶活可达24.32%,较wt_GGT提高了58.33%;经测定,Y280V在pH 11.0条件下的失活速率常数(kd)值仅为5.56×10-3;
(5) 模型分析显示,突变残基V-46、V-502和V-280可分别与位于A链和B链上的多个氨基酸残基产生疏水相互作用,其中V-280与附近氨基酸形成的疏水作用更强,这可能是导致Y280V的pH稳定性最高的主要原因。
| [1] | Roselyne C, Dany H K . Kinetic characterization and identification of the acylation and glycosylation sites of recombinant humanγ-glutamyltranspeptidase[J]. Biochemistry , 2007, 46 (43) : 12253-12262 DOI:10.1021/bi700956c |
| [2] | Toshihiro O, Hideyuki S . Crystal structure of the γ-glutamyltranspetidase precursor protein from E.coli:Structural changes upon autocatalytic processing and implications for the maturation mechanism[J]. J Biom Chem , 2007, 282 : 2433-2439 DOI:10.1074/jbc.M607490200 |
| [3] | Keillor J W, Castonguay R, Lherbet C . Gamma-glutamyltranspeptidase substrate specificity and catalytic mechanism[J]. Method Enzymol , 2005, 401 : 449-467 DOI:10.1016/S0076-6879(05)01027-X |
| [4] | Suzuki H, Izuka S, Miyakawa N . Enzymatic production of theanine, an "umami" component of tea, from glutamine and ethylamine with bacterial γ-glutamyltranspeptidase[J]. Enzyme Microb Tech , 2002, 31 (6) : 884-889 DOI:10.1016/S0141-0229(02)00213-2 |
| [5] | DENG Hai-xia(邓海霞), YAO Zhong(姚忠), ZHOU Zhi(周治) . Enzymatic synthesis of L-theanine a using copper(Ⅱ)-L-glutamine complexes as donor substrate((以L-谷氨酰胺-铜(Ⅱ)配合物为供体酶法制备茶氨酸))[J]. J Chem Ind and Eng(China)(化工学报) , 2008, 59 (12) : 3115-3119 |
| [6] | Kumagai H, Echigo T, Suzuki H . Synthesis of gamma-glutamyl-DOPA from L-glutamine and L-DOPA by gamma-glutamyltranspeptidase of E.coli K-12[J]. Agr Biol Chem , 1988, 52 (7) : 1741-1745 |
| [7] | Suzuki H, Miyakawa N, Kumagai H . Enzymatic production of γ-L-glutamyltaurine through the transpeptidation reaction of γ-glutamyltranspeptidase from E.coli K-12[J]. Enzyme Microb. Tech , 2002, 30 (7) : 883-888 DOI:10.1016/S0141-0229(02)00038-8 |
| [8] | Suzuki H, Kato K, Kumagai H . Development of an efficient enzymatic production of D-glutamyl-L-tryptophan (SCV-07), a prospective medicine for tuberculosis, with bacterial γ-glutamyltranspeptidase[J]. J Biotechnol , 2004, 111 (3) : 291-295 DOI:10.1016/j.jbiotec.2004.04.003 |
| [9] | Suzuki H, Izuka S, Minami H . Use of bacterial γ-glutamyltranspeptidase for enzymatic synthesis of γ-D-glutamyl compounds[J]. Appl Environ Microb , 2003, 69 (11) : 6399-6404 DOI:10.1128/AEM.69.11.6399-6404.2003 |
| [10] | Bridge W J, Zarka M H. Process for the production of γ-glutamylcysteine:US, 11/887667[P]. 2012-07-08. |
| [11] | Roberto F-L . Stabilization of multimeric enzymes:Strategies to prevent subunit dissociation[J]. Enzyme Micro Tech , 2009, 45 (6) : 405-418 |
| [12] | FANG Xin(房鑫), Tian Si-si(田思思), YAO Zhong(姚忠) . Cloning, expression and characterization of γ-glutamyltranspeptidase from E.coli and B.subtilis(大肠杆菌和枯草芽孢杆菌来源γ-谷氨酰转肽酶的克隆表达和性质比较)[J]. J Chem Eng of Chinese Univ(高校化学工程学报) , 2014, 28 (2) : 282-289 |
| [13] | Jose A C B, Jose L R S, Gustavo F . pH dependence of the dissociation of multimeric hemoglobin probed by high hydrostatic pressure[J]. Biophysical Chemistry , 2007, 125 (2-3) : 341-349 DOI:10.1016/j.bpc.2006.09.009 |
| [14] | Torchilin V P, Trubetskoy V S . Stabilization of subunit enzymes by intramolecular crosslinking with bifunctional reagents[J]. Ann N Y Acad Sci , 1984, 434 : 27-30 DOI:10.1111/nyas.1984.434.issue-1 |
| [15] | Mateo C, Palomo J M, Fernandez-Lorente G . Improvement of enzyme activity, stability and selectivity via immobilization techniques[J]. Enzyme Microb Technol , 2007, 40 (6) : 1451-63 DOI:10.1016/j.enzmictec.2007.01.018 |
| [16] | Ahern T J, Casal J I, Petsko G A . Control of oligomeric enzyme thermostability by protein engineering[J]. Proc Natl Acad Sci , 1987, 84 (3) : 675-9 DOI:10.1073/pnas.84.3.675 |
| [17] | Panchanand M, Anshuman D . Mechanistic study of CuZn-SOD from ipomoea carnea mutated at dimer interface:Enhancement of peroxidase activity upon monomerization[J]. Biochimie , 2014, 97 : 181-193 DOI:10.1016/j.biochi.2013.10.014 |
| [18] | Haitao D, Fen G, Danfeng L . Significant improvement of thermal stability of glucose 1-dehydrogenase by introducing disulfide bonds at the tetramerinterface[J]. Enzyme and Microbial Technology , 2013, 53 (6-7) : 365-372 DOI:10.1016/j.enzmictec.2013.08.001 |
| [19] | Bjørk A, Dalhus B, Mantzilas D . Stabilization of a tetrameric malate dehydrogenase by introduction of a disulfide bridge at the dimer-dimer interface[J]. J Mol Biol , 2003, 334 (4) : 811-21 DOI:10.1016/j.jmb.2003.10.006 |
| [20] | Mariana P, Lenin D-R, D.Alejandro F-V . Hydrophobic rpacking of the dimer interface of triosephosphate isomerase by in Ssilico design and directed evolution[J]. Biochemistry , 2008, 47 (20) : 5556-5564 DOI:10.1021/bi702502k |
| [21] | YE Li-jing(叶丽静), FANG Xin(房鑫), WANG Hao-qi(王浩琦) . Effect of surface modification with silane coupling agent on enhancing pzc value and enzyme loading capacity of mesoporous TiO2 whiskers(介孔TiO2晶须材料的硅烷戌改性及其对载体pzc值和酶负载的影响)[J]. CIESC J(化工学报) , 2013, 64 (6) : 2160-2168 |
| [22] | Taniguchi N, Ikeda Y . Gamma-glutamyltranspeptidase catalytic mechanism and gene expression[J]. Adv Enzymol Relat Areas Mol Biol , 1998, 72 : 239-278 |




