20种常见氨基酸作为合成蛋白质的砌块,广泛应用于食品、饲料、药品和化妆品等领域,到2017年,其市场需求已经超过500万吨,仅味之素公司年销售额已接近100亿美元。在早期的氨基酸工业生产中,主要是采用化学合成、酶催化或两者结合的工艺。随着生产菌株的持续改造和过程工程技术的不断进步,微生物发酵法因其良好的经济和环境效益,在谷氨酸、赖氨酸和苏氨酸等大多数氨基酸的工业生产中得到广泛应用(见表 1)。
|
|
表 1 部分氨基酸发酵生产情况 Table 1 Current titers of amino acid fermentation in references |
获得高产菌株是发酵法生产的关键,最初主要是利用诱变育种获得高产菌株。然而诱变育种存在工作量大、突变方向难以掌控、难以累积多个正向突变,且获得的菌株存在回复突变等缺点。因此,到了上世纪80年代,随着重组DNA技术的进步,代谢工程开始运用于高产菌株的构建过程[1],大大缩短了菌株改造的周期。代谢工程改造的重组菌,经过增强限速反应、消除或减弱竞争性代谢途径后,目标产物在细胞内得以更快速的生成。然而,有研究发现,胞内氨基酸浓度过高将制约高产菌株的最终产量,比如胞内谷氨酸浓度过高会对相关合成酶产生负反馈作用[2],而降低其胞内谷氨酸浓度不仅能减弱负反馈作用,还可以提高谷氨酸的产量[3];胞内半胱氨酸浓度过高会导致宿主DNA损伤,对生产菌株产生毒害作用[4];而在丙氨酸发酵生产中胞内丙氨酸含量过高会导致细胞生长抑制[5]。因此,在发现首个氨基酸外向转运蛋白—LysE(赖氨酸转运蛋白)后[6-7],利用外向转运蛋白降低胞内氨基酸的浓度,从而提高重组菌的产量受到广泛关注,各种氨基酸的外向转运蛋白研究陆续开展。
2 氨基酸外向转运蛋白研究及代谢工程应用细胞对氨基酸的运输主要通过细胞膜上的转运蛋白(exporter)完成,根据转运蛋白的结构、运输方式、能量耦合机制和功能特性可以划分不同的家族。Saier等[12]对微生物中69个不同的转运家族进行了系统整理和比较,描绘了氨基酸及其衍生物、糖类、脂质等小分子物质跨膜转运的多样性。其中,至少有24个转运蛋白家族包含有氨基酸及其类似物的转运蛋白。在这些氨基酸的转运蛋白中,有些负责氨基酸的外排,有些则负责氨基酸的吸收,有些还能调控胞内代谢平衡并提高菌株适应性[13]。
自本世纪初以来,在细菌中发现的能够转运氨基酸至胞外的转运蛋白已经超过20种(见表 2),主要来源于大肠杆菌(Escherichia coli)和谷氨酸棒状杆菌(Corynebacterium glutamicum)。不同的外向转运蛋白有各自的底物谱、转运效率及调控机理。
|
|
表 2 细菌中各氨基酸外向转运蛋白的发现 Table 2 Discovery of amino acid exporters in bacteria |
其中,大多数转运蛋白的转运底物不只限于一种,而是能向细胞外转运多种氨基酸及其类似物(图 1),且对不同底物的转运效率也不尽相同。本文根据转运氨基酸的不同种类对其进行分类,从各转运蛋白的发现、研究进展、应用情况分别进行介绍。
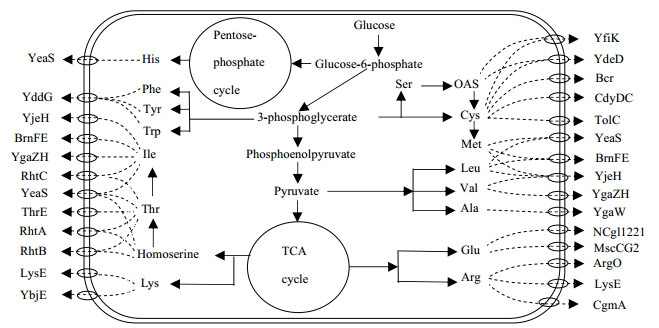
|
图 1 细菌中各氨基酸外向转运蛋白底物谱信息 Fig.1 Substrate specificity of amino acid exporters in bacteria |
目前,世界年产量最高的氨基酸是谷氨酸,我国是世界味精(谷氨酸单钠)主产地。已知的谷氨酸外向转运蛋白包括NCgl1221和MscCG2。它们都是机械敏感型通道(mechanosensitive channel),而这种通道蛋白的转运能力容易受外界溶液极性的影响[14-15]。
2007年,Nakamura等[14]发现谷氨酸棒状杆菌中ncgl1221基因缺失将导致谷氨酸在胞内的累积以及产量的下降,而在C. glutamicum ATCC13869/pL5 k中过表达ncgl1221基因则能将谷氨酸产量从39.3 g·L-1增加至44.6 g·L-1。转运机制表明[35],谷氨酸能以NCgl1221通道蛋白为载体,被动转运出细胞。NCgl1221通道蛋白含有一个N端结构域与一个C端结构域,N端结构域为转运谷氨酸所必需,而C端结构域通过结合特定氨基酸对转运过程起到反馈作用[36-37]。另一个通道蛋白MscCG2与NCgl1221的氨基酸序列同源度为23 %,研究显示MscCG2同样能促进谷氨酸排出胞外,由于通道蛋白能利用阳离子如Ca2+等调节细胞内外电化学梯度,MscCG2与NCgl1221都能增强细胞在低渗透压环境下的生存能力[15, 38]。
胞内谷氨酸浓度与其代谢途径下游的鸟氨酸、瓜氨酸和精氨酸代谢合成速率密切相关(图 2)。因此,通过抑制谷氨酸高产菌株外排谷氨酸,可以使胞内大量的谷氨酸无法转运出胞外从而进入下游代谢途径,提高下游产物的产量。Park等[39]在构建精氨酸生产菌时,敲除ncgl1221基因,使代谢流移向精氨酸,最终L-精氨酸产量达到92.5 g·L-1,这也是目前报道的精氨酸最大产量。类似地,Zhang等[40]通过敲除工程菌ncgl1221基因,使鸟氨酸产量提高22.7 %,达到9.8 g·L-1。
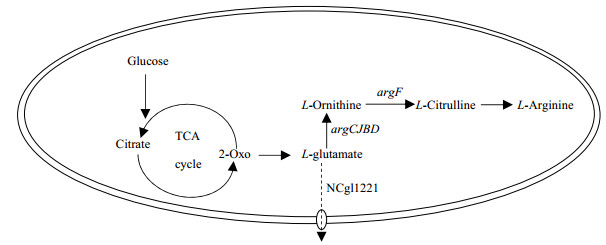
|
图 2 谷氨酸及其下游氨基酸代谢示意图 Fig.2 Metabolism pathway of glutamic acid and related downstream products |
碱性氨基酸包括赖氨酸、精氨酸和组氨酸。目前已知的外向转运蛋白中能转运赖氨酸的有LysE、YbjE,能转运精氨酸的有ArgO、LysE、CgmA,能转运组氨酸的有YeaS。赖氨酸作为重要的食品添加剂,是继谷氨酸之后第二个实现工业化发酵生产的氨基酸。早在1999年,赖氨酸发酵法的产酸率就达到了170 g·L-1[41]。目前,赖氨酸世界年均产量超过220万吨,并且以每年5%~7%的速度增长[42]。
1996年,Vrljic等[7]在转运蛋白研究中,发现过表达LysE导致出发菌株C. glutamicum ATCC13032的赖氨酸排出速率提高4倍,lysE基因敲除菌株则无法排出赖氨酸。底物谱研究发现[43],LysE还能向外转运碱性氨基酸中的精氨酸、鸟氨酸和瓜氨酸。因此,在寻找精氨酸的外向转运蛋白时,Nandineni等[16]通过不同菌株的耐受实验,鉴定了ArgO具有精氨酸转运功能,通过氨基酸序列比对发现ArgO与LysE氨基酸同源度为40%,且ArgO的表达在胞内赖氨酸浓度过高时将受到抑制,ArgP调控其基因转录。将lysE和argO基因都敲除后,Lubitz等[18]发现菌株依然能够转运精氨酸,进一步研究发现了新的精氨酸外向转运蛋白CgmA。
同样在外向转运蛋白研究中,受到Nandineni等对转运蛋白ArgO研究的启发,Pathania等[17]不仅鉴定了大肠杆菌中YbjE(LysO)的赖氨酸高效转运功能,还进一步发现在大肠杆菌中argO与ybjE基因之间存在交叉反馈抑制现象,即ArgO能够转运精氨酸,其基因表达受到赖氨酸抑制;而YbjE能够转运赖氨酸,其基因表达受到精氨酸抑制。这种机制维持了细胞内精氨酸和赖氨酸含量之间的相对平衡。
在氨基酸发酵生产中,LysE能转运的碱性氨基酸最多,在工程菌株育种中应用也最广(表 3)。Ishikawa等[44]在嗜甲基菌(Methylophilus)中导入谷氨酸棒状杆菌来源的lysE基因并与大肠杆菌来源的合成酶基因共表达后,赖氨酸产量达到9.0 g·L-1。Xu等[45]在重组谷氨酸棒状杆菌中过表达LysE,使精氨酸产量提高了13.6 %,达到35.9 g·L-1。Zhang等[40]在谷氨酸棒状杆菌中过表达LysE使重组菌鸟氨酸产量提高了21.8 %,达到18.4 g·L-1。过表达LysE,Lubitz等[18]使重组菌株瓜氨酸产量提高45 %,达到10.5 g·L-1。另外,Ginesye等[46]在重组大肠杆菌中共表达argO基因和argA214基因(argA214起到抑制反馈调节的作用),使精氨酸产量提高1.8倍,达到11.64 g·L-1。
|
|
表 3 外向转运蛋白在碱性氨基酸发酵生产中的应用 Table 3 Applications of exporters in the production of basic amino acids |
芳香族氨基酸主要包括苯丙氨酸、酪氨酸和色氨酸。目前在细菌中发现的芳香族氨基酸的外向转运蛋白只有YddG[20]。Doroshenko等[20]过表达YddG后,发现重组大肠杆菌对芳香族氨基酸及其类似物的耐受增强,利用碱性磷酸酶/绿色荧光蛋白融合分析方法(PhoA/GFP fusion analysis)确定了YddG位于细胞膜上,通过使用强启动子并协同表达htrE基因(编码一种外膜引导蛋白),使重组菌株的芳香族氨基酸产量提高了1.5~3倍。2016年,Tsuchiya等[47]成功解析了YddG蛋白的晶体结构。
目前,发酵生产芳香族氨基酸的产量较低,代谢改造中应用外向转运蛋白的报道很少,集中在色氨酸发酵生产中(表 4)。Liu等[48]过表达YddG使重组菌E. coli MG1655ΔtnaA/pSTV28-trpEDCBA-YddG中L-色氨酸产量提高了12.6 %,达到36.3 g·L-1。Wang等[49]在E. coli MG1655中代谢改造提高色氨酸产量,最终在30 L的发酵罐中发酵38 h积累48.68 g·L-1的L-色氨酸,其中YddG过表达使产量提高15.96 %。
|
|
表 4 外向转运蛋白在色氨酸发酵生产中的应用 Table 4 Applications of exporters in the production of tryptophan |
支链氨基酸包括亮氨酸、异亮氨酸和缬氨酸,它们具有多种生理功能,也是食用醋的添加成分。目前已知的支链氨基酸的外向转运蛋白有BrnFE、YgaZH、YjeH和YeaS四种[21-25],其中BrnFE、YjeH和YeaS也能转运甲硫氨酸。全局调控因子Lrp可以调节brnFE、yeaS和ygaZH基因表达。在大肠杆菌中Lrp会促进ygaZH基因的表达[23],抑制yeaS基因的表达[19];而在谷氨酸棒状杆菌中,Lrp会促进brnFE基因表达[21]。另外,研究显示支链氨基酸在通过YjeH向胞外转运时,互相之间存在竞争性抑制[25]。
Kutukova等[19]发现过表达YeaS(LeuE)使出发菌株亮氨酸、甲硫氨酸和组氨酸产量提高,其中亮氨酸产量提升近3倍,其基因表达受到亮氨酸和L-氨基丁酸的诱导调控。
代谢改造中的相关应用集中在BrnFE和YgaZH的表达强化上(表 5)。BrnFE能够转运甲硫氨酸、缬氨酸、异亮氨酸和亮氨酸[50]。秦天宇等[51]在甲硫氨酸工程菌中过表达BrnFE,使产量提高至1.82 g·L-1,提高了35.8 %。Yin等[52]在工程菌中过表达BrnFE和Lrp后,异亮氨酸产量提高至26.9 g·L-1,提高了11.0 %。陈诚等[53]在工程菌基础上强化BrnFE和Lrp表达,使缬氨酸产量提高了35.6 %,达到38.1 g·L-1。
|
|
表 5 外向转运蛋白在支链氨基酸和甲硫氨酸发酵生产中的应用 Table 5 Applications of exporters in the production of branched chain amino acid and methionine |
大肠杆菌来源的YgaZH与谷氨酸棒状杆菌来源的BrnFE同源度较高,能够转运缬氨酸、异亮氨酸及其类似物。Park等[24]在工程菌株中过表达YgaZH使异亮氨酸产量升至1.11 g·L-1,提高了2.5倍,过表达合成途径的ilvCED基因后异亮氨酸产量升至2.11 g·L-1,提高90 %,继续过表达Lrp后产量再提高了34 %,达到2.83 g·L-1,最终在6 L发酵罐上产量达到9.46 g·L-1。在工程菌中过表达YgaZH与Lrp也能增加缬氨酸产量,从2.38 g·L-1提高到5.25 g·L-1[23]。
2.5 YdeD, YfiK, Bcr, CdyDC和TolC转运蛋白半胱氨酸与乙酰丝氨酸的分子结构很相似,因此当外向转运蛋白的底物特异性不强时往往能对两者都有转运功能。目前已知的半胱氨酸外向转运蛋白有YdeD、YfiK、Bcr、CdyDC和TolC,其中YdeD和YfiK还能够转运乙酰丝氨酸。
通过在大肠杆菌中分别引入20种膜蛋白基因,Dassler等[26]发现YdeD过表达有利于半胱氨酸和乙酰丝氨酸转运至胞外,并影响了细胞内的硫代谢平衡,采用同位素标记的[35S]甲硫氨酸培养细胞,定位了ydeD基因的表达产物在细胞膜上。受此启发,Franke等[27]采用相同方法,证明了YfiK也是一种膜蛋白,结合yfiK基因与cysE基因(cysE可减弱反馈调节的作用)共表达结果和yfiK基因敲除与过表达的研究,鉴定了大肠杆菌YfiK具有向外转运半胱氨酸和乙酰丝氨酸的功能。在探究大肠杆菌中YfiK与YdeD是否存在协同作用的研究中,通过构建E. coli W3110Δyfik/pKP291-ydeD和E. coli W3110ΔydeD/pG2-yfik菌株,发现与对照菌株相比各菌株转运半胱氨酸的能力接近,说明YfiK与YdeD是两种独立的外向转运蛋白,可单独对半胱氨酸进行转运。
与Dassler一样,Yamada等[28]在大肠杆菌中分别引入33种膜蛋白基因(包含ydeD),发现其中8种基因(acrD, acrE-F, bcr, cusA, emrAB, emrKY, ybjYZ, yojIH)过表达后能增强菌株对半胱氨酸的耐受。尽管YdeD与Bcr过表达都能显著提高重组菌的半胱氨酸产量,但Bcr的底物专一性较强,对半胱氨酸的转运效率最高。
CydDC是ABC转运蛋白(ATP-binding cassette-type transporter),Pittman等[29]利用同位素标记的[35S]半胱氨酸证明了其对半胱氨酸的转运功能,由于CydDC直接消耗ATP将半胱氨酸运至胞外,转运机制与前三种不同,还需要进一步研究。
与前四种外向转运蛋白不同,tolC基因编码的是外膜通道蛋白(Outer Membrane Channel)[30, 54],在胞内半胱氨酸被转运到细胞周质中后,外膜通道蛋白可以加速细胞周质中的半胱氨酸转运到外界,从而使氨基酸的外排进一步加快。同时,TolC也是多种疏水性小分子的外向转运蛋白。值得一提的是,由于通道蛋白往往具有较宽的底物谱,利用通道蛋白进行特定小分子的转运是未来的一个潜在研究方向。
在工程菌株应用方面,由于L-半胱氨酸代谢地位的特殊性等原因,微生物发酵生产起步较晚,发酵水平较低,相关转运蛋白的应用报道较少。最近,Liu等[55]代谢改造大肠杆菌生产L-半胱氨酸,发现菌株E. coli JM109-cysE的培养基中未检测出半胱氨酸,而菌株E. coli JM109-cysE-ydeD培养基中检测到半胱氨酸含量45.8 mg·L-1,最终在5 L发酵罐中重组菌株E. coli JM109ΔtnaAΔsdaA/pTrc-serACB-cysE-ydeD产量达到5.1 g·L-1。Stolz等[56]代谢改造谷氨酸棒状杆菌高产L-丝氨酸,通过在发酵中流加葡萄糖积累了36.3 g·L-1的L-丝氨酸,这是国外报道的最高丝氨酸发酵产量。若在高产菌株中强化外向转运,产量可能进一步提高。
2.6 YgaW转运蛋白L-丙氨酸是最小的手性氨基酸,用途广泛。目前工业生产方法包括酶催化法与发酵法,由于高产工程菌的构建,发酵法的生产成本已经低于化学-酶法。
研究丙氨酸外向转运蛋白时,Hori等[31]在丙氨酸敏感型菌株中分别转入11种基因,发现其中4种(yddG, yeaS, ytfF, ygaW)能提高菌株对丙氨酸的耐受,使菌株在含有丙氨酸二肽分子的培养基中正常生长。其中YgaW过表达能使出发菌株的胞内丙氨酸浓度降低,ygaW基因缺失会导致菌株严重的丙氨酸敏感,由此推测YgaW可能是大肠杆菌中丙氨酸外向转运的主要载体。Ihara等[57]发现,当细胞内丙氨酸浓度过高时,胞内丙氨酸能够诱导ygaW基因表达,同时全局调控因子Lrp可以进一步提高ygaW基因的表达,从而调控丙氨酸的胞内含量。而Kim等[58]通过对YgaW蛋白定向改造发现45位和84位氨基酸对转运活性起到重要作用。
丙氨酸外向转运蛋白的研究起步较晚,目前还只在模式菌株中有研究报道。在工程菌中过表达YgaW后重组菌E. coli MG1655/pSTV29-alaD-ygaW产量进一步增加10.2 %,达到1.6 g·L-1[31]。随着丙氨酸发酵产量的快速提高,例如张学礼等[59]通过代谢改造大肠杆菌构建高产L-丙氨酸菌株,发酵产量已达到115 g·L-1,进一步强化外向转运可能获得更高的产量。
2.7 RhtA, RhtB, RhtC和ThrE转运蛋白大肠杆菌中外向转运蛋白RhtA、RhtB和YeaS都能转运苏氨酸和高丝氨酸至胞外,而RhtC仅能转运苏氨酸至胞外。在谷氨酸棒状杆菌中则发现ThrE具有向外转运苏氨酸的功能。
目前苏氨酸产量快速增长,世界年均产量超过33万吨。L-苏氨酸由胞内转运至胞外被认为是微生物发酵生产的限速步骤[60]。在谷氨酸棒状杆菌中最早由Simic等[61]利用转座子突变鉴定了ThrE表达有助于L-苏氨酸向外转运,在工程菌C. glutamicum DR-17中过表达ThrE使苏氨酸产量提高到67 mmol·L-1,提高了37 %。随后在大肠杆菌中Livshits等[32]利用[35S]甲硫氨酸定位了rhtA基因表达产物为膜蛋白,再通过基因敲除与过表达菌株的对照试验,发现RhtA能促进苏氨酸、高丝氨酸和赖氨酸转运至胞外。
在高丝氨酸外向转运蛋白研究中,Zakataeva等[33]发现过表达RhtB能增强重组菌对胞外L-高丝氨酸的耐受,而Kruse等[60]研究发现过表达RhtB后菌株对D-高丝氨酸的耐受并未增强,不仅如此,在工程菌中过表达RhtB与RhtC后,尽管胞外L-苏氨酸产量分别提高了2.9倍和2倍,但是菌株对D-苏氨酸的耐受并未增强。该结果显示RhtB与RhtC存在对转运底物的立体选择性。
值得一提的是,rhtB, rhtC, yeaS基因的表达受到全局调控因子Lrp的调控[62],但通过Lrp增强苏氨酸和高丝氨酸产量的研究目前还相对空缺,大多数研究还只是简单的对转运蛋白进行过量表达(表 6)。
|
|
表 6 外向转运蛋白在苏氨酸和高丝氨酸发酵生产中的应用 Table 6 Applications of exporters in the production of threonine and serine |
在谷氨酸棒状杆菌中,Diesveld等[63]发现过表达RhtC后,胞内苏氨酸含量下降了92 %,最高转运速率提高3.8倍,在工程菌发酵中过表达RhtC使胞外苏氨酸含量提高了56 %,达到6.4 g·L-1。而在大肠杆菌中Livshits等[32]发现rhtA23(rhtA基因-1位点碱基替换)由于基因起始密码子AUG的前一位碱基替换使基因表达强度提高近9倍,苏氨酸产量从18.4 g·L-1提高至36.3 g·L-1,提高近1倍。而Lee等[64]在重组菌E. coli MDS42ΔlacIΔtdh/p-thrABC中利用rhtA23替换tdcC与sstT基因使苏氨酸产量增加近3倍,rhtA基因表达水平较原先提高了4.79倍。
过表达基因rhtA23后,高丝氨酸的积累作用更加显著,重组菌E. coli NZ10/pAL4-rhtA23与E. coli NZ10/pAL4-rhtA相比产量从9.0 g·L-1提高至56.0 g·L-1[32]。这也印证了基因工程中SD序列与起始密码子之间的碱基替换和增减会极大地影响基因表达水平这一论断。此外,相关研究显示RhtA对5-氨基乙酰丙酸也具有非特异性的外向转运功能,Kang等[65]过表达rhtA基因使5-氨基乙酰丙酸产量在工程菌基础上提高了40 %,说明RhtA底物谱较广,可应用于转运多种代谢产物。
2.8 外向转运蛋白研究策略在外向转运蛋白的研究过程中,常见的功能鉴定方法可以总结为:(1)构建丧失特定底物转运能力的菌株,通过导入基因恢复菌株的转运能力;(2)敲除膜蛋白基因后,研究菌株对添加不同底物培养基的敏感性变化,确定敲除基因编码蛋白的转运功能;(3)过表达基因使产物胞外积累量增加,同时胞内含量降低;(4)通过基因BLAST检索,比对氨基酸同源性,筛选与已知转运蛋白功能类似的潜在转运蛋白;(5)通过转录组学预测潜在的外向转运蛋白。
另一方面,通过运用诱导剂与协同基因等方法能有效提升转运蛋白的转运效率。例如YeaS表达受到L-氨基丁酸诱导;Lrp能增强某些转运蛋白的表达水平;rhtA23基因由于转录加快进而使转运效率提高等。通过将有利于表达的正向改造进行整合,将进一步提高转运效率。
3 结语目前在高产菌株中应用外向转运蛋白的代谢改造报道相对较少,一方面因为某些转运蛋白的研究起步较晚、底物谱窄,另一方面是某些氨基酸的发酵水平还相对较低。下一步可以对各氨基酸外向转运蛋白的底物谱进行更加深入地研究,在高产菌株中结合各正向改造进行运用。此外,抑制吸收途径也是增加产量的方法之一。例如敲除编码苏氨酸内向转运蛋白的tdcC基因后工程菌苏氨酸产量提高了15 %[66]。在生产色氨酸的工程菌中敲除mtr、trpP和aroP三个编码色氨酸内向转运蛋白的基因后,色氨酸产量较对照组提高了51.6 %[67]。
随着蛋白质组学以及转录组学等方法的运用,通过分析蛋白结构以及基因表达水平的变化鉴定转运蛋白的功能变得切实可行。研究中已经开始更多地运用结晶学、核磁共振光谱法、低温冷冻电镜技术以及荧光检测技术开展蛋白质结构解析工作,例如Kawakami等[68]最近构建的质谱法高效分析真核细胞膜蛋白结构的实验体系。而越来越多的转运蛋白例如广谱通道蛋白T2SS[69]和类淀粉纤维转运蛋白FapF[70]等结构被解析使得通过底物谱研究结合理性设计获得所需的高效转运蛋白成为可能。在工业育种中,应用转运蛋白可以提高工程菌株的环境耐受性、提高转运的立体选择性、解除生长抑制和简化后续分离步骤。因此相关研究将有助于进一步降低生物法生产成本并扩大生物法生产应用的领域,是未来发展和提高微生物法生产特定化合物的重要方向。
| [1] | Shiio I, Ujigawa K. Enzymes of the glutamate and aspartate synthetic pathways in a glutamate-producing bacterium, Brevibacterium flavum[J]. Journal of Biochemistry, 1978, 84(3): 647-657. DOI:10.1093/oxfordjournals.jbchem.a132170. |
| [2] | Shimizu H, Hirasawa T. Production of glutamate and glutamate-related amino acids:molecular mechanism analysis and metabolic engineering[M].Qerlin: Springer Berlin Heidelberg, 2006: 1-38. |
| [3] | Radmacher E, Stansen K C, Besra G S, et al. Ethambutol, a cell wall inhibitor of Mycobacterium tuberculosis, elicits L-glutamate efflux of Corynebacterium glutamicum[J]. Microbiology, 2005, 151(5): 1359-1368. DOI:10.1099/mic.0.27804-0. |
| [4] | Park S, Imlay J A. High levels of intracellular cysteine promote oxidative DNA damage by driving the fenton reaction[J]. Journal of Bacteriology, 2003, 185(6): 1942-1950. DOI:10.1128/JB.185.6.1942-1950.2003. |
| [5] | Kim S, Ihara K, Katsube S, et al. Characterization of the L-alanine exporter AlaE of Escherichia coli and its potential role in protecting cells from a toxic-level accumulation of L-alanine and its derivatives[J]. MicrobiologyOpen, 2015, 4(4): 632-643. DOI:10.1002/mbo3.2015.4.issue-4. |
| [6] | Stefan B, Reinhard K. Lysine excretion by Corynebacterium glutamicum[J]. European Journal of Biochemistry, 1991, 202(1): 137-143. DOI:10.1111/ejb.1991.202.issue-1. |
| [7] | Vrljic M, Sahm H, Eggeling L. A new type of transporter with a new type of cellular function:L-lysine export from Corynebacterium glutamicum[J]. Molecular Microbiology, 1996, 22(5): 815-826. DOI:10.1046/j.1365-2958.1996.01527.x. |
| [8] | Li Z H, Chen N. Study on the directional breeding of high glutamic-acid production strain GTS190 and the control on its fermentation[J]. Advanced Materials Research, 2012, 573(10): 1029-1033. |
| [9] | SU Yue-wen (苏跃稳). Modification of L-threonine-producing strain and optimization of fermentation process (L-苏氨酸基因工程菌的改造及发酵过程的优化)[D]. Changchun (长春): Jilin University (吉林大学), 2017. |
| [10] | YAO Li-pin (姚丽萍). Metabolic modification of glycometabolism for sucrose to increase L-serine production by Corynobacterium glutamicum(蔗糖利用途径改造促进谷氨酸棒杆菌合成L-丝氨酸)[D]. Wuxi (无锡): Jiangnan University (江南大学), 2016. |
| [11] | ZHANG Xin-wu(张新武), YANG Xiao-ming(杨晓明), LIAN Na-na(廉娜娜), et al. Research on fermentation process with L-tryptophan auxotrophic strains of high-yield(L-色氨酸营养缺陷型高产菌株的发酵工艺研究)[J]. Journal of Food Safety and Quality(食品安全质量检测学报), 2014, 5(12): 4056-4062. |
| [12] | Milton H, Saier Jr. Families of transmembrane transporters selective for amino acids and their derivatives[J]. Microbiology, 2000, 146(8): 1775-1795. DOI:10.1099/00221287-146-8-1775. |
| [13] | Sasaki H, Sato D, Oshima A. Importance of the high-expression of proline transporter PutP to the adaptation of Escherichia coli to high salinity[J]. Biocontrol Science, 2017, 22(2): 121-124. DOI:10.4265/bio.22.121. |
| [14] | Nakamura J, Hirano S, Ito H, et al. Mutations of the Corynebactetium glutamicum NCgl1221 gene, encoding a mechanosensitive channel hornolog, induce L-glutarnic acid production[J]. Applied and Environmental Microbiology, 2007, 73(14): 4491-4498. DOI:10.1128/AEM.02446-06. |
| [15] | Wang Y, Cao G, Xu D, et al. A novel L-glutamate exporter of Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2018, 84(6): 1-15. |
| [16] | Nandineni M G J. Evidence for an arginine exporter encoded by yggA (argO) that is regulated by the LysR-type transcriptional regulator ArgP in Escherichia coli[J]. Journal of Bacteriology, 2004, 186(11): 3539-3546. DOI:10.1128/JB.186.11.3539-3546.2004. |
| [17] | Pathania A, Sardesai A A. Distinct paths for basic amino acid export in Escherichia coli:YbjE (LysO) mediates export of L-lysine[J]. Journal of Bacteriology, 2015, 197(12): 2036-2047. DOI:10.1128/JB.02505-14. |
| [18] | Lubitz D, Jorge J M, PacRez-GarcãA F, et al. Roles of export genes cgmA and lysE for the production of L-arginine and L-citrulline by Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2016, 100(19): 8465-8474. DOI:10.1007/s00253-016-7695-1. |
| [19] | Kutukova E A, Livshits V A, Altman I P, et al. The yeaS (leuE) gene of Escherichia coli encodes an exporter of leucine, and the Lrp protein regulates its expression[J]. Febs Letters, 2005, 579(21): 4629-4634. DOI:10.1016/j.febslet.2005.07.031. |
| [20] | Doroshenko V, Airich L M, Kolokolova A, et al. YddG from Escherichia coli promotes export of aromatic amino acids[J]. Fems Microbiology Letters, 2007, 275(2): 312-318. DOI:10.1111/fml.2007.275.issue-2. |
| [21] | Kennerknecht N, Sahm H, Yen M R, et al. Export of L-isoleucine from Corynebacterium glutamicum:a two-gene-encoded member of a new translocator family[J]. Journal of Bacteriology, 2002, 184(14): 3947-3956. DOI:10.1128/JB.184.14.3947-3956.2002. |
| [22] | Trötschel C, Deutenberg D, Bathe B, et al. Characterization of methionine export in Corynebacterium glutamicum[J]. Journal of Bacteriology, 2005, 187(11): 3786-3794. DOI:10.1128/JB.187.11.3786-3794.2005. |
| [23] | Park J H, Lee K H, Kim T Y, et al. Metabolic engineering of Escherichia coli for the production of L-valine based on transcriptome analysis and in silico gene knockout simulation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(19): 7797-7802. DOI:10.1073/pnas.0702609104. |
| [24] | Park J H, Oh J E, Lee K H, et al. Rational design of Escherichia coli for L-isoleucine production[J]. Acs Synthetic Biology, 2012, 1(11): 532-540. DOI:10.1021/sb300071a. |
| [25] | Liu Q, Liang Y, Zhang Y, et al. YjeH is a novel exporter of l-methionine and branched-chain amino acids in Escherichia coli[J]. Applied and Environmental Microbiology, 2015, 81(22): 7753-7766. DOI:10.1128/AEM.02242-15. |
| [26] | Dassler T, Maier T, Winterhalter C, et al. Identification of a major facilitator protein from Escherichia coli involved in efflux of metabolites of the cysteine pathway[J]. Molecular Microbiology, 2000, 36(5): 1101-1112. DOI:10.1046/j.1365-2958.2000.01924.x. |
| [27] | Franke I, Resch A, Dassler T, et al. YfiK from Escherichia coli promotes export of O-acetyl serine and cysteine[J]. Journal of Bacteriology, 2003, 185(4): 1161-1166. DOI:10.1128/JB.185.4.1161-1166.2003. |
| [28] | Yamada S, Awano N, Inubushi K, et al. Effect of drug transporter genes on cysteine export and overproduction in Escherichia coli[J]. Applied and Environmental Microbiology, 2006, 72(7): 4735-4742. DOI:10.1128/AEM.02507-05. |
| [29] | Pittman M S, Corker H, Wu G, et al. Cysteine is exported from the Escherichia coli cytoplasm by CydDC, an ATP-binding cassette-type transporter required for cytochrome assembly[J]. Journal of Biological Chemistry, 2002, 277(51): 49841-49849. DOI:10.1074/jbc.M205615200. |
| [30] | Wiriyathanawudhiwong N, Ohtsu I, Li Z D, et al. The outer membrane TolC is involved in cysteine tolerance and overproduction in Escherichia coli[J]. Applied Microbiology and Biotechnology, 2009, 81(5): 903-913. DOI:10.1007/s00253-008-1686-9. |
| [31] | Hori H, Yoneyama H, Tobe R, et al. Inducible l-alanine exporter encoded by the novel gene ygaW (alaE) in Escherichia coli[J]. Applied and Environmental Microbiology, 2011, 77(12): 4027-4034. DOI:10.1128/AEM.00003-11. |
| [32] | Livshits V A, Zakataeva N P, Aleshin V V, et al. Identification and characterization of the new gene rhtA involved in threonine and homoserine efflux in Escherichia coli[J]. Research in Microbiology, 2003, 154(2): 123-135. DOI:10.1016/S0923-2508(03)00036-6. |
| [33] | Zakataeva N P, Tokmakova I L, Troshin P V, et al. The novel transmembrane Escherichia coli proteins involved in the amino acid efflux[J]. Febs Letters, 1999, 452(3): 228-232. DOI:10.1016/S0014-5793(99)00625-0. |
| [34] | Simic P, Sahm H, Eggeling L. L-Threonine export:use of peptides to identify a new translocator from Corynebacterium glutamicum[J]. Journal of Bacteriology, 2001, 183(18): 5317-5324. DOI:10.1128/JB.183.18.5317-5324.2001. |
| [35] | Hashimoto K, Murata J, Konishi T, et al. Glutamate is excreted across the cytoplasmic membrane through the NCgl1221 channel of Corynebacterium glutamicum by passive diffusion[J]. Biosci Biotechnol Biochem, 2012, 76(7): 1422-1424. DOI:10.1271/bbb.120366. |
| [36] | Becker M, Krämer R. MscCG from Corynebacterium glutamicum:functional significance of the C-terminal domain[J]. European Biophysics Journal, 2015, 44(7): 577-588. DOI:10.1007/s00249-015-1041-x. |
| [37] | Yamashita C, Hashimoto K, Kumagai K, et al. L-Glutamate secretion by the N-terminal domain of the Corynebacterium glutamicum NCgl1221 mechanosensitive channel[J]. Biosci Biotechnol Biochem, 2013, 77(5): 1008-1013. DOI:10.1271/bbb.120988. |
| [38] | Hashimoto K I, Nakamura K, Kuroda T, et al. The protein encoded by NCgl1221 in Corynebacterium glutamicum functions as a mechanosensitive channel[J]. Biosci Biotechnol Biochem, 2010, 74(12): 2546-2549. DOI:10.1271/bbb.100636. |
| [39] | Park S H, Kim H U, Kim T Y, et al. Metabolic engineering of Corynebacterium glutamicum for L-arginine production[J]. Nature Communications, 2014, 5(8): 4618-4618. |
| [40] | Zhang B, Yu M, Zhou Y, et al. Systematic pathway engineering of Corynebacterium glutamicum S9114 for L-ornithine production[J]. Microbial Cell Factories, 2017, 16(1): 158-158. DOI:10.1186/s12934-017-0776-8. |
| [41] | Eggeling L, Sahm H. l -Glutamate and l -lysine:traditional products with impetuous developments[J]. Applied Microbiology and Biotechnology, 1999, 52(2): 146-153. DOI:10.1007/s002530051501. |
| [42] | Eggeling L, Bott M. A giant market and a powerful metabolism:L-lysine provided by Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2015, 99(8): 3387-3394. DOI:10.1007/s00253-015-6508-2. |
| [43] | Bellmann A, Vrljić M, Pátek M, et al. Expression control and specificity of the basic amino acid exporter LysE of Corynebacterium glutamicum[J]. Microbiology, 2001, 147(7): 1765-1774. DOI:10.1099/00221287-147-7-1765. |
| [44] | Ishikawa K, Toda-Murakoshi Y, Ohnishi F, et al. Medium composition suitable for L-lysine production by Methylophilus methylotrophus in fed-batch cultivation[J]. Journal of Bioscience and Bioengineering, 2008, 106(6): 574-579. DOI:10.1263/jbb.106.574. |
| [45] | Xu M, Rao Z, Yang J, et al. The effect of a LYSE exporter overexpression on l-arginine production in Corynebacterium crenatum[J]. Current Microbiology, 2013, 67(3): 271-278. DOI:10.1007/s00284-013-0358-x. |
| [46] | Ginesy M, Belotserkovsky J, Enman J, et al. Metabolic engineering of Escherichia coli for enhanced arginine biosynthesis[J]. Microbial Cell Factories, 2015, 14(1): 29-29. DOI:10.1186/s12934-015-0211-y. |
| [47] | Tsuchiya H, Doki S, Takemoto M, et al. Structural basis for amino acid export by DMT superfamily transporter YddG[J]. Nature, 2016, 534(7607): 417-420. DOI:10.1038/nature17991. |
| [48] | Liu Q, Cheng Y, Xie X, et al. Modification of tryptophan transport system and its impact on production of L-tryptophan in Escherichia coli[J]. Bioresource Technology, 2012, 114(2): 549-554. |
| [49] | Wang J, Cheng L K, Wang J, et al. Genetic engineering of Escherichia coli to enhance production of L-tryptophan[J]. Applied Microbiology and Biotechnology, 2013, 97(17): 7587-7596. DOI:10.1007/s00253-013-5026-3. |
| [50] | Vogt M, Haas S, Klaffl S, et al. Pushing product formation to its limit:metabolic engineering of Corynebacterium glutamicum for L-leucine overproduction[J]. Metabolic Engineering, 2014, 22(3): 40-52. |
| [51] | QIN Tian-yu (秦天宇). Metabolic engineering of Corynebacterium glutamicum ATCC 13032 to produce L-methionine (代谢工程改造谷氨酸棒杆菌生产L-甲硫氨酸)[D]. Wuxi (无锡): Jiangnan University (江南大学), 2014. |
| [52] | Yin L, Shi F, Hu X, et al. Increasing l-isoleucine production in Corynebacterium glutamicum by overexpressing global regulator Lrp and two-component export system BrnFE[J]. Journal of Applied Microbiology, 2013, 114(5): 1369-1377. DOI:10.1111/jam.2013.114.issue-5. |
| [53] | CHEN Cheng (陈诚). Metabolic engineering of Corynebacterium glutamicum for L-valine production (代谢工程改造谷氨酸棒状杆菌生产L-缬氨酸)[D]. Wuxi (无锡): Jiangnan University (江南大学), 2015. http://cdmd.cnki.com.cn/Article/CDMD-10295-1015523901.htm |
| [54] | Hoffmann C, Leis A, Niederweis M, et al. Disclosure of the mycobacterial outer membrane:cryo-electron tomography and vitreous sections reveal the lipid bilayer structure[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(10): 3963-3967. DOI:10.1073/pnas.0709530105. |
| [55] | Liu H, Fang G, Wu H, et al. L-Cysteine Production in Escherichia coli based on rational metabolic engineering and modular strategy[J]. Biotechnology Journal, 2018, 13(5): e1700695. DOI:10.1002/biot.v13.5. |
| [56] | Stolz M, Peterswendisch P, Etterich H, et al. Reduced folate supply as a key to enhanced L-serine production by Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2007, 73(3): 750-755. DOI:10.1128/AEM.02208-06. |
| [57] | Ihara K, Sato K, Hori H, et al. Expression of the alaE gene is positively regulated by the global regulator Lrp in response to intracellular accumulation of l-alanine in Escherichia coli[J]. Journal of Bioscience and Bioengineering, 2017, 123(4): 444-450. DOI:10.1016/j.jbiosc.2016.11.015. |
| [58] | Kim S, Ihara K, Katsube S, et al. Impact of charged amino acid substitution in the transmembrane domain of L-alanine exporter, AlaE, of Escherichia coli, on the L-alanine export[J]. Archives of Microbiology, 2017, 199(1): 105-114. |
| [59] | ZHANG Xue-li(张学礼), ZHANG Dong-zhu(张冬竹). Construction and application of a strain with high yield of L-alanine(一种高产L-丙氨酸的xz-a26菌株及构建方法与应用): CN, 102329765 A[P]. 2012-01-25. |
| [60] | Kruse D, Krämer R, Eggeling L, et al. Influence of threonine exporters on threonine production in Escherichia coli[J]. Applied Microbiology and Biotechnology, 2002, 59(2-3): 205-210. DOI:10.1007/s00253-002-0987-7. |
| [61] | Simic P, Willuhn J, Sahm H, et al. Identification of glyA (encoding serine hydroxymethyltransferase) and its use together with the exporter ThrE to increase L-threonine accumulation by Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2002, 68(7): 3321-3327. DOI:10.1128/AEM.68.7.3321-3327.2002. |
| [62] | Kutukova E A, Zakataeva N P, Livshits V A. Expression of genes encoding the RhtB family proteins depends on the global regulator lrp[J]. Molecular Biology, 2005, 39(3): 374-378. |
| [63] | Diesveld R, Tietze N, Fürst O, et al. Activity of exporters of Escherichia coli in Corynebacterium glutamicum, and their use to increase L-threonine production[J]. Journal of Molecular Microbiology and Biotechnology, 2009, 16(3-4): 198-207. DOI:10.1159/000142530. |
| [64] | Lee J H, Sung B H, Mi S K, et al. Metabolic engineering of a reduced-genome strain of Escherichia coli for L-threonine production[J]. Microbial Cell Factories, 2009, 8(1): 2-2. DOI:10.1186/1475-2859-8-2. |
| [65] | Zhen K, Yang W, Qiang W, et al. Metabolic engineering to improve 5-aminolevulinic acid production[J]. Bioengineered Bugs, 2011, 2(6): 342-345. DOI:10.4161/bbug.2.6.17237. |
| [66] | Lee K H, Park J H, Kim T Y, et al. Systems metabolic engineering of Escherichia coli for L-threonine production[J]. Molecular Systems Biology, 2007, 3(1): 149-149. |
| [67] | Gu P, Yang F, Li F, et al. Knocking out analysis of tryptophan permeases in Escherichia coli for improving L-tryptophan production[J]. Applied Microbiology and Biotechnology, 2013, 97(15): 6677-6683. DOI:10.1007/s00253-013-4988-5. |
| [68] | Kawakami M, Juge N, Kato Y, et al. Efficient mass spectral analysis of active transporters overexpressed in Escherichia coli[J]. Journal of Proteome Research, 2018, 17(3): 1108-1119. DOI:10.1021/acs.jproteome.7b00777. |
| [69] | Yan Z, Meng Y, Xu D, et al. Structural insights into the secretin translocation channel in the type Ⅱ secretion system[J]. Nature Structural and Molecular Biology, 2017, 24(2): 177-183. DOI:10.1038/nsmb.3350. |
| [70] | Rouse S L, Hawthorne W J, Lambert S, et al. Purification, crystallization and characterization of the Pseudomonas outer membrane protein FapF, a functional amyloid transporter[J]. Acta Crystallographica, 2016, 72(12): 892-896. |




