胰蛋白酶作为一种生产生活上重要的工具酶,在医药科研领域应用非常广泛。如在医学领域,用于胰岛素原转化为胰岛素[1],用作口服药治疗肠胃紊乱,用作消炎药减轻炎症,用作清创剂可清除创伤部位坏死的组织,作为血栓溶解剂治疗血栓紊乱。在食品应用领域,可水解一些食品工业里重要的动植物蛋白,如蚂蚁蛋白、蚕蛹蛋白及植物叶蛋白等。另外,胰蛋白酶也广泛用于多种生物技术过程,例如表面附着细胞的分离[3]、流感病毒的生产[4]、荷尔蒙的水解和用于产生其他蛋白[5]。传统胰蛋白酶的获取,在工业上以从胰液中提取为主,提取成本低廉,但是不同批次产品间的质量不均一,且残留致病原的风险较难避免[6]。因此,工业上急需开发一种高效且质量稳定的胰酶生产方法。同时,在未知蛋白肽段质谱分析中,样品在胰酶酶切处理前,化学变性剂的解折叠处理通常导致样品的后续检测复杂化,不利于样品的快速分析。而高温下进行变性未知蛋白胰酶酶切处理则可避免化学变性剂的使用。因此有必要针对胰酶热稳定性进行探索,以期提高其热稳定性。
目前,通过蛋白质工程的手段提高生物催化剂热稳定性主要包括定向进化及理性设计两类[7]。理性设计作为一种蛋白质工程手段,相较于其他对策,更为有效,具有普适性,通过分析现有的结构信息与生物化学数据提出一种可能有效的突变策略[8~10]。随着计算机算法的发展,多种生物信息学软件被开发,也开始用以分析蛋白结构从而获得其灵活性数据[11, 12]。基因工程技术作为目前一种被广泛应用的获得特异性蛋白的方法已成功地实现了多种重要蛋白产品的表达[10],通过基因工程表达胰蛋白酶,可以规避质量不均一等问题,但是野生型酶还存在一系列的问题,例如活性不高,最适温度还有一定提高的空间等。
Ω-loop结构是一种非常规二级结构,由6~16个氨基酸残基构成,结构类似于希腊字母Ω而得名。相比于α-螺旋、β-折叠和β-转角等结构含有更少的氢键,因此具有更高的灵活性。因而,Ω-loop结构对蛋白质稳定性强弱有着重要作用。Thompson等[13]通过对20个完整基因组进行综合分析后发现,与同源的中温蛋白相比嗜热蛋白具有更短的长度,对结构已解析的蛋白进行序列比对说明短的loop,尤其是表面暴露的短loop是蛋白质耐热性提高的主要贡献者,短的loop可以降低多肽链折叠时的构象熵,可以提高蛋白质的稳定性,因而loop删除可作为一种通用的进化策略以提高蛋白质的稳定性[2]。
活性位点邻近的Ω-loop暴露在蛋白质的表面,基于结构的特殊性,很可能会覆盖活性位点[14],使得底物与活性位点不能很好接触,从而降低了酶的催化活性。删除或截短Ω-loop结构,能在一定程度上提高酶的催化活性。
基于上述知识,作者尝试验证删除或截短暴露在胰蛋白酶表面柔性的Ω-loop,是否能够增加胰蛋白酶的稳定性与活性。根据截去的氨基酸的数目,构建三个突变体R1Q、C1Q和K1Q,并分别研究突变型与野生型的耐温性和活性等相关的酶学性质。
2 材料与方法 2.1 菌株、质粒及试剂重组猪源胰蛋白酶基因由金唯智生物科技(北京)有限公司全基因合成,pPIC9K质粒和GS115毕赤酵母菌株(实验室自备),大肠杆菌Top10感受态细胞购自康为世纪(中国),Phusion® High-Fidelity DNA Polymerases和限制性内切酶SalI购自Thermo-Fisher Scientific。SP SepharoseTM High performance购自GE Healthcare公司。蛋白胨(Peptone)、胰蛋白胨(Tryptone)和酵母提取物(Yeast extract)购自OXIOD公司。其余试剂为进口或国产分析纯。
2.2 组氨酸活性位点附近Ω-loop结构的柔性预测从PDB数据库(Protein Data Bank)中获取野生型胰蛋白酶的三维晶体结构文件(PDB ID: 4AN7),三维结构由VMD软件观察,利用MODELLER v9.11软件补全结构中缺失的残基,通过GROMACS v4.5.5软件进行分子动力学模拟,力场选定Gromos96.1 (53A6)[15],溶剂模型为SPC216,温度设定300 K,常压下进行。体系先后采用最陡下降法(steep descent method)及共轭梯度法(conjugate gradient method)进行能量最小化优化;然后固定蛋白,进行10000步的约束分子动力学模拟,其中静电相互作用力采用Particle Mesh Ewald (PME)算法,范德华力约束截取半径为1 nm;最后进行5000000步的自由分子动力学模拟。利用GROMACS自带的计算工具分析模拟轨迹,g_rmsd程序计算轨迹的均方根方差(root mean square deviation,RMSD)值;g_rmsf程序计算每个残基的均方根涨落(root mean square fluctuation,RMSF)值。
2.3 突变体菌株的构建利用重叠延伸PCR技术,构建R1Q、K3Q和C5Q三株突变,分别删除了组氨酸活性位点邻近的Ω-loop的C端的一个氨基酸、三个氨基酸和五个氨基酸,引物设计如表 1。新的突变基因经EcoRI和NotI限制性内切酶酶切后,用T4DNA连接酶,连接到同样经EcoRI和NotI限制性内切酶酶切后的pPIC9K质粒载体上。通过热激转化法,将质粒导入大肠杆菌TOP10感受态中,转化液涂布至含50 mg·mL-1卡那霉素的LB平板上,37oC过夜培养12 h。从LB平板上挑取单克隆,提取质粒,进行测序鉴定。经测序鉴定正确的重组质粒经SalI酶切线性化处理后,通过电转化的方法导入毕赤酵母GS115菌株,转化液涂布至含G418抗生素的YPD平板上进行多拷贝子的筛选。
| 表 1 引物设计 Table 1 Primers for the site mutation |
从YPD平板上挑取的单克隆接种到50 mL的BMGY培养基中,培养至OD600达到2至6,收集菌体,3000 r·min-1离心5 min,重悬后,将菌体转入BMMY培养基中,进行蛋白的诱导表达。30℃培养96 h,每隔24 h补加甲醇至终浓度为1%(V:V),8000 r·min-1离心30 min回收上清液。利用阴离子交换的方法对蛋白进行纯化。用平衡缓冲液(20 mmol·L-1 citric acid,pH 3.0) 平衡SP SepharoseTM High performance柱后,将过夜透析后的培养液上样,用平衡缓冲液和洗涤缓冲液(20 mmol·L-1 citric acid,0.5 mol·L-1 NaCl,pH 3.0) 进行梯度洗脱,并收集蛋白峰。利用SDS-PAGE及BCA法检测纯化后蛋白的纯度及浓度。
2.5 活性检测胰蛋白酶的活性可以利用N-苯甲酰-L-精氨酸乙酯,简称BAee,作为底物测定胰蛋白酶的活性。胰蛋白酶原的N端有一段寡胎DDDDK为肠激酶的识别位点,测定前,需要肠激酶来激活。激活后,在25 mmol·L-1Tris–HCl, 50 mmol·L-1 NaCl,和2 mmol·L-1 CaCl2缓冲液(pH 7.6) 与底物作用反应。BAEE底物溶液需要在25℃条件下提前水浴处理。3.0 mL BAEE底物溶液与100 mL 1 mmol·L-1 HCl水溶液预混于石英比色皿中。将肠激酶处理过的胰蛋白酶溶液加到盛有上述预混液的比色皿(比色皿光程为1 cm),迅速放至紫外分光光度计检测槽中,开始测定反应液在253 nm处的吸光值变化,根据反应液在253 nm处每分钟吸光度的变化值ΔOD 253·min-1。同时以不含BAEE底物的溶液做对照试验。酶活的计算公式(1) 如下:
| $ \text{BAEE(U}\cdot \text{m}{{\text{L}}^{-1}}\text{)}=\frac{\Delta \text{OD253}\cdot \text{mi}{{\text{n}}^{-1}}\times df}{0.001\times 0.1} $ | (1) |
df为稀释因子。
2.6 热稳定性的测定首先测定胰蛋白酶的半衰期t1/2。将纯化的胰蛋白酶稀释到200 mg·mL-1,50℃下孵育,按0,30,60,120,180 min时间点取出一定样品,冰浴60 s,测定一次酶活。以孵育前的酶活为100%,根据实际酶活计算不同孵育时间点酶残留活性百分比。以酶残留活性自然对数为纵坐标,孵育时间t为横坐标,进行线性拟合。其斜率即为失活速率常数kd,利用(2) 计算其半衰期。
| $ {{t}_{1/2}}=\frac{\ln 2}{{{k}_{\rm{d}}}} $ | (2) |
Tm是衡量蛋白质热动力学稳定性的重要参数,可通过圆二色谱仪进行测定。测定方法为将蛋白质浓度稀释到200 mg·mL-1,以50 mmol-1 Tris–HCl (pH 7.8) 为缓冲液,测定波长为215 nm,温度设定范围为20~95℃,升温速率为1℃·min-1。将观察到的椭圆度利用式(3) 转化为蛋白质的解折叠分数Fd:
| $ {{F}_{\text{d}}}\text{=}\frac{{{\theta }_{\text{T}}}-{{\theta }_{\text{N}}}}{{{\theta }_{\text{D}}}-{{\theta }_{\text{N}}}} $ | (3) |
式是,θT为在特定温度下样品的椭圆度,θN和θD分别为升温过程中前后两个温度点的椭圆度。
2.7 野生型胰蛋白酶和突变型胰蛋白酶最适温度测定将BAEE酶活测定底物溶液分别放置于25~95℃,间隔为5℃的条件下孵育。胰蛋白酶原样品经肠激酶激活后,分别利用上述预孵育的底物溶液在同样温度下进行酶活测定,酶活性最大的温度即为最适反应温度Topt。
3 结果与分析 3.1 结构分析与删除突变的设计在蛋白质的理性设计中,选择突变区域是很重要的一点。选择突变区域不当,会造成酶的稳定性降低,甚至失活。所以在选定酶的突变区域时,需要综合考虑蛋白质的活性、稳定性及灵活性之间的关系[16]。
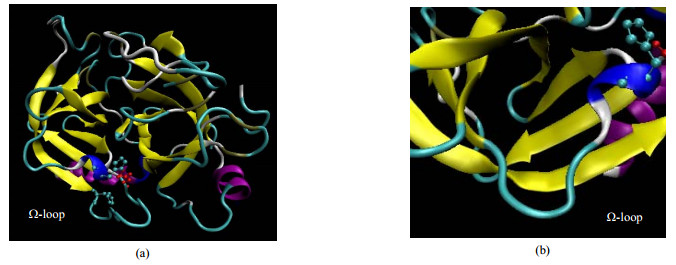
通过VMD软件观察分析猪源胰蛋白酶的三维结构,如图 1(a)所示。Ω-loop包括6个氨基酸和邻近组氨酸活性位点(如图 1(b)所示),位于一个短α-螺旋和β-转角之间。
|
图 1 猪胰蛋白酶的三维结构模型以及猪胰蛋白酶活性区Ω环结构 Fig.1 Three-dimensional (3D) model of porcine trypsin and the Ω-loop in the active region of porcine trypsinogen |
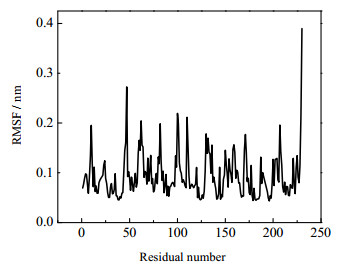
利用GROMACS v4.5.5进行分子动力学模拟,通过计算蛋白质中每个残基的RMSF值来反映构象的灵活度,结构越灵活,柔性越大,相应的RMSF值越高。结果证明,相较于其它的区域,Fragment 41~46(即Ω-loop所在区域)具有较高的柔性,如图 2所示。活性位点邻近的Ω-loop,暴露在蛋白质的表面,可能覆盖活性位点。靠近活性位点的氨基酸为半胱氨酸,半胱氨酸能够合成二硫键,二硫键是共价键,对酶的稳定性起着重要作用,故不能删除整个Ω-loop,需保留半胱氨酸。因而,根据删除氨基酸的数目(分别删除为1,3,5个氨基酸),构建了三种删除突变:R1Q、K3Q及C5Q。
|
图 2 野生型胰蛋白酶的灵活性 Fig.2 Residual flexibility of the wild-type trypsin |
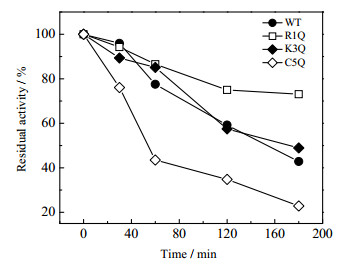
通过测定胰蛋白酶的动力学稳定性,分析截短Ω-loop对胰蛋白酶稳定性的影响。野生型及突变型猪源胰蛋白酶原在肠激酶激活后,迅速转移至50℃条件下孵育,每5 min测定样品的酶活性,得到失活曲线,如图 3所示。
|
图 3 野生型及突变型胰蛋白酶失活曲线比较 Fig.3 Comparison of thermal activation between wild-type and mutant trypsin |
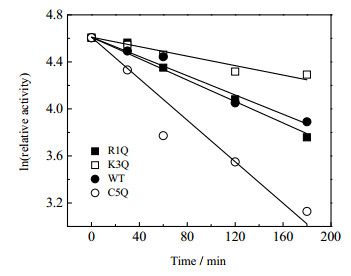
从图中可以看出,野生型酶的失活速率大于R1Q突变酶和K3Q突变酶,但小于C5Q突变酶,在50℃放置180 min后,野生型胰蛋白酶保持了42.8%的残留活性,而R1Q保持了73%的残留活性,K3Q和C5Q分别保持了48.9%和22.8%的残留活性。利用上述失活曲线测定的活性相对值,以相对活性的对数值为纵坐标,以时间为横坐标,利用Origin9.0进行线性拟合,如图 4所示。按照式(3) 计算野生型及突变型胰蛋白酶在50℃条件下的半衰期,野生型的半衰期为155.3 min,R1Q为368.6 min,K3Q和C5Q分别为164.2和86.7 min。突变型R1Q相比于野生型其半衰期提高了213.3 min,K3Q相比于野生型则提高了8.9 min,而C5Q比于野生型其半衰期则降低了68.6 min。
|
图 4 野生型及突变型胰蛋白酶失活半衰期测定 Fig.4 Profiles of half-life inactivation of wild-type and mutant trypsin |
蛋白质Tm值利用圆二色谱分析。在215 nm的波长下检测蛋白质变性椭圆度的变化,结果如图 5所示。野生型胰蛋白酶的Tm值为69℃,突变型R1Q的Tm值为80℃,K3Q的Tm值为70℃,C5Q的Tm值为65℃。结果表明,R1Q的热稳定性要高于野生型胰蛋白酶,K3Q同野生型胰蛋白酶的热稳定性相近,而C5Q则低于野生型胰蛋白酶的热稳定性。该Ω-loop结构区域具有很高的柔性,截除Ω-loop结构中的异亮氨酸,使得Ω-loop变短,短的loop可以降低多肽链折叠时的构象熵,R1Q突变胰蛋白酶的灵活性减弱,从而使蛋白质结构得到稳固,胰蛋白酶的稳定性得到提高,但当删除氨基酸过多时,蛋白质内原有的氢键遭到破坏,由于氢键也是维持蛋白质结构稳定的重要因素,故此导致C5Q的热稳定性远低于野生型胰蛋白酶的热稳定性。
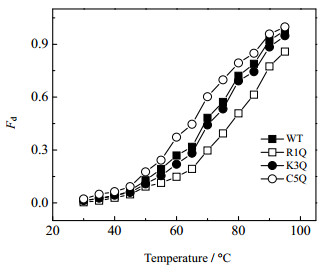
|
图 5 野生型及突变型荧光素酶解折叠曲线 Fig.5 Temperature-induced unfolding of wild-type and mutant trypsin measured by CD spectroscopy |
野生型胰蛋白酶与突变型R1Q, K3Q, C5Q的最适温度分别为75,85,75和50℃,如图 6所示。野生型胰蛋白酶在25~60℃,与R1Q活性相近,但R1Q略有提高,K3Q则明显大于野生型胰蛋白酶的活性,在70~90℃,R1Q和K3Q活性大于野生型胰蛋白酶。而C5Q在25~50℃活性远大于野生型胰蛋白酶,50℃后,酶活开始下降,57℃后则开始低于野生型胰蛋白酶的活性。但可以观察到,三种突变型胰蛋白酶最高活性均大于野生型,这是因为该Ω-loop邻近活性位点,处在活性中心区,暴露于蛋白质表面,覆盖活性位点,减少了活性位点与底物的接触,从而降低了胰蛋白酶的活性,在截短Ω-loop结构后,使得活性位点与底物的接触面积大大提高,从而增强了胰蛋白酶的活性。

|
图 6 最适反应温度测定 Fig.6 Determination of the optimum reaction temperatures |
R1Q突变酶的稳定性得到了很大提高,与野生型相比较,失活半衰期(t1/2)延长了213.3 min,同时酶活也得到了大幅度提高,最高酶活较野生型提高了1.42倍。R1Q的各项实验表征表明胰蛋白酶的稳定性和活性得到了同步提高。
因而,本研究提出的理性设计方法,确定柔性区为改造蛋白热稳定性的相关区域,在不涉及删除活性位点的基础上,通过截除活性位点邻近Ω-loop上的异亮氨酸,实现了酶双功能的共同进化,成功应用于猪源胰蛋白酶的热稳定性和活性的改造,并为其他工业用酶的热稳定性和活性改造提供了借鉴的思路。
| [1] | Kemmler W, Peterson J D, Steiner D F. Studies on the conversion of proinsulin to insulin. I. Conversion in vitro with trypsin and carboxypeptidase B[J]. The Journal of Biological Chemistry, 1971, 246(22): 6786-6791. |
| [2] | Damnjanovic J, Nakang H, Iwasakj Y. Deletion of a dynamic surface loop improves stability and changes kinetic behavior of phosphatidylinositol-synthesizing streptomyces phospholipase D[J]. Biotechnology and Bioengineering, 2014, 111(4): 674-682.DOI:10.1002/bit.25149. |
| [3] | Spier R E. Encyclopedia of cell technology (2 volume set)[M]. New York: John Wiley and Sons, 2000. 351-365. |
| [4] | Kaverin N V, Webster R G. Impairment of multicycle influenza virus growth in Vero (WHO) cells by loss of trypsin activity[J]. Journal of Virology, 1995, 69(4): 2700-2703. |
| [5] | Light A, Duda C T, Odorzynski T W, et al. Refolding of serine proteinases[J]. Journal of Cellular Biochemist, 1986, 31(1): 19-26.DOI:10.1002/(ISSN)1097-4644. |
| [6] | Marcus-Sekura C, Richardson J C, Harston R K, et al. Evaluation of the human host range of bovine and porcine viruses that may contaminate bovine serum and porcine trypsin used in the manufacture of biological products[J]. Biologicals, 2011, 39(6): 359-369.DOI:10.1016/j.biologicals.2011.08.003. |
| [7] | XU Ying-qian(徐英黔), GAN Yi-ru(甘一如), HUANG He(黄鹤). Advances in recombinant protein srugs produced in yeast by gene engineering(酵母中表达基因工程重组蛋白药物的研究进展)[J]. Chemical Industry and Engineering(化学工业与工程), 2005, 22(5): 381-385. |
| [8] | Deng Z, Yang H, Shin H-D, et al. Structure-based rational design and introduction of arginines on the surface of an alkaline α-amylase from Alkalimonas amylolytica for improved thermostability[J]. Applied Microbiology and Biotechnology, 2014, 98(21): 8937-8945.DOI:10.1007/s00253-014-5790-8. |
| [9] | Fang Z, Zhou P, Chang F, et al. Structure-based rational design to enhance the solubility and thermostability of a bacterial laccase Lac15[J]. Plos One, 2014, 9(7): 1-6. |
| [10] | Fei B, Xu H, Cao Y, et al. A multi-factors rational design strategy for enhancing the thermostability of Escherichia coli AppA phytase[J]. Journal of Industrial Microbiology and Biotechnology, 2013, 40(5): 457-464.DOI:10.1007/s10295-013-1260-z. |
| [11] | Van Der Spoel D, Lindahl E, Hess B, et al. GROMACS: fast, flexible, and free[J]. Journal of Computational Chemistry, 2005, 26(16): 1701-1718.DOI:10.1002/(ISSN)1096-987X. |
| [12] | Reetz M T, Carballeira J D, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability[J]. Angewandte Chemie International Edition, 2006, 45(46): 7745-7751.DOI:10.1002/(ISSN)1521-3773. |
| [13] | Thompson M J, Eisenberg D. Transproteomic evidence of a loop-deletion mechanism for enhancing protein thermostability[J]. Journal of Molecular Biology, 1999, 290(2): 595-604.DOI:10.1006/jmbi.1999.2889. |
| [14] | Rejch S, Kress N, Nest B M, et al. Variations in the stability of NCR ene reductase by rational enzyme loop modulation[J]. Journal of Structural Biology, 2014, 185(2): 228-233.DOI:10.1016/j.jsb.2013.04.004. |
| [15] | Scott W R, Hünenberger P H, Tironi I G, et al. The GROMOS biomolecular simulation program package[J]. Journal of Physical Chemistry A, 1999, 103(19): 3596-3607.DOI:10.1021/jp984217f. |
| [16] | Teilum K, Olsen J G, Kragelund B B. Protein stability, flexibility and function[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2011, 1814(8): 969-976.DOI:10.1016/j.bbapap.2010.11.005. |




