骨质疏松症是最常见的一种代谢性骨病,其特征是骨质量及密度减少,骨折风险增加。2010年欧盟研究发现,共有2 200万女性及550万男性患有骨质疏松症,并发现与其相关的350万次脆性骨折的发生[1]。对于骨质疏松症的治疗,每年产生巨大的医疗财政支出[2]。因此关于骨质疏松症病因及治疗方案的探究一直是世界范围内的研究热点。
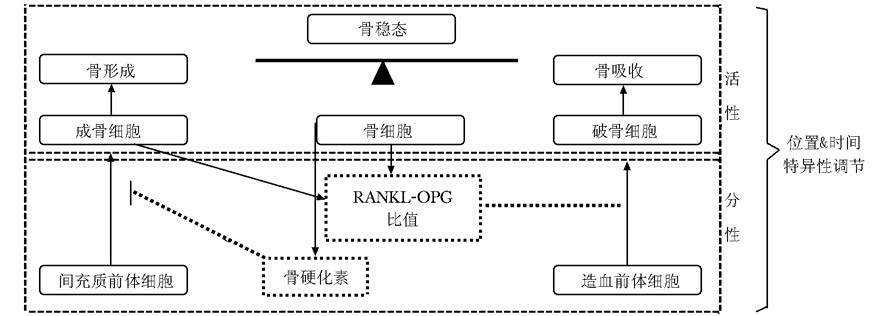
骨质疏松症的发生与骨转换平衡紊乱相关,骨转换的平衡主要依靠破骨细胞的骨吸收作用和成骨细胞的骨形成作用[3,4,5](图1)。成骨细胞的骨形成作用减弱,而破骨细胞的骨吸收作用增强,出现骨转换平衡紊乱,最终导致了骨质疏松症。这种动态平衡受到多种途径的调节,包括维生素D内分泌途径、雌激素内分泌途径、Wnt/β-catenin信号通路和OPG/RANKL/RANK系统等[3]。
很多因素可能增加骨质疏松的风险,如性别、年龄、饮食、运动强度、药物应用及绝经状态等,但目前研究表明,遗传因素是骨质疏松症发病的重要病因之一[6]。随着研究的深入,15种基因已被证实和骨质疏松症的发病有关,包括VDR、ESR1、ESR2、LRP5、LRP4、SOST、GRP177、OPG、RANK、RANKL、COLIA1、SPP1、ITGA1、SP7和SOX6[3]。
表观遗传修饰是一种重要的基因表达调控机制,包括DNA甲基化,组蛋白修饰和非编码RNA等方式。DNA甲基化是可逆的DNA自然化学修饰方式,以S-腺苷-L-甲硫氨酸(S-adenosyl-L-methionine,SAM)为甲基供体,在DNA甲基转移酶(DNA methyltransferases,DNMTs)的作用下将甲基转移到胞嘧啶的C-5上。CpG岛的甲基化会干扰一些转录因子与基因调控区的结合或直接抑制RNA聚合酶活性而抑制基因的表达[7](图2)。甲基化作为一种调控机制可以关闭某种特定的基因,异常的基因甲基化则可能是肿瘤和其他年龄相关性疾病的重要病因[8]。DNA甲基化已经广泛应用于各种肿瘤疾病、糖尿病、骨性关节炎、帕金森等疾病发病机制的研究中。
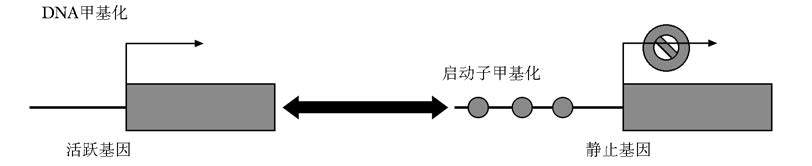
|
| 图2 DNA甲基化表观遗传学修饰 Fig.2 Epigenetic modification of DNA methylation |
目前,有研究表明部分基因的甲基化能够影响成骨细胞和破骨细胞的分化和细胞活性,进而影响骨质疏松症的发生和发展。Delgado-Calle等[9]对髋关节骨质疏松性骨折及髋骨关节炎患者股骨头区域骨细胞进行全细胞基因甲基化测定,发现两组患者中241个CpG位点、228个基因的甲基化水平存在显著差异,这间接证明了骨质疏松症可能与某些特定基因甲基化水平改变相关。Roforth等[10]对青年及老年女性间充质干细胞进行全细胞RNA测序及DNA甲基化水平评估,结果发现启动子具有1 528个序列表达存在差异,而且某些基因甲基化水平存在差异,这反应了年龄相关的骨形成减弱可能与间充质干细胞的甲基化水平相关,这也在一定程度上证明了骨形成减弱导致的骨质疏松症可能与某些基因的甲基化水平相关。目前有较多研究表明,骨质疏松症的发生与一些基因甲基化水平改变相关。本文回顾近年来对骨质疏松症相关基因甲基化水平的研究,从骨转换调节通路相关基因及其他基因的甲基化水平等5个方面进行综述。
雌激素内分泌途径雌激素内分泌途径一直被认为在调节骨量及骨质疏松症的发生中起重要作用。80%的骨质疏松症患者为绝经后妇女,目前普遍认为雌激素水平的降低与绝经后骨质疏松的发生相关。雌激素通过与成骨细胞、破骨细胞表面的雌激素受体结合,调节基因表达,促进成骨细胞增生、分化,抑制破骨细胞的形成及活性,从而起到促进成骨及减少破骨的作用[11]。因此雌激素内分泌途径的某些基因表达发生改变,即能调节该通路的作用,从而导致骨质疏松症发生。
目前关于雌激素受体基因甲基化的研究较多,主要是关于雌激素受体α(estrogen receptor α,ERα)基因甲基化的研究。Penolazzi等[12]在人成骨细胞中发现ERα基因启动子F存在10个特异CpG岛甲基化改变,并且甲基化水平越高基因的转录水平越低,同时成骨细胞的碱性磷酸酶活性也与甲基化水平成负相关。Lambertini等[13] 研究表明,成骨细胞ERα基因的表达,受到启动子F等调节因子的调控。启动子F能够调节ERα mRNA的转录。因此,ERα启动子F基因的甲基化对基因的转录和细胞活性起到反向调控作用。
Lv等[14]研究发现绝经妇女雌激素受体ERα基因启动子A甲基化明显高于未绝经妇女,体外实验证实高半胱氨酸能够提高骨髓间充质干细胞和成骨细胞ERα基因启动子A的甲基化水平,抑制了雌激素受体的表达,从而抑制了干细胞和成骨细胞的生长和分化活性。
以上研究在一定程度上反应了雌激素受体α基因启动子的甲基化水平越高,基因转录水平越低,从而调节骨转换通路,抑制成骨、促进破骨作用,导致骨质疏松症的发生。
Wnt/β-catenin信号通路传统的Wnt/β-catenin信号通路在调节骨量中起到重要作用。当Wnt与跨膜受体家族(frizzled family of receptors,FZD)及低密度脂蛋白受体相关蛋白5/6(low-density lipoprotein receptor-related protein 5/6,LRP5/6)结合为共受体后,Wnt信号通路激活,使β-catenin稳定,稳定的β-catenin转入细胞核,与淋巴增强子结合因子(lymphoid enhancer-binding factor,LEF)及T细胞因子(T cell factor,TCF)结合,影响基因转录。成骨细胞外Wnt因子与膜受体卷曲蛋白 (frizzled,Fz) 结合后,通过经典Wnt通路,激活下游靶基因的转录,促进成骨细胞分化增生[15]因此Wnt通路某些基因表达受到调控,即能调控成骨细胞的增生、分化,影响骨转换。
García-Ibarbia等[16]研究表明骨质疏松性髋部骨折患者较髋骨关节炎患者Wnt相关基因的表达水平明显下降。进一步研究则发现,骨质疏松性骨折患者中成骨细胞核内β-catenin水平较骨关节炎患者低,但二者之间的基因转录水平没有发现明显差别,其降低程度与转录后的β-catenin下调一致,从而导致了Wnt通路的活性降低。更深入的研究表明,二者Wnt通路中FZD10、TBL1X、CSNK1E、WNT8A、CSNK1A1L和SFRP4基因的甲基化水平有明显差异。而通过对两组患者进行全基因组甲基化测定发现,两组患者中241个CpG位点、228个基因的甲基化水平存在显著差异,这些基因大多与细胞分化、骨骼发育相关,其中即包括Wnt通路相关基因[9]。由此可以推断,骨质疏松患者Wnt通路相关基因的甲基化水平改变,能够在转录后下调β-catenin水平,降低Wnt通路的活性,从而抑制成骨细胞的分化、增生,导致骨质疏松症的发生。
Li等[17]的研究表明在地塞米松诱导骨质疏松症的小鼠模型中,间充质干细胞CCAAT增强结合蛋白α(CCAAT/enhancer-binding protein α,C/EBP α)启动子出现明显低甲基化,通过Wnt/β-catenin信号通路使骨形态蛋白-2(bone morphogenetic protein-2,BMP-2)诱导的成骨细胞分化向脂肪细胞分化转变。这表明C/EBP α基因启动子的低甲基化,能够通过调节Wnt/β-catenin信号通路,从而导致骨质疏松症的发生。
通过以上的研究可以发现,某些基因的甲基化水平改变,能够通过Wnt/β-catenin信号通路调节成骨细胞的功能、分化,影响骨转换,从而参与骨质疏松症的产生。Wnt通路中发生甲基化水平改变的基因及其影响调节机制还有待进一步研究。
OPG/RANKL/RANK系统大多数研究表明,骨的重塑能够通过OPG/RANKL/RANK系统调节。破骨细胞中RANKL (receptor activator of NFκB ligand)与它的受体RANK(receptor activator of NFκB)结合,促进破骨细胞的形成及活化[18,19]。骨保护素(osteoprotegerin,OPG) 通过封闭RANKL避免其与RANK结合,从而抑制骨过度吸收[20,21]。OPG/RANKL/RANK系统DNA甲基化的探究,也是目前研究的一个热点。
Kitazawa等[22]对小鼠ST2骨髓间质细胞系进行了研究,发现诱导分化的间质细胞存在RANKL基因甲基化现象,RANKL基因转录起始位点周围的CpG发生甲基化,能够沉默基因启动子的活性,从而抑制RANKL基因的表达,调节骨髓间质细胞的分化多态性,影响成骨细胞的分化。进一步实验研究表明,RANKL基因启动子TATA-box上游的单一CpG位点的甲基化就能够调节细胞和组织特异性RANKL的表达,从而影响破骨细胞的分化[23]。由此,可以发现,骨髓间质细胞的RANKL相关基因位点的甲基化水平改变,能够调节间质细胞分化的多态性,从而调节成骨及破骨细胞分化。
此外,Locklin等[24]发现DNA甲基化酶抑制物5-氮脱氧胞苷(5-Aza-2′-deoxycytidine,AzadC)可以抑制人骨髓中成纤维母细胞DNA甲基化,使成纤维母细胞具有成熟成骨细胞的表型,碱性磷酸酶高表达,从而诱导了成纤维母细胞向成骨细胞分化。Delgado-Calle等[25]发现成骨细胞中RANKL和OPG基因甲基化水平增高会抑制RANKL及OPG表达,应用5-Aza-dC抑制细胞DNA的甲基化水平后,RANKL和OPG表达则相应增加。而RANKL及OPG表达水平的变化,则会影响破骨细胞的形成,从而影响骨稳态。最近Nishikawa等[26]发现,DNA甲基转移酶3a能够通过偶联腺苷甲硫氨酸(S-adenosylmethionine,SAM)介导的代谢途径,抑制抗破骨细胞形成基因的表达,调节破骨细胞分化。研究也发现该代谢过程涉及RANKL通路相关基因甲基化的改变。
以上研究表明,骨髓间质细胞、成纤维母细胞、成骨细胞中OPG/RANKL/RANK通路的某些基因甲基化水平的改变,能影响该通路基因的表达,从而调节成骨细胞及破骨细胞的分化、功能,影响骨转换水平,进而导致骨质疏松的发生。
维生素D内分泌途径维生素D内分泌途径是骨转换中较为重要的一个途径。活性维生素D(1,25-(OH)2D)通过与细胞的维生素D受体(vitamin D receptor,VDR)结合,调节钙磷代谢及骨转换相关蛋白的合成。VDR激素结合区域的错义突变可能导致遗传性VitD抵抗性佝偻病[27]。VDR相关基因是最早研究的骨质疏松相关基因,但对于VDR是否与骨密度相关仍存在较多争议。文献回顾中,未发现VDR相关基因甲基化水平与骨质疏松的关系。
Zhou等[28]通过综述近期对于细胞色素P450相关基因(CYP2R1、CYP24A1、 CYP27A1、CYP27B1)甲基化水平的研究,发现CYP2R1及CYP24A1基因的甲基化水平能够反映不同个体对于维生素D的吸收程度,间接预测了不同个体患维生素D相关骨质疏松症的基因甲基化水平病因。上述研究反映了基因甲基化水平的改变,能够调节维生素D的吸收,进而影响维生素D内分泌途径的调控,调节骨转换。
其他基因甲基化水平与骨质疏松症的关系 Alu序列的甲基化Alu序列是灵长类动物特有的短散在元件(short interspersed elements,SINEs),在基因组中约有120万份拷贝,占基因组的10%以上。Alu序列在人类基因组重组、可变剪切和mRNA转录后的修饰和调控中发挥着特殊的作用。Jintaridth等[29]研究表明,Alu元件的低甲基化水平与绝经后妇女的骨质疏松发生相关,这对于研究全细胞水平低甲基化水平与衰老相关疾病的关系起到提示作用。Rerkasem等[30]研究发现,具有Alu元件高甲基化水平的婴儿,在青少年期具有更快的骨增长速度。这两项研究均反映Alu元件的甲基化水平与骨形成负相关,其低甲基化水平可能会导致骨质疏松。
SOST基因的甲基化骨硬化素(sclerostin)是由骨细胞产生,由SOST基因编码,能够抑制骨形成的物质[31]。Delgado-Calle等[32]研究表明,SOST基因的甲基化能够调节成骨细胞向骨细胞转化过程中硬骨素的表达,从而调节骨形成。因此SOST 基因甲基化水平,可能对骨质疏松的发生起到一定作用。
总结与展望对骨质疏松症相关基因的探究一直是个热点。骨转换平衡相关通路基因,对骨质疏松症的发生产生一定作用。DNA甲基化可以通过调节雌激素内分泌途径、Wnt/β-catenin信号通路、OPG/RANKL/RANK系统、维生素D内分泌途径及其他代谢相关途径中的基因表达,调控成骨、破骨细胞的分化和活性,从而诱导骨质疏松症的发生。人体骨组织中的DNA甲基化水平和相关蛋白的表达反映了表观遗传学在骨质疏松中的作用,这对进一步探索骨质疏松分子水平病因,起到一定的提示作用。但目前的研究并没有明确发生甲基化的基因,与骨转换调节基因的甲基化调控通路机制。
未来的研究方向,一方面可以通过全细胞基因甲基化测定技术,探究骨质疏松的骨组织细胞中不同基因的甲基化水平,找到高甲基化及低甲基化水平的基因或位点,探究基因甲基化对于骨转换的调节机制;另一方面可以通过促进或抑制某些基因甲基化水平,观察骨密度的改变,以期找到对骨质疏松症的可能治疗方案。
| [1] | Hernlund E,Svedbom A,Ivergård M,et al. Osteoporosis in the European Union:medical management,epidemiology and economic burden. A report prepared in collaboration with the International Osteoporosis Foundation(IOF) and the European Federation of Pharmaceutical Industry Associations(EFPIA)[J]. Arch Osteoporos,2013,8:136. |
| [2] | Qu B,Ma Y,Yan M,et al. The economic burden of fracture patients with osteoporosis in western China[J]. Osteoporos Int,2014,25:1853-1860. |
| [3] | Li WF,Hou SX,Yu B,et al. Genetics of osteoporosis:accelerating pace in gene identification and validation[J]. Hum Genet,2010,127:249-285. |
| [4] | Jackson L,Jones DR,Scotting P,et al. Adult mesenchymal stem cells:differentiation potential and therapeutic applications[J]. J Postgrad Med,2007,53:121-127. |
| [5] | Väänänen HK,Zhao H,Mulari M,et al. The cell biology of osteoclast function[J]. J Cell Sci, 2000,113:377-381. |
| [6] | Reppe S,Refvem H,Gautvik VT,et al. Eight genes are highly associated with BMD variation in postmenopausal Caucasian women[J]. Bone,2010,46:604-612. |
| [7] | De Carvalho DD,You JS,Jones PA. DNA methylation and cellular reprogramming[J]. Trends Cell Biol,2010,20:609-617. |
| [8] | Roach HI,Aigner T. DNA methylation in osteoarthritic chondrocytes:a new molecular target[J]. Osteoarthr Cartilage,2007,15:128-137. |
| [9] | Delgado-Calle J,Fernández AF,Sainz J,et al. Genome-wide profiling of bone reveals differentially methylated regions in osteoporosis and osteoarthritis[J]. Arthritis Rheum,2013,65:197-205. |
| [10] | Roforth MM,Farr JN,Fujita K,et al. Global transcriptional profiling using RNA sequencing and DNA methylation patterns in highly enriched mesenchymal cells from young versus elderly women[J]. Bone,2015,76:49-57. |
| [11] | Imai Y. Regulation of bone metabolisms by estrogen/estrogen receptors signaling[J]. Clin Calcium,2013,23:1621-1626. |
| [12] | Penolazzi L,Lambertini E,Giordano S,et al. Methylation analysis of the promoter F of estrogen receptor alpha gene:effects on the level of transcription on human osteoblastic cells[J]. J Steroid Biochem Mol Biol,2004,91:1-9. |
| [13] | Lambertini E,Penolazzi L,Giordano S,et al. Expression of the human oestrogen receptor-alpha gene is regulated by promoter F in MG-63 osteoblastic cells[J]. Biochem J,2003,372:831-839. |
| [14] | Lv H,Ma X,Che T,et al. Methylation of the promoter A of estrogen receptor alpha gene in hBMSC and osteoblasts and its correlation with homocysteine[J]. Mol Cell Biochem,2011,355:35-45. |
| [15] | Kestler HA,Kühl M. From individual Wnt pathways towards a Wnt signalling network[J]. Philos Trans R Soc Lond B Biol Sci,2008,363:1333-1347. |
| [16] | García-Ibarbia C,Delgado-Calle J,Casafont I,et al. Contribution of genetic and epigenetic mechanisms to Wnt pathway activity in prevalent skeletal disorders[J]. Gene,2013,532:165-172. |
| [17] | Li J,Zhang N,Huang X,et al. Dexamethasone shifts bone marrow stromal cells from osteoblasts to adipocytes by C/EBPalpha promoter methylation[J]. Cell Death Dis,2013,4:e832. |
| [18] | Yasuda H,Shima N,Nakagawa N,et al. Osteoclast differentiation factor is a ligand for osteoprotegerin/osteoclastogenesis-inhibitory factor and is identical to TRANCE/RANKL[J]. Proc Natl Acad Sci U S A,1998,95:3597-3602. |
| [19] | Lum L,Wong BR,Josien R,et al. Evidence for a role of a tumor necrosis factor-α(TNF-α)-converting enzyme-like protease in shedding of TRANCE,a TNF family member involved in osteoclastogenesis and dendritic cell survival[J]. J Biol Chem,1999,274:13613-13618. |
| [20] | Simonet WS,Lacey DL,Dunstan CR,et al. Osteoprotegerin:a novel secreted protein involved in the regulation of bone density[J]. Cell,1997,89:309-319. |
| [21] | Kong YY,Yoshida H,Sarosi I,et al. OPGL is a key regulator of osteoclastogenesis,lymphocyte development and lymph-node organogenesis[J]. Nature,1999,397:315-323. |
| [22] | Kitazawa S,Kitazawa R. Epigenetic control of mouse receptor activator of NF-kappa B ligand gene expression[J]. Biochem Biophys Res Commun,2002, 293:126-131. |
| [23] | Kitazawa R,Kitazawa S. Methylation status of a single CpG locus 3 bases upstream of TATA-box of receptor activator of nuclear factor-kappaB ligand(RANKL) gene promoter modulates cell- and tissue-specific RANKL expression and osteoclastogenesis[J]. Mol Endocrinol,2007,21:148-158. |
| [24] | Locklin RM,Oreffo RO,Triffitt JT. Modulation of osteogenic differentiation in human skeletal cells in vitro by 5-azacytidine[J]. Cell Biol Int,1998,22:207-215. |
| [25] | Delgado-Calle J,Sañudo C,Fernández AF,et al. Role of DNA methylation in the regulation of the RANKL-OPG system in human bone[J]. Epigenetics,2012,7:83-91. |
| [26] | Nishikawa K,Iwamoto Y,Kobayashi Y,et al. DNA methyltransferase 3a regulates osteoclast differentiation by coupling to an S-adenosylmethionine-producing metabolic pathway[J]. Nat Med,2015,21:281-287. |
| [27] | Kristjansson K,Rut AR,Hewison M,et al. Two mutations in the hormone binding domain of the vitamin D receptor cause tissue resistance to 1,25 dihydroxyvitamin D3[J]. J Clin Invest, 1993,92:12-16. |
| [28] | Zhou Y,Zhao LJ,Xu X,et al. DNA methylation levels of CYP2R1 and CYP24A1 predict vitamin D response variation[J]. J Steroid Biochem Mol Biol,2014,144:207-214. |
| [29] | Jintaridth P,Tungtrongchitr R,Preutthipan S,et al. Hypomethylation of Alu elements in post-menopausal women with osteoporosis[J]. PLoS One,2013,8:e70386. |
| [30] | Rerkasem K,Rattanatanyong P,Rerkasem A,et al. Higher alu methylation levels in catch-up growth in twenty-year-old offsprings[J]. PLoS One,2015,10:e0120032. |
| [31] | van Bezooijen RL,Roelen BA,Visser A,et al. Sclerostin is an osteocyte-expressed negative regulator of bone formation,but not a classical BMP antagonist[J]. J Exp Med,2004,199:805-814. |
| [32] | Delgado-Calle J,Sañudo C,Bolado A,et al. DNA methylation contributes to the regulation of sclerostin expression in human osteocytes[J]. J Bone Miner Res,2012,27:926-937. |
| (收稿日期:2015-07-13) |